海洋细菌 Agarivorans sp.HZ105的琼胶降解酶系
2015-10-25林伯坤陆国永宋燕谢锐权陈鸿霖胡忠
林伯坤陆国永宋燕谢锐权陈鸿霖胡忠
(1. 汕头大学医学院,汕头 515041;2. 汕头大学理学院生物学系,汕头 515063;3.汕头市雅壳生物科技有限公司,汕头 515041)
海洋细菌 Agarivorans sp.HZ105的琼胶降解酶系
林伯坤1,2陆国永2宋燕2谢锐权3陈鸿霖1胡忠1,2
(1. 汕头大学医学院,汕头 515041;2. 汕头大学理学院生物学系,汕头 515063;3.汕头市雅壳生物科技有限公司,汕头 515041)
通过克隆得到菌株Agarivorans sp. HZ105中3个琼胶酶基因,长度分别为2 988 bp、1 437 bp和1 362 bp,分别编码琼胶酶HZ1、HZ3和HZ4,分别属于糖苷水解酶GH50、GH118和GH16家族。将这些琼胶酶基因与质粒pET-32(a)构建重组表达载体,转化大肠杆菌BL21(DE3),实现了琼胶酶基因的重组原核表达,制备了重组酶,研究了琼胶酶的酶解产物。琼胶酶HZ1降解琼脂糖以及高聚合度新琼寡糖(聚合度为8、10、12和14)得到新琼二糖和新琼四糖;琼胶酶HZ3降解琼脂糖的终产物是高聚合度新琼寡糖;琼胶酶HZ4降解琼脂糖和高聚合度新琼寡糖为新琼四糖和新琼六糖。因此推测菌株HZ105主要先用琼胶酶HZ3和HZ4降解琼脂糖为较高聚合度的新琼寡糖,随后这些寡糖被琼胶酶HZ1和HZ2(课题组先前报道的另一个琼胶酶)降解为低聚合度新琼寡糖。首次研究报道了Agarivorans属中能产生4个琼胶酶的细菌菌株及其琼胶降解酶系,丰富了有关细菌降解琼胶酶体系及其中各琼胶酶作用的研究和认识,也有利于菌株HZ105琼胶酶的有效开发应用。
琼胶酶;基因克隆;Agarivorans;酶系;降解产物
海洋来源的功能性低聚糖的研发正受到越来越多的重视。琼胶是很多红藻细胞壁的主要成分。琼胶寡糖经大量试验证实具有多种生物活性:可促进小鼠肠道有益菌群生长,有望作为益生元[1];具有抗氧化能力[2];抑制血管形成[3];具有皮肤保湿和美白功能,也可作为化妆品添加剂[4]。琼脂糖是琼胶的主要成分,琼胶酶(Agarase)是能够降解琼脂糖的水解酶,根据其作用方式不同,可以把琼胶酶分为两类:α-琼胶酶和β-琼胶酶。α-琼胶酶裂解琼脂糖的α,1-3糖苷键,生成琼寡糖(agarooligosaccharides)系列;β-琼胶酶裂解琼脂糖的β,1-4糖苷键,生成新琼寡糖(neoagarooligosaccharides,NA)系列。生产琼胶寡糖是琼胶酶的一个重要应用。
至今绝大多数研究报道的琼胶酶都来自海洋细菌。其中Agarivorans属细菌是重要的一类琼胶降解细菌,目前该属中已有多个菌株的琼胶酶基因得到研究报道,包括菌株Agarivorans sp. JAMB-A11的琼胶酶AgaA11[5],菌株Agarivorans sp. JA-1的琼胶酶AgaJA1[6]和AgaJA2[7],菌株Agarivorans sp. LQ48的琼胶酶AgaA[8],菌株Agarivorans albus YKW-34的琼胶酶AgaB34[9],菌株Agarivorans sp. AG17的琼胶酶AG17[10],菌株Agarivorans sp. QM38的琼胶酶AgaD01[11]等。但是这些报道都是单独对其中一个琼胶酶进行研究,而且Agarivorans属的这些琼胶酶存在基因序列和酶性质的多样性。虽然目前已有比较多的琼胶酶得到研究,少数产琼胶酶细菌也已得到基因组测序,但这些产琼胶酶细菌中,其琼胶酶降解系统有被研究得很少,尤其是Agarivorans属细菌。
高产琼胶酶海洋细菌Agarivorans sp.HZ105由本课题组从厦门附近海域海底沉积物中筛选得到,并发现该菌株能产多个琼胶酶[12]。本课题组前期已经研究了菌株HZ105中的一个琼胶酶HZ2[13],本文继续研究该菌株的其他3个琼胶酶,阐述菌株HZ105的琼胶降解酶体系,以期丰富对细菌降解琼胶的酶体系的研究和认识,促进对细菌降解琼胶酶体系中各琼胶酶作用的理解,也有利于菌株HZ105琼胶酶的应用开发。
1 材料与方法
1.1 材料
1.1.1 培养基和试剂 LB培养基:10 g 胰蛋白胨,5 g 酵母提取物,10 g NaCl,加双蒸水至1 000 mL,pH7.0,121℃/20 min高压灭菌。3,5-二硝基水杨酸(DNS)试剂配方:1% DNS,0.2% 苯酚,0.05%亚硫酸钠,1% NaOH。细菌总DNA提取试剂盒、DNA胶回收试剂盒和质粒提取试剂盒均购自广州东盛生物公司。
1.1.2 菌株和载体 产琼胶酶菌株Agarivorans sp.HZ105由本课题组从厦门附近海域海底沉积物中筛选得到。克隆和表达宿主菌分别是大肠杆菌E.coli DH5α和BL21(DE3)。表达载体为pET-32a(+)。
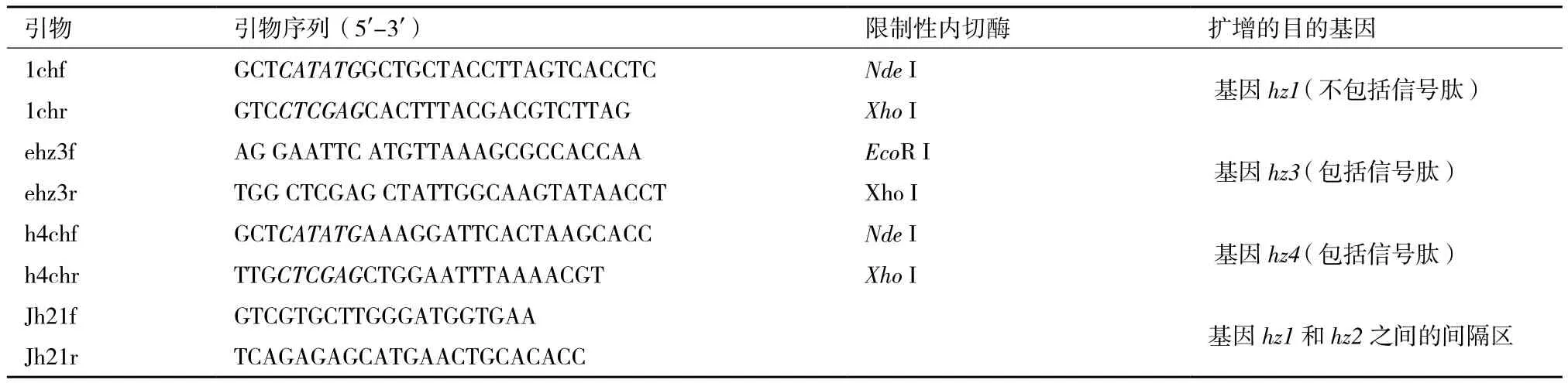
表1 菌株HZ105琼胶酶基因克隆表达引物
1.2 方法
1.2.1 基因克隆和表达载体构建 将菌株Agarivorans sp.HZ105培养于2216E液体培养基中,离心收集菌体,使用试剂盒提取细菌总DNA。使用表1的引物扩增菌株HZ105的3个琼胶酶基因hz1、hz3和hz4。按照表1中限制性内切酶对引物进行切割,并连接经同样酶切的表达载体pET-32a(+),连接产物转化大肠杆菌E.coli DH5α。经验证连接正确的重组载体转化大肠杆菌表达宿主BL21(DE3),最后测序验证琼胶酶基因是否正确连接到载体上。
1.2.2 琼胶酶基因的原核表达 将带有重组表达载体的重组菌接种于LB液体培养基中,37℃,150 r/min培养至生长期加入终浓度为1 mmol/L的IPTG,随后置于16℃继续振荡培养6 h以上。
1.2.3 重组琼胶酶的制备 用SDS-PAGE检测重组蛋白在大肠杆菌中的表达部位。对于在大肠杆菌胞内表达的重组琼胶酶,则用6 000 r/min 5 min离心收集诱导的重组菌(100 mL),加入20 mL的Tris-Cl缓冲液(20 mmol/L,pH8.0)重悬菌体,冰浴中超声破碎至澄清。破碎液12 000 r/min离心10 min,上清即作为重组琼胶酶初酶液,-20℃保存备用;对于在大肠杆菌胞外表达的重组琼胶酶,则用12 000 r/min 5 min离心诱导的重组菌发酵液,取上清;用硫酸铵沉淀蛋白,蛋白沉淀中加入适量的Tris-Cl缓冲液(20 mmol/L,pH8.0)溶解,经用同样的Tris-Cl缓冲液多次透析后,用聚乙二醇20000浓缩,浓缩液经12 000 r/min离心10 min,上清即作为重组琼胶酶初酶液,-20℃保存备用。
1.2.4 DNS法测定琼胶酶活性 琼胶酶降解琼脂,产生还原性末端,与3,5-二硝基水杨酸(DNS)结合,发生变色反应,颜色变化与还原糖的生成量成正比,即与酶活成正比[14]。通过这种有颜色的产物在540 nm处有特殊吸收值,故根据540 nm光吸收值查对标准曲线确定还原糖产生量,定为酶的活力单位。定义每毫升(mL)酶液每分钟内产生1 μmol还原糖的酶量为1 U。标准曲线的制作以D-半乳糖作为还原糖。
每100 μL粗酶液加入900 μL的含0.25%琼脂的Tris-Cl缓冲液(20 mmol/L,pH8.0),42℃温浴15 min,对照样品沸水浴中灭活10 min。加入DNS试剂 2 mL,沸水浴中显色15 min,快速冷却中止反应,加蒸馏水定容至10 mL。OD540nm处测吸光值,对比标准曲线,计算酶活。
1.2.5 重组琼胶酶的最适反应温度 考虑到反应温度可能对琼胶酶降解琼脂糖影响较大,因此需要测定各个琼胶酶的最适反应温度。设置温度为30-70℃,间隔5℃。每100 μL酶液加入900 μL的含0.25%琼脂的Tris-Cl缓冲液(20 mmol/L,pH8.0),在各个温度下反应15 min,DNS法测定琼胶酶活性。
1.2.6 重组琼胶酶降解琼脂糖的产物分析 将琼胶酶酶液1 mL与1 mL的底物(含1.0%琼脂糖的Tris-Cl缓冲液,20 mmol/L,pH8.0)混合,在最适合反应温度下温浴24 h,12 000 r/min离心10 min,上清即作为薄层层析的样品。作琼胶酶对琼脂糖的降解谱分析时,即在不同间隔时间(5 min、15 min、30 min、1 h、2 h、12 h、24 h)取样,沸水浴5 min终止反应,离心,上清待用于上样。
薄层层析板使用前置于110℃烘箱中干燥1 h。在层析板上距离底部1 cm处用铅笔划出底线,将样品10 μL点于底线上,用风筒吹干,置于自制层析缸中展层。展开剂配方为正丁醇∶冰乙酸∶水= 2∶1∶1(体积比)。待前沿线到达离层析板顶端1 cm处时停止展层,烘箱中干燥。最后往层析板喷洒10%的硫酸(体积比),85℃烘箱中显色。
由于市面上缺乏琼胶寡糖标准品,因此本试验利用本课题组所获得的琼胶酶制备琼胶寡糖用作标准品,具体方法参照文献[13]所述。
1.2.7 重组琼胶酶降解琼胶寡糖的研究 将琼胶寡糖与等体积的琼胶酶酶液混合,在最适合反应温度下温浴12 h,12 000 r/min离心5 min,上清即作为薄层层析的样品。
2 结果
2.1 菌株HZ105的琼胶酶基因克隆
本课题组在前期研究中对菌株HZ105琼胶酶进行了串联质谱分析[12],发现菌株HZ105的琼胶酶与菌株Aagarivorans sp. JAMB-A11的一个琼胶酶AgaA匹配分数最高,经序列比对发现后者又与菌株Vibrio sp.JT0107和Vibrio sp. PO-303的琼胶酶具有较高的序列相似性,因此尝试以报道的这些相近琼胶酶基因序列设计引物对菌株HZ105的基因组进行PCR扩增,并由此得到了4个琼胶酶基因(hz1,hz2,hz3,hz4),经测序它们的长度分别是2 988,2 868,1 437 和1 362 bp。将这4个基因的序列提交到GenBank,登录号分别是HQ625020、HQ625021、HQ625022和HQ625023。此前本课题组已经研究报道了菌株HZ105的琼胶酶基因hz2及其编码的琼胶酶HZ2的性质[13],本研究将描述菌株HZ105其他3个琼胶酶的性质。
2.2 琼胶酶基因序列分析
将获得的菌株HZ105的琼胶酶基因序列(hz1,hz3,hz4)在NCBI网站上进行Blastn比对,结果发现这4个琼胶酶基因均与一些报道的β-琼胶酶基因序列具有较高的相似性。其中基因hz1、hz3均有与同为Agarivorans属的琼胶酶基因具有97%以上的相似性,HZ1与Agarivorans sp. QM38的agaD01只有2个氨基酸差别,但目前未见有关琼胶酶agaD01性质的报道。而在Blastn搜索结果中hz4只与菌株Pseudoalteromonas sp. CY24的agaA基因、Vibrio sp. PO-303的agaD基因匹配。本研究首次报道Agarivorans属细菌中单一菌株含有4个琼胶酶基因。鉴于菌株HZ105的4个琼胶酶基因均与菌株Vibrio sp. PO-303的琼胶酶基因序列有较高的相似性,而Vibrio sp. PO-303 还拥有第5个琼胶酶基因AgaA,因此尝试以该基因序列设计多对引物对菌株HZ105基因组进行PCR扩增,但均未获成功,说明菌株HZ105可能不拥有与菌株Vibrio sp. PO-303的琼胶酶基因AgaA相似的基因。
据报道,菌株Vibrio sp.JT0107的琼胶酶基因agaB位于agaA的上游[16],而本文菌株HZ105的基因hz2、hz1分别与agaB、agaA有较高的相似性,因此设计引物Jh21(表1)以扩增两者的间隔区。测序发现,基因hz2和hz1间隔74 bp,与菌株Vibrio sp. JT0107的基因agaB和agaA的间隔序列(Genbank登录号=D14721)有100%相似性,但两菌株的琼胶酶基因并没有100%相似性。
琼胶酶基因(hz1,hz3,hz4)分别编码995 aa、478 aa、453 aa,分别命名为琼胶酶HZ1、HZ3和HZ4,用DNAStar软件推测的分子量大小分别是107.4、50.9和50.8 kD。按照氨基酸序列相似性以及CAZy数据库的分类[17],琼胶酶HZ1、HZ3和HZ4分别应属于糖苷水解酶GH50、GH118和GH16家族。
2.3 琼胶酶基因的重组原核表达
将琼胶酶基因hz1、hz3和hz4按照表1中的限制性酶酶切位点与质粒pET-32(a)连接,成功构建了重组表达载体,并转入大肠杆菌BL21(DE3)中,获得了重组表达菌,并制备了具有较高活性的重组琼胶酶。其中琼胶酶HZ1不包括信号肽序列,经SDS-PAGE检测,主要在大肠杆菌胞内表达;HZ3和HZ4则包含信号肽序列,经SDS-PAGE检测,主要在大肠杆菌胞外表达。
2.4 重组琼胶酶的最适反应温度
由图1可知,琼胶酶HZ1的最适合反应温度是60℃;琼胶酶HZ3的最适合反应温度在40-45℃之间;琼胶酶HZ4在35-50℃之间的酶活力均比较接近,最适温度是50℃。

图1 不同温度对琼胶酶活性影响
2.5 重组琼胶酶降解琼脂糖和琼胶寡糖的产物分析
根据琼胶酶对琼脂糖降解不同时间的产物的薄层层析分析,可知琼胶酶HZ1降解琼脂糖的产物在开始阶段是聚合度较高的寡糖,最终产物是新琼二糖(NA2)和新琼四糖(NA4)(图2-A)。

图2 琼胶酶HZ1对琼脂糖和琼胶寡糖的降解
由图2-B可知,往NA4添加琼胶酶HZ1后,NA4没有被降解,说明HZ1不能降解四糖,这与HZ1降解琼脂糖的终产物有NA4相符合。但HZ1可以降解NA6为NA4和NA2,也可以将聚合度为8/10/12/14的新琼寡糖混合物降解为NA4和NA2。
琼胶酶HZ3降解琼脂糖的主要产物是NA8、NA10、NA12和NA14(图3),它不能进一步降解这些寡糖,也不能降解NA4和NA6(图未展示)。

图3 琼胶酶HZ3对琼脂糖的降解
而琼胶酶HZ4在开始阶段对琼脂糖的降解类似琼胶酶HZ1,也是些聚合度较高的寡糖,最终产物是NA4和NA6(图4-A)。HZ4降解聚合度为8/10/ 12/14的寡糖混合物得到NA4和NA6(图4-B)。

图4 琼胶酶HZ4对琼脂糖和琼胶寡糖的降解
3 讨论
琼胶酶HZ1的最适合反应温度是60℃,这在目前所报道的琼胶酶中是比较高的。菌株Agarivorans sp. AG17 的琼胶酶AG17与HZ1差别7个氨基酸,AG17的最适合反应温度是65℃[10],与HZ1比较接近。而与HZ1分别差别4个、9个氨基酸的AgaA11(来自Agarivorans sp. JAMB-A11)、AgaJA1(来自 Agarivorans sp. JA-1)的最适合反应温度都是40℃[5,6],这与HZ1差别比较大。与琼胶酶HZ3氨基酸序列相似性高的Pseudoalteromonas sp. CY24的 agaB是 40℃[18],Vibrio sp. PO-303的 agaC是35℃[19],三者比较接近。而与HZ4分别差别2个、10个氨基酸的agaA(来自Pseudoalteromonas sp. CY24)、agaD(来自Vibrio sp. PO-303)的最适合反应温度都是40℃[20,21]。
与HZ1序列接近的琼胶酶中,AgaJA1(来自 Agarivorans sp. JA-1)[6]和 菌 株Agarivorans sp. AG17 的琼胶酶AG17[10]降解琼脂糖的终产物也主要是NA2和NA4,与HZ1类似。AgaA11(来自Agarivorans sp. JAMB-A11)的终产物则是NA2[5]。而AgaA11和AgaJA1均可将NA4和NA6降解为NA2,与HZ1不同。菌株Agarivorans sp. AG17 的琼胶酶AG17可以将NA6降解为NA4和NA2,这与HZ1类似。
与HZ3序列接近的琼胶酶中,Pseudoalteromonas sp. CY24的agaB降解琼脂糖的终产物主要是NA8和NA10[18],还有少量的NA4和NA6。Vibrio sp. PO-303的agaC产物主要是NA4、NA6和NA8[19]。agaB和agaC都不能降解NA8,但它们都可以降解NA10。但本研究中的HZ3却与之有所区别,并不能降解NA8、NA10、NA12和NA14。
与HZ4序列接近的琼胶酶中,Pseudoalteromonas sp. CY24的agaA降解琼脂糖的终产物也是NA4和NA6,降解NA8得到NA4和NA6[20],与HZ4一样。Vibrio sp. PO-303的agaD的产物主要是NA4,还有少量的NA2和NA6,还能够将NA6降解为NA4和NA2[21],这与HZ4不一样。
虽然菌株HZ105的琼胶酶与其他菌株的琼胶酶具有较高的序列相似性,但这些琼胶酶在部分性质上存在较大差异。目前已有研究表明琼胶酶的部分氨基酸残基的改变导致酶性质差异。如Han等[22]研究发现菌株Flammeovirga sp. MY04的一个β-琼胶酶AgaG4的催化区里面有一个额外的肽段Asn(246)-Gly(302),这个肽段的切除导致该酶不能降解新琼八糖。因此有待进行突变进一步确认是否是少量的氨基酸差别导致了菌株HZ105的琼胶酶与其他菌株的琼胶酶的性质差异。
随着基因测序技术的发展,到目前为止已有少数产琼胶酶细菌得到基因组测序。但这些基因组得到测序的菌株中,其琼胶酶降解系统被研究得很少。最近,Yasuike等[23]报道了Agarivorans属琼胶降解菌中第一个基因组,但是并未提及基因组中的琼胶酶基因。菌株Saccharophagus degradans 2-40 是目前已得到基因组测序并且其琼胶降解体系得到较全面研究的菌株。Ekborg等[24]鉴定了该菌株的5个琼胶酶,其中2个属于糖水解酶GH50家族,1个属于GH16家族,2个属于GH86家族;最终通过研究认为该菌株的琼胶降解系统包括:一个胞外的内切GH16家族琼胶酶,一个位于细菌表面的GH50家族琼胶酶,一个外切的GH86家族琼胶酶,以及一个新琼二糖水解酶。
Liu等[25,26]最近先后报道了菌株Agarivorans gilvus WH0801的两个琼胶酶AgWH50A和AgWH-50C,这两个酶均属于GH50家族。而本研究报道了菌株Agarivorans sp. HZ105中琼胶酶HZ2之外的其他3个琼胶酶,HZ1降解琼脂糖得到NA2和NA4;HZ2降解琼脂糖得到NA4[6];HZ1和HZ2均可以将聚合度为6/8/10/12/14的新琼寡糖降解为NA2和NA4;HZ3降解琼脂糖的终产物是聚合度为8/10/12/14的新琼寡糖;HZ4降解琼脂糖和高聚合度新琼寡糖为NA6和NA4。因此,推测菌株HZ105主要先用GH118家族琼胶酶HZ3和GH16家族琼胶酶HZ4降解琼脂糖为较高聚合度的新琼寡糖,随后这些寡糖被GH50家族琼胶酶HZ1和HZ2降解为低聚合度寡糖。
4 结论
本文首次报道了Agarivorans属中能产生4个琼胶酶的细菌-菌株HZ105。琼胶酶HZ3和HZ4降解琼脂糖为较高聚合度的新琼寡糖,随后这些寡糖被琼胶酶HZ1和HZ2降解为低聚合度寡糖。本课题组首次较系统地研究了Agarivorans属细菌中的琼胶降解酶系,丰富了对细菌降解琼胶的酶体系的认识,也有利于菌株HZ105琼胶酶的应用开发。
[1] Hu B, Gong QH, Wang Y, et al. Prebiotic effects of neoagarooligosaccharides prepared by enzymatic hydrolysis of agarose[J]. Anaerobe, 2006, 12(5-6):260-266.
[2] Wang JX, Jiang XL, Mou HJ, et al. Anti-oxidation of agar oligosaccharides produced by agarase from a marine bacterium[J]. J Appl Phycol, 2004, 16(5):333-340.
[3] 陈海敏, 严小军, 王峰, 等. 琼胶寡糖抑制血管形成作用的研究[J]. 营养学报, 2007, 29(4):405-407.
[4]Lee DG, Jang MK, Lee OK, et al. Over-production of a glycoside hydrolase family 50 β-agarase from Agarivorans sp. JA-1 in Bacillus subtilis and the whitening effect of its product[J]. Biotechnol Lett,2008, 30(5):911-918.
[5] Ohta Y, Hatada Y, Ito S, et al. High-level expression of a neoagarobiose- producing-agarase gene from Agarivorans sp. JAMB-A11 in Bacillus subtilis and enzymic properties of the recombinant enzyme[J]. Biotechnol Appl Bioc, 2005, 41(Pt 2):183-191.
[6] Lee DG, Park GT, Kim NY, et al. Cloning, expression, and characterization of a glycoside hydrolase family 50 β-agarase from a marine Agarivorans isolate[J]. Biotechnol Lett, 2006, 28(23):1925-1932.
[7] Lee DG, Jeon MJ, Lee SH. Cloning, expression, and characterization of a glycoside hydrolase family 118 beta-agarase from Agarivorans sp. JA-1[J]. J Microbiol Biotechnol, 2012, 22(12):1692-1697.
[8]Long M, Yu Z, Xu X. A novel beta-agarase with high pH stability from marine Agarivorans sp. LQ48[J]. Mar Biotechnol, 2010, 12(1):62-69.
[9] Fu XT, Pan CH, Lin H, et al. Gene cloning, expression, and characterization of a beta-agarase, agaB34, from Agarivorans albus YKW-34[J]. J Microbiol Biotechnol, 2009, 19(3):257-264.
[10] Nikapitiya C, Oh C, Lee Y, et al. Characterization of a glycoside hydrolase family 50 thermostable beta agarase AG17 from marine bacteria Agarivorans sp. AG17[J]. Fish Aquat Sci, 2010, 13(1):36-48.
[11] Du Z, Wang J, Yang L, et al. Identification of a marine agarolytic bacterium Agarivorans albus QM38 and cloning and sequencing its beta-agarase genes[J]. Acta Oceanol Sin, 2011, 30(1):118-124.
[12]Hu Z, Lin BK, Xu Y, et al. Production and purification of agarase from a marine agarolytic bacterium Agarivorans sp.HZ105[J]. J Appl Microbiol, 2009, 106(1):181-190.
[13]Lin BK, Lu GY, Zheng YD, et al. Gene cloning, expression and characterization of a neoagarotetraose-producing β-agarase from a marine bacterium Agarivorans sp.HZ105[J]. World J Microb Biot, 2012, 28(4):1691-1697.
[14]Miller GL. Use of dinitrosaIicyIic acid reagent for determination of reducing sugar[J]. Anal Chem, 1959, 31(3):426-428.
[15]Dong J, Tamaru Y, Araki T. A unique beta-agarase, AgaA, from a marine bacterium, Vibrio sp. strain PO-303[J]. Appl Microbiol Biot, 2007, 74(6):1248-1255.
[16]Sugano Y, Matsumoto T, Noma M. Sequence analysis of the agaB gene encoding a new β-agarase from Vibrio sp. strain JT0107[J]. Bioch Bioph Acta, 1994, 17(1):105-108.
[17]Cantarel BL, Coutinho PM, Rancurel C, et al. The Carbohydrate-Active EnZymes database(CAZy):an expert resource for Glycogenomics[J]. Nucleic Acids Res, 2009, 37:D233-238.
[18]Ma C, Lu X, Shi C, et al. Molecular cloning and characterization of a novel β-Agarase, AgaB, from marine Pseudoalteromonas sp. CY24[J]. J Biol Chem, 2007, 282(6):3747-3754.
[19]Dong J, Hashikawa S, Konishi T, et al. Cloning of the novel gene encoding beta-agarase C from a marine bacterium, Vibrio sp. strain PO-303, and characterization of the gene product[J]. Appl Environ Microb, 2006, 72(9):6399-401.
[20]Lu X, Chu Y, Wu Q, et al. Cloning, expression and characterization of a new agarase-encoding gene from marine Pseudoalteromonas sp.[J]. Biotechnol Lett, 2009, 31(10):1565-1570.
[21] Dong J, Tamaru Y, Araki T. Molecular cloning, expression and characterization of a β-agarase gene, agaD, from a marine bacterium, Vibrio sp.strain PO-303[J]. Biosci Biotech Bioch, 2007, 71(1):38-46.
[22] Han WJ, Gu JY, Liu HH, et al. An extra peptide within the catalytic module of a β-Agarase affects the agarose degradation pattern[J]. J Biol Chem, 2013, 288:9519-9531.
[23] Yasuike M, Nakamura Y, Kai W, et al. Draft genome sequence of Agarivorans albus strain MKT 106T, an agarolytic marine bacterium[J]. Genome Announc, 2013, 1(4):e00367-13.
[24] Ekborg NA, Larry ET, Atkinson GL, et al. Genomic and proteomic analyses of the agarolytic system expressedby Saccharophagus degradans 2-40[J]. Appl Environ Microb, 2006, 72(5):3396-3405.
[25] Liu N, Mao X, Du Z, et al. Cloning and characterisation of a novel neoagarotetraose- forming-beta-agarase, AgWH50A from Agarivorans gilvus WH0801[J]. Carbohyd Res, 2014, 388:147-151.
[26] Liu N, Mao X, Yang M, et al. Gene cloning, expression and characterization of a new beta-agarase, AgWH50C, producing neoagarobiose from Agarivorans gilvus WH0801[J]. World J Microb Biot,2014, doi:10.1007/s11274-013-1591-y.
(责任编辑 李楠)
The Agar-degrading Enzymatic System of Marine Bacterium Agarivorans sp.HZ105
Lin Bokun1,2Lu Guoyong2Song Yan2Xie Ruiquan3Chen Honglin1Hu Zhong1,2
(1. Medical College,Shantou University,Shantou 515041;2. Department of Biology,Science College,Shantou University,Shantou 515063;3. Shantou Yuccor Biotechnology co. ltd,Shantou 515041)
Three agarase genes(hz1, hz2, hz3)of 2 988 bp, 1 437 bp and 1 362 bp respectively, were cloned from strain Agarivorans sp.HZ105. These three agarase genes(hz1, hz2, hz3)encoded agarase HZ1, HZ3 and HZ4 and belonged to glycoside hydrolase family GH50, GH118 and GH16, respectively. The agarase genes of strain HZ105 were linked with vector pET-32a(+)and then were transfered to Escherichia coli BL21(DE3). These agarase genes were successfully expressed in E. coli cells and their recombinant agarases were prepared. Agarose degradations of the recombinant agarases were studied. Agarase HZ1 could degrade agarose and neoagarooligosaccharides with high degrees of polymerization(8, 10, 12 and 14)into neoagarobiose and neoagarotetraose. Agarase HZ3 digested agarose to produce neoagarooligosaccharides with high degrees of polymerization, while agarase HZ4 decomposed agarose and neoagarooligosaccharides with high degrees of polymerization to yield neoagarotetraose and neoagarohexaose. Therefore, strain HZ105 might produce firstly agarases HZ3 and HZ4 to degrade agarose into neoagarooligosaccharides with high degrees of polymerization, and the products of agarases HZ3 and HZ4 would be then digested into neoagarooligosaccharides with low degrees of polymerization by agarase HZ1 and another agarase(HZ2)from strain HZ105 reported previously by our research group. Strain HZ105 is the first reported strain that produces four agarases and this study is the first report of the agar-degrading enzymatic system in the genus Agarivorans. The results could enrich the knowledge about the bacterial agar-degrading enzymatic system and the roles of the agarases in the system. The application of agarases from strain HZ105 should also benift from this study.
agarase;gene cloning;Agarivorans;enzymatic system;degradation products
10.13560/j.cnki.biotech.bull.1985.2015.01.024
2014-05-23
国家自然科学基金项目(31200077,41476150),中国博士后科学基金项目(2013M531871),广东省自然科学基金项目(S2012040006279),广东省科技计划项目(2011B031100006)
林伯坤,男,博士,研究方向:微生物学生化与分子;E-mail:biotech_1@stu.edu.cn
胡忠,男,教授,研究方向:微生物学,E-mail:hzh@stu.edu.cn;陈鸿霖,男,教授,研究方向:微生物学;E-mail:hlchen@hku.hk
