鹌鹑肠道微生物PCR-DGGE方法的优化
2015-10-25张振华鲍王波余冉王长永刘燕
张振华鲍王波余冉王长永刘燕
(1.环境保护部南京环境科学研究所,南京 210042;2.东南大学能源与环境学院,南京 210096)
鹌鹑肠道微生物PCR-DGGE方法的优化
张振华1鲍王波2余冉2王长永1刘燕1
(1.环境保护部南京环境科学研究所,南京 210042;2.东南大学能源与环境学院,南京 210096)
通过对鹌鹑肠道微生物总DNA不同提取方法的效率进行比较,探讨DNA质量以及PCR-DGGE反应条件对研究鹌鹑肠道微生物多样性的影响。本试验利用常规的饱和苯酚/氯仿法和3种试剂盒(QIAGEN试剂盒、上海生工试剂盒、天根试剂盒)方法提取鹌鹑肠道微生物总DNA,分别以细菌V3可变区引物和V6-V8可变区引物以及不同退火温度进行PCR-DGGE分析。QIAGEN试剂盒法提取的肠道微生物总DNA的 A260/A280值在1.8-1.9之间,浓度约200 ng/μL,DGGE微生物多样性条带均匀度均高于其他3种提取方法。此外,利用不同引物扩增的DGGE图谱发现,以V6-V8可变区引物扩增的DGGE图谱条带均匀,分离充分,较V3可变区更能反映鹌鹑肠道微生物种群的多样性;同时,随PCR退火温度的升高,V6-V8可变区的微生物多样性指数下降,V3可变区的微生物多样性则无明显变化。
鹌鹑;肠道微生物;总DNA提取;变性凝胶梯度电泳(DGGE)
DIO: 10.13560/j.cnki.biotech.bull.1985.2015.01.011
动物肠道内定居着种类多数量庞大的微生物群,对宿主的生理和健康起着极其重要的作用。据研究统计,每克家禽盲肠内容物中约含微生物种类500-600种,数量107-1011个[1,2]。随着动物肠道生态学研究的发展,单纯的培养筛选法已无法满足对肠道微生物多样性研究的需要,分子生物学技术克服了传统的微生物分离、培养和分类方法的局限性,迅速发展成为肠道微生物多样性研究的常用技术[3]。目前,针对鸟类肠道微生物的现代分子生物学技术包括了16S rRNA基因克隆文库、变性梯度凝胶电泳(Denatured gradient gel electrophoresis,DGGE)、 核酸分子杂交以及实时荧光定量PCR技术[4]。
由于DGGE技术能快速、有效的分析许多环境中优势菌群的特异性差异,因而也非常适于动物肠道微环境中的微生物群落多样性分析研究[5,6]。
鹌鹑不仅是一种高产特禽,而且被国内外广泛作为环境及日粮毒性实验中的模式鸟类展开相关研究。目前,有关动物肠道微生物的分子生态学研究主要集中在牛、猪、鸡等主要的经济畜禽动物,针对鹌鹑肠道微生物分子生态学方面的研究较少,大都参考传统的平板菌落技术法来分离和测定其肠道主要微生物菌群的种类与数量[7]。因此,建立一套针对鹌鹑肠道微环境的PCR-DGGE方法对研究其微生物多样性有重要意义。
不同研究者发现,DNA提取方法、PCR退火温度及引物种类对PCR-DGGE法研究肠道微生物菌群多样性有一定影响[8-10]。在DNA提取过程中,细胞裂解不彻底、抽提过程造成的DNA链断裂及PCR抑制剂去除不彻底等因素将对PCR扩增效率产生影响[9]。PCR退火温度也是影响DGGE条带丰富度的重要因素。Sipos1等[11]的研究表明,在保证PCR条带特异性的前提下,应尽可能降低退火温度,以提高微生物多样性指数。目前,基于16S rRNA基因序列的V1,V3,V1-V3,V6-V8可变区引物进行的肠道微生物多样性研究都有报道[12-16],其中V3和V6-V8可变区由于其DGGE图谱的条带分离效果好,条带数目和亮度较好而被广泛使用[17,18]。
本研究取鹌鹑肠道为试验材料,比较了4种方法对肠道微生物总DNA的提取结果,研究了V3、V6-V8可变区引物以及不同退火温度对PCR-DGGE分析结果的影响,旨在建立鹌鹑肠道微生物总DNA的最佳提取方法与PCR-DGGE优化条件,以便能更深刻地揭示鹌鹑肠道微生物菌群种类和丰度。
1 材料与方法
1.1 鹌鹑肠道食糜样品的获取
约90天龄的健康雌性日本鹌鹑于无菌条件下解剖,将3只鹌鹑直肠内容物取出,混合后置于1.5 mL无菌EP管,-80℃冻存备用(肠道样品由南京中医药大学提供)。
1.2 鹌鹑肠道微生物总DNA的提取及检测
CTAB提取法:参考文献[19]。准确称取鹌鹑直肠内容物0.2 g,加入700 μL CTAB抽提液、15 μL SDS、10 μL蛋白酶K,混匀后常温裂解20 min;56℃温浴2-3 h,10 000 r/min 离心10 min;提取上清液至新的离心管中,加入5 μL的RNase A(10 mg/mL),于37℃下水浴1 h,12 000 r/min 离心10 min;吸取上层水相于新的EP管中,加等体积苯酚/氯仿/异丙醇抽提2次直至界面清晰,异丙醇沉淀及70%乙醇洗涤,最后加入30 μL的1×TE溶解,-20℃保存备用。
试剂盒提取:准确称取0.1 g鹌鹑直肠内容物,加500 μL PBS,冰上破碎20 s,10 000 r/min离心10 min,离心弃上清液,随后按试剂盒说明书操作,提取的DNA于-20℃保存备用。
DNA检测:取1 μL DNA,核酸定量仪测定其浓度及A260/A280值的结果判定。
1.3 PCR扩增及DGGE图谱多样性分析
V3区引物序列为:341 F(5'-CCTACGGGAGGCAGCAG-3');534 R(5'-ATTACCGCGGCTGCTGG -3')[17]。V6-V8区引物序列:U968(5'-AACGCGAAGAACCTTAC-3');L1401(5'-CGGTGTGTACAAGACCC-3')[18]。GC夹子(CGCCCGCCGCGCCCCGCG CCCGGCCCGCCGCCCCCGCCCC)。
PCR体系参考Zhou[18]研究方法,25 μL体系:2.5 μL 10×PCR buffer(Mg2+free),2 μL dNTP Mix,左右引物各0.5 μL,1.5 μL MgCl2,0.3 μL Taq酶,DNA模板50 ng,ddH2O 17 μL。反应条件:94℃预变性3 min;94℃变性30 s,3个不同温度下退火30 s,72℃延伸1 min,35个循环;72℃保持5 min。退火温度设置如下梯度:V3区:55℃、60℃、65℃;V6-V8区:50℃、55℃、60℃。扩增产物5 μL进行1.5%的琼脂糖电泳,1×TAE电泳液,紫外凝胶成像仪(Bio-Rad,USA)成像。Gelred染料,DL2000 Marker。
DGGE参考Torok等[20]试验方法,使用8%的聚丙烯酰胺(37.5∶1 acrylamide∶bisacrylamide)凝胶,预试验确定变性范围:V3区:40%-60%;V6-V8区:30%-50%(100%的变性剂中含有7 mol/L的尿素和40%的去离子甲酰胺),蠕动泵自动灌胶。每条泳道加反应体系样20 μL,1×TAE电泳液,60℃,200 V预跑10 min,随后60 V电泳17 h,SYBR Green I染色40 min后扫描成像。DGGE图谱使用Quantity one软件分析后将条带信息转移到Excel工作表中进行后期多样性分析。Quantity one软件自动计算出每个条带的丰度值,由此计算出每个泳道条带数S,并计算每条泳道的Shanoon指数H和均匀度E。其中:Pi=ni/N,ni为第i个条带的丰度值,N为第i个条带的泳道上所有条带丰度之和[4]。
2 结果
2.1 四种方法提取的鹌鹑肠道微生物总DNA浓度及纯度比较
如图1所示,在使用相同肠道样本情况下,不同方法获得鹌鹑肠道微生物总DNA浓度表明,天根试剂盒法>QIAGEN试剂盒法>酚-氯仿法>生工试剂盒法;纯度(A260/A280值)表明,QIAGEN试剂盒法和生工试剂盒法最优,天根试剂盒法和酚-氯仿法则表现出不同程度的污染。
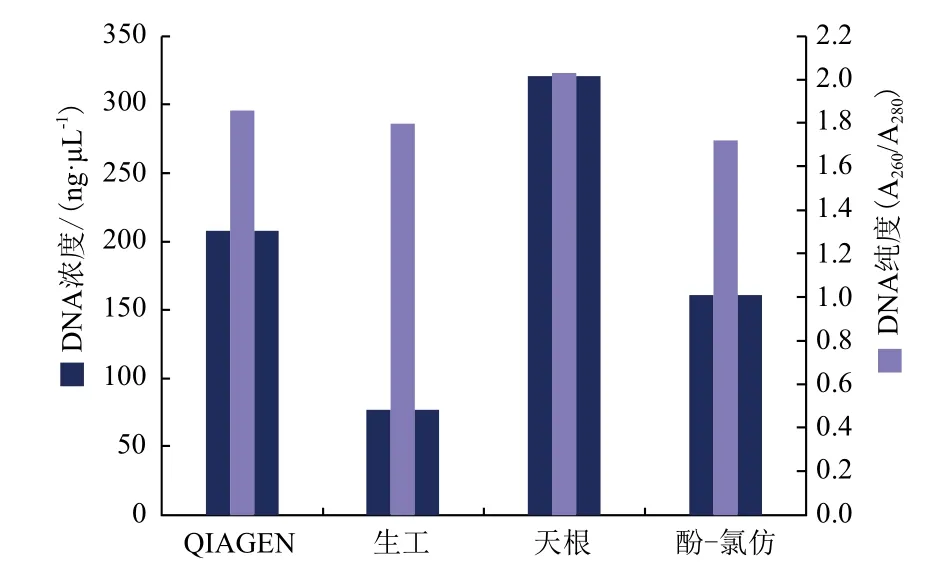
图1 四种方法提取的鹌鹑肠道总DNA浓度和纯度
对4种提取方法的鹌鹑肠道微生物总DNA分别在55℃、60℃、65℃退火温度下,以16S rRNA V3和V6-V8可变区引物进行扩增。结果如图2所示,4种方法提取的鹌鹑肠道微生物总DNA都得到预期大小的目的条带。酚-氯仿法处理样品的目的条带亮度较低,说明其DNA模板中含有某些PCR抑制因子[21]。V6-V8可变区引物的扩增产物都出现了引物二聚体,而V3可变区引物的扩增产物只在酚-氯仿法提取的DNA模板中出现。退火温度对4种提取方法提取的鹌鹑肠道微生物总DNA的PCR扩增效率无显著影响。
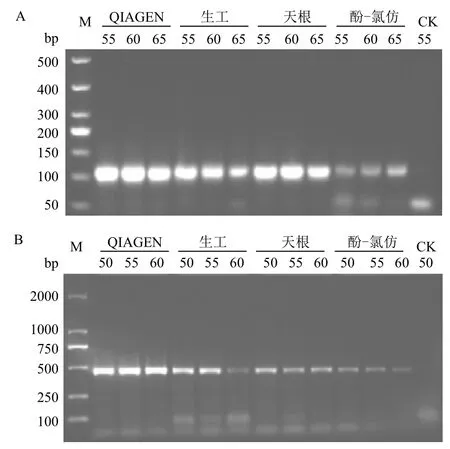
图2 比较不同提取方法引物及退火温度下的PCR产物琼脂糖电泳图
2.2 DGGE图谱及微生物多样性分析
为进一步探讨不同提取方法、退火温度以及引物对鹌鹑肠道微生物多样性的影响,试验对鹌鹑肠道微生物总DNA的PCR产物进行了DGGE电泳分析。以V3可变区引物扩增的DGGE电泳图谱以及戴斯系数分析结果,如图3-A所示。QIAGEN试剂盒法和天根试剂盒法提取的DNA模板PCR扩增产物得到的DGGE条带数较多,3种不同退火温度下,平均值分别为21和27。而生工试剂盒法与酚-氯仿法提取的总DNA模板PCR-DGGE图谱条带数平均值分别为11和18。在3种不同退火温度条件下,4种提取方法得到的PCR-DGGE图谱条带数并未随退火温度升高而减少。在60℃和65℃退火温度条件下,4种提取方法的条带丰富度较高且相对稳定。Shannon指数分析结果(表1)表示,在V3可变区,4种提取方法的微生物多样性指数从高到低分别为天根试剂盒法、QIAGEN试剂盒法、酚-氯仿法和生工试剂盒法,最佳退火温度为60℃。
以V6-V8可变区引物扩增的DGGE电泳图谱以及戴斯系数分析结果,如图3-B所示。QIAGEN试剂盒法和生工试剂盒法提取的DNA模板PCR扩增产物得到的DGGE条带数较多,3个不同退火温度下,平均值分别为20和27。天根试剂盒法与酚-氯仿法提取的总DNA模板PCR-DGGE图谱条带数平均值分别为16和13。和V3可变区分析结果不同的是,四种提取方法的PCR-DGGE图谱的条带数都随着退火温度升高而减少。表1的Shannon指数分析也说明,在V6-V8可变区,50℃退火温度条件下,4种提取方法的微生物多样性指数最高。

图3 比较不同提取方法引物下的DGGE指纹图谱

表1 比较不同提取方法引物及退火温度下的微生物多样性指数分析
比较V3可变区和V6-V8可变区的PCR-DGGE图谱可以发现,V3可变区的微生物种类大多聚集于泳道上部,且不同条带的丰度值差异较大,均匀度E值都在0.90左右。V6-V8可变区的微生物种类则均匀的分布于整个泳道,且不同条带的丰度值差异较小,均匀度E值均在0.99左右。
3 讨论
PCR-DGGE作为一种现代分子生物学技术,给研究动物肠道微生物多样性提供了一种准确、高效的方法[9]。本试 验以鹌鹑肠道为研究材料,探讨了提取肠道微生物总DNA的4种方法,V3、V6-V8可变区通用引物以及不同退火温度对PCR-DGGE分析结果的影响。
试验表明,QIAGEN试剂盒法提取的鹌鹑肠道微生物总DNA浓度及纯度均较高,PCR-DGGE分析结果表明微生物多样性指数较高,整体表现最优;天根试剂盒法和生工试剂盒法次之;酚-氯仿法提取的鹌鹑肠道微生物总DNA有蛋白或酚类物质污染,对PCR扩增有一定抑制作用,但都能基本满足PCR-DGGE分析要求。
研究不同引物对PCR-DGGE的影响发现,V6-V8可变区较V3可变区更能反映鹌鹑肠道微生物种群的多样性。可能由于鹌鹑肠道微生物种类相对较少而种间水平丰富,而V3可变区较短(190 bp左右),DNA链之间碱基差异小,因此很难将同属不同种的优势菌群有效分离,导致在DGGE图谱上显示某些条带丰度值很高,均匀度下降。而V6-V8可变区大小在450 bp左右,条带能较均匀的分布在DGGE图谱上,均匀度较高。Hong等[17]用V3可变区通用引物研究猪肠道微生物时发现,由于扩增片段太短,导致很难将细菌种类有效区分。Yu等[5]研究肠道微生物多样性时也发现V6-V8区的E值也高于V3区。
综上所述,就鹌鹑肠道微生物多样性的PCRDGGE分析方法而言,QIAGEN试剂盒法对肠道微生物总DNA的提取效果最好;V6-V8可变区的PCRDGGE图谱条带清晰、分布均匀、分离充分,能更客观的反映鹌鹑肠道微生物菌群结构;以V6-V8可变区进行PCR扩增时,可通过降低退火温度来增加DGGE条带数量。
4 结论
本研究以鹌鹑肠道微生物总DNA为模板,比较4种DNA提取方法、两对16S rRNA可变区引物、3个退火温度对PCR-DGGE法研究鹌鹑肠道微生物多样性的影响。研究结果表明,V6-V8可变区引物及55℃退火温度更能客观的反映鹌鹑肠道微生物菌群结构。
[1] Salanitro J, Fairchilds I, Zgornicki Y. Isolation, culture characteristics, and identification of anaerobic bacteria from the chicken cecum[J]. Applied Microbiology, 1974, 27(4):678-687.
[2] Kelley RW. Comparison of media for isolation of poultry intestinal bacteria[J]. Appl Environ Microbiol, 1983, 46(2):421-424.
[3] Kocherginskaya SA, Aminov RI, White BA. Analysis of the rumen bacterial diversity under two different diet conditions using denaturing gradient gel electrophoresis, random sequencing, and statistical ecology approaches[J]. Anaerobe, 2001, 7(3):119-134.
[4] Ahir V, Singh K, Tripathi A, et al. Study of Bacterial Diversity in Poultry Gut Using Denaturing Gradient Gel Electroresis[J]. Iranian Journal of Applied Animal Science, 2012, 2(3):227-232.
[5] Yu Z, Morrison M. Comparisons of different hypervariable regions of rrs genes for use in fingerprinting of microbial communities by PCR-denaturing gradient gel electrophoresis[J]. Appl Environ Microbiol, 2004, 70(8):4800-4806.
[6] Ferris M, Muyzer G, Ward D. Denaturing gradient gel electrophoresis profiles of 16S rRNA-defined populations inhabiting a hot spring microbial mat community[J]. Appl Environ Microbiol, 1996, 62(2):340-346.
[7] 王书全. 合生素对肉用鹌鹑免疫功能和肠道菌群的影响[J].中国农业通报,2011, 27(11):39-42.
[8] Beutler E, Gelbart T, Kuhl W. Interference of heparin with the polymerase chain reaction[J]. BioTechniques, 1990, 9(2):166.
[9] Monteiro L, Gras N, Vidal R, et al. Detection of Helicobacter pylori DNA in human feces by PCR:DNA stability and removal of inhibitors[J]. Journal of Microbiological Methods, 2001, 45(2):89-94.
[10] Mahony J, Chong S, Jang D, et al. Urine specimens from pregnant and nonpregnant women inhibitory to amplification of Chlamydia trachomatis nucleic acid by PCR, Ligase chain reaction, and transcription-mediated amplification:identification of urinary substances associated with inhibition and removal of inhibitory activity[J]. Journal of Clinical Microbiology, 1998, 36(11):3122-3126.
[11] Sipos R, Székely AJ, Palatinszky M, et al. Effect of primer mismatch, annealing temperature and PCR cycle number on 16S rRNA gene-targetting bacterial community analysis[J]. FEMS Microbiology Ecology, 2007, 60(2):341-350.
[12] Simpson JM, McCracken VJ, Gaskins HR, et al. Denaturing gradient gel electrophoresis analysis of 16S ribosomal DNA amplicons to monitor changes in fecal bacterial populations of weaning pigs after introduction of Lactobacillus reuteri strain MM53[J]. Appl Environ Microbiol, 2000, 66(11):4705-4714.
[13] De Boever P, Wouters R, Vermeirssen V, et al. Development of a six-stage culture system for simulating the gastrointestinal microbiota of weaned infants[J]. Microbial Ecology in Health and Disease, 2001, 13(2):111-123.
[14] Mai V, Katki HA, Harmsen H, et al. Effects of a controlled diet and black tea drinking on the fecal microflora composition and the fecal bile acid profile of human volunteers in a double-blindedrandomized feeding study[J]. The Journal of Nutrition, 2004,134(2):473-478.
[15] Favier CF, Vaughan EE, De Vos WM, et al. Molecular monitoring of succession of bacterial communities in human neonates[J]. Appl Environ Microbiol, 2002, 68(1):219-226.
[16] Nübel U, Engelen B, Felske A, et al. Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel electrophoresis[J]. Journal of Bacteriology, 1996, 178(19):5636-5643.
[17] Hong TTT, Passoth V, Lindberg JE. Bacterial diversity at different sites of the digestive tract of weaned piglets fed liquid diets[J]. Asian-Aust J Anim Sci, 2011, 24:834-843.
[18] Zhou H, Gong J, Brisbin J, et al. Appropriate chicken sample size for identifying the composition of broiler intestinal microbiota affected by dietary antibiotics, using the polymerase chain reactiondenaturing gradient gel electrophoresis technique[J]. Poultry Science, 2007, 86(12):2541-2549.
[19] Vaughan EE, Heilig HG, Ben-Amor K, et al. Diversity, vitality and activities of intestinal lactic acid bacteria and bifidobacteria assessed by molecular approaches[J]. FEMS Microbiology Reviews, 2005, 29(3):477-490.
[20] 王书全. 合生素对肉用鹌鹑免疫功能和肠道菌群的影响[J].中国农业通报, 2011, 27(11):39-42.
[21] 白雪, 付晶, 白秀娟. 鹌鹑肠道微生物总DNA的最佳提取方法[J].黑龙江畜牧兽医, 2012, 5:24-29.
(责任编辑 狄艳红)
Optimization Method for the Extraction of Total DNA from the Intestinal Microbes in Quails
Zhang Zhenhua1Bao Wangbo2Yu Ran2Wang Changyong1Liu Yan1
(1. Nanjing Institute of Environmental Sciences,MEP,Nanjing 210042;2. Department of Energy and Environment,Southeast University,Nanjing 210096)
Comparing the effects of total DNA extracted by different methods from intestinal microbes in quails, the study aimed at investigating the influences of DNA quality and PCR-DGGE conditions on intestinal microbial diversity of quail. In this study, the experiment used four methods to extract the DNA. The first was a conventional method for using saturated phenol/chloroform, other three commercially available DNA extraction kits(QIAGEN, SHENGGONG, TIANGEN)were assessed. The ribosomal gene sequences were amplified with V3 and V6-V8 hypervariable region universal primers at different annealing temperature and subjected to denaturing gradient gel electrophoresis(DGGE). The QIAGEN DNA Kit generated the greatest bacterial species eveness and DNA quantity and quality in compare with other methods(A260/A280=1.8, 200 ng/μL). The resulting DGGE profiles were substantially different in terms of the number and relative intensity of the amplification products. The V6-V8 region produced better DGGE profiles than V3 region. The QIAGEN DNA Kit and V6-V8 hypervariable region universal primers will be used in future studies of intestinal microbial diversity of quail.
quail;ntestinal microbes;DNA extraction;DGGE
2014-06-09
转基因生物新品种培育重大专项(2013ZX08011-003,2014ZX08011-003,2013ZX08012-005,2014ZX08012-005),环保公益行业专项(201309038)
张振华,男,博士,研究方向:微生物学;E-mail:zzh@nies.org
刘燕,女,副研究员,研究方向:生物安全与生物多样性保护;E-mail:liuyan@nies.org
