植物非生物胁迫相关转录因子研究方法
2015-10-25宿明星孙颖颢施鹤李秋莉
宿明星 孙颖颢 施鹤 李秋莉
(辽宁师范大学生命科学学院 辽宁省植物生物技术重点实验室,大连116081)
植物非生物胁迫相关转录因子研究方法
宿明星 孙颖颢 施鹤 李秋莉
(辽宁师范大学生命科学学院 辽宁省植物生物技术重点实验室,大连116081)
转录因子是一类能够与启动子区域顺式作用元件特异性结合的蛋白质,是一大类转录调控因子,也是植物中最大的基因家族之一。转录因子可以调节众多下游基因的表达,对植物的生长发育、形态建成、激素调节,以及抵抗多种生物和非生物胁迫具有重要作用。结合近年来转录因子的研究进展,归纳总结了植物非生物胁迫相关转录因子研究的主要策略和方法,包括转录因子结构域、亚细胞定位、转录激活作用、转录因子复合体以及转录因子功能的研究,为植物转录因子的相关研究提供理论和方法的参考。
非生物胁迫;转录因子;瞬时表达;转录激活;转基因植物
DIO: 10.13560/j.cnki.biotech.bull.1985.2015.01.008
转录因子(Transcription factor,TF)又称反式作用因子,是直接或间接与基因启动子区域顺式作用元件发生特异性结合、对基因的转录起调节作用的一类蛋白质。转录因子一般具有4个功能结构域:DNA结合区(DNA-binding domain,DBD),是转录因子上识别并结合顺式作用因子的一段氨基酸序列,可与靶位点特异性结合;寡聚化位点区(Oligomerization site,OS),是不同转录因子间发生相互作用的功能域;核定位信号区(Nuclear location signal,NLS),可将转录因子定位于细胞核内的某些区域;转录调控区(Transcription regulation domain,TRD),决定着转录因子对靶基因的调控特征,分为转录激活域(Activation domain,AD)和转录抑制域(Repression domain,RD),对靶基因的转录起激活或抑制作用。
转录因子在植物的生长发育、形态建成及对生物和非生物胁迫中起着重要的作用,在植物基因表达调控系统中转录因子一直备受关注,我们结合近年来非生物胁迫相关转录因子的研究进展,归纳、总结了非生物胁迫相关转录因子研究的主要方法和策略,以期为植物转录因子的鉴定、功能分析及靶基因研究提供理论和技术上的参考。
1 转录因子结构域分析
1.1 生物信息学分析转录因子保守结构域
转录因子序列通常具有保守性,这些保守的结构域(Conserved domains,CD)在进化过程中氨基酸序列变化很小,因此可以利用生物信息学方法对序列进行分析,预测转录因子的结构域。随着生物信息学技术的发展,各种分子生物学数据库提供了大量的核苷酸和氨基酸序列,常用的大型生物数据库包括NCBI、EBI和DDBJ等都为转录因子的研究提供了大量的数据。转录因子有其自己的数据 库, 如TRANSFAC(http://www.gene-regulation. com/pub/ databases.html)和PlnTFDB(http://plntfdb. bio.unipotsdam.de/v3.0/),为转录因子的鉴定、结构和功能预测提供了新的方法[1]。植物转录因子数据库 PlantTFDB database(http://planttfdb.cbi.pku.edu. cn/)中包含多种植物特有的转录因子。Kato等[2]将ANAC036基因的核苷酸序列输入NCBI数据库中,进行在线Blast(http://blast.ncbi.nlm.nih.gov/Blast. cgi),预测ANAC036氨基酸序列具有典型的NAM保守结构域,确定为NAC类转录因子。Lu等[3]研究的SbSNAC1转录因子,是从PlantTFDB数据库中提取出的编码NAC类的转录因子,SbSNAC1转录因子具有典型的NAC结构域,具有抵抗干旱的作用。Zhao等[4]以DREB转录因子保守的AP2结构域为筛选标准,从苹果(Malus pumila Mill.)基因组数据库(http:// genomics. research. iasma. it/)中获得多个苹果中DREB转录因子,并构建了本地BLASTP搜索,为后续对苹果中DREB转录因子家族全基因组分析及表达特性分析奠定基础。模式生物拟南芥(Arabidopsis thaliana L.)和水稻(Oryza sativa L.)等有自己的转录数据库,如拟南芥的转录数据库RARTF(http://rarge.gsc.riken.jp/rartf/)和AGRIS(http://arabidopsis.med.ohio-state.edu/AtTFDB/), 为转录因子的研究提供了大量的数据。Sun等[5]利用水稻基因组数据库(http:// rice. plantbiology. msu. edu/),对分离出的ONAC122和ONAC131进行BLASTn和BLASTp比对,并对ORF(Open reading frame)进行分析,结果显示两基因N末端都具有高度保守的NAC结构域,并包含NAC的5个亚结构域,C末端具有特异性,确定ONAC122和ONAC131属于NAC转录因子家族。本实验室Li等[6]利用生物信息学分析辽宁碱蓬(Suaeda liaotungensis K.)中的一个NAC转录因子SlNAC1发现,其具有NAM结构域,属于NAC家族中的TIP亚家族,Yang等[7]利用此方法分析SlNAC2发现,其具有NAM结构域,属于NAC家族中的ATAF亚家族。目前,对转录因子结构域分析的方法比较局限,生物信息学分析是最常见也是最普遍的方法。
1.2 定点突变分析转录因子结构域的功能位点
定点突变(Site-directed mutagenesis)是在体外增添、缺失或者替换DNA序列中特定碱基的技术,是分析蛋白质中特定的单个或成簇的氨基酸功能的一种方法,也是分析转录因子结构域功能的常见方法。定点突变研究转录因子结构域可在DNA水平和蛋白质水平两个方面进行。DNA水平进行定点突变的方法主要有:(1)寡核苷酸介导的基因突变,是指以含有突变碱基的寡核苷酸片段为引物,在DNA聚合酶的催化下启动DNA复制,该方法保真度高,但操作复杂且周期长;(2)盒式突变,是利用一段含突变序列的人工合成寡核苷酸片段取代野生型基因中的相应序列,该方法简单易行且突变效率高,但需合成多条引物且受到酶切位点的限制;(3)PCR介导的基因突变,是在基因的5'和3'末端产生突变,PCR延伸后再利用另一对引物进行重叠延伸进行基因的定点突变,该方法操作简单突变率高,但后续工作复杂且Taq DNA聚合酶保真性偏低。蛋白质水平进行定点突变的方法主要有两种:丙氨酸置换法和基团置换法。由于丙氨酸疏水性小,侧链碳原子少,因此可作为侧链缺失突变的类似物;基团置换法中用带有突出侧链的氨基酸替代野生型残基的位置,如将Arg替换为Asp,这种方法可以消除原先的接触,又可在空间或化学上破坏蛋白质之间的相互作用。Duan等[8]利用DNA定点突变技术鉴定出AtWRKY1-C的DNA结合结构域位于 β2和β3链上。 Tavares和Ling等[9,10]利用氨基酸定点突变的方法鉴定AtMYB30转录因子的Cys49和Cys53是亚硝基化作用位点,并且是DNA结合域的关键位点。定点突变的方法能够鉴定转录因子结构域中的功能位点,是分析转录因子结构域的重要方法。
2 转录因子亚细胞定位分析
真核生物转录过程主要发生在细胞核中,因而转录因子一般定位在细胞核中,但由于线粒体和叶绿体中也含有少量的DNA和RNA,转录过程在线粒体、叶绿体中也有发生,因此转录因子也可能定位在线粒体或叶绿体中。植物蛋白质亚细胞定位的研究方法有很多,如融合报告基因定位法,免疫组织化学定位法,共分离标记酶辅助定位法,蛋白质组学定位技术等。此外生物信息技术的利用是蛋白质亚细胞定位预测的常用方法。在植物转录因子的研究中,生物信息学分析以及融合报告基因定位法是预测和鉴定转录因子亚细胞定位最常见的方法。
2.1 生物信息学分析转录因子的亚细胞定位
蛋白质的N端或C端有一段指导蛋白质寻靶的氨基酸序列,称为信号序列,可以指导蛋白质的亚细胞定位,如核定位信号(Nuclear localization signal,NSL)能指导蛋白质定位于细胞核中。因此根据蛋白质中的信号序列,利用生物信息学分析可以预测蛋白质的亚细胞定位。主要预测蛋白质亚细胞定位的软件有WoLF-PSORT(http:// wolfpsort. seq. cbrc. jp/)、NNPSL(http://predict.sanger.ac.uk. nnps)、SubLoc(http:// www.bioinfo.tsinghua. edu.cn/ SubLoc)和TargetP(http://www.cbs.dtu.dk/services/ TargetP/),为蛋白质亚细胞定位提供了大量的实验数据。Puranik等[11]利用WoLF-PSORT预测SiNAC转录因子上存在核定位信号、C末端存在α折叠,预测SiNAC的亚细胞定位位于细胞核和质膜上,经实验验证SiNAC转录因子同时存在于细胞核和质膜上。生物信息学分析转录因子的亚细胞定位操作简便,快捷。
2.2 融合报告基因定位法分析转录因子的亚细胞定位
绿色荧光蛋白(Green fluorescent protein,GFP)在488 nm的激发光下会发出绿色荧光,将目的转录因子基因与绿色荧光蛋白基因串联,形成融合表达载体,基因枪法将表达载体导入洋葱表皮细胞中,在共聚焦显微镜下通过观察GFP的位置,从而确定转录因子的亚细胞定位。常用的表达载体有pEGAD、pCAMBIA1302等。Puranik等[11]利用pCAMBIA1302载体分别构建GFP-SiNAC Full length、缺失C末端的GFP-SiNACΔC1-158表达载体,在CaMV35S启动子作用下,以 GFP为对照,基因枪法瞬时转化至洋葱表皮细胞,融合表达48 h,共聚焦显微镜下观察绿色荧光蛋白的位置,在明场、488 nm激发光和DAPI染色下,GFP-SiNAC Full length定位在细胞膜和细胞核中,GFP-SiNACΔC1-158定位在细胞核中(图1),确定SiNAC上存在核定位信号,C末端与膜定位密切联系。Liu等[12]构建TaPIMP1-GFP融合表达载体,基因枪法转化至洋葱表皮细胞,结果显示 TaPIMP1蛋白质仅定位在细胞核中,属于核蛋白。Sun等[13]亚细胞定位试验表明胁迫相关的一个MYB转录因子定位在细胞核中。本实验室Li等[6]构建GFP-SlNAC1融合表达载体,基因枪法转化至洋葱表皮细胞,结果显示GFP-SlNAC1定位在细胞核中;Yang等[7]构建GFP-SlNAC2融合表达载体,基因枪法转化至洋葱表皮细胞,SlNAC2-GFP同样定位在细胞核中。该方法灵敏度高,并且对细胞无毒害,可广泛利用。由于基因枪瞬间转化设备十分昂贵,在一定程度上阻碍了该方法的广泛应用,也可采用农杆菌介导进行转化,但转化效率较低。
3 转录因子转录激活作用分析
转录因子的转录激活活性通常在酵母细胞中进行检测。常用方法是构建转录激活表达载体,转化至酵母菌中,在营养缺陷型培养基上培养,能够生长的酵母菌具有转录激活活性,通过β-半乳糖苷酶染色可进一步确定转录因子的转录激活作用。常用的载体有pGBKT7、pDBLeu等。为检验转录因子具体是哪一段序列具有转录激活活性,可构建多个转录激活载体。Peng等[14]为了鉴定CarNAC5转录因子的激活域位置,分别构建了pDBLeu-F、pDBLeu-N和pDBLeu-C表达载体,以pDBLeu为对照,转入酵母菌AH109中,在His单缺培养基上仅pDBLeu-F和pDBLeu-C的酵母菌才能够生长(图2),β-半乳糖苷酶染色表明CarNAC5转录因子的激活域位于C末端。Sun等[15]根据 AtbZIP1转录因子上的模体不同,分别构建pGBKT7-AtbZIP1,pGBKT7-AtbZ1,pGBKT7-AtbZ2、pGBKT7-AtbZ3及 pGBKT7-AtbZ4,以pGBKT7-AtDREB1A作为阳性对照,用醋酸锂介导法转化将表达载体转化至酵母菌中,在Trp单缺培养基上带有表达载体的酵母细胞均能正常生长,而在Trp-His-Ade三缺培养基上只有pGBKT7-AtbZIP1、pGBKT7-AtbZ1、pGBKT7-AtbZ3能生长,将酵母菌进行β-半乳糖苷酶染色,pGBKT7-AtbZIP1蓝色最深,转录激活最强;pGBKT7-AtbZ1、pGBKT7-AtbZ3颜色较浅,活性相对弱,结果显示AtbZIP1转录因子的N末端和靠近C端的第92-145个氨基酸对转录激活活性是十分必要的。本实验室Li等[6]利用此方法鉴定SlNAC1具有转录激活活性,Yang等[7]鉴定SlNAC2具有转录激活活性。该方法简单易行,无需分离纯化蛋白,且酵母菌属于真核生物,表达的蛋白质处于自然构象,克服了体外研究蛋白质时的非自然构象,因而灵敏度高,并便于检测。该方法的缺点是一方面会出现待鉴定转录因子可能与酵母内源转录因子相互作用从而激活报告基因表达的假阳性结果;另一方面是酵母菌表达的融合蛋白对细胞有毒或蛋白不能在细胞中稳定表达而造成的假阴性结果。
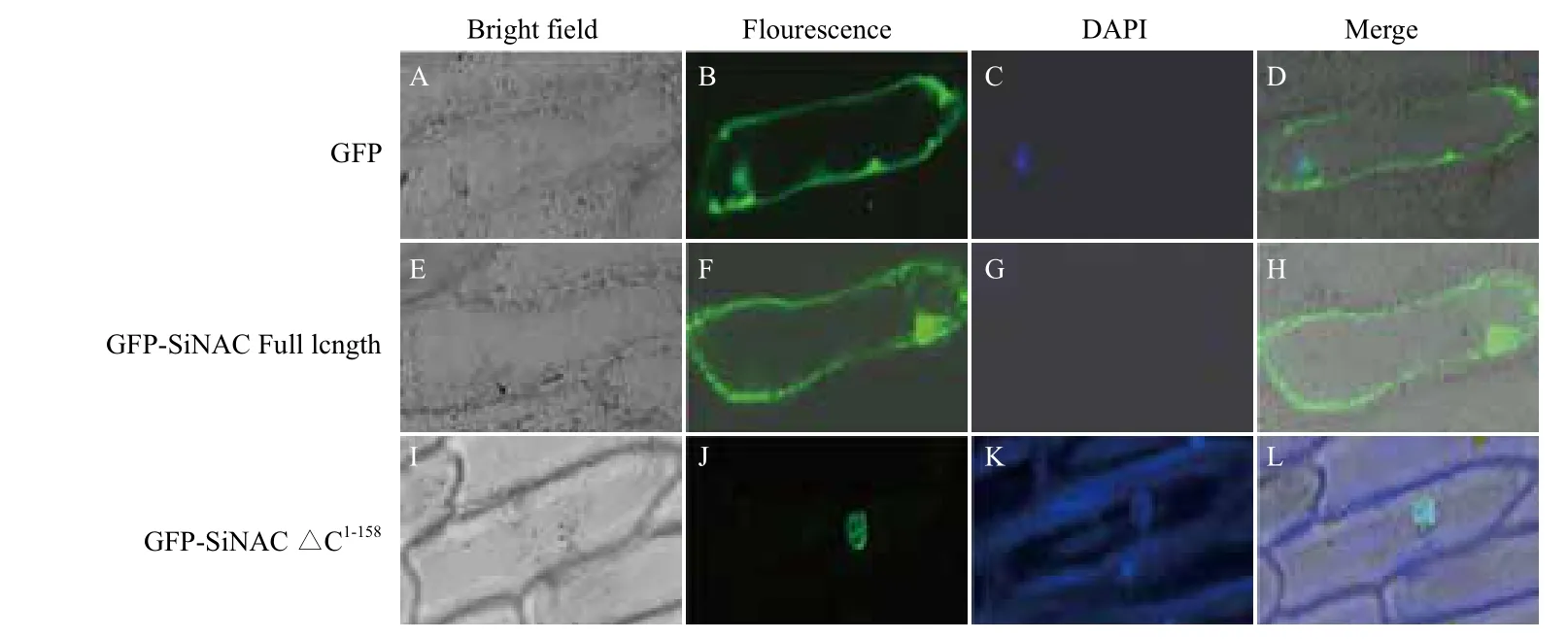
图1 SiNAC在洋葱表皮细胞中的亚细胞定位分析[11]

图2 CarNAC5转录激活活性分析[14]
4 转录因子复合体研究
转录因子作为一种蛋白质,在细胞内并不都是独立存在、单独发挥作用的,它往往与其他蛋白质结合,它们在结构上相互作用,最后以复合体的形式行使功能。鉴定蛋白质与蛋白质之间相互作用的方法有很多,其中酵母双杂交、双分子荧光互补以及免疫共沉淀技术(Co-Immunoprecipitation,Co-IP)、Pull Down等方法最常见,此外还有串联亲和纯化技术、寡沉淀等多种技术。这些方法均可用于植物转录因子复合体研究。
4.1 酵母双杂交
Fields和Song[16]于1989年建立酵母双杂交系统,用于真核生物蛋白质之间相互作用的研究。该方法是将转录因子的DNA结合结构域和转录激活结构域分别与待研究蛋白质融合表达,若蛋白之间存在相互作用,则两个结构域在空间上靠近可激活报告基因的表达(图3)。郑甲成等[17]利用酵母双杂交技术以TaAIDFa为诱饵,从cDNA文库中筛选到84个与TaAIDFa相互作用的序列,Blast比对获得4类候选蛋白,经同源性分析预测与逆境胁迫相关。Li等[18]利用酵母双杂交鉴定拟南芥中RCAR1/PYL9与一个R2R3-MYB类型的AtMYB44之间具有相互作用,促进AtMYB44转录因子发挥作用。这种方法灵敏度高、高通量化,但由于酵母菌内源蛋白质可能被激活而存在着假阳性,且要求待测蛋白为核内蛋白,若在核外发挥作用不能激活报告基因的表达,导致假阴性结果。1998年Karimova等、Ladant等和姜茜等[19-21]分别于1998年、2000年和2008年建立细菌双杂交,弥补了酵母双杂交的缺陷。

图3 酵母双杂交原理示意图
4.2 双分子荧光互补
双分子荧光互补(Bimoleeular fluoreseence complementation,BiFC)方法是将两个不发光的荧光互补片段与待测蛋白融合表达,在待测蛋白的相互作用下,驱动荧光片段重新组装,恢复荧光特性。Li 等和Walter等[18,22]利用BiFC方法鉴定出拟南芥中RCAR1/PYL9与一个R2R3-MYB类型的AtMYB44之间具有相互作用,促进AtMYB44转录因子发挥作用。Grinberg等[23]利用该技术研究3个转录因子家族Myc、Max和Mad在活细胞中蛋白质的相互作用,观察到不同转录因子蛋白间竞争性二聚体的形成。该技术可检测体内蛋白发挥作用的时间、强度及位置等,其缺点是存在假阳性和假阴性结果。
4.3 免疫共沉淀
免疫共沉淀是利用抗原-抗体之间特异性识别并结合的特点来研究体内蛋白质间相互作用的一种方法,是确定两种蛋白质在完整细胞内生理性相互作用的有效方法。其基本原理是在细胞裂解液中加入转录因子蛋白的抗体,孵育后加入与抗体特异结合的Protein A或G,Protein A或G结合于Agarose珠上,若细胞中存在与转录因子结合的目的蛋白,就可以形成“转录因子-抗体-Protein A或G-Agarose珠-目的蛋白”复合物,变性后经免疫印迹或质谱检测就可鉴定目的蛋白。Kawarazaki 等[24]利用免疫共沉淀鉴定AtSRC2与AtRbohF的N末端相互作用。Weis等[25]鉴定拟南芥中的95个蛋白质与GFP相作用。该方法优点是可以分离天然状态下相互作用的蛋白质,缺点是对于低亲和力和瞬间的蛋白质—蛋白质相互作用的复合体可能检测不到,因而可能存在假阴性。
5 转录因子功能研究
对非生物胁迫相关转录因子功能的分析的方法很多,包括通过生物信息学构建系统进化树对其功能进行预测、胁迫条件下鉴定基因表达特性、在基因缺失和过表达条件下检测植株的表型变化及生理变化、检测过表达植株基因表达变化分析转录因子调控的下游效应基因等。综合这些方法,可以对转录因子功能进行全面的分析。
5.1 构建系统进化树预测转录因子功能
系统进化树已经是公认的一种生物进化模型,其主要是依据DNA或氨基酸的相似度构建的。根据已知序列在线Blast比对的结果,找出与已知基因相似度较高的相关基因,利用构建生物进化树的软件,根据亲缘关系大小构建系统进化树,在进化树中找出与该基因亲缘关系最相近的基因,通过相关基因文献的阅读,推测已知基因可能具有的功能。构建系统进化树的方法主要有两大类,距离法和离散特征法。距离法中有UPGM、邻接法(Neighbor-Joining,NJ)等。距离法是根据距离的类聚算法,运算量不大,但在进化树中无法确定进化分支的时间。离散特征法根据最优原则不同,可分为最大似然法(Maximum likelihood,ML)及最大简约法(Maximum parsimony,MP),这种进化树最大的优点就是树中有进化的时间。常用构建生物系统进化树的软件有PHYLIP(http://evolution.genetics.washinton.edu/phylip/software.html)、PAUP(ftp://onyx.si.edu/paup)、MEGA(http://bioinfo.weizmann.ac.il/ databases/info/ mega.sof) 及TreeView(http://taxonomy.zoology.gla. ac.uk/rod/treeview.html)等。Li等[26]将EsDREB2B与来自鹰嘴豆(Cicer arietinum L.)中的CAP2有高达80%的相似度,且具有DREB转录因子典型的缬氨酸(V14)和谷氨酸(E19),EsDREB2B属于AP2/ERF蛋白亚家族,其他成员还包括CAP2、GmDREB2B、OsDREB2B和AtDREB2A,这几个转录因子均具有抵抗干旱的作用,因此预测了EsDREB2B也具有抵抗干旱的作用。Lu等[27]将ZmSNAC1基因序列输入NCBI数据库中进行在线Blast,找出与ZmSNAC1基因相似的序列,利用ClustalW软件将这些相关序列进行多序列比对,同时根据比对结果构建了系统进化树,结果显示ZmSNAC1基因与OsNAC044、OsSNAC1基因和的相似度较高,经查阅文献,OsNAC044、OsSNAC1基因具有抵抗干旱、高盐、低温等非生物胁迫的作用,进而推测ZmSNAC1基因也可能具有抵抗非生物胁迫的功能,为后续实验奠定了基础。Sun等[28]利用ClustalW软件将 ENAC1与大豆、水稻等多个NAC转录因子序列进行比对并构建了系统进化树,结果显示ENAC1转录因子与OsDREB1A同源性最高。Liu等[29]利用系统进化树将苎麻(Boehmeria nivea L.)中32个全长的NAC转录因子与其他物种中的47个功能性的NAC蛋白同源关系进行了分析,结果显示这79个NAC蛋白质可分为8组。Cenci等[30]对香蕉(Musa acuminata L.)中的NAC转录因子家族进行了全基因组分析,并构建系统进化树对其亲缘关系进行分析。本实验室Li等[6]构建进化树表明SlNAC1与拟南芥中AtNAC014亲缘关系最近,通过阅读AtNAC014相关文献,预测SlNAC1响应非生物胁迫。Yang等[7]的进化树结果显示SlNAC2与PaNAC1亲缘关系最大,通过阅读PaNAC1相关文献,预测SlNAC2响应非生物胁迫。目前,通过构建系统进化树预测转录因子功能,这一方法已经十分成熟,操作简单且预测结果可信度高,但该方法要求转录因子序列中要含有保守的结构域,否则预测结果将不准确。
5.2 表达特性分析推测转录因子功能
转录因子是否与植物抗逆相关,需要对逆境条件下转录因子基因的表达特性进行分析。利用半定量PCR(RT-PCR)和实时定量PCR(qRT-PCR)技术可以检测胁迫条件下转录因子基因在不同组织、器官中随胁迫时间变化的相对表达量,进而确定转录因子是否与胁迫相关以及与哪种胁迫相关。Peng等[31]提取不同时间、不同条件胁迫处理下鹰嘴豆中CarNAC3基因的RNA,反转录获得cDNA,以cDNA为模板利用半定量PCR和实时定量PCR技术进行检测,结果表明CarNAC3基因在盛开的花朵中相对表达量高,而在开花后15 d的植株的根、茎、叶中的相对表达量较低。在胁迫条件下,CarNAC3基因表达量有明显的升高,且在高温1 h、干旱12 h、创伤1 h、水杨酸12 h、生长素1 h等胁迫处理下的相对表达量最大,因此CarNAC3基因与高温、干旱等非生物胁迫相关。Tian等[32]分别在低温胁迫下对红掌中的AaDREB1、AaWRKY1和AaNAC1等基因进行半定量PCR和实时定量PCR分析其表达量的变化,结果表明随低温处理时间加长,以上基因的相对表达量上升,有的在24 h表达量最高。Hu等[33]利用qRT-PCR技术分别检测杨树(Populus trichocarpa)中25个NAC转录因子在芽尖、叶、韧皮部、微分化木质部以及根的成熟区和根尖的相对表达量,表明NAC转录因子在不同组织器官中表达量有明显差异。本实验室Li[6]利用qRT-PCR技术分析辽宁碱蓬SlNAC1基因在非生物胁迫下的表达,SlNAC1基因受盐、干旱、低温及ABA诱导表达,推测SlNAC1基因与响应非生物胁迫有关。Yang等[7]利用qRT-PCR技术分析辽宁碱蓬SlNAC2基因在非生物胁迫下的表达,SlNAC2基因受盐、干旱、低温及ABA诱导表达,推测SlNAC2基因与响应非生物胁迫有关。半定量PCR和实时定量PCR在检测基因表达特性中应用比较广泛,为研究提供可靠的数据。该方法中材料RNA的提取十分关键,且相对于RT-PCR,qRT-PCR检测结果更为准确,对分析鉴定转录因子抵抗生物及非生物胁迫响应提供重要证据。
5.3 基因缺失表型分析鉴定转录因子功能
基因缺失引起的表型变化最能体现基因的生理功能。基因功能缺失突变体的获得通常采用插入法和反义RNA抑制法等。插入法将一段DNA插入到目的基因中,使目的基因不表达,包括转座子插入法、T-DNA插入法和同源重组插入法。最常使用的是T-DNA插入法,T-DNA是质粒上一段容易转移到其他基因上的DNA,插入到基因中使目的基因无法表达,在拟南芥中应用十分广泛。Lacroix等[34]利用T-DNA插入法破坏WRKY17 基因,发现WRKY17 突变体植株农杆菌更敏感,表明WRKY17 基因在农杆菌侵染中发挥积极的作用。Lu等[35]将T-DNA插入到ATAF1基因,破坏其结构,以标签基因 COR47、ERD10、KIN1、RD22 和RD29A的表达量变化检测ATAF1的功能,结果显示在干旱胁迫下ataf1-1突变体植株标签基因在2 h、3 h和5 h相对表达量更高,证明ATAF1转录因子在干旱胁迫中发挥重要的作用。Paul等[36]检测到插入了T-DNA的OsTEF1水稻,降低了60%-80%的分蘖,而且出现根部生长阻滞、对高盐更为敏感。T-DNA插入法存在其局限性,若目的基因编码区序列过短,T-DNA插入的概率较小,插入到非编码区或内含子中不一定会引起基因表达的完全缺失,只是使基因表达水平下降。T-DNA插入法主要局限性在于植物中有一定数量的基因是多拷贝的,有的甚至紧密地串联在一起,通常不易获得纯合突变体。
反义RNA抑制技术又称为RNA干扰(RNAi)技术,是将目的基因全部或部分的反向序列转入植物中,转录时就会与目的基因转录产物按照碱基互补配对原则形成杂交双链,阻止目的基因的表达,从而获得功能缺失突变体植株。与RNAi相比,人工miRNA(Artificial microRNA,amiRNA)来源于RNA一个短的茎环,只产生一个单一的小的RNA,能更精准的抑制相关靶基因的表达。目前已有Web MicroRNA designer(http://wmd3.weigelworld.org)和THE RNAi WEB(http://www.rnaiweb.com)工具用于amiRNA的设计。Song等[37]为检测OsNAC5转录因子对非生物胁迫的响应,利用RNAi技术将水稻中OsNAC5基因敲除,与野生型水稻相对照,在干旱、高盐、冷等胁迫条件下,敲除OsNAC5的水稻植株对胁迫具有更强的抵抗能力。基因缺失表型分析只适用于模式植物,对于一些非模式植物操作有困难。
5.4 基因过表达表型分析鉴定转录因子功能
基因功能增加通常采用过表达法,又称超表达法,是指在目的基因前加一个组成型的强启动子,如CaMV 35S启动子,转化植物,使转录因子在转基因植物中大量表达,从而促进下游基因的大量表达,从而改变转基因植物的性状,分析该转录因子的抗逆功能。Bouaziz等[38]利用基因过表达的方法证明,StDREB1基因的过量表达能提高转基因马铃薯对盐胁迫的抵抗力,在盐胁迫下植株能健康生长。Takaskki等[39]以CaMV35S启动子驱动OsNAC5转录因子基因,转化至野生型拟南芥中过量表达,过表达的拟南芥植株在250 mmol/L NaCl溶液胁迫处理下,比较野生型抵抗力更强,证明OsNAC5具有抵抗高盐胁迫的作用。Xiong 等[40]研究在OsMYB48-1基因过表达情况下,水稻增强了对干旱和高盐胁迫的响应。Yu等[41]将鹰嘴豆中CarNAC2基因过表达,结果显示过表达转基因拟南芥抗旱能力更强,但对种子的发芽具有延缓作用。本实验室获得的过表达SlNAC1基因拟南芥的盐、干旱、低温耐受性增强[6]、过表达SlNAC2基因拟南芥[7]的抗盐性增强,表明SlNAC1、SlNAC2能提高植物抗非生物胁迫的能力。
基因过表达鉴定植物的表型已经得到了广泛的应用,但也存在着一定的局限性。由于许多转录因子都是以蛋白复合体的形式行使功能,只过量表达其中的一个蛋白质,植物一般不会发生明显的表型变化,另外,由于有些基因的表达是转录因子和其他因素共同作用的结果,因此转录因子的过量表达也并不一定总是引起下游基因的变化。
5.5 下游相关基因分析鉴定转录因子功能
转录因子行使生理功能的过程十分复杂,涉及转录因子所调控的下游基因和与其相互作用的其他转录因子,对转录因子下游基因的研究可以进一步明确转录因子的功能。
5.5.1 染色质免疫沉淀-DNA基因芯片分析 染色质免疫沉淀-DNA基因芯片(Chromatin immunoprecipitation-gene chip)是研究蛋白质与DNA相互作用的有效方法,可用于转录因子相关靶基因的筛选。染色质免疫沉淀是在活细胞状态下用甲醇固定蛋白质-DNA复合物,用酶切或超声波将复合物随机切断并分为一定大小的片段,利用特异性的抗体结合此复合体上的转录因子使其沉淀,通过水解释放复合体中的DNA,即可得到目的DNA片段。利用基因芯片与目的DNA进行杂交,可以获得DNA序列信息。Zhu等[42]利用染色质免疫沉淀-DNA基因芯片相结合的技术成功鉴定拟南芥和水稻中与BZR1转录因子直接作用的靶基因。
5.5.2 酵母单杂交分析 酵母单杂交也可以鉴定转录因子调控的下游相关基因。其原理是将编码待测转录因子的cDNA与AD形成融合表达载体并导入酵母菌细胞中,该基因产物若能与顺式作用原件(cis)结合,就能激活Pmin启动子,使报告基因得以表达,从而筛选出与已知转录因子相结合的顺式作用元件(图4)。酵母单杂交方法是在细胞内分析鉴定转录因子与DNA结合的有效方法,在基因表达调控机制的研究中是一个很有价值的工具,通过酵母单杂交可以在大范围内确定蛋白质与DNA的相互作用,实现高通量筛选。该方法虽然能快速分析转录因子的DNA结合特性和转录调控特性,但在综合分析其功能具有一定局限性,因此需要与植株体内功能分析相配合。

图4 酵母单杂交分析原理示意图
5.5.3 过表达植株基因表达变化分析 利用实时定量PCR分析转录因子基因过表达植株中已知胁迫相关基因的表达变化,可以分析该转录因子调控的下游基因。Xiao等[43]利用实时定量PCR技术检测PeMYB2基因过表达拟南芥中与干旱胁迫相关的NXH1、SOS1、RD29A和 COR15A基因表达量有明显上升,可推测PeMYB2基因与NXH1、SOS1、RD29A和 COR15A表达相关。Yu等[41]研究CarNAC2基因过表达时,RD22、RD29A、COR15A、KIN1在干旱情况下表达量也明显上升,表明这4个基因可能是CarNAC2转录因子作用的下游基因。转录因子的超表达可能使转录因子与不相关启动子中亲和力较低的结合位点结合,从而激活一些不相关基因的表达,因此很难估计哪些基因是受目的转录因子的直接调控,哪些基因受其间接调控。
基因芯片(Gene chip)可以分析转录因子基因过表达植株中基因表达的变化。构建野生型植株、转录因子基因过表达植株的基因芯片,通过筛选分析转录因子基因过表达植株中上调或下调表达的基因,经qRT-PCR验证后,确认转录因子调控的下游基因。
6 展望
转录因子在植物生长发育以及抵抗非生物胁迫方面发挥着重要作用。对转录因子进行深入、细致的研究,更好地理解转录因子的结构和功能显得尤为重要。随着生物技术的不断发展,转录因子的研究方法将不断丰富,将极大推动植物非生物胁迫相关转录因子的研究。
[1]王传琦, 孔稳稳, 李晶. 植物转录因子最新研究方法[J]. 生物技术通讯, 2013(1):118-123.
[2]Kato H, Motomura T, Komeda Y, et al. Overexpression of the NAC transcription factor family gene ANAC036 results in a dwarf phenotype in Arabidopsis thaliana[J]. Journal of Plant Physiology,2010, 167(1):571-577.
[3]Lu M, Zhang DF, Shi YS, et al. Expression of SbSNAC1, a NAC transcription factor from sorghum, confers drought tolerance to transgenic Arabidopsis[J]. Plant Cell Tiss, 2013, 115(3):443-455.
[4]Zhao T, Liang D, Wang P, et al. Genome-wide analysis and expression profiling of the DREB transcription factor gene family in Malus under abiotic stress[J]. Molecular Genetics and Genomics,2012, 287(5):423-436.
[5]Sun L, Zhang H, Li D, et al. Functions of rice NAC transcriptional factors, ONAC122 and ONAC131, in defense responses against Magnaporthe grisea[J]. Plant Molecular Biology, 2012, 81(1-2):41-56.
[6]Li XL, Yang X, Hu YX, et al. A novel NAC transcription factor from Suaeda liaotungensis K. enhanced transgenic Arabidopsis drought,salt, and cold stress tolerance[J]. Plant Cell Rep, 2014, 33(5):767-778.
[7]Yang X, Hu YX, Li XL, et al. Molecular characterization and function analysis of SlNAC2 in Suaeda liaotungensis K. [J]. Gene,2014, 543(2):190-197.
[8]Duan MR, Nan J, Liang YH, et al. DNA binding mechanism revealed by high resolution crystal structure of Arabidopsis thaliana WRKY1 protein[J]. Nucleic Acids Res, 2007, 35(4):1145-1154.
[9]Tavares CP, Vernal J, Delena RA, et al. S-nitrosylation influences the structure and DNA binding activity of AtMYB30 transcription factor from Arabidopsis thaliana[J]. Biochimica et Biophysica Acta, 2014, 1844(2014):810-817.
[10] Ling MM, Robinson BH. Approaches to DNA mutagenesis:an overview[J]. Anal Biochem, 1997, 254(2)157-178.
[11] Puranik S, Bahadur RP, Srivastava PS, Prasad M. Molecular cloning and characterization of a membrane associated NAC family gene,SiNAC from foxtail mill[Setaria italica(L. )P. Beauv][J]. Mol Biotechnol, 2011, 49(2):138-150.
[12] Liu HX, Zhou XY, Dong N, et al. Expression of a wheat MYB gene in transgenic tobacco enhances resistance to Ralstonia solanacearum, and to drought and salt stresses[J]. Functional & Integrative Genomics, 2011, 11(3):431-443.
[13] Sun P, Zhu X, Huang X, Liu JH. Overexpression of a stress-responsive MYB transcription factor of Poncirus trifoliata confers enhanced dehydration tolerance and increases polyamine biosynthesis[J]. Plant Physiol Biochem, 2014, 78(1):71-79.
[14] Peng H, Cheng HY, Yu XW, et al. Characterization of a chickpea(Cicer arietinum L. )NAC family gene, CarNAC5, which is both developmentally- and stress-regulated[J]. Plant Physiol Biochem, 2009, 47(11-12):1037-1045.
[15] Sun XL, Li Y, Cai H, et al. The Arabidopsis AtbZIP1 transcription factor is a positive regulator of plant tolerance to salt, osmotic and drought stresses[J]. Journal of Plant Research, 2012, 125(3):429-438.
[16] Fields S, Song O. A novel genetic system to detect protein-protein interactions[J]. Nature, 1989, 340(6230):245-246.
[17]郑甲成, 徐兆师, 胡银岗, 等. 小麦DREB转录因子TaAIDFa的互作蛋白筛选[J]. 麦类作物学报, 2010, 30(2):189-193.
[18]Li D, Li Y, Zhang L, et al. Arabidopsis ABA receptor RCAR1/ PYL9 interacts with an R2R3-Type MYB transcription factor,AtMYB44[J]. Int J Mol Sci, 2014, 15(5):8473-8490.
[19]Karimova G, Pidoux J, Ullmann A, et al. A bacterial two-hybrid system based on a reconstituted signal transduction pathway[J]. Proc Natl Acad Sci USA, 1998, 95(10):5752-5756.
[20]Ladant D, Karimova G. Genetic systems for analyzing proteinprotein interactions in bacteria[J]. Res Microbiol, 2000, 151(9):711-720.
[21]姜茜, 贾凌云. 蛋白质相互作用研究的新技术与新方法[J].中国生物化学与分子生物学报, 2008, 24(10):974-979.
[22]Walter M, Chaban C, Schutze K, et al. Visualization of protein interaetions in living plant cells using bimolecular fluorescence complementation[J]. The Plant Journal, 2004, 40:428-438.
[23]Grinberg AV, Hu CD, Kerppola TK. Visualization of Myc/Max/Mad family dimers and the competition for dimerization in living cells[J]. Mol Cell Biol, 2004, 24(10):4294-4308.
[24]Kawarazaki T, Kimura S, Iizuka A, et al. A low temperatureinducible protein AtSRC2 enhances the ROS-producing activity of NADPH oxidase AtRbohF[J]. Biochim Biophys Acta, 2013,1833(12):2775-2780.
[25]Weis C, Pfeilmeier S, Glawischnig E, et al. Co-immunoprecipitationbased identification of putative BAX INHIBITOR-1-interacting proteins involved in cell death regulation and plant-powdery mildew interactions[J]. Mol Plant Pathol, 2013, 14(8):791-802.
[26]Li XH, Zhang DY, Li HY, et al. EsDREB2B, a novel truncated DREB2-type transcription factor in the desert legume Eremospartonsongoricum, enhances tolerance to multiple abiotic stresses in yeast and transgenic tobacco[J]. BMC Plant Biology, 2014, 14:44.
[27]Lu M, Ying S, Zhang DF, et al. A maize stress responsive NAC transcription factor, ZmSNAC1, confers enhanced tolerance to dehydration in transgenic Arabidopsis[J]. Plant Cell Rep, 2012,31(9):1701-1711.
[28]Sun H, Huang X, Xu XJ, et al. ENAC1, a NAC Transcription factor,is an early and transient response regulator induced by abiotic stress in rice(Oryza sativa L. )[J]. Molecular Biotechnology,2012, 52(2):101-110.
[29]Liu T, Zhu S, Tang Q, Tang S. Identification of 32 full-length NAC transcription factors in ramie(Boehmeria nivea L. Gaud)and characterization of the expression pattern of these genes[J]. Molecular Genetics and Genomics, 2014, 289(4):675-684.
[30]Cenci A, Guignon V, Roux N, Rouard M. Genomic analysis of NAC transcription factors in banana(Musa acuminata)and definition of NAC orthologous groups for monocots and dicots[J]. Plant Molecular Biology, 2014, 85(1-2):63-80.
[31]Peng H, Cheng HY, Chen C, et al. A NAC transcription factor gene of chickpea(Cicer arietinum), CarNAC3, is involved in drought stress response and various developmental processes[J]. Journal of Plant Physiology, 2009, 166(17):1934-1945.
[32]Tian DQ, Pan XY, Yu YM, et al. De novo characterization of the Anthurium transcriptome and analysis of its digital gene expression under cold stress[J]. BMC Genomics, 2013, 14:827.
[33]Hu RB, Qi G, Kong YZ, et al. Comprehensive analysis of NAC domain transcription factor gene family in Populus trichocarpa[J]. BMC Plant Biology, 2010, 10:145.
[34]Lacroix B, Citovsky V. A mutation in negative regulator of basal resistance WRKY17 of Arabidopsis increases susceptibility to Agrobacterium-mediated genetic transformation[J]. F1000Res,2013, 2(33):doi:10. 12688/f1000.
[35]Lu PL, Chen N, An R, et al. A novel drought-inducible gene,ATAF1, encodes a NAC family protein that negatively regulates the expression of stress-responsive genes in Arabidopsis[J]. Plant Molecular Biology, 2007, 663(2):289-305.
[36]Paul P, Awasthi A, Rai AK, et al. Reduced tillering in Basmati rice T-DNA insertional mutant OsTEF1 associates with differential expression of stress related genes and transcription factors[J]. Functional & Integrative Genomics, 2012, 12(2):291-304.
[37]Song SY, Chen Y, Chen J, et al. Physiological mechanisms underlying OsNAC5-dependent tolerance of rice plants to abiotic stress[J]. Planta, 2011, 234(2):331-345.
[38]Bouaziz D, Pirrello J, Charfeddine M, et al. Overexpression of StDREB1 transcription factor increases tolerance to salt in transgenic potato plants[J]. Molecular Biotechnology, 2013, 54(3):803-817.
[39]Takasaki H, Maruyama K, Kidokoro S, et al. The abiotic stressresponsive NAC-type transcription factor OsNAC5 regulates stressinducible genes and stress tolerance in rice[J]. Mol Genet Genomics, 2010, 284(3):173-183.
[40]Xiong H, Li J, Liu P, et al. Overexpression of OsMYB48-1, a novel MYB-related transcription factor, enhances drought and salinity tolerance in rice[J]. PLoS One, 2014, 9(3):e92913.
[41]Yu XW, Peng H, Liu YM, et al. CarNAC2, a novel NAC transcription factor in chickpea(Cicer arietinum L. ), is associated with drought-response and various developmental processes in transgenic Arabidopsis[J]. Plant Biol, 2014, 57(1):55-66.
[42] Zhu JY, Sun Y, Wang ZY. Genome-wide identification of transcription factor-binding sites in plants using chromatin immunoprecipitation followed by microarray(ChIP-chip)or sequencing(ChIP-seq)[J]. Plant Signalling Networks, 2012, 876(1):173-188.
[43]Xiao DC, Zhang ZJ, Xu YW, et al. Cloning and functional analysis of Phyllostachys edulis MYB transcription factor PeMYB2[J]. Yi Chuan, 2013, 35(10):1217-1215.
(责任编辑 狄艳红)
Research Methods of Abiotic Stress-related Transcription Factors in Plants
Su Mingxing Sun Yinghao Shi He Li Qiuli
(Key Laboratory of Plant Biotechnlogy of Liaoning Province, College of Life Sciences, Liaoning Normal University,Dalian 116081)
Transcription factor is a kind of proteins which bind to the cis element region of the promoters specifically and a kind of transcription regulatory factors, and it is also one of the biggest gene families in plants. Many transcription factors can regulate the expression of downstream genes, and play important roles on plant growth and development, morphogenesis, hormone adjustment, and resistance to a variety of biological and abiotic stress. In combination with the research progress of transcription factors in recent years, we have summarized main strategies and methods of transcription factors to the abiotic stress, including the transcription factor domain structure, subcellular localization and transcriptional activation, transcription factor complex and functions of transcription factors, to provide related research theories and methods to plant transcription factors for reference.
abiotic stress;transcription factor;transient expression;transcriptional activation;transgenic plants
2014-04-14
国家自然科学基金项目(31340052),辽宁省自然科学基金项目(2013020069)
宿明星,女,硕士研究生,研究方向:植物分子生物学与基因工程;E-mail:sumingxing8866@163.com
李秋莉,女,博士,教授,研究方向:植物分子生物学与基因工程;E-mail:skyliqiuli@163.com
