长链非编码RNA在血液系统肿瘤中作用的研究进展
2015-10-22胡婉莉高艾
胡婉莉,高艾
1. 首都医科大学基础医学院2012级7年制临床医学3班, 北京 100069;
2. 首都医科大学公共卫生学院, 北京 100069
长链非编码RNA在血液系统肿瘤中作用的研究进展
胡婉莉1,高艾2
1. 首都医科大学基础医学院2012级7年制临床医学3班, 北京 100069;
2. 首都医科大学公共卫生学院, 北京 100069
随着分子生物学技术的飞速发展,人们对于长链非编码RNA(Long non-coding RNA,lncRNA)的研究越来越深入。lncRNA不仅在生物体正常生物活动中不可或缺,还在许多疾病尤其是肿瘤中扮演重要角色。已有的研究表明多种lncRNA与血液系统肿瘤密切相关,具有影响抑癌基因p15表达、p53蛋白功能,以及与miRNA相互作用参与疾病等功能。本文综述了血液系统肿瘤相关的lncRNA并着重介绍与p15、p53、miRNA有关的lncRNA以及它们的相互作用在疾病中发挥的功能,以期能够全面了解血液系统肿瘤相关lncRNA的作用特点,为血液系统肿瘤的研究、诊断以及治疗提供新的思路。
lncRNA;白血病;p15;p53;miRNA
长链非编码RNA(Long non-coding RNA,lncRNA)是长度大于200nt、无明显蛋白质编码功能的RNA[1,2]。根据编码lncRNA的基因在基因组中的位置,lncRNA可分为3类:基因间lncRNA(Long intergenic RNA,lincRNA),天然反义lncRNA以及内含子lncRNA[3]。
lncRNA是具有重要生物学功能的生物大分子,对于基因表达水平的调控具有重要作用。它主要通过表观遗传学、转录水平以及转录后水平三方面调控基因表达[4]。大量研究表明,在许多疾病尤其是肿瘤中存在着特定lncRNA的表达水平异常的现象。这一现象提示lncRNA可能是疾病发生的一个重要因素。而在血液系统肿瘤中也存在着许多表达异常的lncRNA,如ANRIL[5]、MALAT1[6]、Gas5[6]等等,提示lncRNA可能在血液系统肿瘤的发生发展扮演重要角色。随着研究的飞速发展,越来越多lncRNA在血液系统肿瘤的作用正在逐渐被揭示。本文将以与血液系统肿瘤相关的lncRNA为重点展开综述,以期全面了解这些lncRNA在疾病中的作用机制,为血液系统肿瘤的研究、诊断以及治疗提供新的思路。
1 lncRNA的功能
lncRNA是具有重要功能的生物大分子,在许多生物过程中如染色体失活、染色体修饰、基因印记、细胞分化调控、细胞周期调控等发挥关键作用。在这些过程中,lncRNA的功能主要表现在3个方面:表观遗传学修饰、转录水平调控以及转录后调控[4]。根据其效应机制的不同,lncRNA的多种复杂作用可以分为:(1) 作为信号分子,在特定时间和特定组织中表达,调控某些基因的表达;(2) 作为诱饵分子,吸附特定miRNA,调控这些miRNA靶基因表达,这种lncRNA也被称作竞争性内源RNA(Competing endogenous RNA,ceRNA);(3) 作为引导分子,引导与RNA结合蛋白结合的复合体到达调控位点,通过某些途径调节有关基因的表达;(4) 作为支架分子,为相关分子的装配提供中央平台等4种作用机制[3]。lncRNA通过这些机制发挥正常的生物功能,一旦lncRNA的表达异常,lncRNA的功能可能也会随之出现异常,引发疾病。
lncRNA在人类疾病尤其是癌症发生发展中有重要作用[7]。流行病学研究发现,某些lncRNA的异常表达参与多种肿瘤发生,并影响肿瘤转移及预后[8]。例如,目前研究较为明确的HOX基因转录反义RNA(HOX transcript antisense RNA,HOTAIR),它在多种癌症中出现异常高表达[9~11]。体内外研究证明过表达HOTAIR还与乳腺癌转移密切相关[12],并且HOTAIR对于肝癌患者的术后预后评估以及耐药性也存在一定影响[13]。lncRNA对于肿瘤的诊断、治疗以及预后都有着重要意义。不仅如此,目前已有越来越多证据表明lncRNA在血液系统肿瘤中也发挥至关重要的作用。
2 lncRNA在血液系统肿瘤中的作用
lncRNA与血液系统肿瘤关系密切,许多白血病中存在lncRNA表达异常。Isin等[6]挑选5种lncRNA(TUG1、lincRNA-p21、MALAT1、HOTAIR和Gas5),通过分析对比它们在慢性淋巴细胞白血病(Chronic lymphoblastic leukemia,CLL)、多发性骨髓瘤(Multiple myeloma,MM)中与正常人的血浆循环lncRNA的表达水平差异,发现在疾病中这5种lncRNA表达水平与正常人存在差异。lincRNA-p21在CLL中表达低于正常;在MM中MALAT1、HOTAIR、Gas5表达低于正常,TUG1高于正常。研究者认为这5种lncRNA与CLL或MM存在很大关联,并可作为潜在的生物标记。Zhang等[14]发现在21例急性T淋巴细胞白血病(Acute T lymphocyte leukemia,T-ALL)患者中有11例患者的肿瘤细胞中存在一种新的lncRNA(T-ALL-R-lncR1),而人的正常组织中并未检测出这种lncRNA,提示它的异常表达可能促进T淋巴细胞白血病的发生。他们还发现抑制T-ALL-R-lncR1的表达促使T-ALL Jurkat细胞易发生Par-4介导的细胞凋亡;促进Par-4/THAP1蛋白质复合体的形成,激活caspase-3并增加促细胞凋亡Smac蛋白的表达。这表明抑制T-ALL-R-LncR1可能作为潜在治疗T-ALL的一种手段。
lncRNA在血液系统肿瘤中的作用方式复杂多样,例如通过染色质重塑、表观遗传学沉默等方式抑制抑癌基因的表达或激活癌基因等[15]。高艾课题组报道了lncRNA与苯血液毒性的关系,发现lncRNA NR_045623和NR_028291通过免疫调控的方式参与苯血液毒性的发生[16]。目前大量lncRNA在血液系统肿瘤中的功能已经被揭示(表1)。其中,很多lncRNA具有重要的调控基因表达功能,如ANRIL具有调控p15基因的表达的功能。有的lncRNA构成一个p53调控网络,调控或受p53调控,并影响p53信号通路[17],如MEG3影响鼠双微体基因2(Murine double minute2, MDM2)基因表达,下调MDM2表达,增加p53蛋白[18];有的lncRNA与miRNA互相作用影响miRNA靶基因的表达。已有研究表明在白血病中存在p15基因表达异常,p53基因缺失或异常甲基化。miRNA也是白血病发生的一个重要因素。影响p15基因表达、p53蛋白功能
以及与miRNA之间的相互作用可能是lncRNA、p15、p53或miRNA影响疾病发生的关键。
2.1调控p15基因表达的lncRNA分子
p15位于染色体9p21,编码的p15蛋白与细胞周期蛋白D1(Cylin D1)竞争性结合细胞周期蛋白依赖性激酶4/6(CDK4/6),抑制Cyclin-CDK复合体形成,阻遏细胞从G1期进入S期,调控细胞周期[19]。p15基因功能缺失会导致细胞周期调控紊乱,引起细胞无限增值,导致肿瘤的形成[20]。已有许多研究发现在白血病中存在p15基因失活。Rosu-Myles等[21]发现急性髓系白血病(Acute myelogenous leukemia,AML)和骨髓异常增生综合征(Myelodysplastic syndrome,MDS)中存在p15基因沉默。郑瑞玑等[22]认为p15基因失活可以作为急性白血病疾病进展、复发以及预后的指标之一。MDS患者的p15出现异常甲基化易转化发展为急性白血病[21,23]。p15的基因沉默、基因失活或者DNA甲基化会影响p15的正常表达和功能,而有些lncRNA则具有通过表观遗传学调控影响p15表达的功能。
2.1.1p15反义编码的非编码RNA
Yu等[24]发现p15反义编码的一个非编码RNA (p15 antisense,p15AS),具有调控p15基因表达的功能。他们在16例急性白血病患者(5例急性淋巴细胞白血病(Acute lymphoblastic leukemia,ALL)和11例AML)中检测到69%的患者存在p15AS表达水平增高,同时p15表达水平降低。进一步的研究发现p15AS能够调控p15甲基化引起p15基因沉默,促进急性白血病的发生。
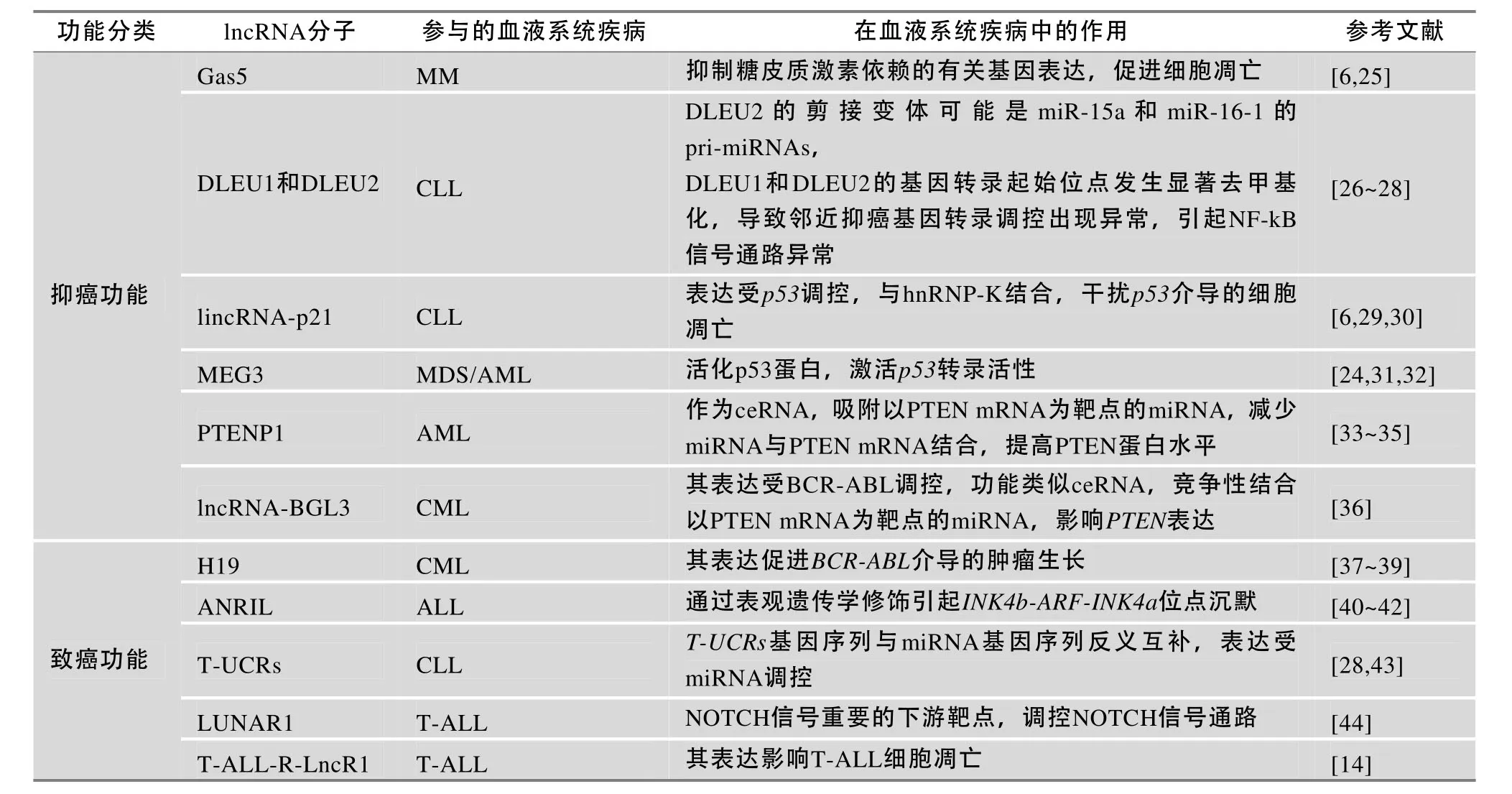
表1 与血液系统疾病相关的lncRNA
2.1.2lncRNA ANRIL
INK4位点反义非编码RNA(Antisense non-coding RNA in the INK4 locus,ANRIL),也被称作CDKN2B-AS[40],在神经母细胞瘤、ALL、黑色素瘤等肿瘤中均发现ANRIL的表达异常[5]。ANRIL能够调控INK4b-ARF-INK4a位点表达。近40%的癌症中存在INK4b-ARF-INK4a位点的调控异常[45]。ANRIL与多梳蛋白抑制复合体2(Polycomb repressive complex 2,PRC2)亚单位SUZ12结合,募集复合物至p15,引起H3K27三甲基化,通过组蛋白修饰引起p15基因表达抑制[41]。此外,ANRIL基因多态性也与ALL的发生有关。Iacobucci等[42]通过对费城染色体呈阳性的ALL和AML患者的研究,认为ANRIL基因rs564398位点的多态性可以引起CDKN2A/B表达谱的改变,促进细胞异常增殖,增加ALL易感性。ANRIL还能与多梳抑制复合物1 (Polycomb repressive complex 1,PCR1)亚单位 CBX7(Chromobox7)结合,招募PCR1至INK4a/ARF位点,引起H3K27的三甲基化,介导基因沉默[31],调控INK4a/ARF的表达。
2.2影响p53功能的lncRNA分子
ANRIL不仅调控p15基因表达,还能通过影响INK4a/ARF位点的表达间接影响p53蛋白的功能。ANRIL与INK4b-ARF-INK4a位点、MDM2、以及p53蛋白具有相互调控作用的网络,共同影响疾病的发生发展(图1)。p53基因具有调控细胞周期、促进DNA损伤修复以及诱导细胞凋亡等作用,有超过一半肿瘤存在p53基因异常[46]。在血液系统肿瘤中也存在p53基因异常。张建清等[47]发现在50例复杂核型异常的白血病患者中有48%患者存在p53基因缺失,而24例非复杂核型异常的患者中p53缺失率为4%,提示p53基因缺失与白血病复杂核型相关。此外林东军等[48]研究发现38.7%的ALL和急性非淋巴细胞性白血病的患者存在p53基因第1启动子异常甲基化,而正常对照组中未检测到p53基因甲基化。p53基因的缺失或异常甲基化,影响p53表达、影响p53蛋白发挥正常功能。研究发现多种lncRNA能影响p53的表达。例如在胃癌中lncRNA H19导致p53的部分失活[49],linc-ROR通过与核不均一核糖核蛋白-I(hnRNP-I)相互作用抑制p53翻译[50]等。不仅如此还有许多其他的lncRNA可以直接或间接影响p53蛋白功能或表达受p53调控,它们共同构成一个p53调控网络,影响p53信号通路[17]。有人将这些lncRNA分为两组:一组lncRNA的表达受p53调节,作为p53的一种效应器(effector),对p53信号通路有影响,如lincRNA-p21、H19;另一组lncRNA作为一种p53的调节器(regulator),如MALAT1和MEG3,而这种调节主要是转录后调节[17]。在血液系统肿瘤中也存在这种p53调控网络的影响。
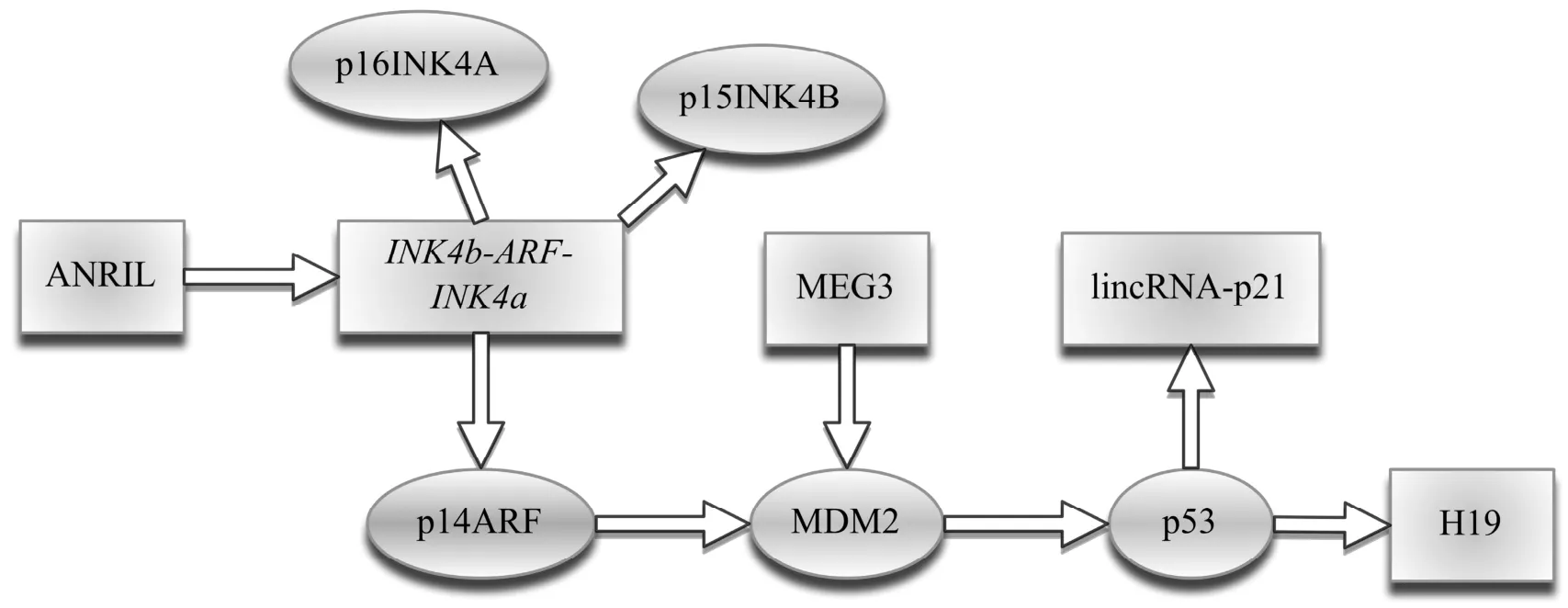
图1 ANRIL、INK4b-ARF-INK4a位点、MDM2以及p53蛋白之间的相互调控作用的网络ANRIL、MEG3、lincRNA-p21、H19是与白血病相关的lncRNA分子:ANRIL具有调控INK4b-ARF-INK4a位点表达的功能[31,41]。INK4b-ARF-INK4a位点编码蛋白p14ARF、p15INK4B、p16INK4A。p14ARF蛋白具有结合MDM2,稳定p53蛋白功能[51]。MEG3能够下调MDM2的表达,增强p53稳定性[18]。lincRNA-p21的表达受p53调控,它能够干扰p53调节的细胞凋亡基因的表达,影响p53依赖的转录反应[29,30]。lncRNA H19表达受p53调控,同时H19能导致p53基因的部分失活[17,49].
2.2.1lncRNA MEG3
母系表达基因3(Maternally expressed gene 3,MEG3)位于染色体14q 32.3,表达的lncRNA MEG3与人类多种肿瘤密切相关。在多种正常组织特别是脑组织中MEG3呈高表达,但一些肿瘤细胞中几乎不表达MEG3,提示MEG3可能具有抑癌作用[52]。后续研究发现多种肿瘤细胞均不表达MEG3(包括脑组织、膀胱、骨髓、乳腺、子宫、结肠、肝脏、肺、脑膜和前列腺的肿瘤组织或者癌细胞株),这似乎是一个普遍现象[52]。而MEG3的启动子高甲基化会下调MEG3的表达,可能促进肿瘤发生[53]。Benetatos等[53]在对MDS和AML患者的MEG3、SNRPN启动子异常甲基化的研究中发现约有48%AML和35%MDS的患者存在MEG3异常甲基化。然而这与世界卫生组织(WHO)基于预后评分系统分类、核型或WHO疾病亚型之间没有显著相关性。尽管如此,MEG3的高甲基化与AML病人的总体生存率降低存在联系,提示MEG3与血液系统肿瘤的联系值得进一步研究。
Zhou等[54]发现人类癌细胞株转染MEG3引起p53蛋白显著增加。通常p53蛋白水平降低是由于泛素-蛋白酶体通路导致的快速降解,p53泛素化主要由MDM2泛素连接酶引起,抑制MDM2的功能对于维持p53稳定性十分重要。而MEG3具有下调MDM2表达的功能,导致p53活化,增强依赖p53的转录。在p53缺乏的情况下,MEG3能够抑制细胞增殖。提示MEG3具有抑制肿瘤发生的作用[18,54,55]。朱娟娟等[56]发现在SK-Hep-1肝癌细胞中MEG3过表达能够激活p53转录活性。这些研究提示MEG3可能与AML或MDS有关,具有抑癌作用,并且能够通过具有下调MDM2表达,增强p53的功能。
2.2.2lincRNA-p21
受p53诱导表达的lincRNA-p21定位于6p21.2,能够与核不均一核糖核蛋白-k(hnRNP-K)结合,干扰p53调节的细胞凋亡基因的表达[29,30],影响p53依赖的转录反应,是调控细胞凋亡的一个关键因素。Isin等[6]挑选并分析5种lncRNA(TUG1、lincRNA-p21、MALAT1、 HOTAIR和Gas5)对比它们在CLL、MM与正常人血浆中的含量差异,发现在疾病中它们的表达水平与正常人存在差异。其中lincRNA-p21在 CLL中的表达水平低于正常水平,提示这可能与CLL的发生存在联系。
综上所述,与血液系统肿瘤相关的lincRNA-p21和MEG3共同参与p53的调控网络:MEG3增强p35活性,激活p53发生转录;lincRNA-p21表达受p53调控,影响p53依赖的转录反应。当lincRNA-p21或MEG3表达出现异常时,p53调控网络可能出现异常,影响p53功能以及p53信号通路,参与白血病的发生。清晰地了解lincRNA-p21或MEG3与p53的调控网络的关系,有助于全面认识它们在血液系统肿瘤中发挥的功能。而这些lncRNA也可能作为白血病潜在治疗靶点。
2.3与miRNA相互作用的lncRNA分子
miRNA属于非编码RNA的一种,在人类各种疾病尤其是血液系统肿瘤中具有重要作用。lncRNA与miRNA的关系十分密切。有些lncRNA是miRNA的前体,如lncRNA H19是miR-675的前体[57];有些lncRNA与miRNA结合产生作用。Salmena等[58]提出的ceRNA假说中提到lncRNA通过自身miRNA应答元件(miRNA response elements,MREs)竞争性结合miRNA,调控有关基因的表达。人们对于miRNA的认识比较全面,大量资料表明在血液系统肿瘤发生过程中,多种miRNA发挥重要功能。而在血液系统肿瘤中,一些lncRNA也与这些miRNA存在相互作用,影响白血病发生的过程(表2)。
2.3.1DLEU1、DLEU2与miR-15/-16
miR-15/-16是最早发现的与血液系统肿瘤有关的miRNA,是导致CLL发生的重要因素之一[59],它们具有极强的NF-kB通路诱导活性,而NF-kB通路异常可能引起CLL的发生[26]。DLEU1与DLEU2也是两个与CLL相关的lncRNA,定位于13q14.3[30]。Dal Bo等[60]研究发现,在50%的CLL患者中存在DLEU1和DLEU2的表达水平下降,这与患者预后不良有关,而95%(58/61)的CLL患者DLEU1和DLEU2的基因转录起始位点发生显著去甲基化,导致邻近抑癌基因转录调控出现异常,进而引起NF-kB信号通路异常[26]。有人认为DLEU1和DLEU2在CLL中发挥的作用可能与miR-15/-16密切相关,DLEU2的剪接变体是miR-15a和miR-16-1的pri-miRNAs[27]。这将lncRNA与miR-15a和miR-16-1联系在一起,提示我们DLEU1和DLEU2的表达水平下降可能影响miR-15a和miR-16-1的表达,影响miRNA的功能,引起CLL的发生。

表2 血液系统肿瘤中与miRNA相互作用的lncRNA分子
2.3.2PTENP1
PTEN的假基因PTENP1的转录物是一种ceRNA,也属于lncRNA。它具有多个与PTEN mRNA相同的MREs,能够作为诱饵分子吸附大量以PTEN mRNA为靶点的miRNA,如miR-17,miR-19b,miR-20a。其中miR-19b,miR-20a具有降低PTEN和PTENP1的表达丰度的功能[33]。若lncRNA PTENP1的表达水平升高,吸附更多miRNA,则与PTEN mRNA与miRNA的结合将减少,降低转录后抑制,提高PTEN蛋白水平[34]。而PTEN基因缺失或表达异常,能够影响血液系统肿瘤的发生或预后[34]。PTEN丢失会加速T-ALL的发生,产生多克隆肿瘤[71]。在CLL中PTEN出现表达下调,导致预后不良[72]。Panuzzo等[73]发现在慢性髓性白血病(Chronic myeloid leukemia,CML)中,BCR-ABL融合基因通过MEK依赖途径下调PTEN的表达。PTEN通过非基因组功能的缺失参与发病机制。而影响PTEN表达的因素很多,其中包含miRNA和假基因PTENP1[72]。PTENP1通过吸附以PTEN mRNA为靶点的miRNA,促进PTEN蛋白的表达,可能影响白血病的发生。王翠翠等[35]发现在急性白血病患者的骨髓细胞中,PTEN和PTENP1的表达水平下降而且两者的表达呈正相关,也说明PTENP1的表达可能通过吸附以PTEN mRNA为靶点的miRNA,影响PTEN表达并参与急性白血病的发生。
2.3.3lncRNA-BGL3
lncRNA-BGL3是BCR-ABL融合基因介导CML发生的一个关键因素,它的功能类似于ceRNA。lncRNA-BGL3具有与PTEN mRNA相同的MREs,是miR-17,-93,-20a,-20b,-106a,-106b的重要靶点。同时这些miRNA能抑制PTEN表达。lncRNA-BGL3通过竞争性结合miRNA以影响PTEN表达[36]。而在CML中,BCR-ABL融合基因通过MEK依赖途径下调PTEN的表达,PTEN也能通过非基因组功能的缺失影响疾病发生[73]。BCR-ABL融合基因还通过c-Myc依赖的DNA甲基化方式抑制lncRNA-BGL3表达[36],而lncRNA-BGL3表达下降,也会减少竞争性结合以PTEN mRNA为靶点的miRNA的结合,影响PTEN蛋白的表达,影响CML的发生。
总之,PTENP1和lncRNA-BGL3均可结合以PTEN mRNA为靶点的miRNA,而这些miRNA中有的可以抑制PTEN的表达。所以PTENP1和lncRNA-BGL3可以通过这种方式影响PTEN的表达。如果PTENP1或lncRNA-BGL3的表达下降,就可能会影响PTEN的功能,导致白血病的发生。另外,lncRNA-BGL3的表达受BCR-ABL的调控,而BCR-ABL可以下调PTEN的表达。因此BCR-ABL、PTENP1、lncRNA-BGL3可能共同相互作用于白血病,研究它们的相互作用有利于了解疾病的发生机制。
2.3.4T-UCRs
转录超保守区域(Transcribed ultraconserved regions,T-UCRs)是一类完全保守的基因序列。它们中的大部分可以转录RNA且不翻译蛋白质,有人认为属于lncRNA[28,74]。Calin等[56]发现T-UCRs在肿瘤及白血病中发挥重要作用。T-UCRs常位于基因脆性位点并且它们在癌症中的差异表达发生于该癌症相关的癌症相关基因组区域(Cancer-associated genomic regions,CAGRs)。研究认为T-UCRs在CLL中的表达受到miRNA的表达调控。他们通过对一系列T-UCRs分析检测,发现在CLL中存在5种T-UCRs(uc.269A,uc.160,uc.215,uc.346A,uc.348)表达与有关miRNA表达呈负相关。随后他们通过序列对比,发现这5种T-UCRs中有3种T-UCRs的序列与相关miRNA序列存在反义互补,提示在CLL中T-UCRs与miRNA可相互作用参与疾病的发生。
3 结语与展望
lncRNA是长度大于200 nt、无明显蛋白质编码功能的RNA[1,2]。它具有重要生物学功能,在染色体失活、染色体修饰、基因印记、细胞分化调控、细胞周期调控等过程发挥关键作用。lncRNA主要表现在表观遗传学修饰、转录水平调控以及转录后调控这3方面,调控相关基因表达[4]。在血液系统肿瘤中有些lncRNA可以调控p15基因表达,有些lncRNA调控或受p53蛋白调控,构成一个p53调控网络,调控相关基因表达,影响p53信号通路,有些lncRNA与miRNA相互作用,影响miRNA与靶基因的作用。本文重点综述这些lncRNA,并将它们与p15基因表达、p53调控网络以及miRNA参与疾病的发生联系在一起。这对于了解疾病的发生机制,寻找治疗靶点以及疾病的诊断具有很大的帮助。
随着对lncRNA的研究更加深入,最近有研究发现骨骼肌中存在一种能编码微肽的lncRNA,研究者将这包含46个氨基酸的微肽命名为myoregulin[75]。此外,还有学者发现在植物的miRNA初级转录物(pri-miRNAs)中具有短的开放阅读框序列,能够编码一种调节多肽,促进miRNA生成[76]。这颠覆了我们以往对于非编码RNA不能编码蛋白质的概念,毫无疑问地表明目前对于lncRNA的了解还不够全面[77]。lncRNA与血液系统肿瘤有着密切联系,更加深入研究lncRNA对于血液系统肿瘤发生发展的研究、诊断与治疗都有着重要意义。
[1] Derrien T, Johnson R, Bussotti G, Tanzer A, Djebali S, Tilgner H, Guernec G, Martin D, Merkel A, Knowles DG, Lagarde J, Veeravalli L, Ruan XA, Ruan YJ, Lassmann T, Carninci P, Brown JB, Lipovich L, Gonzalez JM, Thomas M, Davis CA, Shiekhattar R, Gingeras TR, Hubbard TJ, Notredame C, Harrow J, Guigó R. The GENCODE v7 catalog of human long non coding RNAs: analysis of their gene structure, evolution, and expression. Genome Res, 2012, 22(9): 1775–1789.
[2] Esteller M. Non-coding RNAs in human disease. Nat Rev Genet, 2011, 12(12): 861–874.
[3] 杨峰, 易凡, 曹慧青, 梁子才, 杜权. 长链非编码RNA研究进展. 遗传, 2014, 36(5): 456–468.
[4] 李灵, 宋旭. 长链非编码RNA在生物体中的调控作用.遗传, 2014, 36(3): 228–236.
[5] Zhang XQ, Sun S, Pu JKS, Tsang ACO, Lee D, Man VOY, Lui WM, Wong STS, Leung GKK. Long non-coding RNA expression profiles predict clinical phenotypes in glioma. Neurobiol Dis, 2012, 48(1): 1–8.
[6] Isin M, Ozgur E, Cetin G, Erten N, Aktan M, Gezer U, Dalay N. Investigation of circulating lncRNAs in B-cell neoplasms. Clin Chim Acta, 2014, 431: 255–259.
[7] Derrien T, Guigó R, Johnson R. The long non-coding RNAs: a new (p)layer in the "Dark Matter". Front Genet, 2011, 2: 107.
[8] 邓齐文, 许晔琼, 王书奎. 长链非编码RNA多态性与肿瘤相关性的研究. 医学研究生学报, 2014, 27(3): 303–306.
[9] Kim K, Jutooru I, Chadalapaka G, Johnson G, Frank J, Burghardt R, Kim S, Safe S. HOTAIR is a negative prognostic factor and exhibits pro-oncogenic activity in pancreatic cancer. Oncogene, 2013, 32(13): 1616–1625.
[10] Yang Z, Zhou L, Wu LM, Lai MC, Xie HY, Zhang F, Zheng SS. Overexpression of long non-coding RNA HOTAIR predicts tumor recurrence in hepatocellular carcinoma patients following liver transplantation. Ann Surg Oncol, 2011, 18(5): 1243–1250.
[11] Chen FJ, Sun M, Li SQ, Wu QQ, Ji L, Liu ZL, Zhou GZ, Cao G, Jin L, Xie HW, Wang CM, Lv J, De W, Wu M, Cao XF. Upregulation of the long non-coding RNA HOTAIR promotes esophageal squamous cell carcinoma metastasis and poor prognosis. Mol Carcinog, 2013, 52(11): 908–915.
[12] Gupta RA, Shah N, Wang KC, Kim J, Horlings HM, Wong DJ, Tsai MC, Hung T, Argani P, Rinn JL, Wang YL, Brzoska P, Kong B, Li R, West RB, van de Vijver MJ, Sukumar S, Chang HY. Long non-coding RNA HOTAIR reprograms chromatin state to promote cancer metastasis. Nature, 2010, 464(7291): 1071–1076.
[13] 庹磊, 李苏卿, 汪春梅, 曹秀峰. 长链非编码RNA HOTAIR在肿瘤中的研究进展. 医学综述, 2014, 20(16): 2925–2927.
[14] Zhang L, Xu HG, Lu C. A novel long non-coding RNA T-ALL-R-LncR1 knockdown and Par-4 cooperate to induce cellular apoptosis in T-cell acute lymphoblastic leukemia cells. Leuk Lymphoma, 2014, 55(6): 1373–1382.
[15] Han BW, Chen YQ. Potential pathological and functional links between long noncoding RNAs and hematopoiesis. Sci Signal, 2013, 6(289): re5.
[16] Bai WL, Yang J, Yang GX, Niu PY, Tian L, Gao A. Long non-coding RNA NR_045623 and NR_028291 involvedin benzene hematotoxicity in occupationally benzene-exposed workers. Exp Mol Pathol, 2014, 96(3): 354–360.
[17] Zhang AL, Xu M, Mo YY. Role of the lncRNA-p53 regulatory network in cancer. J Mol Cell Biol, 2014, 6(3): 181–191.
[18] Zhou YL, Zhang X, Klibanski A. MEG3 noncoding RNA: a tumor suppressor. J Mol Endocrinol, 2012, 48(3): R45–R53.
[19] 陈恩, 肖建华. p15基因的研究进展. 肿瘤学杂志, 2009, 15(3): 246–247.
[20] Uchida T, Kinoshita T, Saito H, Hotta T. CDKN2 (MTS1/p16INK4A) gene alterations in hematological malignancies. Leuk Lymphoma, 1997, 24(5–6): 449–461.
[21] Rosu-Myles M, Wolff L. p15Ink4b: dual function in myelopoiesis and inactivation in myeloid disease. Blood Cells Mol Dis, 2008, 40(3): 406–409.
[22] 郑瑞玑, 马旭东. INK4系列抑癌基因纯合子缺失、甲基化与白血病预后的实验研究. 白血病·淋巴瘤, 2010, 19(4): 230–233.
[23] 张耀, 宋陆茜, 吴凌云, 常春康, 李晓. 骨髓增生异常综合征患者p15INK4B基因甲基化与疾病进展的Meta分析. 肿瘤防治研究, 2013, 40(6): 525–530.
[24] Yu WQ, Gius D, Onyango P, Muldoon-Jacobs K, Karp J, Feinberg AP, Cui HM. Epigenetic silencing of tumour suppressor gene p15 by its antisense RNA. Nature, 2008, 451(7175): 202–206.
[25] Kino T, Hurt DE, Ichijo T, Nader N, Chrousos GP. Noncoding RNA gas5 is a growth arrest- and starvation-associated repressor of the glucocorticoid receptor. Sci Signal, 2010, 3(107): ra8.
[26] Garding A, Bhattacharya N, Claus R, Ruppel M, Tschuch C, Filarsky K, Idler I, Zucknick M, Caudron-Herger M, Oakes C, Fleig V, Keklikoglou I, Allegra D, Serra L, Thakurela S, Tiwari V, Weichenhan D, Benner A, Radlwimmer B, Zentgraf H, Wiemann S, Rippe K, Plass C, Döhner H, Lichter P, Stilgenbauer S, Mertens D. Epigenetic upregulation of lncRNAs at 13q14.3 in leukemia is linked to the In Cis downregulation of a gene cluster that targets NF-kB. PLoS Genet, 2013, 9(4): e1003373.
[27] Lerner M, Harada M, Lovén J, Castro J, Davis Z, Oscier D, Henriksson M, Sangfelt O, Grandér D, Corcoran MM. DLEU2, frequently deleted in malignancy, functions as a critical host gene of the cell cycle inhibitory microRNAs miR-15a and miR-16-1. Exp Cell Res, 2009, 315(17): 2941–2952.
[28] Calin GA, Liu CG, Ferracin M, Hyslop T, Spizzo R, Sevignani C, Fabbri M, Cimmino A, Lee EJ, Wojcik SE, Shimizu M, Tili E, Rossi S, Taccioli C, Pichiorri F, Liu XP, Zupo S, Herlea V, Gramantieri L, Lanza G, Alder H, Rassenti L, Volinia S, Schmittgen TD, Kipps TJ, Negrini M, Croce CM. Ultraconserved regions encoding ncRNAs are altered in human leukemias and carcinomas. Cancer Cell, 2007, 12(3): 215–229.
[29] Huarte M, Guttman M, Feldser D, Garber M, Koziol MJ, Kenzelmann-Broz D, Khalil AM, Zuk O, Amit I, Rabani M, Attardi LD, Regev A, Lander ES, Jacks T, Rinn JL. A large intergenic noncoding RNA induced by p53 mediates global gene repression in the p53 response. Cell, 2010, 142(3): 409–419.
[30] Huarte M, Rinn JL. Large non-coding RNAs: missing links in cancer?. Hum Mol Genet, 2010, 19(R2): R152–R161.
[31] Yap KL, Li SD, Muñoz-Cabello AM, Raguz S, Zeng L, Mujtaba S, Gil J, Walsh MJ, Zhou MM. Molecular interplay of the noncoding RNA ANRIL and methylated histone H3 lysine 27 by polycomb CBX7 in transcriptional silencing of INK4a. Mol Cell, 2010, 38(5): 662–674.
[32] Zhou Y, Zhang X, Klibanski A. MEG3 noncoding RNA: a tumor suppressor. J Mol Endocrinol, 2012, 48(3): R45–R53.
[33] Poliseno L, Salmena L, Zhang JW, Carver B, Haveman WJ, Pandolfi PP. A coding-independent function of gene and pseudogene mRNAs regulates tumour biology. Nature, 2010, 465(7301): 1033–1038.
[34] Milella M, Falcone I, Conciatori F, Incani UC, Del Curatolo A, Inzerilli N, Nuzzo CMA, Vaccaro V, Vari S, Cognetti F, Ciuffreda L. PTEN: multiple functions in human malignant tumors. Front Oncol, 2015, 5: 24.
[35] 王翠翠, 怀磊, 张翠萍, 贾玉娇, 李其辉, 陈一瑞, 田征,唐克晶, 邢海燕, 王敏, 王建祥. 抑癌基因PTEN与其假基因PTENP1在急性白血病细胞中的表达及其相关性研究. 中华血液学杂志, 2012, 33(11): 896–901.
[36] Guo G, Kang Q, Zhu X, Chen Q, Wang X, Chen Y, Ouyang J, Zhang L, Tan H, Chen R, Huang S, Chen JL. A long noncoding RNA critically regulates Bcr-Abl-mediated cellular transformation by acting as a competitive endogenous RNA. Oncogene, 2015, 34(14): 1768–1779.
[37] Gabory A, Jammes H, Dandolo L. The H19 locus: role of an imprinted non-coding RNA in growth and development. BioEssays, 2010, 32(6): 473–480.
[38] Bock O, Schlué J, Kreipe H. Reduced expression of H19 in bone marrow cells from chronic myeloproliferative disorders. Leukemia, 2003, 17(4): 815–816.
[39] Guo GJ, Kang QZ, Chen QH, Chen ZL, Wang J, Tan L, Chen JL. High expression of long non-coding RNA H19 is required for efficient tumorigenesis induced by Bcr-Abl oncogene. Febs Lett, 2014, 588(9): 1780–1786.
[40] Hannou SA, Wouters K, Paumelle R, Staels B. Functional genomics of the CDKN2A/B locus in cardiovascular and metabolic disease: what have we learned from GWASs?.
Trends Endocrinol Metab, 2015, 26(4): 176–184.
[41] Kotake Y, Nakagawa T, Kitagawa K, Suzuki S, Liu N,Kitagawa M, Xiong Y. Long non-coding RNA ANRIL is required for the PRC2 recruitment to and silencing of p15INK4Btumor suppressor gene. Oncogene, 2011, 30(16): 1956–1962.
[42] Iacobucci I, Sazzini M, Garagnani P, Ferrari A, Boattini A, Lonetti A, Papayannidis C, Mantovani V, Marasco E, Ottaviani E, Soverini S, Girelli D, Luiselli D, Vignetti M, Baccarani M, Martinelli G. A polymorphism in the chromosome 9p21 ANRIL locus is associated to Philadelphia positive acute lymphoblastic leukemia. Leuk Res, 2011, 35(8): 1052–1059.
[43] Garzon R, Liu SJ, Fabbri M, Liu ZF, Heaphy CEA, Callegari E, Schwind S, Pang JX, Yu JH, Muthusamy N, Havelange V, Volinia S, Blum W, Rush LJ, Perrotti D, Andreeff M, Bloomfield CD, Byrd JC, Chan K, Wu LC, Croce CM, Marcucci G. MicroRNA-29b induces global DNA hypomethylation and tumor suppressor gene reexpression in acute myeloid leukemia by targeting directly DNMT3A and 3B and indirectly DNMT1. Blood, 2009, 113(25): 6411–6418.
[44] Trimarchi T, Bilal E, Ntziachristos P, Fabbri G, Dalla-Favera R, Tsirigos A, Aifantis I. Genome-wide mapping and characterization of Notch-regulated long noncoding RNAs in acute leukemia. Cell, 2014, 158(3): 593–606.
[45] Heuston EF, Lemon KT, Arceci RJ. The beginning of the road for non-coding RNAs in normal hematopoiesis and hematologic malignancies. Front Genet, 2011, 2: 94.
[46] 谢咪雪, 谢彦晖. P53家族成员及相互作用与白血病关系的研究进展. 中国实验血液学杂志, 2013, 21(5): 1331–1335.
[47] 张建清, 王志林. p53基因异常在白血病患者中的研究.临床和实验医学杂志, 2010, 9(21): 1603–1605.
[48] 林东军, 郑永江, 方志刚, 范蕊芳, 刘加军, 林玉辉. 急性白血病p53基因P1启动子区域DNA甲基化研究. 中国病理生理杂志, 2008, 24(7): 1327–1330.
[49] Yang F, Bi JW, Xue XC, Zheng LM, Zhi KK, Hua JD, Fang GE. Up-regulated long non-coding RNA H19 contributes to proliferation of gastric cancer cells. FEBS J, 2012, 279(17): 3159–3165.
[50] Zhang AL, Zhou NJ, Huang JG, Liu Q, Fukuda K, Ma D, Lu ZH, Bai CX, Watabe K, Mo YY. The human long non-coding RNA-RoR is a p53 repressor in response to DNA damage. Cell Res, 2013, 23(3): 340–350.
[51] 陈芳, 陈杰. ARF蛋白——p53蛋白的重要调控因子. 国外医学: 生理病理科学与临床分册, 2003, 23(6): 555–557.
[52] Zhang X, Gejman R, Mahta A, Zhong Y, Rice KA, Zhou YL, Cheunsuchon P, Louis DN, Klibanski A. Maternally expressed gene 3, an imprinted noncoding RNA gene, is associated with meningioma pathogenesis and progression. Cancer Res, 2010, 70(6): 2350–2358.
[53] Benetatos L, Hatzimichael E, Dasoula A, Dranitsaris G, Tsiara S, Syrrou M, Georgiou I, Bourantas KL. CpG methylation analysis of the MEG3 and SNRPN imprinted genes in acute myeloid leukemia and myelodysplastic syndromes. Leuk Res, 2010, 34(2): 148–153.
[54] Zhou YL, Zhong Y, Wang YY, Zhang X, Batista DL, Gejman R, Ansell PJ, Zhao J, Weng C, Klibanski A. Activation of p53 by MEG3 non-coding RNA. J Biol Chem, 2007, 282(34): 24731–24742.
[55] Zhang X, Rice K, Wang YY, Chen W, Zhong Y, Nakayama Y, Zhou YL, Klibanski A. Maternally expressed gene 3 (MEG3) noncoding ribonucleic acid: isoform structure, expression, and functions. Endocrinology, 2010, 151(3): 939–947.
[56] 朱娟娟, 付汉江, 朱捷, 张胜权, 郑晓飞. 非编码基因MEG3稳定表达细胞株建立及其对p53转录活性的影响.军事医学, 2011, 35(6): 424–427.
[57] Cai XZ, Cullen BR. The imprinted H19 noncoding RNA is a primary microRNA precursor. RNA, 2007, 13(3): 313–316. [58] Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language? Cell, 2011, 146(3): 353–358.
[59] 耿凌云, 王欣. MicroRNA在慢性淋巴细胞白血病中的研究进展. 中国实验血液学杂志, 2014, 22(1): 255–258.
[60] Dal Bo M, Rossi FM, Rossi D, Deambrogi C, Bertoni F, Del Giudice I, Palumbo G, Nanni M, Rinaldi A, Kwee I, Tissino E, Corradini G, Gozzetti A, Cencini E, Ladetto M, Coletta AM, Luciano F, Bulian P, Pozzato G, Laurenti L, Forconi F, Di Raimondo F, Marasca R, Del Poeta G, Gaidano G, Foà R, Guarini A, Gattei V. 13q14 deletion size and number of deleted cells both influence prognosis in chronic lymphocytic leukemia. Genes Chromosomes Cancer, 2011, 50(8): 633–643.
[61] Pekarsky Y, Croce CM. Role of miR-15/16 in CLL. Cell Death Differ, 2015, 22(1): 6–11.
[62] Mraz M, Kipps TJ. MicroRNAs and B cell receptor signaling in chronic lymphocytic leukemia. Leuk Lymphoma, 2013, 54(8): 1836–1839.
[63] Thai TH, Calado DP, Casola S, Ansel KM, Xiao CC, Xue YZ, Murphy A, Frendewey D, Valenzuela D, Kutok JL, Schmidt-Supprian M, Rajewsky N, Yancopoulos G, Rao A, Rajewsky K. Regulation of the germinal center response by microRNA-155. Science, 2007, 316(5824): 604–608.
[64] San José-Enériz E, Román-Gómez J, Jiménez-Velasco A, Garate L, Martin V, Cordeu L, Vilas-Zornoza A, Rodríguez-Otero P, Calasanz MJ, Prósper F, Agirre X. MicroRNA expression profiling in Imatinib-resistant Chronic Myeloid Leukemia patients without clinically significant ABL1-mutations. Mol Cancer, 2009, 8(1): 69–72.
[65] Cho WCS. MicroRNAs: potential biomarkers for cancerdiagnosis, prognosis and targets for therapy. Int J Biochem Cell Biol, 2010, 42(8): 1273–1281.
[66] Stamatopoulos B, Meuleman N, Haibe-Kains B, Saussoy P, Van Den Neste E, Michaux L, Heimann P, Martiat P, Bron D, Lagneaux L. microRNA-29c and microRNA-223 down-regulation has in vivo significance in chronic lymphocytic leukemia and improves disease risk stratification. Blood, 2009, 113(21): 5237–5245.
[67] Mi SL, Lu J, Sun M, Li ZJ, Zhang H, Neilly MB, Wang YG, Qian ZJ, Jin J, Zhang YM, Bohlander SK, Le Beau MM, Larson RA, Golub TR, Rowley JD, Chen JJ. MicroRNA expression signatures accurately discriminate acute lymphoblastic leukemia from acute myeloid leukemia. Proc Natl Acad Sci USA, 2007, 104(50): 19971–19976.
[68] Pulikkan JA, Dengler V, Peramangalam PS, Peer Zada AA, Müller-Tidow C, Bohlander SK, Tenen DG, Behre G. Cell-cycle regulator E2F1 and microRNA-223 comprise an autoregulatory negative feedback loop in acute myeloid leukemia. Blood, 2010, 115(9): 1768–1778.
[69] Garzon R, Garofalo M, Martelli MP, Briesewitz R, Wang LS, Fernandez-Cymering C, Volinia S, Liu CG, Schnittger S, Haferlach T, Liso A, Diverio D, Mancini M, Meloni G, Foa R, Martelli MF, Mecucci C, Croce CM, Falini B. Distinctive microRNA signature of acute myeloid mia bearing cytoplasmic mutated nucleophosmin. Proc Natl Acad Sci USA, 2008, 105(10): 3945–3950.
[70] Li ZJ, Lu J, Sun M, Mi SL, Zhang H, Luo RT, Chen P, Wang YG, Yan M, Qian ZJ, Neilly MB, Jin J, Zhang YM, Bohlander SK, Zhang DE, Larson RA, Le Beau MM, Thirman MJ, Golub TR, Rowley JD, Chen JJ. Distinct microRNA expression profiles in acute myeloid leukemia with common translocations. Proc Natl Acad Sci USA, 2008, 105(40): 15535–15540.
[71] Martelli AM, Lonetti A, Buontempo F, Ricci F, Tazzari PL, Evangelisti C, Bressanin D, Cappellini A, Orsini E, Chiarini F. Targeting Signaling Pathways in T-cell acute lymphoblastic leukemia initiating cells. Adv Biol Regul, 2014, 56: 6–21.
[72] Zou ZJ, Zhang R, Fan L, Wang L, Fang C, Zhang LN, Yang S, Li YY, Li JY, Xu W. Low expression level of phosphatase and tensin homolog deleted on chromosome ten predicts poor prognosis in chronic lymphocytic leukemia. Leuk Lymphoma, 2013, 54(6): 1159–1164.
[73] Panuzzo C, Crivellaro S, Carrà G, Guerrasio A, Saglio G, Morotti A. BCR-ABL promotes PTEN downregulation in chronic myeloid leukemia. PLoS One, 2014, 9(10): e110682.
[74] Johnsson P, Lipovich L, Grandér D, Morris KV. Evolutionary conservation of long non-coding RNAs; sequence, structure, function. Biochim Biophys Acta, 2014, 1840(3): 1063–1071.
[75] Anderson DM, Anderson KM, Chang CL, Makarewich CA, Nelson BR, McAnally JR, Kasaragod P, Shelton JM, Liou J, Bassel-Duby R, Olson EN. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance. Cell, 2015, 160(4): 595–606.
[76] Lauressergues D, Couzigou JM, San Clemente H, Martinez Y, Dunand C, Bécard G, Combier JP. Primary transcripts of microRNAs encode regulatory peptides. Nature, 2015, 520(7545): 90–93.
[77] 井深, 张惠荣. 长链非编码RNA研究现状与趋势的文献计量分析. 中华医学图书情报杂志, 2012, 21(9): 54–56.
(责任编委: 杨昭庆)
The role of long non-coding RNAs in hematologic malignancies
Wanli Hu1, Ai Gao2
1. School of Basic Medical Sciences, Long Duration Clinical Expertise 2012 Grade 3 Class, Capital Medical University, Beijing 100069, China;
2. School of Public Health, Capital Medical University, Beijing 100069, China
With the rapid development of molecular biology technology, researchers have got much deeper understanding of the role of long non-coding RNA (lncRNA). It is not only indispensable for biological processes, but also plays an important role in human diseases especially in tumor. Previous studies have shown that a variety of lncRNAs are closely associated with hematologic malignancies. These lncRNAs are involved in diseases through diverse functions including affecting the expression of tumor suppressor gene p15, regulating p53 protein function, and interacting with miRNAs. In this review, we summarize the hematological tumor-associated lncRNAs and focus on p15, p53 and miRNA-related lncRNAs as well as the role of their interaction in hematological malignancies, which may provide a comprehensive understanding of the role of hematological tumor-associated lncRNAs and some insights for research, diagnosis and treatment of hematologic malignancies.
lncRNA; leukemia; p15; p53; miRNA
2015-06-04;
2015-09-07
国家自然科学基金项目(编号:81472957、81172639),北京市自然科学基金项目(编号:7142020)和北京市青年拔尖人才项目(编号:CIT&TCD201404187)资助
胡婉莉,本科在读。E-mail:736508060@qq.com
高艾, 博士,副教授,硕士研究生导师;研究方向:环境因素致造血系统损伤的表观遗传机制。E-mail: gaoai0980@163.com
10.16288/j.yczz.15-261
网络出版时间: 2015-9-119:54:28
URL: http://www.cnki.net/kcms/detail/11.1913.R.20150911.0954.006.html
