稻瘟病菌单加氧酶lpmo M1基因的克隆及生物信息学分析
2015-09-29谭海东马君燕李悝悝张建平尹恒中国科学院大连化学物理研究所辽宁大连6023中国科学院大学北京00049大连大学医学院辽宁大连6622
张 鑫,赵 勇,谭海东,马君燕,3,李悝悝,张建平,尹恒*(.中国科学院大连化学物理研究所,辽宁大连6023;2.中国科学院大学,北京00049;3.大连大学医学院,辽宁大连6622)
稻瘟病菌单加氧酶lpmo M1基因的克隆及生物信息学分析
张鑫1,2,赵勇1,谭海东1,马君燕1,3,李悝悝1,张建平1,尹恒1*
(1.中国科学院大连化学物理研究所,辽宁大连116023;2.中国科学院大学,北京100049;3.大连大学医学院,辽宁大连116622)
多糖裂解单加氧酶(LPMO)是一类新型的可以与水解酶系协同降解纤维素、几丁质和淀粉等难溶多糖的酶。以稻瘟病菌(Magnaporthe grisea70-15)为研究对象,采用逆转录-聚合酶链反应克隆得到多糖裂解单加氧酶基因lpmo M1,成功构建了真核表达载体pPICZαA-lpmo M1。生物信息学分析表明该基因编码区长819 bp,编码272个氨基酸,预测该基因的理论分子质量为28.85 ku,等电点为7.66;结构预测显示存在7个O-糖基化位点和16个磷酸化位点,没有N-糖基化位点。通过生物软件Vector NTI对不同来源的LPMO的同源性进行分析,结果显示稻瘟病菌LPMO M1与其他LPMO同源性最高仅为41%;系统进化分析发现稻瘟病菌(Magnaporthe grisea70-15)的多糖裂解单加氧酶LPMO M1与黄孢原毛平革菌(Phanerochaete chrysosporium)的同类酶Gh61D亲缘关系最近。
纤维素;多糖裂解单加氧酶;稻瘟病菌;真核表达载体;生物信息学分析
随着能源需求的增长、储备的减少以及全球温室效应的加剧,寻求可替代化石燃料的可再生能源已亟不可待[1]。除太阳能和风能等洁净能源外,含量丰富的生物质能源成为人们关注的焦点[2]。木质纤维素是自然界中分布极其广泛且数量极多的生物质资源,全球每年纤维素的产量约为8×1013kg[3-4],合理地利用这类资源将会对能源危机的缓解具有重大意义[5-6]。目前高效、环保的利用纤维素等难溶多糖是该类生物质开发和利用的一大瓶颈,纤维素的利用主要通过化学法和酶法,与化学法相比,酶法具有设备简单、反应条件温和及环境友好等优点。酶法主要是通过由内切葡聚糖酶、纤维二糖水解酶和β-1,4-D-葡萄糖苷酶组成的酶系降解纤维素。由于传统的纤维素酶系均属于糖苷水解酶家族,它们对于底物结晶区的降解效率较低,且成本较高,这使得传统的纤维素酶系难以满足工业应用的需求,限制了纤维素等生物质的高效利用[7-9]。
2010年VAAJE-KOLSTAD G团队报道了一个可以破坏几丁质晶体结构的酶,它主要通过氧化作用破坏糖苷键,为几丁质酶的进一步作用奠定了基础,最终得到可溶性寡糖或单糖[10-11]。该酶即为多糖裂解单加氧酶(lytic polysaccharide monooxygenases,LPMO),是一类在二价金属离子和还原剂存在的条件下氧化破坏难溶多糖晶体结构的酶。它的出现提高了难溶多糖的降解效率,使得生物质的高效转化成为可能。LPMO的分类原则有3种,根据氨基酸序列可以分为AA9家族、AA10家族和AA11家族;根据作用底物可以分为以纤维素为底物的LPMO、以几丁质为底物的LPMO和以淀粉为底物的LPMO[12];根据作用机制可以分为PMO1、PMO2和PMO3[13]。近年来,科学家们对LPMO展开了深入地研究,但是仍然有很多尚待解决的问题,所以寻找新的酶源对LPMO后续的研究和早日实现生物质的高效转化有重要意义。
稻瘟病菌(Magnaporthe grisea70-15)是水稻的一种常见致病菌,研究表明该菌的致病性可能与其分泌的纤维素酶和果胶酶可以破坏水稻的细胞壁有关[14]。该菌株的基因组全序列已经测序完成,根据CAZy数据库注释Magnaporthe grisea70-15有24个AA9家族基因,这为新型酶源的获得提供了可能。该文为研究稻瘟病菌中LPMO的性质与功能,以LPMO M1为研究对象,成功构建了酵母表达载体,并且对其进行了生物信息学方面的分析,为后续研究LPMO的功能奠定基础。
1 材料与方法
1.1材料与试剂
1.1.1菌株和载体
大肠杆菌(EscherichiacoliTop10)、毕赤酵母X-33(Pichia pastorisX-33)、水稻稻瘟病菌(Magnaporthe grisea70-15):西北农林科技大学农药研究所;克隆载体pEASY-BluntZero Cloning Vector:大连万泽有限公司;表达载体pPICZαA:美国Invitrogen公司。
1.1.2培养基
LB培养基:胰蛋白胨1%,酵母提取物0.5%,NaCl 1%,固体培养时加入2%琼脂,用于转化子筛选时加入100μg/mL氨苄青霉素。
马铃薯葡萄糖琼脂(potato dextrose agar,PDA)固体培养基:马铃薯20%,葡萄糖2%,琼脂2%。
1.1.3酶和试剂
1.2仪器与设备
ML 104/02电子天平:梅特勒-托利多国际贸易(上海)有限公司;LRH系列生化培养箱、HWS24型电热恒温水浴锅:上海一恒科技有限公司;QHZ-98A全温振荡培养箱:太仓市华美生化仪器厂;SW-CJ-2FD洁净工作台:苏净集团苏州安泰空气技术有限公司;精科752型紫外分光光度计;Gene Pulser XcellTM电穿孔系统:美国BIO-RAD公司;DYY-6C型电泳仪:北京市六一仪器厂。
1.3方法
1.3.1稻瘟病菌多糖裂解单加氧酶基因lpmo M1的克隆
将稻瘟病菌(Magnaporthe grisea70-15)接种在PDA培养基,28℃培养7 d后使用真菌RNA提取试剂盒提取稻瘟病菌RNA、宝生物工程(大连)有限公司的cDNA第一链合成试剂盒合成cDNA。根据GeneBank中公布的稻瘟病菌的LPMO M1基因序列(登录号:XM_003709062.1)和酵母表达载体pPICZαA设计引物,为了保证得到含有天然N端的LPMO M1,设计引物时去除LPMO M1的信号肽序列,上游引物5′-GACCTCGAGAAAAGACACTACAACTTCGAGT CCCTC-3′,下游引物:5′-CTTGCGGCCGCGTAGTGTGCG GAGCGGCG-3′[15],下划线标记的序列为酶切位点,分别为XhoI和NotI,粗体部分为载体kex2信号酶裂解位点。以稻瘟病菌Magnaporthe grisea70-15cDNA为模板,扩增目的基因的编码区,聚合酶链反应(polymerase chain reaction,PCR)体系(50 μL):ddH2O 34.3 μL,5×buffer 10 μL,脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)4μL,上游引物(100μmol/L)0.1μL,下游引物(100μmol/L)0.1 μL,PrimeSTARRHS DNA Polymerase 0.5 μL,真菌模板基因组DNA 1 μL。PCR反应程序:98℃预变性3 min,98℃变性10 s,55℃退火5 s,72℃延伸2 min,共30个循环,72℃延伸5 min。PCR产物经1%琼脂糖凝胶电泳检测并用琼脂糖凝胶回收纯化试剂盒回收,然后与克隆载体pEASY-Blunt Zero Cloning vector连接,转入E.coliTop10感受态细胞中,在LB/Amp平板上筛选阳性克隆。菌落PCR检测是否有插入片段,挑取单菌落,在LB/Amp液体培养基中培养过夜,提取质粒,用XhoI和NotI进行双酶切鉴定,送华大基因(北京)测序,采用Vector NTI软件分析测序结果。
1.3.2稻瘟病菌多糖裂解单加氧酶基因lpmo M1的表达载体的构建、鉴定及转化
将测序正确的阳性克隆37℃过夜培养,菌液经质粒小量抽提试剂盒提取质粒后,用XhoI和NotI在37℃双酶切2 h后经1%琼脂糖凝胶电泳检测回收酶切后的目的片段,再与经过相同双酶切的pPICZαA酵母表达载体用T4DNA连接酶室温连接20 h。将重组质粒转入E.coliTop10感受态细胞,菌落PCR和双酶切鉴定酵母表达载体是否构建成功,并送至华大基因(北京)测序,构建成功的重组质粒命名为pPICZαA-lpmo M1。
将测序正确的质粒pPICZαA-lpmoM1用限制性内切酶SacI线性化,然后转入毕赤酵母X-33感受态细胞,并对转化子进行菌落PCR鉴定,以检测该表达载体是否转入毕赤酵母中。1.3.3多糖裂解单加氧酶基因及其编码蛋白生物信息学分析
通过生物在线网站预测LPMO M1的分子质量、氨基酸组成和等电点等;根据SignalP对该蛋白信号肽进行预测;在NCBI网站对其结构域进行预测分析;采用生物在线网站NetNGlyc 1.0 Server和NetOGlyc 3.1 Serve对该蛋白中存在的N-糖基化位点和O-糖基化位点进行了预测;利用NetPhos 2.0 Server预测其磷酸化位点[16-17]。通过生物学软件Vector NTI分析多糖裂解单加氧酶的序列同源性,并通过MEGA 6.0软件的ML法分析AA9家族LPMO的系统进化关系[18]。
2 结果与分析
2.1稻瘟病菌多糖裂解单加氧酶基因lpmo M1的克隆
以Magnaporthe grisea70-15 cDNA为模板,PCR扩增目的基因,琼脂糖凝胶电泳结果见图1。由图1可知,在750~1 000 bp之间有清晰的条带,大小与预测一致。将其连接至克隆载体pEASY-Blunt Zero Cloning Vector中,双酶切结果表明,酶切后的片段与目的片段大小一致,结果见图2。测序后与稻瘟病菌多糖裂解单加氧酶lpmo M1基因序列进行比对,结果表明有1个碱基发生同义突变,说明获得的基因编码区序列是正确的,可以构建表达载体。

图1 多糖裂解单加氧酶基因lpmo M1扩增产物的电泳检测Fig.1 Agarose gel analysis of PCR amplification products of lpmo M1gene
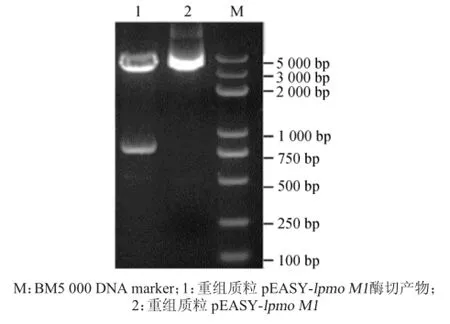
图2 lpmo M1基因重组质粒pEASY-lpmo M1的双酶切鉴定Fig.2 Double digestion of recombinant plasmid pEASY-lpmo M1of lpmo M1
2.2稻瘟病菌多糖裂解单加氧酶基因lpmo M1表达载体的构建
将目的片段与用相同酶切回收后的表达载体pPICZαA连接,电击(电压2000V,电容25μF,电阻200Ω)转入E.coli Top10,挑取阳性克隆采用菌落PCR和双酶切方法鉴定重组表达载体。通过PCR鉴定,所用引物为检测引物,目的片段大小为1 000 bp左右(图3);另外通过双酶切鉴定显示重组质粒经过XhoI和NotI双酶切,得到大小分别为3 593 bp和819 bp的条带(图4),与预期估计条带大小相符。将构建好的载体送华大基因测序,其测序结果与克隆序列一致,开放阅读框架正确。结果表明稻瘟病菌多糖裂解单加氧酶基因lpmo M1酵母表达载体构建成功。
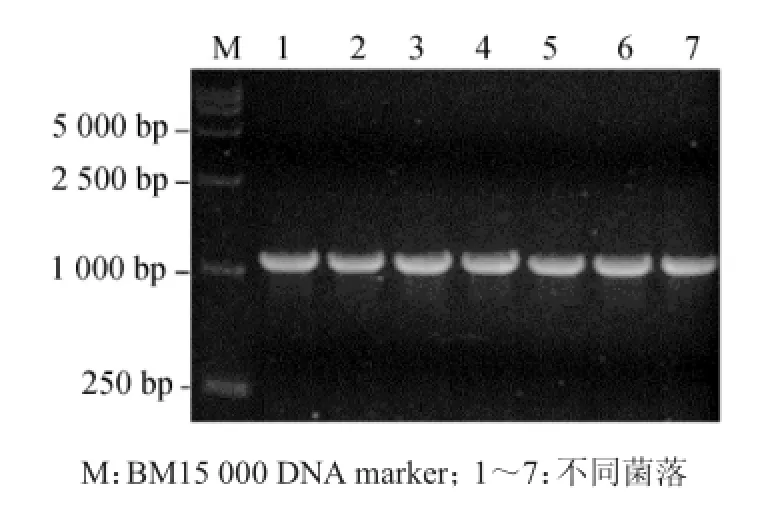
图3 稻瘟病菌多糖裂解单加氧酶基因重组质粒pPICZαA-lpmo M1菌落PCR鉴定Fig.3 Colony PCR identification of recombinant plasmid pPICZαA-lpmo M1ofMagnaporthe griseaLPMO

图4 稻瘟病菌多糖裂解单加氧酶基因重组质粒pPICZαA-lpmo M1双酶切鉴定Fig.4 Double digestion of recombinant plasmid pPICZαA-lpmo M1of Magnaporthe griseaLPMO
2.3稻瘟病菌多糖裂解单加氧酶基因lpmo M1的表达载体转入毕赤酵母X-33

图5 毕赤酵母转化子菌落PCR鉴定Fig.5 PCR identification ofPichia pastoristransformant
将构建好的酵母表达载体pPICZαA-lpmoM1通过电击转化的方法转化至毕赤酵母X-33中,在含有博来霉素和山梨醇的酵母浸出粉胨葡萄糖培养基上筛选阳性克隆,挑取单菌落进行菌落PCR,PCR产物经琼脂糖凝胶电泳检测,得到大小为1500bp和2200bp的条带(图5),结果表明酵母表达载体pPICZαA-lpmo M1成功导入毕赤酵母X-33中,可用于下一步多糖裂解单加氧酶的表达、纯化以及活性研究。
2.4稻瘟病菌多糖裂解单加氧酶LPMO M1的生物信息学分析
2.4.1稻瘟病菌多糖裂解单加氧酶lpmo M1基因及其编码氨基酸序列分析
对多糖裂解单加氧酶lpmo M1基因序列进行分析,结果表明该基因序列长度为819bp,编码272个氨基酸,理论分子质量为28.85ku,等电点为7.66,分子式为C1285H1956N350O391S7,原子总数为3 989,总平均亲水性为-0.322,说明lpmo M1亲水性较强。在NCBI网站对其结构域进行预测分析表明,该多糖裂解单加氧酶只有1个结构域(AA9)。通过SignalP软件分析得知LPMO M1的信号肽切割位点位于21位的A(丙氨酸)。通过在线分析软件NetNGlyc1.0和YinOYang1.2对多糖裂解单加氧酶LPMO M1进行N-糖基化位点和O-糖基化位点预测。结果表明,该氨基酸序列没有N-糖基化位点;分别在184、208、211、217、226、227和248位存在7个潜在的O-糖基化位点。用NetPhos 2.0在线分析软件对其磷酸化位点进行预测,结果显示当阈值为0.5时,其氨基酸序列存在16个潜在的磷酸化位点(9个丝氨酸位点、5个苏氨酸位点和2个酪氨酸位点)。
2.4.2不同真菌来源的多糖裂解单加氧酶的同源比对及系统进化分析

图6 LPMO M1与AA9家族LPMO的氨基酸序列比对Fig.6 The amino acid sequence comparison of LPMO M1 and other LPMOs in AA9 family
利用Vector NTI将LPMO M1的氨基酸序列与不同真菌来源的多糖裂解单加氧酶的氨基酸序列比对[10],分析显示上述LPMO来源于不同的种属,由于种属间的差异性导致其同源性较低(图6)。稻瘟病菌多糖裂解单加氧酶LPMO M1与Gh61D、Gh61E、LPMO9D、Gh61B和Cel61A的同源性分别为41%、36%、36%、27%和22%。这些酶均属于AA9家族,该家族酶的活性位点是由2个组氨酸和1个酪氨酸构成的铜离子结合位点(图6中用*标注),这3个氨基酸在该类酶中是高度保守的[19]。利用MEGA 6.0对15个不同来源的LPMO进行系统进化分析,从稻瘟病菌LPMO M1与其他真菌来源LPMO的系统进化树可以看出(图7),不同来源的LPMO根据氧化位点的不同形成2个明显的分支,其中Ⅰ号分支由氧化C-1位的PMO1和氧化C-4位的PMO2组成,Ⅱ号分支由氧化C-1位和C-4位的PMO3组成[13]。稻瘟病菌(Magnaporthe grisea70-15)多糖裂解单加氧酶LPMO M1位于Ⅰ号分支,属于PMO1家族,与黄孢原毛平革菌(Phanerochaete chrysosporium)的Gh61D亲缘关系最近[20]。

图7 稻瘟病菌LPMO M1与其他真菌来源LPMO的系统进化树Fig.7 The phylogenetic tree analysis of LPMO M1 fromMagnaporthe griseawith other fungus
3 结论
纤维素虽然是自然界中含量最丰富的生物质资源,但是结构稳定和难以降解的特性大大限制了纤维素等生物质的高效利用。多糖裂解单加氧酶是最新发现的可以氧化降解结晶多糖的一类酶,这为阐明自然界中生物质的降解机制提供了新的视角[10],并且为提高生物质的降解效率,实现生物质的高效转化奠定了基础。
采用RT-PCR扩增得到稻瘟病菌中的一个多糖裂解单加氧酶基因序列,并且成功构建真核表达载体pPICZαA-lpmo M1,其携带的基因在宿主中可以稳定遗传。利用多种生物信息学软件对稻瘟病菌多糖裂解单加氧酶LPMO M1的基因序列和氨基酸序列进行分析;通过Vector NTI生物分析软件对不同真菌来源的LPMO的氨基酸序列的同源性进行分析,结果表明该酶与其他同类酶同源性较低。PMO与来源于Phanerochaete chrysosporium的LPMO(Gh61D)的亲缘关系最近,该基因在毕赤酵母X-33中的表达量较低,后续将通过密码子优化、表达条件优化以及筛选高拷贝的转化子等方法提高表达量,为后续实验奠定基础。稻瘟病菌中含有丰富的LPMO,深入地研究每个多糖裂解单加氧酶的性质对于早日阐明稻瘟病菌降解纤维素的机制和提高纤维素的降解效率具有重要意义。
[1]PANWAR N L,KAUSHIK S C,KOTHARI S.Role of renewable energy sources in environmental protection:A review[J].Renew Sust Energ Rev,2011,15(3):1513-1524.
[2]KARAKASHEVD,THOMSENAB,ANGELIDAKII.Anaerobic biotechnological approaches for production of liquid energy carriers from biomass[J].Biotechnol Lett,2007,29(7):1005-1012.
[3]GELFAND I,SAHAJPAL R,ZHANG X,et al.Sustainable bioenergy production from marginal lands in the US Midwest[J].Nature,2013,493(7433):514-517.
[4]JORDAN D B,BOWMAN M J,BRAKER J D,et al.Plant cell walls to ethanol[J].Biochem J,2012,442(2):241-252.
[5]HORN S J,VAAJE-KOLSTAD G,WESTERENG B,et al.Novel enzymes for the degradation of cellulose[J].Biotechnol Biofuels,2012,5(1):89-93.
[6]HEMSWORTH G R,DAVIES G J,WALTON P H.Recent insights into copper-containing lytic polysaccharide mono-oxygenases[J].Curr Opin Struct Biol,2013,23(5):660-668.
[7]HORN S J,SIKORSKI P,CEDERKVIST J B,et al.Costs and benefits of processivity in enzymatic degradation of recalcitrant polysaccharides[J]. Proc Natl Acad Sci USA,2006,103(48):18089-18094.
[8]高美荣.斜卧青霉多糖单加氧酶(PMOs)的功能研究[D].济南:山东大学硕士论文,2013.
[9]王健,袁永俊,张驰松.纤维素发酵产酒精研究进展[J].中国酿造,2006,25(6):9-13.
[10]VAAJE-KOLSTAD G,WESTERENG B,HORN S J,et al.An oxidative enzyme boosting the enzymatic conversion of recalcitrant polysaccharides[J].Science,2010,330(6001):219-222.
[11]施贤卫,张伟涛,张小飞,等.Actinosynnema mirumDSM43827溶解性多糖单加氧酶的异源表达和酶学性质表征[J].中国生物工程杂志,2014(7):3.
[12]AGGER J W,ISAKSEN T,VARNAI A,et al.Discovery of LPMO activity on hemicelluloses shows the importance of oxidative processes in plant cell wall degradation[J].Proc Natl Acad Sci USA,2014,111(17):6287-6292.
[13]VU V V,BEESON W T,PHILLIPS C M,et al.Determinants of regioselective hydroxylation in the fungal polysaccharide monooxygenases[J]. J Am Chem Soc,2014,136(2):562-565.
[14]游树鹏,韩献忠.稻瘟菌的胞外纤维素酶和果胶酶活性的研究[J].安徽农业科学,1991(4):355-358.
[15]WESTERENG B,ISHIDA T,VAAJE-KOLSTAD G,et al.The putative endoglucanase PcGH61D fromPhanerochaete chrysosporiumis a metaldependent oxidative enzyme that cleaves cellulose[J].PLoS One,2011,6(11):e27807.
[16]王乃栋,张丽霞,向勤锃,等.茶树多酚氧化酶基因的生物信息学分析及原核表达[J].茶业科学,2011,31(1):33-39.
[17]周井祥,李新伟,王好,等.锦鲤疱疹病毒_CJ株ORF81基因的克隆及生物信息学分析[J].水产学报,2011,35(12):1780-1786.
[18]赵海龙,孟庆玲,乔军,等.少孢节丛孢菌XJ_A1几丁质酶基因的克隆及生物信息学分析[J].西北农业学报,2015,24(3):26-32.
[19]LEGGIO L L,WELNER D,DE MARIA L.A structural overview of GH61 proteins-fungal cellulose degrading polysaccharide monooxygenases[J].Comp Struct Biotechnol J,2012,2(3):1-8.
[20]刘艳敏,卢彪,沈玺龙,等.枯草芽孢杆菌BJ3-2精氨酸脱羧酶基因印水平speA的克隆与序列分析[J].中国酿造,2014,33(5):39-43.
Cloning and bioinformatic analysis oflpmo M1fromMagnaporthe oryzae
ZHANG Xin1,2,ZHAO Yong1,TAN Haidong1,MA Junyan1,3,LI Kuikui1,ZHANG Jianping1,YIN Heng1*
(1.Dalian Institute of Chemical Physics,Chinese Academy of Sciences,Dalian 116023,China;2.University of Chinese Academy of Sciences,Beijing 100049,China;3.Medical College of Dalian University,Dalian 116622,China)
Lytic polysaccharide monooxygenases(LPMO)is a new type of enzymes that acts synergistically with hydrolase system to degrade recalcitrant polysaccharide,such as cellulose,chitin and starch.In order to study the function of LPMO fromMagnaporthe grisea70-15,lpmo M1was cloned by RT-PCR and its eukaryotic expression vector pPICZαA-lpmo M1was constructed.Bioinformatic analysis showed that the coding region of lpmo M1was 819 bp,which encoded 272 amino acids,and the theory molecule weight was predicted as 28.85 ku and the soelectric point was 7.66. The structure predicted that it had 7 O-glycosylation sites and 16 phosphorylation sites,but no N-glycosylation sites.Homologous analysis by Vector NTI software indicated that LPMO M1 had a low homology with other LPMOs of the different organisms.The phylgenetic analysis results showed that LPMO M1 had a most close phylogenetic relationship with the LPMO fromPhanerochaete chrysosporium.
cellulose;lytic polysaccharide monooxygenases;Magnaporthe grisea;eukaryotic expression vector;bioinformatic analysis
Q933
A
0254-5071(2015)11-0035-06
10.11882/j.issn.0254-5071.2015.11.009
2015-09-23
国家自然科学基金(31370811);辽宁省自然科学基金(2015020690);中科院青年创新促进会(2015144)
张鑫(1990-),女,硕士研究生,研究方向为生物质利用。
尹恒(1982-),男,研究员,博士,研究方向为天然产物与糖工程。
