大鳞副泥鳅部分线粒体基因的序列分析研究
2015-08-22韩晓磊朱绿定王富凯韩曜平朱庆华
韩晓磊,王 丹,徐 涛,朱绿定,王富凯,韩曜平,朱庆华
(1.常熟理工学院 生物与食品工程学院,江苏 常熟 215500;2.常州市武进区前黄镇农技农机站,江苏 常州 213100;3.常州市秋余水产养殖专业合作社,江苏 常州 213100)
大鳞副泥鳅部分线粒体基因的序列分析研究
韩晓磊1,王丹2,徐涛1,朱绿定3,王富凯1,韩曜平1,朱庆华3
(1.常熟理工学院 生物与食品工程学院,江苏 常熟 215500;2.常州市武进区前黄镇农技农机站,江苏 常州 213100;3.常州市秋余水产养殖专业合作社,江苏 常州 213100)
通过PCR和DNA测序技术,获得了大鳞副泥鳅(Paramisgurnus dabryanus)线粒体基因组的部分序列,所得4段序列长度共计11552 bp,包括5个蛋白质编码基因(ND1、ND2、ND4、ND4L、ND6和Cytb),14个tRNA基因.本研究获得序列的碱基组成为A 28.9%、C 27.7%、G 15.7%、T 27.7%,可看出(A+T)>(C+G);测出的蛋白编码基因的碱基都有一个明显的现象,就是G的含量很明显的少于其他碱基,而C的含量都比较高,并且(A+T)>(C+G),这样的结果表现出明显的反G偏倚和碱基组成偏好性.将大鳞副泥鳅Cytb序列与GenBank其他6种鱼类的Cytb序列进行%对,表明大鳞副泥鳅与洛东江高丽鳅和俄罗斯泥鳅亲缘关系最近.用NJ法构建7种鱼类的进化关系树,结果表明:大鳞副泥鳅与洛东江高丽鳅和俄罗斯泥鳅最先聚类,再与其他鳅科鱼类相聚,最后与鲤科鱼类斑马鱼相聚,结果与传统分类一致.
大鳞副泥鳅;线粒体DNA;NADH还原酶复合体;Cytb
大鳞副泥鳅(Paramisgurnus dabryanus)隶属于鲤形目,鳅科,花鳅亚科,副泥鳅属,主要分布于四川、江苏、浙江、台湾,辽宁、黑龙江等地,具有很高的食用价值,俗称“水中人参”[1-3].大鳞副泥鳅现已成为我国出口韩国的重要水产品之一,且在国内市场的需求量也在不断攀升.作为一种优良的养殖品种,它是中国近年养殖规模发展较快的名优鱼类,许多省份已开始规模化养殖生产[4-7].动物mtDNA是共价闭合的双链DNA分子,核酸序列和组成比较保守,基因的排列顺序比较稳定而且紧密,无重组和单拷贝.由于mtDNA含有丰富的进化信息,具有长度短、含量丰富、母性遗传和进化速度快等特点,已成为研究动物起源进化以及群体遗传分化的理想对象[8-13].本实验采用PCR技术,获得大鳞副泥鳅部分线粒体DNA的序列,为大鳞副泥鳅的系统分类提供分子水平的证据,并对其种属水平的分类研究进行了初步探讨,同时也为今后大鳞副泥鳅的资源保护、开发和养殖提供相应的理论依据.
1 材料与方法
1.1实验样品
大鳞副泥鳅,取自常州市武进区水产推广站大鳞副泥鳅良种场,运输到实验室后进行活体宰杀,迅速采集肌肉组织后放于-80℃冰箱保存备用.
1.2实验方法
1.2.1基因组DNA提取
基因组DNA提取参照《分子克隆试验指南》[14].用Eppendorf公司生产的Bio Photometer核酸检测仪检测DNA溶液浓度与纯度,并将浓度稀释至30 ng/μL,置-20℃保存备用.
1.2.2线粒体DNA基因片段的扩增
从GenBank下载了与大鳞副泥鳅亲缘关系较近的3种鳅科鱼类(Cobitis striata,Misgurnus anguillicaudatus,Misgurnus nikolskyi)线粒体基因组全序列并采用DNAMAN5.0进行比对,寻找保守区域,设计了7对PCR引物(表1),引物合成由上海生工完成.用PE9600型PCR仪进行PCR扩增反应.PCR反应的总体积为50 μL,其中包括5 μL的10×Reaction Buffer,1 μL dNTP mix(10 mmol/L),引物F和R各1 μL(25 μmol/L),0.4 μL Taq DNA聚合酶(5 U/μL),1 μL总DNA(约100 ng),加灭菌双蒸水至终体积.PCR反应程序如下:94℃预变性5 min,94℃变性30 s,55℃退火30 s,72℃延伸45 s,运行35个循环,然后再延伸10 min.最后将PCR扩增产物在2%琼脂糖凝胶电泳(内有溴化乙锭)中电泳,以DL2000 marker为分子量标记,电泳结束后在凝胶成像系统中拍照.
1.2.3DNA序列测定

表1 大鳞副泥鳅线粒体扩增引物设计
将PCR产物割胶纯化(纯化试剂盒购自中科开瑞生物技术公司),然后送往上海英骏生物工程公司进行序列测定,测序在ABI3730测序仪上进行.
1.2.4数据分析
所获得的序列经校对后,采用ClustalX1.83软件进行比对,用MEGA5.1软件进行分析.根据Kimura双参数模型计算遗传距离,用NJ法构建系统发生树,采用bootstrap检验,重复次数为1000次.
2 结果与分析

表2 大鳞副泥鳅部分线粒体基因
2.1大鳞副泥鳅部分线粒体DNA序列
本实验共测定拼接完成4段大鳞副泥鳅的线粒体基因,拼接后长度分别为2790 bp、2861 bp、1859 bp和4042 bp,其中包括6个蛋白质编码基因(ND1、ND2、ND4、ND4L、ND6和Cytb,14个tRNA基因,也注明了各个基因的起始位点和终止位点(表2).
2.2蛋白质编码基因的序列分析
鱼类线粒体基因组共编码13个蛋白质,包括:细胞色素b(Cytb),2个ATP酶的亚单位(ATPase8和ATPase6),3个细胞色素C氧化酶的亚单位(COI,Ⅱ,Ⅲ),7个NADH还原酶复合体的亚单位(ND1,2,3,4,4L,5和6).我们获得了部分蛋白质编码基因(ND1、ND2、ND4、ND4L、ND6和Cy⁃tb),分析了其碱基组成及密码子的使用情况.
2.2.1ND1基因
大鳞副泥鳅ND1基因的序列长度为972 bp(表2).碱基T、C、A、G分别是30.5%、27.6%、27.5%、14.5%;密码子第三位上碱基T、C、A、G分别是29.6%、23.8%、41.7%、4.9%(表3).比较ND1基因总的碱基组成和第三位点的碱基组成,发现A的含量都比较高.比较ND1基因三个位点的碱基组成,发现第一位点C含量稍高;第二位点T含量最高,为40.4%,存在反G偏移,含量为11.4%;第三位点A含量最高,为41.7%,存在反G偏移,含量为4.9%.
将大鳞副泥鳅ND1基因核苷酸序列翻译为氨基酸序列时,在所有密码子中,UUG,CCG,UAG,CAG,AAG,UGU,UGC,CGG,AGU,AGA,AGG都没有使用到.使用频率最高的是CUA密码子(30.0),其次是AUU 和GCC密码子(都是18.0);GGG,UGG,GAU,UAA,GCG,ACG,CUG密码子使用情况均为1.0.由于篇幅有限,此处及下文的密码子使用情况表格均未列出.
2.2.2ND2基因
大鳞副泥鳅ND2基因的序列长度为1047 bp(表2).碱基T、C、A、G分别为25.7%、30.0%、30.5%、13.8%;密码子第三位上碱基T、C、A、G含量分别为38.4%、34.7%、15.8%、11.2%(表3).比较ND2基因总的碱基组成和第一位点和第三位点的碱基组成,发现C的含量都比较高,第一位点C含量很高,为30.0%,存在反G偏移,只有8.0%;第三位点T含量最高,为38.4%.C为34.7%,G为11.2%,存在反G偏移.
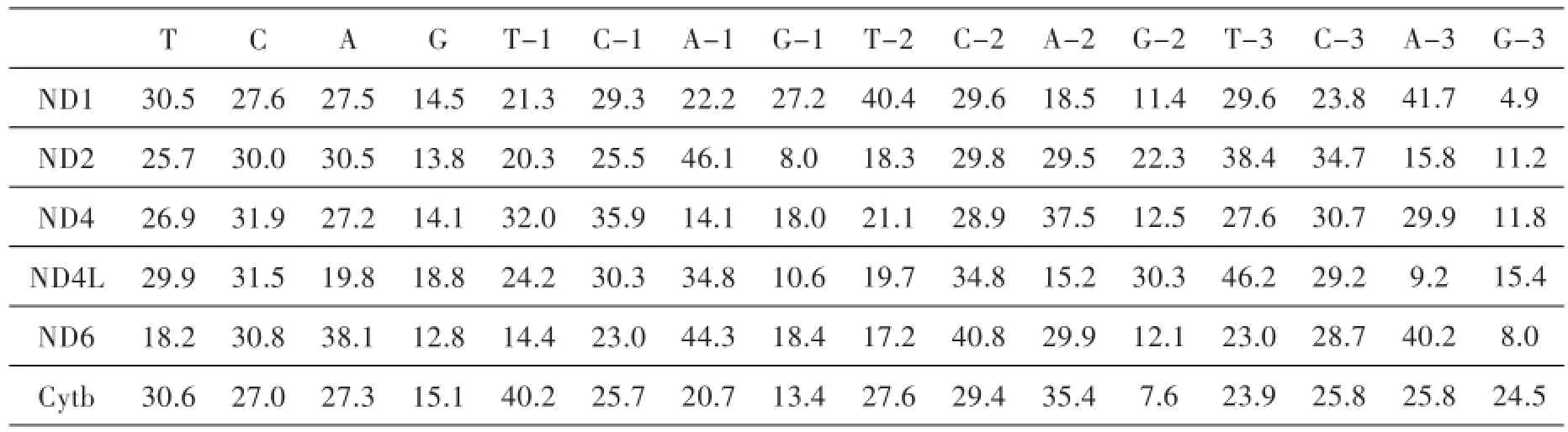
表3 大鳞副泥鳅ND1基因密码子各位置的碱基组成及含量(%)
将大鳞副泥鳅ND2基因核苷酸序列翻译为氨基酸序列时,在所有密码子中,CCG,UCG,GCG,UAG,GAA,GAG,CGA,CGG,GGA都没有使用到.使用频率最高的是CCU密码子(28.0),其次是AAC密码子(25.0);GUU,GUA,GCA,GAU,CGU密码子使用情况均为1.0.
2.2.3ND4基因
大鳞副泥鳅ND4基因的序列长度为383 bp(表2).碱基T、C、A、G含量分别为26.9%、31.9%、27.2%、14.1%;密码%第三位上碱基T、C、A、G含量分别为27.6%、30.7%、29.9%、11.8%(表3).比较ND4基因总的碱基组成和第一、二、三位点的碱基组成,发现C的含量都比较高,第一位点C含量很高,为35.9%,存在反G偏移,含量为18.0%;第二位点A含量最高,C为28.8%,存在反G偏移,含量为12.5%;第三位点C含量最高,为30.7%,G为11.8%,存在反G偏移.
将大鳞副泥鳅ND4基因核苷酸序列翻译为氨基酸序列时,在所有密码子中,CCG,CGG,AUU,AGU,AGC,AUG,ACG,AGG,GGC,GUA,GUG,GCG,GAG,GGG,UCG,UGA,CGU都没有使用到.使用频率最高的是CAA,CCC密码子(6.0),其次是CUA,CCA密码子(5.0);CUC,AUA,AAA,AAG,GCA,GAA,CGC,CGA,GGU密码子使用情况均为1.0.
2.2.4ND4L基因
大鳞副泥鳅ND4L基因的序列长度为197 bp(表2).碱基T、C、A、G含量分别为29.9%、31.5%、19.8%、18.8%;密码子第三位上碱基T、C、A、G含量为46.2%、29.2%、9.2%、15.4%(表3).比较ND4L基因总的碱基组成和第三位点的碱基组成,发现C的含量都比较高.比较ND4L基因三个位点的碱基组成,发现第一位点C含量很高,为30.3%,存在反G偏移,含量为10.6%;第三位点T含量最高,为46.2%,C为29.2%,存在反G偏移,含量为15.4%.
将大鳞副泥鳅ND4L基因核苷酸序列翻译为氨基酸序列时,在所有密码%中,UUU,UCC,UUA,UUG,UCG,UAG,CAU,CGU,CCC,CUA,CAA,CGA,CGG,AGU,AUA,AAA,AUG,ACG,AAG,GUU,GGU,GUC,GCC,GAC,GUA,GCA,GAA,GGA,GUG,GCG,GAG,GGG都没有使用到.使用频率最高的是CCU密码子(8.0),其次是AUU密码子(5.0);UUC,CUU,CUC,UCA,CCA,CCG,ACC,ACA,CAG,AAC,GAU,UGU,UGG,AGA密码子使用情况均为1.0.
2.2.5ND6基因
大鳞副泥鳅ND6基因的序列长度为522 bp(表2).各碱基T、C、A、G含量分别为18.2%、30.8%、38.1%、12.8%;密码子第三位上碱基T、C、A、G含量为23.0%、28.7%、40.2%、8.0%(表3).比较ND6基因总的碱基组成和第二、三位点的碱基组成,发现C的含量都比较高.第二位C含量最高,为40.8%,存在反G偏移,含量为12.1%;第三位A含量最高,为40.2%,C的含量为28.7%,存在反G偏移,含量为4.9%.
将大鳞副泥鳅ND6基因核苷酸序列翻译为氨基酸序列时,在所有密码子中,UUU,UGU,UGC,UGA,CGU,CUG,CCG,CAG,ACG,GUA,GUG,GG%都没有使用到.使用频率最高的是AAA密码子(13.0),其次是AUU和CCC密码子(都是11.0);UCU,UUC,UUA,UUG,UGG,CUU,CAU,CUC,CGG,AGU,AUC,AUG,AGG,GAU,GUC,GGC,GGA,GCG密码子使用情况均为1.0.
2.2.6Cytb基因
大鳞副泥鳅Cytb基因的序列长度为1142 bp(表2).各碱基T、C、A、G含量分别为30.6%、27.0、27.3、15.1%(表3).比较Cytb基因总的碱基组成和第一、二位点的碱基组成,发现C的含量都比较高.比较Cytb基因三个位点的碱基组成,发现第一位点C含量很高,为25.7%,存在反G偏移,含量为13.4%;第二位点C含量明显高于G,为29.4%,存在反G偏移,含量为7.6%.
将大鳞副泥鳅Cytb基因核苷酸%列翻译为氨基酸序列时,在所有密码子中,GUC,GUA,GCG,CGU,AGG,GGC都没有使用到.使用频率最高的是UUC密码子(19.0),其次是AUU密码子(5.0);CGC,CGA,CGG,AGU,AGC,GGA密码子使用情况均为1.0.
2.3基于Cytb的大鳞副泥鳅系统分类分析
2.3.1大鳞副泥鳅Cytb与其他物种的同源性比较
利用DNAMAN5.0软件,将大鳞副泥鳅Cytb的DNA序列与泥鳅,丘氏益秀朝鲜鳅,洛东江高丽鳅,俄罗斯泥鳅,后鳍花鳅和斑马鱼6个物种分别进行比对,得到大鳞副泥鳅与其他物种的同源性系数.从表10中可知,大鳞副泥鳅与其他几种鳅科鱼类的碱基同源性均在为85%~88%之间,以洛东江高丽鳅同源性最高,丘氏益秀朝鲜鳅同源性最低.
2.3.2不同物种间的遗传距离
利用MEGA5.1软件计算出各个物种间的遗传距离,结果见表11(左下角为遗传距离,右上角为同源性系数).由表可知,大鳞副泥鳅与洛东江高丽鳅之间的遗传距离最小,为0.147,其次是俄罗斯泥鳅,为0.159;与斑马鱼的遗传距离最大,为0.303. 2.%.3基于大鳞副泥鳅Cytb序列的系统进化树
利用ClustalX1.83和MEGA5.1软件,将大鳞副泥鳅Cytb序列与其他6个物种进行比对.再用MEGA5.1软件中的NJ法构建分子进化树(图1).以斑马鱼为外类群,其余6个物种为内群,从所构建的系统树可以看出,内群的6个物种分为2个支系,第1支系由丘氏益秀朝鲜鳅、后鳍花鳅和泥鳅组成;第2支系由大鳞副泥鳅、俄罗斯泥鳅和洛东江高丽鳅组成,这六种鱼类都是鳅科,但分属于不同的属.大鳞副泥鳅与洛东江高丽鳅的亲缘关系最近,与丘氏益秀朝鲜鳅亲缘关系最远.
3 讨论

表10 大鳞副泥鳅与其他物种Cytb同源性比较
3.1MtDNA编码基因的序列特征分析
NADH还原酶复合体亚单位ND1、ND2、ND4、ND4L和ND6以及Cytb蛋白质编码基因中,都很明显存在反G偏移.ND1的第一、三位,ND2第一、三位,ND4第一、二、三位,ND4L第一、三位,ND6第二、三位,分析这几个基因的碱基组成发现,C的含量比较高,G的含量相对较低.分析这个基因三个位点的碱基组成发现,第一、二、三位存在的反G偏移概率较高,第四位碱基反G概率较低,说明第一、二、三位点对核苷酸有使用偏好.在脊椎动物中,第三位的反G偏移主要是因为该位的碱基在进化上选择压力比其他两位小,表现出明显的反G偏倚和碱基组成偏好性.
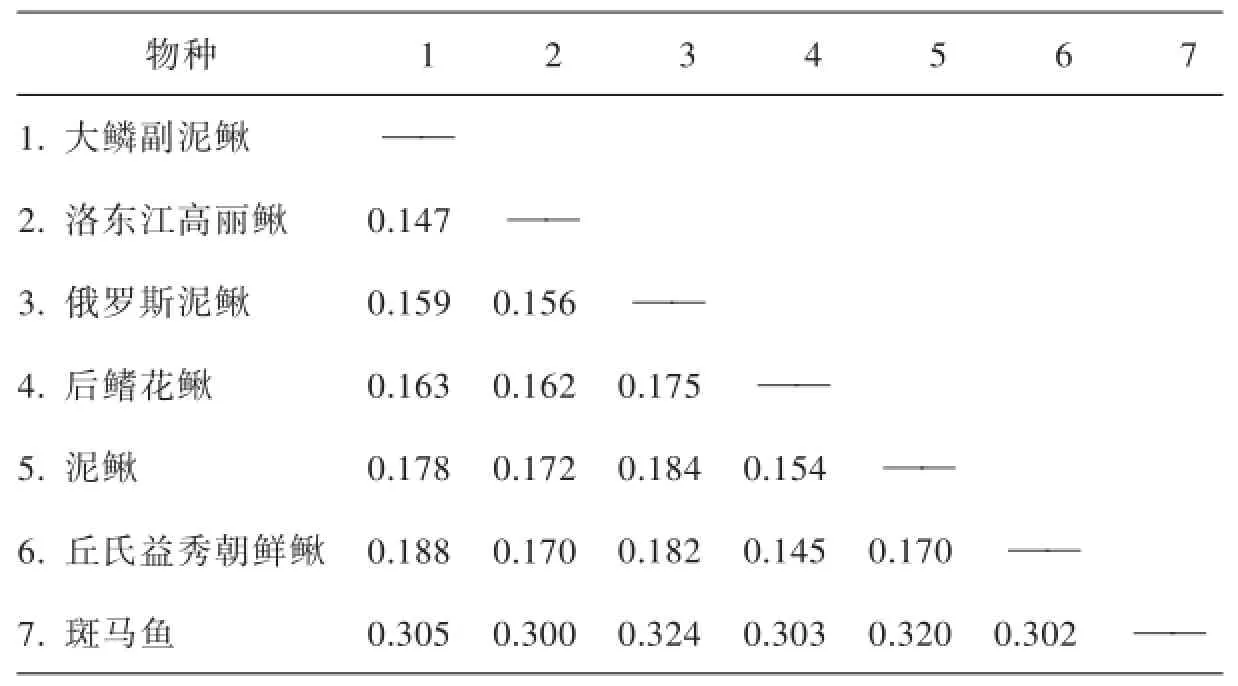
表11 几种鳅科鱼类遗传距离比较
3.2大鳞副泥鳅Cytb在系统分类中的适用性
从表11的各个物种间的遗传距离可知,大鳞副泥鳅与洛东江高丽鳅、俄罗斯泥鳅、后鳍花鳅泥鳅、泥鳅、丘氏益秀朝鲜鳅的遗传距离较接近(分别是0.147、0.159、0.163、0.178、0.188),与斑马鱼遗传距离较远(0.305).另外表10中计算出的大鳞副泥鳅与洛东江高丽鳅、俄罗斯泥鳅、后鳍花鳅泥鳅、丘氏益秀朝鲜鳅的碱基同源性分别为88%、87%、86%、85%、85%,显示大鳞副泥鳅与洛东江高丽鳅、俄罗斯泥鳅、后鳍花鳅泥鳅、丘氏益秀朝鲜鳅的关系较近,与斑马鱼的碱基同源性为68%,关系较远.认为这是基于两种算法有所差别所致,且相差很小.
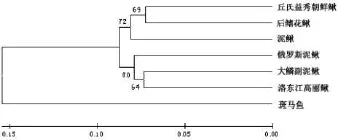
图1 由邻接法构建的系统树(基于Cytb序列)
进化树结果显示:以斑马鱼为外群,其余6个物种为内群,主要分为2个支系,第1支系由大鳞副泥鳅与洛东江高丽鳅首先聚为一群,再与俄罗斯泥鳅聚合,BCL值均为80%;第2支系由后鳍花鳅泥鳅、丘氏益秀朝鲜鳅、泥鳅组成,首先由后鳍花鳅泥鳅、丘氏益秀朝鲜鳅组成一群,再与泥鳅聚合,BCL值为72%.
随着研究的深入,以mtDNA中完整的基因序列或多个基因序列协同作用而获得的遗传信息来探讨鱼类的系统发育,将是今后研究的发展方向,它已在多种鱼类的系统发育中得到应用[15-16].本研究基于CYTB的几种鱼系统分类与传统形态学分类结果相符[1-2],可见Cytb基因与mtDNA中16SrRNA、12SrRNA和D-loop区等是鱼类系统进化中有效的分子标记手段.
[1]孟庆闻,苏锦祥,缪学祖.鱼类分类学[M].北京:中国农业出版社,1995.
[2]倪勇,伍汉霖.江苏鱼类志[M].北京:中国农业出版社,2006.
[3]吴焱,时月,凌去非,等.苏州地区泥鳅与大鳞副泥鳅肌肉微量元素含量比较[J].水产养殖,2011,32(10):%1-43.
[4]Ham L,Xu Y.Design of generalized E L Gamal type distal signature schemes based on discrete logarithm[J].Electron Lett,1994,30 (24):2025-2026.
[5]田功太,陈有光,段登选,等.大鳞副泥鳅人工繁育技术研究[J].河北渔业,2010(6):34-36.
[6]赵林斌.泥鳅苗种规模化繁育与高效生态养殖技术[J].中国水产,2007(5):40-42.
[7]张刚.洪泽湖大鳞副泥鳅苗种规模化繁育技术[J].中国水产,2004(3):48-50.
[8]Curole J P,Kocher T D.Mitogenomics:digging deeper with complete mitochondrial genomes[J].Tree,1999,14:394-398.
[9]Wallace D C.Mitochandrial DNA variation in human evolution,degenerative disease,and aging[J].Am J Hum Genet,1995,57: 201-223.
[10]Zhang Ya-Ping,Oliver A Ryder.Mitochondfial DNA sequence evolution in the Arctoidea[J].Proceedings of the National Academy of Sciences,1993,90(20):9557-9561.
[11]Russo CA,Takezaki N,Nei M.Efficiencies of different genes and different tree-building methods in recovering a known verte⁃brate phylogeny[J].Molecular biology and evolution,1996,13(3):525-536.
[12]Pollock D D,Eisen J A,Doggett N A,et al.A case for evolutionary genomics and the comprehensive examination of sequence bio⁃diversity[J].Molecular biology and evolution,2000,17(12):1776-1788.
[13]Roques S,Fox C J,Villasana M I,et al.The complete mitochondrial genome of the whiting,Merlangius merlangus and the had⁃dock,Melanogrammus aeglefinus:a detailed genomic comparison among closely related species of the Gadidae family[J].Gene, 2006,383:12-23.
[14]Sambrook J,Fritsch E F,Maniatic T.分子克隆试验指南[M].2版.金东雁,黎孟枫,译.北京:科学出版社,1996.
[15]肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J].水生生物学报,2000,24(4):384-391.
[16]郭新红,刘少军,刘巧,等.鱼类线粒体DNA研究新进展[J].遗传学报,2004,31(9):983-1000.
Sequence Analysis of Several Mitochondrial Genes in Paramisgurnus dabryanus
HAN Xiao-lei1,WANG Dan2,XU Tao1,ZHU Lv-ding3,WANG Fu-kai1,HAN Yao-ping1,ZHU Qing-hua3
(1.School of Biology and Food Engineering,Changshu Institute of Technology,Changshu 215500,China;2.Qianhuang Town Agricultural Machinery Station of Wujin District,Changzhou 213100,China;3.Qiuyu Aquiculture Professional Cooperative,Changzhou 213100,China)
This research obtained a total of 11556bp fragment of the Paramisgurnus dabryanus mitochondrial gene by using PCR method and analyzed the base composition and codon usage of protein genes in this frag⁃ment.The sequence contains 5 protein-encoding genes(ND1,ND2,ND4,ND4L and ND6)and one tRNA gene 14tRNA gene.The results showed that the average contents of A,T,C and G bases were 28.9%、27.7%、15.7% and 27.7%respectively,indicating a clear preference for base composition.The codon usage,base composition of ND1,ND2,ND4,ND4L,ND6 and Cytb genes and amino acid composition of the encoded proteins were ana⁃lyzed in this study,providing a basis for the study of genetic variation on P.dabryanus.
Paramisgurnus dabryanus;Mitochondrial DNA;NADH dehydrogenase;Cytb
Q959.468
A
1008-2794(2015)02-0110-06
2014-12-09
常州市科技支撑计划(农业)项目“泥鳅种质资源保存与提纯复壮技术研究”(CE20132014)
通讯联系人:韩晓磊,实验师,硕士,研究方向:淡水水生生物学,E-mail:hanxiaolei0724@163.com.
