大白菜CBL4基因的比较分析
2015-08-18李利斌纪复勤刘立锋李化银高建伟曹齐卫
李利斌+纪复勤+刘立锋+李化银+高建伟+曹齐卫

摘 要:分析了大白菜基因组中3个与拟南芥CBL4直系同源的基因,并进行了比较分析。结果表明,大白菜3个CBL4基因在序列和结构上非常保守,但在顺式调控元件上有所不同,暗示其在功能上存在分化。
关键词:大白菜;CBL4;遗传进化;序列分析;顺式元件
中图分类号:S634.101 文献标识号:A 文章编号:1001-4942(2015)06-0005-04
Comparative Analysis of CBL4 Genes in Chinese Cabbage
Li Libin1, Ji Fuqin2, Liu Lifeng1, Li Huayin1, Gao Jianwei1*, Cao Qiwei1*
(1.Vegetable Research Institute,Shandong Academy of Agricultural Sciences/National Improvement Center for
Vegetables, Shandong Branch/Key Laboratory of Greenhouse Vegetables Biology of Shandong Province,Jinan 250100, China;
2. Wanfeng Seed Limited Cooperation of Ningyang County in Shandong Province, Ningyang 271400,China)
Abstract Three genes in Chinese cabbage genome was comparatively analyzed with CBL4 orthologous gene of Arabidopsis. The results showed that 3 CBL4 genes of Chinese cabbage were very conserved in sequence and structure, but different in cis-elements,which suggested they were different in functions.
Key words Chinese cabbage; CBL4;Genetic evolution; Sequence analysis;Cis-element
植物钙调神经磷酸酶 B 类似蛋白(Calcineurin B like protein, 简称CBL)与钙调神经磷酸酶 B 类似蛋白互作的蛋白激酶(CBL-interacting protein kinase, 简称CIPK) 组成的信号途径简称CBL-CIPK途径,在植物钙信号转导和非生物逆境应答中具有重要功能,从低等的苔藓到高等的十字花科植物拟南芥、油菜及禾本科水稻等农作物以及木本植物杨树等物种中普遍存在[1~5]。CBL家族起到钙感受器的作用,与CIPK互作来调节下游基因的表达以应答不同的逆境信号[6]。有关CBL的功能在拟南芥中研究得最为清楚,其中CBL4/SOS3基因在植物高盐胁迫应答中具有重要功能,而且存在复杂的调控机制[7]。CBL4/SOS3可以与SOS2/CIPK24互作调控SOS1(一种钠离子质子泵)的表达和介导钙依赖的微丝重组从而调节植物盐胁迫的抗性[8,9];也可以通过与CIPK6互作介导钾离子通道AKT2定位在细胞膜上[10];在低盐胁迫下还可以通过调控生长素的重新分布来调节侧根的发育[11]。拟南芥CBL4/SOS3编码的蛋白具有结合钙离子的EF手结构,还有豆蔻酰化位点,与钙离子的结合和豆蔻酰化是其在盐胁迫应答中行使功能所必需的[12~14]。而且在CBL4/SOS3的碳末端存在保守的磷酸化位点,可被SOS2/CIPK24等磷酸化[15]。突变体的研究表明钙可以调节拟南芥CBL4/SOS3对盐的敏感性[12],拟南芥CBL4/SOS3可以调节离子平衡和氧化胁迫反应[16],而且它还受MYB73的负向调节[17];突变体研究和基因定位表明水稻和大麦的CBL4/SOS3基因在盐胁迫应答中具有与拟南芥CBL4/SOS3相似的功能[18,19],过表达玉米的CBL4基因可以提高拟南芥的抗逆性[20]。大白菜是我国重要的特产蔬菜,其有关CBL4/SOS3的研究尚缺乏报道。笔者在解析大白菜CBL家族基因的基础上[21],发现有三个大白菜CBL4基因与AtCBL4/SOS3直系同源。本文对这三个大白菜CBL4/SOS3类基因的染色体定位、基因结构、编码序列及其顺式调控元件进行了系统分析,为进一步研究其功能奠定了基础。
1 研究方法
利用拟南芥的CBL4基因序列在大白菜基因组数据库中(BRAD:http://brassicadb.org/brad/)搜索比对大白菜的基因组序列,寻找大白菜的CBL1基因序列,对鉴定出的CBL4基因进行结构分析和遗传进化分析,并对启动子序列中的顺式元件进行预测。结构分析利用GSDS(http://gsds. cbi. pku. edu. cn /) 在线进行,遗传进化分析利用MEGA4.1 进行,采用Bootstrap test-Neighbor Joining 方法,重复运算500 次,顺式元件预测采用PlantCARE 软件进行在线分析。亚细胞定位预测在WoLF PSORT(http://wolfpsort.org/)中进行。
2 结果与分析
2.1 大白菜CBL4基因的鉴定和特征分析
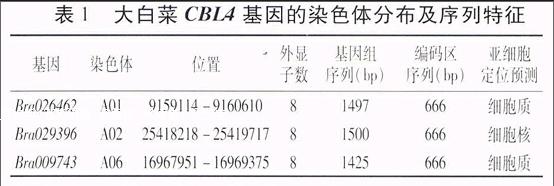
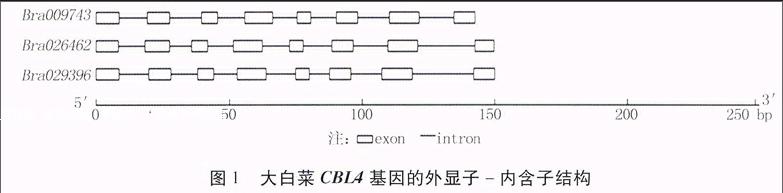
笔者利用生物信息学方法从大白菜基因组中鉴定出三个拟南芥CBL4的同源基因Bra026462、Bra029396和Bra009743,分别位于大白菜染色体A01、A02和A06上。大白菜三个CBL4的基因组序列中含有8个外显子,而且这8个外显子的大小和排列比较保守;预测的编码区序列均为666 bp,基因组序列大小略有不同,其编码蛋白预测定位于细胞核或细胞质中(表1)。endprint
表1 大白菜CBL4基因的染色体分布及序列特征
基因 染色体 位置 外显
子数 基因组
序列(bp) 编码区
序列(bp) 亚细胞
定位预测
Bra026462 A01 9159114-9160610 8 1497 666 细胞质
Bra029396 A02 25418218-25419717 8 1500 666 细胞核
Bra009743 A06 16967951-16969375 8 1425 666 细胞质
图1 大白菜CBL4基因的外显子-内含子结构
2.2 大白菜CBL4基因的遗传进化和序列比对
遗传进化分析是揭示基因功能的重要线索。通过对大白菜CBL4和拟南芥CBL基因的进化分析发现,大白菜的CBL4基因 Bra026462、 Bra029396和Bra009743是拟南芥CBL4/SOS3的直系同源基因(图2)。序列比对发现这三个基因非常保守(图3),三者的核苷酸序列一致性为92.94%,与拟南芥CBL4/SOS3的一致性分别为87.61%、85.99%和85.55%;三者预测编码蛋白的序列一致性为92.74%,与AtCBL4的一致性分别为82.67%、78.22%和81.78%,与AtCBL8的序列一致性分别为58.22%、58.67%和58.22%。这说明大白菜的三个CBL4类基因可能具有相似的生物学功能。
图2 大白菜三个CBL4基因预测编码蛋白的序列比对
图3 大白菜CBL4基因与拟南芥CBL
基因的遗传进化分析
2.3 大白菜CBL4基因的顺式调控元件和功能预测
转录水平的调控是植物基因表达的主要调控步骤,植物基因表达是自身顺式调控元件与转录因子相互作用的结果。因此,植物基因上游顺式调控元件的分析对于揭示基因的功能具有重要意义。由表2可知,大白菜三个CBL4基因均含有多个激素和逆境应答顺式调控元件,而且各不相同,说明它们在功能上存在分化。Bra026462可能响应茉莉酸甲酯和水杨酸信号,受干旱、热胁迫调控表达;Bra029396 可能响应茉莉酸甲酯、赤霉素和水杨酸信号,可能应答干旱和冷胁迫;Bra009743可能响应脱落酸、茉莉酸甲酯和水杨酸信号,应答热胁迫、干旱和冷胁迫。
表2 大白菜CBL4基因的顺式调控元件
基因
ABRE
CGTCA
-motif GARE
-motif
HSE LTR MBs TCA
-element
Bra026462〖3〗 1 2 2 1
Bra029396 2 1 2 1 2
Bra009743 1 1 1 2 2 1
注:ABRE为脱落酸应答元件;CGTCA-motif为茉莉酸甲酯应答元件;GARE-motif为赤霉素应答元件;HSE为热胁迫应答元件;LTR为冷胁迫应答元件;MBs为干旱诱导表达元件;TCA-element为水杨酸应答元件。
3 结论
本文从大白菜基因组中鉴定出三个与拟南芥CBL4直系同源的基因Bra026462、Bra029396和Bra009743,在结构和序列上十分保守,推测是由于基因扩增的结果,可能在盐胁迫应答过程中具有相似的功能。但是,它们分布在不同的染色体上,而且在其上游序列中含有的顺式调控元件的数目和类型不尽相同,亚细胞定位预测的结果也不一样。因此,笔者认为大白菜三个CBL4基因Bra026462、Bra029396和Bra009743在功能上有所分化,具体情况需要进一步研究。
参 考 文 献:
[1] Kolukisaoglu U, Weinl S, Blazevic D, et al. Calcium sensors and their interacting protein kinases: genomics of the Arabidopsis and rice CBL-CIPK signaling networks[J]. Plant Physiology,2004,134(1):43-58.
[2] Kleist T J, Spencley A L, Luan S. Comparative phylogenomics of the CBL-CIPK calcium-decoding network in the moss Physcomitrella, Arabidopsis, and other green lineages[J]. Front. Plant Sci., 2014,5:187.
[3] Gong D, Guo Y, Schumaker K S, et al. The SOS3 family of calcium sensors and SOS2 family of protein kinases in Arabidopsis[J]. Plant Physiology,2004,134(3):919-26.
[4] Zhang H, Yang B, Liu W Z, et al. Identification and characterization of CBL and CIPK gene families in canola (Brassica napus L.)[J]. BMC Plant Biology,2014, 14:8.
[5] Tang R J, Liu H, Bao Y, et al. The woody plant poplar has a functionally conserved salt overly sensitive pathway in response to salinity stress[J]. Plant Mol. Biol., 2010,74(4-5):367-380.endprint
[6] Sánchez-Barrena M J, Martínez-Ripoll M, Albert A. Structural biology of a major signaling network that regulates plant abiotic stress: the CBL-CIPK mediated pathway[J]. Int. J. Mol. Sci., 2013,14(3):5734-5749.
[7] Ji H, Pardo J M, Batelli G, et al. The Salt Overly Sensitive (SOS) pathway: established and emerging roles[J]. Molecular Plant, 2013, 6(2):275-286.
[8] Qiu Q S, Guo Y, Dietrich M A, et al. Regulation of SOS1, a plasma membrane Na+/H+ exchanger in Arabidopsis thaliana, by SOS2 and SOS3[J]. Proc. Natl. Acad. Sci. USA, 2002,99(12):8436-8341.
[9] Ye J, Zhang W, Guo Y. Arabidopsis SOS3 plays an important role in salt tolerance by mediating calcium-dependent microfilament reorganization[J]. Plant Cell Report, 2013, 32(1):139-148.
[10]Held K, Pascaud F, Eckert C, et al. Calcium-dependent modulation and plasma membrane targeting of the AKT2 potassium channel by the CBL4/CIPK6 calcium sensor/protein kinase complex[J]. Cell Research,2011, 21(7):1116-1130.
[11]Zhao Y, Wang T, Zhang W, et al. SOS3 mediates lateral root development under low salt stress through regulation of auxin redistribution and maxima in Arabidopsis[J]. New Phytologist,2011,189(4):1122-1134.
[12]Horie T, Horie R, Chan W Y, et al. Calcium regulation of sodium hypersensitivities of sos3 and athkt1 mutants[J]. Plant Cell Physiology,2006, 47(5):622-633.
[13]Sánchez-Barrena M J, Martínez-Ripoll M, Zhu J K, et al. The structure of the Arabidopsis thaliana SOS3: molecular mechanism of sensing calcium for salt stress response[J]. Journal of Molecular Biology,2005, 345(5):1253-1264.
[14]Ishitani M, Liu J, Halfter U, et al. SOS3 function in plant salt tolerance requires N-myristoylation and calcium binding[J]. Plant Cell,2000,12(9):1667-1678.
[15]Halfter U, Ishitani M, Zhu J K. The Arabidopsis SOS2 protein kinase physically interacts with and is activated by the calcium-binding protein SOS3[J]. Proc. Natl. Acad. Sci. USA,2000, 97(7):3735-3740.
[16]Zhu J, Fu X, Koo Y D, et al. An enhancer mutant of Arabidopsis salt overly sensitive 3 mediates both ion homeostasis and the oxidative stress response[J]. Molecular Cell Biology,2007, 27(14):5214-5224.
[17]Kim J H, Nguyen N H, Jeong C Y,et al. Loss of the R2R3 MYB, AtMyb73, causes hyper-induction of the SOS1 and SOS3 genes in response to high salinity in Arabidopsis[J]. Plant Physiology,2013,170(16):1461-1465.
[18]Rivandi J, Miyazaki J, Hrmova M, et al. A SOS3 homologue maps to HvNax4, a barley locus controlling an environmentally sensitive Na+ exclusion trait[J]. Journal of Experimental Botany,2011,62(3):1201-1216.
[19]Martínez-Atienza J, Jiang X, Garciadeblas B, et al. Conservation of the salt overly sensitive pathway in rice[J]. Plant Physiology,2007,143(2):1001-1012.
[20]Wang M, Gu D, Liu T, et al. Overexpression of a putative maize calcineurin B-like protein in Arabidopsis confers salt tolerance[J]. Plant Molecular Biology,2007,65(6):733-746.
[21]李利斌,王殿峰,刘立锋,等. 大白菜CBL家族基因的鉴定和遗传进化分析[J].山东农业科学,2009(5):4-7.endprint
