福州野生蕉FeSOD 家族基因的克隆及在低温胁迫下的表达分析
2015-07-05赖恭梯谢晓清林玉玲赖钟雄
冯 新,徐 蕾,赖恭梯,谢晓清,林玉玲,赖钟雄
(福建农林大学 园艺植物生物工程研究所,福州350002)
植物在生长发育过程中往往会受到各种生物和非生物胁迫的影响,从而诱发细胞内活性氧(ROS)的代谢失衡,产生氧化胁迫,引起细胞膜、蛋白、DNA 等氧化损伤进而导致细胞代谢紊乱,严重时细胞死亡。超氧化物歧化酶(superoxide dismutase,SOD)是抗氧化酶系统的第一道防线,能快速地催化超氧阴离子自由基发生歧化反应生成分子氧和过氧化氢,过氧化氢再经过氧化氢酶或愈创木酚等过氧化物酶作用生成水,从而解除细胞内的氧化胁迫,保护植物免受氧化损伤[1]。根据酶活性中心金属离子的不同,植物SOD 分为Cu/ZnSOD、MnSOD 和Fe-SOD。3种类型的SOD 具有不同的亚细胞定位,如Cu/ZnSOD 主要存在于细胞质、叶绿体和过氧化物酶体中,MnSOD 主要存在于线粒体中,而FeSOD主要存在于叶绿体和细胞质中[2]。由于超氧阴离子自由基不能透过磷脂膜,不同亚细胞定位的SOD 在各自的位置上行使相应的活性氧清除功能,以保证生物体各种代谢的正常进行。FeSOD 是唯一一种存在于厌氧古细菌中的SOD 同工酶,因而在进化上被认为是最古老的一类SOD,目前已在原核生物和植物中鉴定到FeSOD,但未在动物中发现[3]。FeSOD蛋白通常以同源二聚体或同源四聚体的形式存在,其酶活性仅受H2O2抑制。近年研究表明,FeSOD活性还受铁伴侣蛋白CPN20的直接调控[3-4]。
植物FeSOD 家族基因一般有1~3个成员,目前已从拟南芥、油菜、小麦和杨树等多种植物中克隆得到[5-9]。其基因组DNA 通常含有6~8 个内含子[9-10]。已有的研究表明FeSOD 家族基因参与植物的体胚发生过程,且不同成员表达模式存在差异[8,11]。单宁伟[12]研究发现,失水胁迫处理切花月季可诱导其花瓣产生3条新的FeSOD 酶条带。转录水平研究表明FeSOD 基因的表达受光照、低温、NaCl和百草枯等逆境胁迫的诱导[13-14],此外,还受生长素、脱落酸、赤霉素和激动素等生长调节因子的调控[15]。在玉米中过表达拟南芥FeSOD 基因能有效提高玉米的耐寒和抗氧化能力,并使其生长速率加快[16]。因此,FeSOD 在植物的抗逆中也发挥重要作用。
中国作为香蕉的起源地之一,蕴藏着丰富的野生蕉资源,本实验室经多年考察发现福建分布着大量的野生蕉资源,其中位于福州北峰海拔300~500 m 的二倍体野生香蕉(命名为福州野生蕉),在自然野生状态下能耐受冬季-2 ℃以下低温,是香蕉抗寒育种的重要种质资源[17]。而香蕉FeSOD 家族基因及其与抗寒性的关系至今未见报道。本研究以福州野生蕉为材料,克隆FeSOD 家族基因的不同成员,分析不同成员的基因结构、蛋白的结构域、基本理化性质、亚细胞定位等特征,并借助qRT-PCR 技术检测不同成员在低温胁迫过程中的表达情况,以期为探讨香蕉的抗氧化能力与抗寒之间的关系奠定基础,为香蕉的抗寒遗传改良提供科学依据。
1 材料和方法
1.1 植物材料与胁迫处理
本研究以福州野生蕉(Musaspp.)的组培苗为材料,选取植株大小和长势一致的组培苗进行不同温度(28 ℃、20 ℃、13 ℃、4 ℃和0 ℃)处 理,处 理 时间为36h,进行2次重复处理,处理结束后收集叶片于液氮中速冻,之后保存于-80 ℃超低温冰箱用于后续研究。
1.2 核酸提取与cDNA 合成
分别采用Column Plant RNAOUT 2.0试剂盒(TIANDZ,China)和CTAB法[18]进行香蕉总RNA和DNA 的提取。选择经紫外分光光度计检测OD260/280在1.8~2.0之间,且凝胶电泳显示其完整性良好的核酸备用。采用Thermo Scientific RevertAid First Strand cDNA Synthesis Kit(Fermentas,EU)将总RNA 逆转录为带AP(GGCCACGCGTCGACTAGTACTTTTTTTTTTTTTTTTTT)接头的cDNA,作为ORF和3′-UTR 扩增的模板。采用SMARTTMRACE cDNA Amplification kit(Takara,Japan)进行cDNA 逆转录,用于5′-UTR 扩增。

表1 基因克隆及qRT-PCR引物序列信息Table 1 Primers used for gene cloning and qRT-PCR
1.3 引物设计及PCR扩增
根据GenBank上已知的植物FeSOD 基因序列和小果野生蕉(Musa acuminata var.DH-Pahang,AA group)基因组数据库(http://banana-genome.cirad.fr/)中注释为FeSOD 的序列信息,经序列多重比对分析,在FeSOD 各基因成员的保守区分别设计3′-RACE 和5′-RACE 引物,用于扩增3′末端和5′末端,经测序后,在DNAMAN 上进行序列拼接,根据拼接全长预测最大ORF 区,设计引物进行ORF验证分析,本研究所用的引物信息见表1。以DNA 为 模 板,FSD1B-vF 和FSD1B-vR 为 引 物,扩增MuFSD1B 的gDNA 序列,用以分析可变剪接转录本的类型和基因结构。PCR 反应程序为:94℃预变性3min;94 ℃变性30s,Tm(根据引物具体温度设定)退火30s,72 ℃延伸时间根据片段大小设定,共35个循环;72 ℃继续延伸10min。PCR 扩增产物经1.0%琼脂糖凝胶电泳分离后,回收,连接到pMD 18-T(Takara,Japan)载体,并选取阳性克隆子送测。
1.4 基因序列分析
采用DNAMAN 6.0软件进行核苷酸序列比对和拼接。使用在线软件对福州野生蕉FeSOD 基因及其推导的氨基酸进行同源性(NCBI,http://www.ncbi.nlm.nih.gov/)、氨 基 酸 基 本 理 化 性 质(http://web.expasy.org/protparam/)、亚 细 胞 定 位(WoLF PSORT,http://wolfpsort.org/;Soft Berry,http://linux1.soft berry.com/;)、N 端目标序列(Predotar,https://urgi.versailles.inra.fr/predotar/predotar.html)、磷 酸 化 位 点(NetPhos 2.0,http://www.cbs.dtu.dk/services/NetPhos/)、功 能 位 点(Prosite,http://www.predict protein.org/)、保 守 结 构 域(SMART,http://smart.embl-heidelberg.de/)、二 级结构(Psired,http://bioinf.cs.ucl.ac.uk/)和三级结构(Swissmodel,http://swissmodel.expasy.org/)的预测与分析。采用Mega 5.02 软件的邻位相连法(Neighbor Joining)构建植物FeSOD 氨基酸的分子系统进化树。采用ClustalX(1.83)软件进行氨基酸序列的多重比对分析。
1.5 亚细胞定位分析
根据在线数据库预测分析显示福州野生蕉Fe-SOD 家族各基因成员均定位于叶绿体的可能性最大,因而本研究以MuFSD1A 为代表进行亚细胞定位验证。根据MuFSD1A 基因ORF 序列设计带酶切位点NcoⅠ的上游引物MuFSD1A-subF(CAT-下划线标示酶切位点,粗体标示保护性碱基,下同),带酶切位点SpeⅠ的下游引物MuFSD1A-subR扩增MuFSD1A 基因片段并连接到pMD18-T(Takara),经测序后,双酶切,连接到pCAMBIA1302载体GFP的N 端,构建融合表达载体35S∷MuFSD1AGFP,采用冻融法导入农杆菌EHA105中,并用注射法侵染烟草叶片。同时用水和pCAMBIA1302 空载体注射的烟草叶片分别作为阴性和阳性对照,3d后于激光共聚焦显微镜(Olympus FV1000)下观察35S∷MuFSD1A-GFP的定位情况并拍照。
1.6 福州野生蕉FeSOD 各基因成员在不同温度处理下的qRT-PCR分析
以Chen等[19]筛选的在非生物胁迫下表达较稳定的CAC(clathrin adaptor complexes medium)基因作为本研究定量分析的内参基因,引物序列为CAC-QF(AACTCCTATGTTGCTCGCTTATG)和CAC-QR(GGCTACTACTTCGGTTC TTTCAC),扩增片段148bp。以经不同温度处理植株的cDNA为模板,在基因的特异位置设计定量引物(表1),分析福州野生蕉各FeSOD 基因在不同温度处理下的表达情况。根据SYBR ExScript试剂盒(Takara,Japan)说明书配制荧光定量PCR 反应液,在Lihgt-Cycler480仪器上进行PCR 扩增,重复3 次。PCR反应条件为:95 ℃预 变 性30s;95 ℃变性10s,57℃退火30s,72 ℃延伸30s,共40个循环。首先,以不同处理的cDNA 模板混合样的15倍稀释液为模板,经qRT-PCR扩增后分析各基因的扩增曲线、融解曲线(60~95 ℃)和PCR 产物的凝胶电泳条带,以确定引物的特异性。随后,在样品扩增的同时,对不同处理的cDNA 模板混合样进行5倍梯度系列稀释,分别对FeSOD 各基因成员和内参基因CAC进行扩增,获得标准曲线和扩增效率。数据采用Excel和geNORM(version 3.5)[20]软件进行分析。
2 结果与分析
2.1 福州野生蕉FeSOD 家族基因不同成员cDNA全长序列的克隆与分析
以福州野生蕉叶片总RNA 逆转录的cDNA 为模板,经RACE扩增得到2条大小分别为661bp和868bp的3′-末端(图1,A、B),以及2条大小分别为690bp和439bp的5′-末端(图1,C、D)。在DNAMAN 上对已获得的3′-末端序列和5′-末端序列进行比对及拼接,发现可能存在2个基因成员,进一步设计ORF 验证引物,经RT-PCR 扩增得到分别为903bp和1 211bp的2个片段(图1,E、F),TA 克隆后送测。测序结果显示,这2 条序列与DNAMAN 拼接结果一致。将这2条序列的核苷酸和推导的氨基酸序列分别在NCBI上进行Blast分析,结果显示,与数据库中已知的百脉根(Lotus japonicus)、拟 南 芥(Arabidopsis thaliana)、葡 萄(Vitis vinifera)、蓖麻(Ricinus communis)、水稻(Oryza sativa)和龙眼(Dimocarpus longan)等的FeSOD/FeSOD 的序列有较高的同源性,因此推断已成功获得福州野生蕉FeSOD 的2 个基因成员,分别命名为MuFSD1A(登录号为JX844026)和MuFSD1B(登录号为KJ786318)。
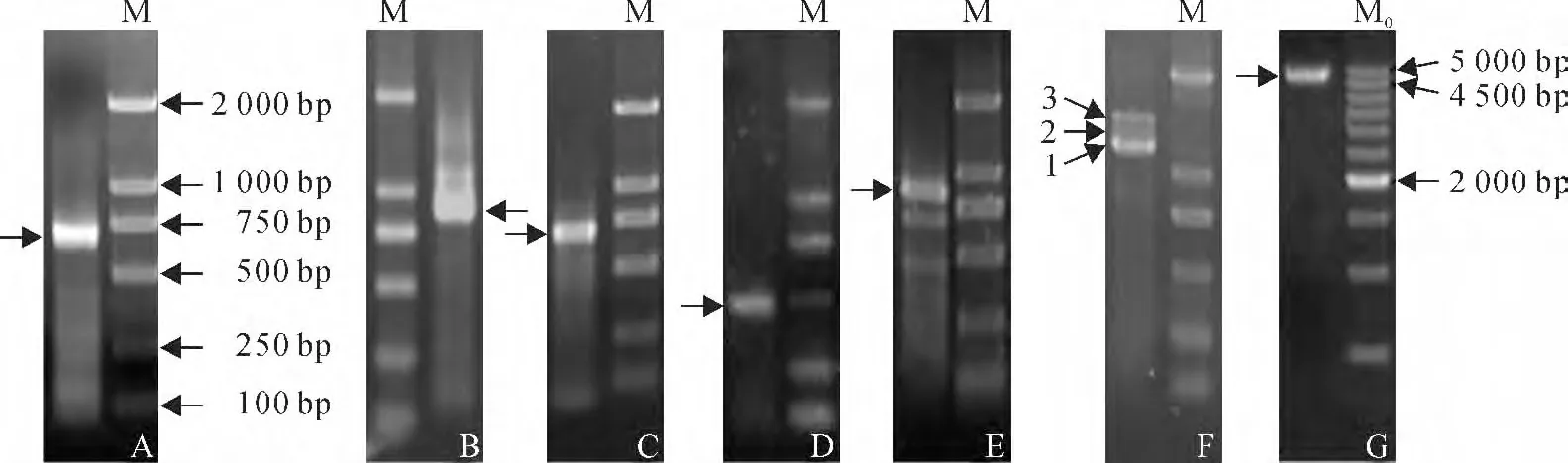
图1 MuFSD1A 和MuFSD1B 的PCR 扩增M.D2000DNA ladder(Solarbio);Mo.500bp DNA ladder marker(Takara);A.MuFSD1A-3′-末端;B.MuFSD1B-3′-末端;C.MuFSD1A-5′-末端;D.MuFSD1B-5′-末端;E.MuFSD1A-ORF验证;F.MuFSD1B-ORF验证,1为正常转录本,2和3为可变剪接转录本;G.MuFSD1B-gDNA 扩增;箭头所指为特异条带Fig.1 PCR amplification of MuFSD1Aand MuFSD1B M.D2000DNA ladder(Solarbio);Mo.500bp DNA ladder marker(Takara);A.MuFSD1A-3′-end;B.MuFSD1B-3′-end;C.MuFSD1A-5′-end;D.MuFSD1B-5′-end;E.MuFSD1A-ORF verification;F.MuFSD1B-ORF verification,1is normal transcript,2and 3are variant transcripts;G.MuFSD1B-gDNA amplification;arrows show the specific bands
MuFSD1A 的cDNA 全长为1 277bp,开放阅读框为903bp,编码300 个氨基酸,5′-UTR 为114 bp,3′-UTR 为260bp;MuFSD1B 的cDNA 全长为1 378bp,开 放 阅 读 框 为783bp,编 码2 6 0个 氨 基酸,5′-UTR 为133bp,3′-UTR 为462bp。与小果野生蕉(Musa acuminata DH-Pahang,AA group)基因组中注释为FeSOD 的序列进行比对发现,MuFSD1A 开放阅读框的cDNA 序列与小果野生蕉基因组中编号为GSMUA_Achr11P23560的FeSOD序列长度一致,且相似性达到97.79%。而MuFSD1B 的ORF 序列与基因组中注释为FeSOD的另外4 条序列(编号分别为GSMUA_Achr10P-27220、27190、27230 和27210)有较高的相似性(图2),推测它们为同源基因。但本研究获得的福州野生蕉MuFSD1B 的ORF 序列比小果野生蕉的长,编码1个更长的蛋白。
2.2 福州野生蕉FeSOD 可变剪接转录本与基因结构分析
在验证MuFSD1B 的ORF 时,获得了2 个比MuFSD1B 的ORF 条带略长的转录本(图1,F)。经测序后,与MuFSD1B 的ORF 序列比对显示,这2条转录本的3′端分别多出了1段(87bp)和2段(87bp和172bp)的序列。为了进一步分析这2条转录本的特征,以福州野生蕉DNA 为模板,MuFSD1B-vF 和MuFSD1B-vR 为 引 物,扩 增 得 到MuFSD1B 基因4 982bp的gDNA 片段(图1,G),测序结果分析表明开放阅读框的gDNA 序列为4 189bp。与gDNA序列比对结果表明,这2条转录本分别是由MuFSD1B 基因在转录过程中的第7个内含子和第7、8个内含子共同驻留产生的可变剪接转录本,因而将这2条转录本分别命名为MuFSD1Bvariant1(登录号为KJ786319)和MuFSD1B-variant2(登录号为KJ786320)。

图2 福州野生蕉MuFSD1B 与小果野生蕉FeSOD 基因的序列比对GSMUA_Achr10P27220、27190、27230和27210为小果野生蕉基因组中注释为FeSOD 的序列。颜色由深到浅表示序列间一致性由高到低Fig.2 Sequence alignment of MuFSD1Band FeSODs from M.acuminata DH-Pahang GSMUA_Achr10P27220,27190,27230and 27210are sequences from the genome database of M.acuminata DH-Pahang,which are annotated as FeSOD.Colors vary from dark to light means that the identities of sequences are from high to low
由于MuFSD1A 的ORF序列与小果野生蕉的GSMUA_Achr11P23560 基因高度相似,因而采用GSMUA_Achr11P23560的gDNA 序列(4 075bp)为参考序列分析MuFSD1A 的外显子和内含子组成。如图3显示,MuFSD1A 基因由8个外显子和7个内含子组成,最长和最短的外显子分别为210 bp和24bp,而最长和最短的内含子分别为2 281 bp和72bp;MuFSD1B 基因由9个外显子和8个内含子组成,最长和最短的外显子分别为189bp和24bp,而最长和最短的内含子分别为1 804bp 和79bp。MuFSD1A 和MuFSD1B 的起始密码子和终止密码子均为ATG 和TGA,而可变剪接转录本MuFSD1B-variant1 和MuFSD1B-variant2 均 由于第7个内含子驻留导致终止密码子提前,变为TAA,因而都具有7个外显子和6个内含子。所有福州野生蕉FeSOD 基因成员的内含子剪切位点均符合真核生物内含子剪接规律,即“GT-AG”规则。
2.3 福州野生蕉FeSOD氨基酸序列比对与生物信息学分?析
虽然MuFSD1A 基因和MuFSD1B 基因推导的氨基酸序列的相似性比较低,仅有33.33%,但与来自拟南芥的3 个FeSOD(AtFSD1、AtFSD2 和AtFSD3)的多重序列比对结果显示(图4),MuFSD1A 和MuFSD1B 均具有植物FeSOD 保 守的金属结合位点和区别于MnSOD 的特征氨基酸,且SMART 软件分析显示这2个成员均含有Sod_Fe_N 结构域和Sod_Fe_C 结构域。这些结果表明本研究所获得的MuFSD1A 和MuFSD1B这2个成员均具有FeSOD 的基本功能和特性。
采用生物信息学方法进一步对所获得的这2个FeSOD 成员进行理化性质和结构特征的分析,以了解其蛋白的生物学活性及潜在的功能。结果(表2)表明,MuFSD1A 和MuFSD1B 均为亲水蛋白,但MuFSD1A 为酸性不稳定蛋白,而MuFSD1B 为碱性稳定蛋白。亚细胞定位结果均显示MuFSD1A和MuFSD1B 定位于叶绿体的可能性最大。Predotar v1.03 分 析 结 果 亦 显 示 MuFSD1A 和MuFSD1B均具有导向叶绿体的N 端目标序列,这也验证了亚细胞预测结果的准确性,说明福州野生蕉的FeSOD 主要是参与叶绿体中超氧阴离子自由基的清除。NetPhos 2.0软件预测显示MuFSD1A具有16个磷酸化位点(丝氨酸位点10个,苏氨酸位点3个,酪氨酸位点3个),而MuFSD1B具有14个磷酸化位点(丝氨酸位点7个,苏氨酸位点2个,酪氨酸位点5个)。此外,MuFSD1A 具有6个蛋白激酶C磷酸化位点,6个酪蛋白激酶Ⅱ磷酸化位点,3个N 端酰基化位点,1个酰胺化位点和1个Mn/Fe-SOD 特 征 位 点;而MuFSD1B 具 有2 个cAMP 和CGMP依赖性蛋白激酶磷酸化位点,4个蛋白激酶C磷酸化位点,1个酪蛋白激酶Ⅱ磷酸化位点,1个N 端酰基化位点和1个Mn/Fe-SOD 特征位点。以上磷酸化位点分析结果表明,福州野生蕉FeSOD 中存在丰富的磷酸化位点,且不同成员间磷酸化位点及类型的差异可能与它们在香蕉不同的发育进程或逆境胁迫中受到不同因子的调控(磷酸化或去磷酸化)有关。二级结构分析显示MuFSD1A 蛋白由42.67%α-螺旋,6.33%β-折叠和51.00%无规则卷曲 组 成,而MuFSD1B 蛋 白 由50.77% α-螺 旋,6.93%β-折叠和42.31%无规则卷曲组成。以PDB数据库中相似性最高的FeSOD(编号为1unf.1)为模板构建福州野生蕉FeSOD 蛋白的三级结构模型亦显示,MuFSD1A 和MuFSD1B 的三级结构存在明显差异(图5)。综上,本研究获得的福州野生蕉MuFSD1A 和MuFSD1B这2个成员在蛋白的理化性质,磷酸化位点及类型,二级和三级结构均存在明显差异,暗示它们的具体功能可能存在差异。

图3 福州野生蕉FeSOD 家族基因的基因结构Fig.3 Genomic structure of FeSODfamily genes from the wild banana in Fuzhou
2.4 福州野生蕉FeSOD的系统进化分析

图4 福州野生蕉与拟南芥FeSOD 氨基酸序列的多重比对▼表示保守的金属键结合位点氨基酸;*表示FeSOD催化活性中心的氨基酸酪氨酸;◆表示FeSOD区别于MnSOD的特征氨基酸,而Sod_Fe_N 和Sod_Fe_C 结构域分别用单和双直线表示。MuFSD1A 和MuFSD1B为福州野生蕉FeSOD;AtFSD1、AtFSD2和AtFSD3为拟南芥FeSODFig.4 Multiple sequence alignment of the FeSOD amino acids from the wild banana in Fuzhou and A.thaliana▼represents residues essential for conserved metal ligand binding sites;*represents tyrosines essential for catalytic activity;◆represents residues important for distinguishing FeSOD from MnSOD;Sod_Fe_N and Sod_Fe_C domains are marked with single and double lines,respectively.MuFSD1Aand MuFSD1Bare FeSODs from the wild banana in Fuzhou;AtFSD1,AtFSD2and AtFSD3are FeSODs fromArabidopsis thaliana

表2 福州野生蕉FeSOD的基本理化性质Table 2 Basic parameters of FeSOD from the wild banana in Fuzhou
为了分析福州野生蕉中FeSOD 不同成员的进化关系,采用Mega 5.02对MuFSD1A、MuFSD1B和来自NCBI中已知的其他17种植物FeSOD 基因的氨基酸序列进行聚类分析(图6),结果显示植物FeSOD 基因的氨基酸序列可分为两大分支(Ⅰ和Ⅱ),各分支的FeSOD 可能具有不同的基因起源。Ⅰ和Ⅱ大分支中均包含2个小分支,即双子叶植物分支和单子叶植物分支。福州野生蕉MuFSD1A位于Ⅰ大分支的单子叶植物类中,与马蹄莲FeSOD的亲缘关系最近;而MuFSD1B 聚到Ⅱ大分支的单子叶植物类中,与海枣FeSOD 的亲缘关系最近,其次为手参、二穗短柄草、小麦、水稻和玉米的,与双子叶植物的亲缘关系较远。香蕉2个成员聚类到不同的大分支中,这与它们氨基酸间的低一致性相符合(33.33%),暗示它们来自不同的基因祖先。
2.5 福州野生蕉FeSOD 基因的亚细胞定位
将水(阴性对照)、pCAMBIA1302(阳性对照)和35S∷MuFSD1A-GFP分别注射烟草叶片进行瞬时表达,25 ℃培养3d后于激光共聚焦显微镜下观察福州野生蕉MuFSD1A 蛋白的亚细胞定位情况。结果(图版Ⅰ)表明,阴性对照未见GFP绿色荧光信号(图版Ⅰ,A),但保卫细胞中叶绿体的自发红色荧光信号可以观察到(图版Ⅰ,B、D);阳性对照可以观察到在叶绿体、细胞质、细胞膜和细胞核上均有GFP绿色荧光信号(图版Ⅰ,E、H),同时可观察到叶绿体的自发红色荧光信号(图版Ⅰ,F),及其与绿色荧光信号叠加(图版Ⅰ,H);转35S∷MuFSD1AGFP的烟草可在其保卫细胞的叶绿体上观察到明显的GFP绿色荧光信号和自发红色荧光信号(图版Ⅰ,I、J、L),此外还观察到在其细胞质中也存在比较弱的GFP绿色荧光信号(图版Ⅰ,I、L),说明福州野生蕉MuFSD1A的蛋白主要定位在叶绿体中,这与生物学信息学的预测结果一致。

图5 福州野生蕉FeSOD 的三级结构Fig.5 Tertiary structure of FeSOD from the wild banana in Fuzhou

图6 福州野生蕉与其他植物FeSOD 基因氨基酸序列的进化树分析进化树分支点的数字表示Bootstrap验证中基于1 000次重复该节点可信度的百分比,各物种FeSOD的GenBank登录号列在括号内。标尺表示进化距离Fig.6 Phylogenetic tree based on the amino acid sequences of FeSODfrom the wild banana and other plants Numbers at the nodes represent the reliability percent of Bootstrap values based on 1 000replications.The GenBank accession numbers of FeSOD from various plants are in brackets.The bar represents evolutionary distances
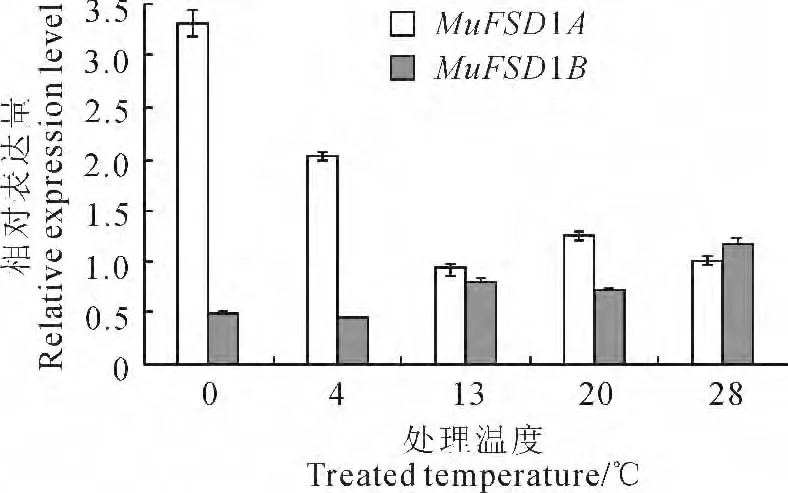
图7 福州野生蕉FeSOD 在不同温度处理下的定量表达Fig.7 qRT-PCR analyses of FeSODunder different temperature treatments
2.6 福州野生蕉FeSOD 家族基因在不同温度条件下的表达分析
福州野生蕉具有较强的抗寒能力,而植物抗逆能力被认为与其抗氧化能力密切相关,分析福州野生蕉FeSOD 家族基因对低温的应答模式有助于了解该类型基因在香蕉抗寒中的作用。由于MuFSD1B 可 变 剪 接 转 录 本MuFSD1B-variant1和MuFSD1B-variant2的特异序列(即所包含的驻留内含子序列)存在较多的同种或2种碱基连续结构,本研究分析MuFSD1B 基因所有转录本类型的总体表达模式。qRT-PCR 结果显示(图7),当处理温度高于香蕉生长的限制温度13 ℃时,MuFSD1A和MuFSD1B 基因相对表达量均变化不大,当温度低于13℃时,MuFSD1A 基因表达量随着温度的下降持续上升,在4 ℃时的表达量是28 ℃时2倍,而温度降到0 ℃时,其表达量是28 ℃时3 倍以上。MuFSD1B 基因表达量在4 ℃时下降到28 ℃时的0.5倍以下,之后保持基本不变。以上结果说明福州野生蕉FeSOD 家族基因参与低温应答,且不同成员发挥不同的功能,其中MuFSD1A 基因受低温诱导,且表达强烈,可能在香蕉抗寒中起主要作用。
3 讨 论
3.1 福州野生蕉FeSOD 家族基因具有2个成员
植物FeSOD 家族基因在不同物种中的成员数量不同,如拟南芥中存在3 个FeSOD 基因(AtFSD1,AtFSD2,AtFSD3)[5],杨树中亦存在3个FeSOD 基因(PtFSD2.1,PtFSD2.2,PtFSD3)[9],龙眼中存在2个FeSOD 基因(DlFSD1a,DlFSD1b)[8],巴尔干苔苣中仅有1个(HrFSD1)[21]。小果野生蕉基因组中注释为FeSOD 的基因序列共有5条[22]。其中GSMUA_Achr11P23560基因与福州野生蕉MuFSD1A 的一致性高达97.79%,说明该基因成员在香蕉不同类群间极为保守。另外4条基因GSMUA_Achr10P27190(678bp,complete)、27210(144bp,fragment)、27220(717bp,complete)和27230(198bp,fragment)均与福州野生蕉MuFSD1B 基因(ORF为783bp)同源。染色体定位表明小果野生蕉的GSMUA_Achr1 0 P-27190、27210、27220和27230基因在10号染色体上成簇排列,且它们的cDNA 序列和gDNA序列间均具有高度的相似性,说明它们是由基因重复而来,不同长度的ORF可能是由各重复基因间的转录和翻译起始位点不同导致。曾在这4条基因cDNA 的特异5′端设计引物尝试扩增福州野生蕉中可能存在的不同翻译起始位点的FeSOD 基因,但均未得到任何条带,而在它们的保守区设计引物经RACE和RT-PCR验证仅获得1条序列(MuFSD1B),推测该基因成员的转录调控机制在香蕉不同类群中存在差异。同样的,本研究克隆得到的MuFSD1B 的gDNA 序列与小果野生蕉的4条gDNA序列也高度相似,因而可以确定福建野生蕉中仅存在此1成员。综上,福州野生蕉FeSOD 家族基因由MuFSD1A 和MuFSD1B2个成员组成。
3.2 低温胁迫下MuFSD1A 和MuFSD1B 基因的功能差异
温度低于13℃即可限制香蕉生长,而中国香蕉的主产区处于香蕉栽培的北缘区域,易受到冬季低温的影响而造成减产或品质下降[23]。已有研究表明香蕉的抗寒能力与其SOD 等抗氧化酶的活性变化密切相关[24-25]。而叶绿体中的光合作用是受低温影响最明显的过程之一[26]。叶绿体型FeSOD 主要参与清除光合传递链上产生的过多ROS,保证光合作用的顺利进行,进而提高植株的抗氧化能力[27]。福 州 野 生 蕉 的2 个FeSOD 成 员(MuFSD1A 和MuFSD1B)主要定位在叶绿体,但其氨基酸序列间的相似性低。系统进化分析表明MuFSD1A 和MuFSD1B 基因来自不同的基因祖先,可能存在功能分化。Kliebenstein 等[5]研究表明拟南芥中的AtFSD2和AtFSD3 基因对光胁迫、Ozone介导的氧化胁迫和UV-B胁迫表现出不同的应答模式,说明植物FeSOD 家族不同成员的具体功能存在差异。已有研究表明低温等逆境条件能改变相关蛋白的磷酸化水平进而调控其活性或亚细胞定位进一步参 与 逆 境 应 答[28-29]。福 州 野 生 蕉MuFSD1A 和MuFSD1B蛋白的理化性质、磷酸化位点的数量和类型、二级结构及三级结构上均存在明显差异,说明它们生物学活性存在一定差异,且可能通过不同的磷酸化或去磷酸化方式,改变酶活性和蛋白构像,参与由低温引起的各种氧化胁迫应答过程。李林玲等[30]研究发现银杏叶绿体型GbFeSOD 基因的表达受4 ℃低温诱导。过表达分析表明转拟南芥Fe-SOD 基因的玉米植株比非转基因表现出更耐寒的能 力[16]。福 州 野 生 蕉MuFSD1A 和MuFSD1B 基因在温度低于13℃时的转录表达变化明显,说明它们 参与低 温 应 答,这 与 李 琳 玲[30]和Tsang 等[13]的研究结果一致。但MuFSD1A 和MuFSD1B 基因的表达模式存在明显差异,其中MuFSD1A 基因显著被低温诱导表达,且随着胁迫温度的降低表达量不断增加,而MuFSD1B 基因的表达受到抑制,说明福州野生蕉FeSOD 不同成员在低温胁迫下可能行使不同的功能,其中MuFSD1A 基因可能在香蕉抗寒中起主要作用,需进一步通过过表达或RNA干扰进行验证。
3.3 内含子驻留可能是香蕉FeSOD 基因可变剪接的普遍方式
可变剪接是基因转录调控的一种重要机制,通过不同的剪接方式可从同一个mRNA 前体产生各种mRNA 转录本,进而翻译出具有不同生物学功能的蛋白亚型,在生物体的发育、组织分化及应对环境变化等过程中发挥重要作用[31-32]。基因的可变剪接现象普遍存在于各种真核生物中,但其普遍性和特性在不同物种间存在明显差异[33]。近年的研究表明,植物SOD 家族基因的部分成员也存在多种可变剪接现象[8]。其中FeSOD 基因的可变剪接转录本在 水 稻、龙 眼 和 荔 枝 中 均 有 报 道[8,34-35]。龙 眼 的DlFSD1b基因存在4个可变剪接转录本(DlFSD1bvariant1~4),分别由内含子驻留、外显子被选择性切除及前面这2个种剪接模式共同作用产生,而荔枝可变剪接转录本LcFe-SOD7b和水稻可变剪接转录本OsFe-SODb均是由内含子驻留产生。福州野生蕉的2 个 可 变 剪 接 转 录 本(MuFSD1B-variant1 和MuFSD1B-variant2)均是由内含子驻留产生,说明内含子驻留剪接模式是香蕉FeSOD 基因可变剪接转录本形成的普遍方式。
此外,Feng等[34]研究发现虽然水稻的可变剪接转录本OsFeSODb产生的是一个截短蛋白,但其氨基酸序列含有植物FeSOD 4个保守金属结合位点中的3个和区别于MnSOD 的所有特征氨基酸,且原核表达结果表明该截短蛋白具有SOD活性,能行使SOD功能。MuFSD1B-variant1和MuFSD1B-variant2转录本翻译的蛋白均由于内含子驻留而提前终止,产生一个228aa的截短蛋白,有意思的是,这2个转录本翻译的蛋白序列完全相同,且与水稻的OsFeSODb类似,同样拥有FeSOD 的3个保守金属结合位点和所有的FeSOD 特征氨基酸,因而,推测在同为单子叶植物的香蕉中,这2个可变剪接转录本翻译的蛋白很可能也具有SOD 活性,但需要进一步通过原核表达验证。
[1] KARUPPANAPANDIAN T,MOON J C,KIM C,et al.Reactive oxygen species in plants:their generation,signal transduction,and scavenging mechanisms[J].Australian Journal of Crop Science,2011,5(6):709-725.
[2] PILON M,RAVET K,TAPKEN W.The biogenesis and physiological function of chloroplast superoxide dismutases[J].Biochimica et Biophysica Acta,2011,1 807(8):989-998.
[3] ALSCHER R G,ERTURK N,HEATH L S.Role of superoxide dismutases(SODs)in controlling oxidative stress in plants[J].Journal of Experimental Botany,2002,53(372):1 331-1 341.
[4] KUO W Y,HUANG C H,LIU A C,et al.CHAPERONIN 20mediates iron superoxide dismutase(FeSOD)activity independent of its cochaperonin role in Arabidopsis chloroplasts[J].New Phytologist,2013,197(1):99-110.
[5] KILEBENSTEIN D J,MONDE R A,LAST R L.Superoxide dismutase in Arabidopsis:an eclectic enzyme family with disparate regulation and protein localization[J].Plant Physiology,1998,118(2):637-650.
[6] YANG Y Y(杨鸯鸯),LI Y(李 云),DING Y(丁 勇),et al.Cloning of Cu/Zn-superoxide dismutase of Brassica napus and its induced expression by Sclerotinia sclerotiorum[J].Acta Agronomica Sinica(作物学报),2009,35(1):71-78(in Chinese).
[7] LI J(李 娟),YANG L X(杨立新),ZHENG W Y(郑文寅),et al.Cloning and sequence analysis of stress tolerance relative gene FeSOD in wheat(Triticum aestivum)[J].Journal of Nuclear Agricultural Sciences(核农学报),2013,27(8):1 111-1 117(in Chinese).
[8] LIN Y L,LAI Z X.Superoxide dismutase multigene family in longan somatic embryosa comparison of CuZn-SODFe-SODand Mn-SOD gene structure,splicing,phylogeny,and expression[J].Molecular Breeding,2013,32(3):595-615.
[9] MOLINA-RUEDA J J,TSAI C J,KIRBY E G.The populus superoxide dismutase gene family and its responses to drought stress in transgenic poplar overexpressing apine cytosolic glutamine synthetase(GS1a)[J].PLOS One,2013,8(2):E56421.doi:56410.51371/journal.pone.0056421.
[10] KATYSHEV A I,ROGOZIN I B,KONSTANTINOV Y M.Identification of new superoxide dismutase transcripts in plants by EST analysis:Alternative polyadenylation and splicing events[C]//Proceedings of the Fifth International Conference on Bioinformatics of Genome Regulation and Structure,2006:61-64.
[11] BAGNOLI F,CAPUANA M,RACCHI M L.Developmental changes of catalase and superoxide dismutase isoenzymes in zygotic and somatic embryos of horse chestnut[J].Australian Journal of Plant Physiology,1998,25(8):909-913.
[12] 单宁伟.超氧化物歧化酶(SODs)与切花月季‘Samantha’失水胁迫耐性间的关联[D].北京:中国农业大学,2004.
[13] TSANG E W,BOWLER C,HEROUART D,et al.Differential regulation of superoxide dismutases in plants exposed to environmental stress[J].The Plant Cell,1991,3(8):783-792.
[14] LIU Y L(刘亚丽),YANG G Y(杨桂燕),ZHANG K M(张凯敏).The analysis of 2 Tamarix hispida FeSOD genes’clone and expression under salt stress[J].Journal of West China Forestry Science(西部林业科学),2014,43(4):31-36(in Chinese).
[15] KUREPA J,HEROUART D,VAN M M,et al.Differential expression of CuZn-and Fe-superoxide dismutase genes of tobacco during development,oxidative stress,and hormonal treatments[J].Plant &Cell Physiology,1997,38(4):463-470.
[16] VAN B F,SLOOTEN L,STASSART J M,et al.Overproduction of Arabidopsis thaliana FeSOD confers oxidative stress tolerance to transgenic maize[J].Plant &Cell Physiology,1999,40(5):515-523.
[17] LAI ZH X(赖钟雄),CHEN Y(陈 源),LIN Y L(林玉玲),et al.Discovery and taxonomy of wild banana(Musaspp.,‘AA’Group)in Fuzhou[J].Subtropical Agriculture Research(亚热带农业研究),2007,3(1):1-5(in Chinese).
[18] STEWART C N,VIA L E.A rapid CTAB DNA isolation technique useful for RAPD fingerprinting and other PCR applications[J].Bio.Techniques,1993,14(5):748-750.
[19] CHEN L,ZHONG H Y,KUANG J F,et al.Validation of reference genes for RT-qPCR studies of gene expression in banana fruit under different experimental conditions[J].Planta,2011,234(2):377-390.
[20] VANDESOMPELE J,DE PRETER K,PATTYN F,et al.Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J].Genome Biology,2002,3(7):research0034.0031-0034.0011.
[21] APOSTOLOVA E,RASHKOVA M,ANACHKOV N,et al.Molecular cloning and characterization of cDNAs of the superoxide dismutase gene family in the resurrection plant Haberlea rhodopensis[J].Plant Physiology and Biochemistry,2012,55:85-92.
[22] D′HONT A,DENOEUD F,AURY J M,et al.The banana(Musa acuminata)genome and the evolution of monocotyledonous plants[J].Nature,2012,488(7410):doi:10.1038/nature11241.
[23] LI N(李 娜),HUO ZH G(霍治国),HE N(贺 楠),et al.Climatic risk zoning for banana and litchi’s chilling injury in south China[J].Chinese Journal of Applied Ecology(应用生态学报),2010,21(5):1 244-1 251(in Chinese).
[24] ZHOU B Y(周碧燕),LIANG L F(梁立峰),HUANG H B(黄辉白),et al.Effect of low temperature and paclobutrazol on superoxide dismutase and abscisic acid of banana(Musaspp.)[J].Acta Horticulturae Sinica(园艺学报),1995,22(4):331-335(in Chinese).
[25] YANG Q S,WU J H,LI C Y,et al.Quantitative proteomic analysis reveals that antioxidation mechanisms contribute to cold tolerance in Plantain(Musa paradisiaca L.;ABB Group)seedlings[J].Molecular &Cellular Proteomics,2012,11(12):1 853-1 869.
[26] HE J(何 洁),LIU H X(刘鸿先),WANG Y R(王以柔),et al.Low temperature and photosynthesis of plants[J].Plant Physiology Communications(植物生理学通讯),1986,(2):1-6(in Chinese).
[27] ARISI A C,CORNIC G,JOUANIN L,et al.Overexpression of iron superoxide dismutase in transformed poplar modifies the regulation of photosynthesis at low CO2partial pressures or following exposure to the prooxidant herbicide methyl viologen[J].Plant Physiology,1998,117(2):565-574.
[28] GUO J W(郭军伟),WEI H M(魏慧敏),WU SH F(吴守锋),et al.Effects of low temperature on the distribution of excitation energy in photosystem and the phosphorylation of thylakoid membrane proteins in rice[J].Acta Biophysica Sinica(生物物理学报),2006,22(3):197-202(in Chinese).
[29] WANG W(王 尉),GUO N(郭 宁),XU M ZH(徐明照),et al.Expression in vitro of OsPIN1agene and analysis of phosphorylation sites[J].Chinese Journal of Tropical Crops(热带作物学报),2014,35(8):1 528-1 532(in Chinese).
[30] LI L L(李琳玲),CHENG H(程 华),XU F(许 峰),et al.Molecular cloning,characterization and expression of iron superoxide dis-mutase gene fromGinkgo bilobaJ.Journal of Fruit Science果树学报2009266840-846in Chinese.
[31] GUO X Q(郭小勤),LI D B(李德葆).Pre-mRNA alternative splicing in plants[J].Journal of Agricultural Biotechnology(农业生物技术学报),2006,14(5):809-815(in Chinese).
[32] PAN J W(潘教文),LI D Q(李德全).Cellular localization of components of mitogen-activated protein kinase(MAPK)cascades and alternative splicing[J].Chinese Journal of Biochemistry and Molecular Biology(中国生物化学与分子生物学报),2010,26(5):393-400(in Chinese).
[33] KEREN H,LEV-MAOR G,AST G.Alternative splicing and evolution:diversification,exon definition and function[J].Nature Reviews Genetics,2010,11(5):345-355.
[34] FENG W,HONGBIN W,BING L,et al.Cloning and characterization of a novel splicing isoform of the iron-superoxide dismutase gene in rice(Oryza sativa L.)[J].Plant Cell Reports,2006,24(12):734-742.
[35] LIAN C L(练从龙),LAI ZH X(赖钟雄),LU B G(卢秉国),et al.Cloning and bioinformatics analysis of Fe-SODgene from embryogenic callus of an ancient litchi tree[J].Chinese Journal of Tropical Crops(热带作物学报),2014,35(1):74-81(in Chinese).

图版Ⅰ 35S∷MuFSD1A-GFP蛋白在烟草中的亚细胞定位A、E和I为488nm 激发光下GFP蛋白的绿色荧光信号;B、F和J为561nm 激发光下叶绿体的自发红色荧光信号;C、G 和K 为烟草细胞的明场图;D、H 和L为叠加图;A~D为用水处理的阴性对照;E~H 为用pCAMBIA1302处理的阳性对照;I~L 为35S∷MuFSD1A-GFP处理。箭头1.叶绿体;箭头2.细胞质;箭头3.细胞膜;箭头4.细胞核。PlateⅠ Subcellular localization of 35S∷MuFSD1A-GFP protein in tobacco cells Fig.A,E and I images present the green fluorescence of GFP under excitation wavelength of 488nm;Fig.B,F and J images present the auto-fluorescence of chloroplast under excitation wavelength of 561nm;Fig.C,G and K images present outlook of tobacco cells;Fig.D,H and L images are the merged images.Fig.A~D are negative controls treated with water;Fig.E~H are positive controls treated with pCAMBIA1302;Fig.I~L are treated with 35S∷MuFSD1A-GFP.Arrow 1is chloroplast;Arrow 2is cytoplasm;Arrow 3is cellular membrane;Arrow 4is nucleus.
