粘虫转录组中SSR位点的信息分析
2015-06-18胡艳华李敏张虎芳李生才王青赵惠玲
胡艳华,李敏,,张虎芳*,李生才,王青,赵惠玲
(1.山西农业大学 农学院,山西 太谷 030801;2.太原师范学院 生物系,山西 太原 030031)
粘虫转录组中SSR位点的信息分析
胡艳华1,李敏1,2,张虎芳1*,李生才1,王青2,赵惠玲2
(1.山西农业大学 农学院,山西 太谷 030801;2.太原师范学院 生物系,山西 太原 030031)
粘虫Mythimnaseparata(Walker)是一种迁飞性害虫,严重危害玉米、水稻、小麦等粮食作物。SSR是指以1~6个核苷酸为基本重复单位的串联重复DNA序列。SSR位点的信息分析为粘虫扩散、迁飞和交配等行为分子机制的研究以及粘虫的综合防治奠定理论基础。本研究基于高通量测序获得的粘虫转录组数据,利用软件msatcommander发掘粘虫SSR位点。结果从20 776条转录组Unigenes中共搜索出400个SSR,分布于372条Unigenes中。在粘虫转录组SSR中,三核苷酸重复的数量最为丰富,有271个;其次是二核苷酸和单核苷酸重复,分别是70个和49个;四至六核苷酸重复的数量都很少,共10个。粘虫转录组SSR共包含24种重复基元,其中CCG/CGG是优势重复基元类型,有69个;其次是AAG/CTT,有57个。CG/CG有18个,在二核苷酸重复基元中所占的比例达到25.7%。此研究发掘到的SSR位点将为粘虫遗传图谱的构建、遗传多样性分析、亲缘关系分析等提供丰富的分子标记。
粘虫;转录组;SSR;重复类型;重复基元
简单重复序列(Simple Sequence Repeat,SSR)又称微卫星(Microsatellites DNA),是指以1~6个核苷酸为基本重复单位的串联重复序列,如 An、(AT)n、(TGG)n和(ATTC)n等重复[1]。SSR广泛存在于真核生物和部分原核生物核基因组中,是一种有效的基于DNA长度多态性的分子标记,具有高度多态性、杂合性高、分布广泛、变异丰富、呈共显性遗传和检测快速方便诸多优点,已被广泛应用于遗传图谱绘制、种群遗传结构和遗传多样性分析、基因定位与克隆、分子标记辅助育种、亲缘关系分析等研究领域[2,3]。
粘虫Mythimnaseparata(Walker)属于鳞翅目夜蛾科,具有迁飞性和暴发性,是粮食作物的主要害虫,严重威胁我国农业生产和粮食安全,在亚洲和澳洲其它国家也常发生危害[4]。2012年三代粘虫在我国暴发成灾,发生面积和危害程度为近十年之最[5]。粘虫SSR标记位点的开发有利于构建粘虫的遗传图谱,探究其种群遗传结构和遗传多样性,从分子水平探究粘虫扩散、迁飞和交配等行为,为制定科学合理的检测及防控措施奠定理论基础。
传统的从基因组DNA序列中开发SSR的方法存在步骤繁琐、成本高、效率低以及耗时长等问题。随着新一代高通量测序技术的发展,从转录组数据库中搜索并鉴定SSR位点为SSR位点的开发提供了一种经济、高效的途径和丰富的遗传学资源。同时,由于基于转录组开发的SSR位于蛋白质的编码区域,这些编码区中包含着已知或者未知的基因功能,因而可用于鉴定功能基因,从而可进一步进行表型研究。
至今,基于转录组对EST-SSR位点的开发已有相关报道。Bai et al[7]通过对温带臭虫(Cimex lectulariusLinnaeus)的转录组数据库筛选得到370个SSR,其中三核苷酸重复的数量最丰富。Wei et al[8]基于嗜虫书虱(Liposcelisentomophila(Enderlein))转录组筛选得到1 110个EST-SSR,并成功设计出231对引物。Xu et al[9]从白背飞虱(Sogatellafurcifera(Horváth))转录组数据中搜索得到7 291个SSR位点,并且预测有7.26%的蛋白质编码序列位于这些SSR中。Duan et al[10]从绿豆象(CallosobruchuschinensisLinnaeus)转录组数据中搜索并鉴定出6 303个SSR遗传标记,并从中筛选出20对高度多态性的SSR遗传标记已经成功用于分析绿豆象种群的遗传多样性。Sun et al[11]从 褐 飞 虱 (Nilaparvatalugens(Stål))转录组中发掘得到 465个 SSR,将含有SSR的序列与NCBI中nonredundant(nr)蛋白质数据库比对发现37.2%的序列是已知功能的基因,62.4%的基因功能未知。对鳞翅目昆虫的研究报道中,Xie et al[12]从小菜蛾(PlutellaXylostella(Linnaeus))转录组中发掘出2 351个SSR位点,三核苷酸重复的数量最丰富。Li et al[13]基于二点委夜蛾(Athetislepigone(Möschler))转录组发掘到2 819个SSR,分布于2 411条序列中。Zhu et al[14]从细梢小卷蛾(RhyacionialeptotubulaLiu et Bai)转录组中发掘到1 450个SSR位点,其中(ATC)n是出现最多的重复基元。Pascual et al[15]从甜菜夜蛾(Spodopteraexigua(Hübner))转录组中发掘到351个SSR位点,25%的SSR位于预测的开放阅读框(ORFs)中。
本研究通过新一代高通量测序技术对粘虫进行转录组测序,利用从头组装软件获得粘虫的Unigenes,通过搜索软件对其EST-SSR位点进行大批量搜索,并对其进行分析,以便了解粘虫转录组EST-SSR位点的组成和分布特征,为后续粘虫的SSR引物的设计、遗传多样性分析以及功能基因的研究提供有效的分子标记。
1 材料与方法
1.1 试虫来源和总RNA提取
试虫引自西北农林科技大学无公害农药研究服务中心饲养多年的粘虫,饲养期间不接触任何药剂。取粘虫卵、1~6龄幼虫、蛹和成虫,采用trizol法抽提粘虫总RNA。利用Nanodrop 2000分光光度计检测总RNA质量,当总RNA的浓度大于等于200mg·L-1、OD260/280值在1.8~2.2之间时,表明所提取的RNA符合转录组测序要求。
1.2 转录组测序及序列组装拼接
总RNA样品达到转录组测序要求后采用华大基因Illumina HiSeq2000测序仪进行高通量深度测序。对测序得到的原始序列进行去接头、去低质量读段和去重复等处理,之后使用组装软件Trinity[16]对这些短序列进行从头de nove组装,最终得到尽可能长的非冗余序列Unigenes。
1.3 粘虫全转录组EST-SSR的发掘
利用msatcommander软件[17]对粘虫全转录组Unigenes序列进行SSR搜索,搜索标准如表1,符合上述搜索标准的在本次研究中被定义为SSR。
2 结果与分析
2.1 总RNA质量检测结果
经Nanodrop 2000分光光度计检测粘虫总RNA质量后,检测结果为:总RNA浓度为831.5 mg·L-1,OD260/280值为2.01,表明粘虫总RNA样品质量很高,能够满足转录组测序的要求。
2.2 原始序列组装结果
粘虫转录组原始序列经组装拼接以后获得了20 776条Unigenes。这些Unigenes的总长度为14 002 443bp,平均长度为674bp。Unigenes序列长度均大于等于200bp,最长的Unigenes 6 868bp。这些Unigenes序列用于后续的SSR搜索。
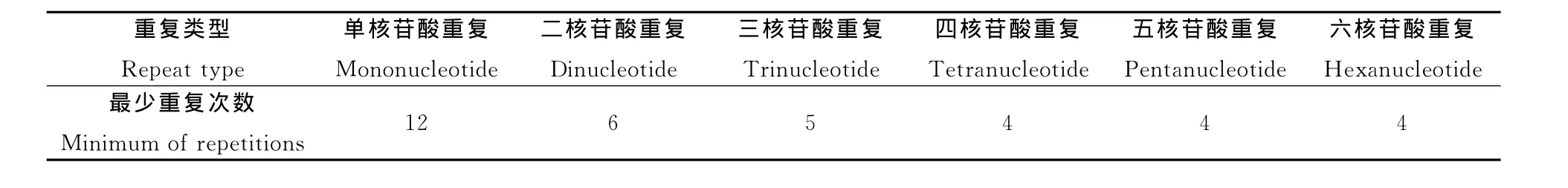
表1 发掘粘虫转录组SSR的搜索标准Table 1 The search criteria of SSR fromMythimnaseparatatranscriptome
2.3 粘虫EST-SSR位点数量分布特征
粘虫转录组Unigenes序列经msatcommander软件搜索,得到400个SSR,占总Unigenes数量的1.925%,即出现频率。这400条SSR位点分布于372条Unigenes中,发生频率为1.790%,发生频率指含有SSR的Unigenes数量与总Unigenes数量的比值。其中,含单个SSR位点的Unigenes有365条,含2个SSR位点的Unigenes有10条,含3个SSR位点的Unigenes有5条。从分布情况看,粘虫转录组Unigenes中平均每35 006bp(35kb)就出现1个SSR,即平均距离,但不同重复类型间差异较大。
从粘虫转录组中共搜索到六种核苷酸重复类型,出现数量最多的是三核苷酸重复类型,占总SSR数量的67.75%,其次是二核苷酸重复,占总SSR数量的17.50%,数量最少的是五核苷酸重复,只占总数的0.25%(表2)
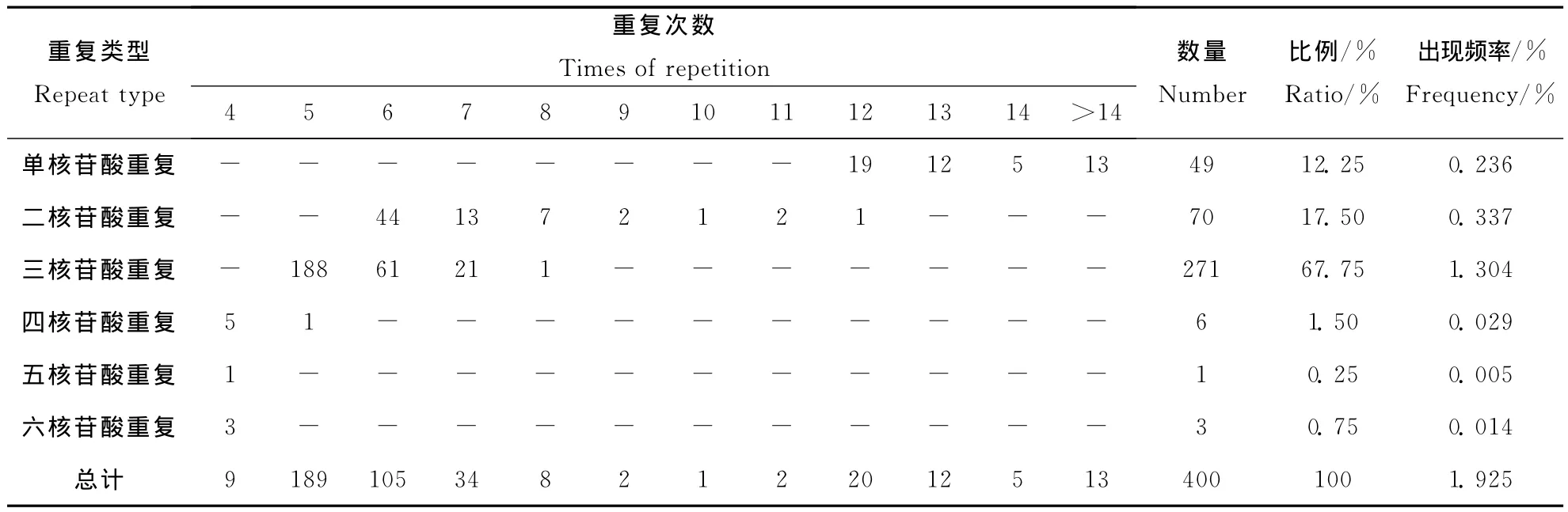
表2 粘虫EST-SSR位点的数量与分布Table 2 The amount and distribution of the EST-SSR inMythimnaseparata
另外,不同核苷酸重复的重复次数有所差异,单核苷酸重复基元的重复次数主要集中在12~13次,二、三核苷酸重复主要集中在5~7次,四、五、六核苷酸重复基元的重复次数主要以4次为主(表2)。
2.4 粘虫EST-SSR重复基元的分布特征
在400个粘虫EST-SSR位点中,共有24种重复基元出现,其中单、二、三、四、五及六核苷酸重复基元的种类分别是2、4、10、5、1和2(表3)。其中,出现频率最多的重复基元是CCG/CGG,其次是AAG/CTT(图1)。单核苷酸重复基元中,A/T是优势基元,占单核苷酸重复的87.75%;二核苷酸重复基元中,出现的次数最多的是AC/GT,占二核苷 酸 重 复 的 55.71%,其 次 是 CG/CG,占25.71%。三核苷酸重复基元中,最高为CCG/CGG,占三核苷酸重复的25.46%;四、五和六核苷酸重复基元类型数量最少,总计占总SSR数量的2.5%(表3)。
3 讨论
本研究通过生物信息学方法,从粘虫全转录组数据库20 776条Unigenes中共发掘出400个EST-SSR,出现频率为1.93%,这比从黑翅土白蚁(Odontotermesformosanus(Shiraki))(9.98%)[18]、扶桑绵粉蚧(PhenacoccussolenopsisTinsley)(6.33%)[19]、烟 粉 虱 (Bemisiatabaci(Gennadius))(5.07%)[20]和嗜虫书虱(2.03%)[8]转录组中发掘的SSR出现频率要低,但比黄粉虫(TenebriomolitorLinnaeus)(1.67%)[21]、云南切梢小蠹(TomicusyunnanensisKirkendall and Faccoli)(1.29%)[22]和 葱 地 种 蝇 (DeliaantiguaMeigen)(1.12%)[23]的SSR 出现频率要高。鳞翅目昆虫中,细梢小卷蛾SSR的出现频率是3.09%,高于粘虫SSR的出现频率[14]。分析原因,除了由于SSR搜索方法或标准有所差异外,最根本的原
因可能是物种本身的差异。
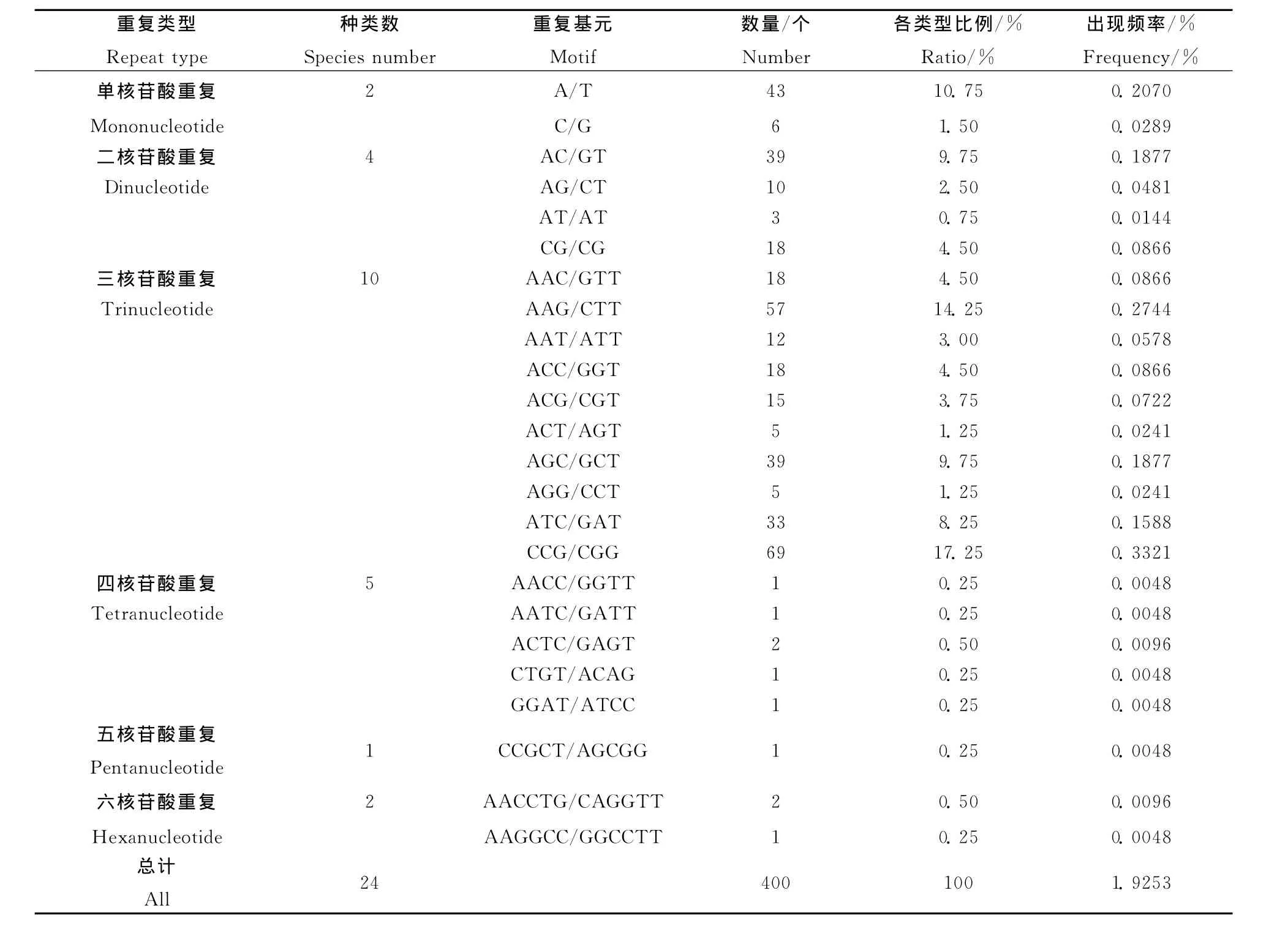
表3 粘虫EST-SSR重复基元的分布特征Table 3 Distribution Characteristics of EST-SSR's motif types inMythimnaseparata

图1 粘虫转录组SSR中不同重复类型及不同重复基元的分布Fig.1 Distribution of SSR among different nucleotide types found in the transcriptome of Mythimnaseparata
粘虫EST-SSR的种类较为丰富,一至六核苷酸重复类型都有出现,其中数量最多的重复类型是三核苷酸重复序列。这与二点委夜蛾、灰飞虱(Laodelphaxstriatellus(Fallén))和大豆蚜(A-phisglycinesMatsamura)等昆虫的EST-SSR以三核苷 酸 重 复 为 主 类 似[13,24,25],而 与 扶 桑 绵 粉蚧[19]、黄 粉 虫[21]、黑 翅 土 白 蚁[20]、蠋 蝽 (Arma chinensisFallou)[26]的 EST-SSR 分布不同。其中扶桑绵粉蚧和黄粉虫EST-SSR中都是单核苷酸重复序列占主导地位,黑翅土白蚁和蠋蝽ESTSSR中占主导地位的是二核苷酸重复。由此可见不同物种间EST-SSR类型的分布存在着差异。大部分昆虫中三核苷酸重复序列数量最丰富,可能是为了防止蛋白质编码时发生移码突变[11]。
粘虫EST-SSR主要重复基元以1~3核苷酸为主,占总EST-SSR的99%以上。单核苷酸重复中A/T是主要的重复基元,这与已研究的90%以上昆虫的单核苷酸重复以A/T为主类似。在二核苷酸重复类型中,AC/GT重复基元出现的频率最高,与鳞翅目昆虫细梢小卷蛾[14]AC/GT为优势重复基元相似。但与褐飞虱[12]和黄粉虫[21]以 AG/CT为主导重复基元以及与桔小实蝇[9]、扶桑绵粉蚧[19]、云南切梢小蠹[22]以 AT/AT 为主导重复基元不同。三核苷酸重复中,CCG/CGG重复基元占绝对优势,鳞翅目昆虫二点委夜蛾中(CGC)n和(GCC)n的数量最多[24];而在扶桑绵粉蚧[19]、黄粉虫[21]和 德 国 小 蠊 (Blattellagermanica(Linnaeus))[27]中 AAT/ATT 是优势基元;云南切梢小蠹[22]中AAT/ATT和ATC/GAT含量最多;在桔小实蝇[8]中,AGC/GCT 最常见;在灰飞虱[24]中AAC/GTT数量最丰富;在褐飞虱[11]中 AAG/CTT占主导地位,而CCG/CGG的含量却是最低的,仅占褐飞虱三核苷酸重复基元的1.83%。
值得注意的是,粘虫二核苷酸重复基元中GC/GC所占的比例达到25.7%,仅仅低于重复基元AC/GT。该结果与鳞翅目昆虫二点委夜蛾[13]和细梢小卷蛾[14]中GC/GC重复基元很常见相类似。以往对昆虫EST-SSR的研究中,GC/GC的含量都极低甚至没有。例如在灰飞虱[24]和褐飞虱[12]EST-SSR搜索中均没有发现GC重复基元;在桔小实蝇转录组SSR中含有3个GC/GC重复基元[8],云南切梢小蠹转录组中仅有2个[22]。粘虫、二点委夜蛾和细梢小卷蛾同属鳞翅目,GC/GC重复基元是否与鳞翅目的某些特殊功能有关系,今后需进一步研究。
4 结论
本研究基于粘虫的转录组数据发掘获得大批量SSR,并对SSR数量分布相关特性及可用性进行了阐述和评估,表明通过转录组数据发掘SSR的方法是高效可行的。研究结果为粘虫遗传图谱的构建、种群多样性分析以及特定基因的定位奠定了基础,这对于探究粘虫扩散、迁飞和交配等行为的分子机制以及粘虫的综合防治都具有重要意义。同时,也为鳞翅目其它种类基于转录组数据发掘SSR提供参考。
[1]Powell W,Machray GC,Provan J.Polymorphism revealed by simple sequence repeats[J].Trends in Plant Science,1996,1(7):215-222.
[2]Varshney RK,Graner A,Sorrells ME.Genic microsatellite markers in plants:features and applications[J].Trends in Biotechnology,2005,23(1):48-55.
[3]Li YC,Korol AB,Fahima T,et al.Microsatellites:genomic distribution,putative functions and mutational mechanisms:a review[J].Molecular Ecology,2002,11(12):2453-2465.
[4]江幸福,张蕾,程云霞,等.我国粘虫发生危害新特点及趋势分析[J].应用昆虫学报,2014,51(6):1444-1449.
[5]姜玉英,李春广,曾娟,等.我国粘虫发生概况:60年回顾[J].应用昆虫学报,2014,51(4):890-898.
[6]Zalapa JE,Cuevas H,Zhu H,et al.Using next-generation sequencing approaches to isolate simple sequence repeat(SSR)loci in the plant sciences[J].American Journal of Botany,2012,99(2):193-208.
[7]Bai XD,Mamidala P,Rajarapu SP,et al.Transcriptomics of the Bed Bug(Cimexlectularius)[J].PLoS One,2011,6(1):e16336.
[8]Wei DD,Chen EH,Ding TB,et al.De Novo Assembly,Gene Annotation,and Marker Discovery in Stored-Product PestLiposcelisentomophila(Enderlein)Using Transcriptome Sequences[J].PLoS One,2013,8(11):e80046.
[9]Xu Y,Zhou W,Zhou Y,et al.Transcriptome and Comparative Gene Expression Analysis ofSogatellafurcifera(Horváth)in Response to Southern Rice Black-Streaked Dwarf Virus[J].PLoS One,2012,7(4):e36238.
[10]Duan CX,Li DD,Sun SL,et al.Rapid Development of Microsatellite Markers forCallosobruchuschinensisUsing Illumina Paired-End Sequencing[J].PLoS One,2014,9(5):e95458.
[11]Sun JT,Zhang YK,Ge C,et al.Mining and characterization of sequence tagged microsatellites from the brown planthopperNilaparvata lugens[J].Journal of Insect Science,2011,11:134.
[12]Xie W,Lei YY,Fu W.Tissue-Specific Transcriptome Profiling ofPlutellaXylostellaThird Instar Larval Midgut[J].International Journal of Biological Science,2012,8(8):1142-1155.
[13]Li LT,Zhu YB,Ma JF,et al.An Analysis of theAthetislepigoneTranscriptome from Four Developmental Stages[J].PLoS One,2013,8(9):e73911.
[14]Zhu JY,Li YH,Yang S,et al.De novo Assembly and Characterization of the Global Transcriptome forRhyacionialeptotubulaUsing Illumina Paired-End Sequencing[J].PLoS One,2013,8(11):e81096.
[15]Pascual L,Jakubowska AK,Blanca JM,et al.The transcriptome ofSpodopteraexigualarvae exposed to different types of microbes[J].Insect Biochemistry and Molecular Biology,2012,42:557-570.
[16]Grabherr MG,Haas BJ,Yassour M,et al.Trinity:reconstructing a full-length transcriptome without a genome from RNA-Seq data[J].Nature Biotechnology,2013,29(7):644-652.
[17]Faircloth BC.Msatcommander:detection of microsatellite repeat arrays and automated,locus-specific primer design[J].Molecular Ecology Resources,2008,8(1):92-94.
[18]Huang Q,Sun P,Zhou X,et al.Characterization of Head Transcriptome and Analysis of Gene Expression Involved in Caste Differentiation and Aggression inOdontotermesformosanus(Shiraki)[J].PLoS One,2012,7(11):e50383.
[19]罗梅,张鹤,宾淑英,等.基于转录组数据高通量发掘扶桑绵粉蚧微卫星引物[J].昆虫学报,2014,57(4):395-400.
[20]Xie W,Meng QS,Wu QJ,et al.Pyrosequencing theBemisiatabaciTranscriptome Reveals a Highly Diverse Bacterial Community and a Robust System for Insecticide Resistance[J].PLoS One,2012,7(4):e35181.
[21]朱家颖,吴国星,杨斌.基于转录组数据高通量发掘黄粉甲微卫星引物(英文)[J].昆虫学报,2013,56(7):724-728.
[22]袁远,张丽芳,吴国星,等.云南切梢小蠹微卫星的高通量发掘[J].环境昆虫学报,2014,36(2):166-170.
[23]Zhang YJ,Hao YJ,Si FL,et al.The de novo Transcriptome and Its Analysis in the Worldwide Vegetable Pest,Deliaantiqua(Diptera:Anthomyiidae)[J].G3-Genes Genomes Genetics,2014,4(5):851-859.
[24]Zhang F,Guo H,Zheng H,et al.Massively parallel pyrosequencing-based transcriptome analyses of small brown planthopper(Laodelphaxstriatellus),a vector insect transmitting rice stripe virus(RSV)[J].BMC Genomics,2010,11:303.
[25]Bai XD,Zhang W,Orantes L,et al.Combining Next-Generation Sequencing Strategies for Rapid Molecular Resource Development from an Invasive Aphid Species,Aphisglycines[J].PLoS One,2010,5(6):e11370.
[26]Zou D,Coudron TA,Liu C,et al.Nutrigenomics inArmachinensis:Transcriptome Analysis ofArmachinensisFed on Artificial Diet and Chinese Oak Silk MothAntheraeapernyiPupae[J].PLoS One,2013,8(4):e60881.
[27]Zhou X,Qian K,Tong Y,et al.De Novo Transcriptome of the Hemimetabolous German Cockroach (Blattellagermanica)[J].PLoS One,2014,9(9):e106932.
The Information Analysis of SSR Loci in theMythimnaseparate(Walker)Transcriptome
Hu Yanhua1,Li Min1,2,Zhang Hufang1*,Li Shengcai1,Wang Qing2,Zhao Huiling2
(1.CollegeofAgriculture,ShanxiAgriculturalUniversity,TaiguShanxi030801,China;2.DepartmentofBiology,TaiyuanNormalUniversity,TaiyuanShanxi030031,China)
Mythimnaseparata(Walker)is a kind of migratory pests and causes serious damage to the crops such as corn,wheat,rice and so on.Simple sequence repeat(SSR)is tandemly repeated motif of 1~6nucleotide.The information analysis of SSR loci in theMythimnaseparate(Walker)establishes a theoretical basis for the research of molecular mechanisms such as its diffusion,journey and mating behavior,as well as its integrated control.Based on the constructed transcriptome database inMythimnaseparata,the SSR loci were explored by the software msatcommander.In total,400SSR loci were explored from 20 776transcriptome unigenes,and they were distributed in 372unigenes.Among these SSR loci,trinucleotide were the most abundant repeats,of which were 271,followed by dinucleotide and mononucleotide repeats,of which were 70and 49,respectively.Tetra-、penta-and hexanucleotide were 10in all.There were 24 kinds of repeated motif types inMythimnaseparatatranscriptome SSR.CCG/CGG was the most advantages repeat motif types(69),then AAG/CTT (57).There were 18CG/CG repeated motif types which accounted for 25.3%of the dinucleotide repeat motif types.The SSR loci which were discovered in this study would benefit a lot for the construction of gene mapping,the research of genetic diversity and the parentage analysis.
Mythimnaseparata;Transcriptome;SSR;Repeat type;Motif type
S433.4
A
1671-8151(2015)05-0484-06
10.13842/j.cnki.issn1671-8151.2015.05.007
2015-04-12
2015-05-21
胡艳华(1990-),女(汉),山西洪洞人,硕士研究生,研究方向:农业昆虫与害虫防治
*通讯作者:张虎芳,教授,硕士生导师。Tel:0354- 6288225;E-mail:zh_hufang@sohu.com
国家自然科学基金(31440078);中国博士后研究经费(134845);山西省高等学校大学生创新创业训练项目(2014376);太原师范学院大学生创新创业训练项目(20140413)
(编辑:武英耀)
