流感病毒RNA聚合酶PA亚基:潜在抗流感药物靶点
2015-06-09连雯雯刘艾林杜冠华
连雯雯,刘艾林,2,3,杜冠华,2,3
(1.中国医学科学院北京协和医学院药物研究所,2.药物靶点研究与新药筛选北京市重点实验室;3.天然药物活性物质与功能国家重点实验室,北京 100050)
◇讲座与综述◇
流感病毒RNA聚合酶PA亚基:潜在抗流感药物靶点
连雯雯1,刘艾林1,2,3,杜冠华1,2,3
(1.中国医学科学院北京协和医学院药物研究所,2.药物靶点研究与新药筛选北京市重点实验室;3.天然药物活性物质与功能国家重点实验室,北京 100050)
流感是世界范围内的严重传染性疾病,目前抗流感药物面临的主要问题是病毒耐药性和对高致病性流感病毒的效价低。流感病毒RNA聚合酶是病毒在宿主细胞内完成复制和转录过程的关键性酶,其中PA亚基通过内切酶活性为流感病毒的转录过程提供引物,成为潜在抗流感药物靶点。该文对PA亚基的结构、功能及内切酶抑制剂类抗流感药物研究进展进行概述,为PA亚基的深入研究及针对此靶点的抗流感药物发现提供信息指导。
流感病毒;PA亚基氨基端;PA亚基羧基端;内切酶;金属离子结合位点;高通量筛选模型
流行性感冒(简称流感)是全球性严重传染性疾病,每年大约有20%的儿童和5%的成年人患有伴随临床症状的甲型或乙型流感。目前用于预防和治疗流感的化学药物仅有两种类型:M2离子通道抑制剂(amantadine和rimantadine)和NA抑制剂(zanamivir和oseltamivir)。这两类药物面临的最大问题是病毒耐药性问题,特别是M2离子通道抑制剂由于出现广泛耐药性而不被推荐用于临床[1-3],其次这两类药物对高致病性流感病毒如H5N1的效果不理想。近几年,高致病性禽流感病毒H5N1频频出现,发现高效、广谱的抗流感药物成为新药研发紧迫而重要的任务。
流感病毒进入宿主细胞后,在细胞核内完成复制(vRNA-cRNA-vRNA)和转录(vRNA-mRNA)过程,这两个过程都是由流感病毒编码的RNA聚合酶催化。RNA聚合酶由PB1、PB2和PA亚基组成,PB1亚基主要参与病毒基因组的复制过程;PB2亚基主要负责与宿主pro-mRNA帽状结构结合,协助完成内切酶的剪切过程;而关于RNA聚合酶内切酶活性存在PB1还是PB2亚基的问题一直存在争议,直到2009年Yuan和Dias两个课题组同时揭示PA亚基氨基端的晶体结构,并确认RNA聚合酶的内切酶活性存在于PA亚基的氨基端,结束了关于内切酶活性存在哪个亚基的争论。由于病毒蛋白质的合成依赖宿主细胞的翻译体系,流感病毒mRNA需要同时具备可供宿主细胞翻译体系识别的5′帽状结构和3′-poly(A)尾,其中5′帽状结构是通过RNA聚合酶PA亚基的内切酶活性从宿主细胞pro-mRNA的5′端剪切得到的10~13个核苷酸(即cap-snatching)[4-5],是流感病毒转录起始所必须的。cap-snatching是流感病毒生命周期中的一个关键事件,宿主细胞中不存在类似的事件和相应的酶,因此针对cap-snatching的内切酶抑制剂可以选择性阻断流感病毒的转录过程,对宿主细胞不造成影响,同时PA亚基氨基端和羧基端晶体结构的揭示,以及氨基酸序列的高度保守性,使PA亚基成为潜在的抗流感药物靶点。本文对PA亚基的结构、功能及内切酶抑制剂类抗流感药物研究进展进行概述,为PA亚基的深入研究及针对此靶点的抗流感药物发现提供信息指导。
1 RNA聚合酶PA亚基的结构
流感病毒RNA聚合酶以PB1亚基为中心,PA亚基的C端和PB2亚基的N端分别与PB1亚基的N端和C端结合,构成RNA聚合酶,后者与核蛋白、vRNA一起构成核糖核蛋白体RNP[5-7]。流感病毒的基因组是由8个单负链RNA片段组成,PA亚基由第3个RNA片段编码,含有716个氨基酸,分子质量约为83.4 ku,在胰蛋白酶的作用下,可以被水解为2个片段:N端约为25 ku的结构域(氨基酸残基1~212或213,简称PAN)和C端约为55 ku的结构域(氨基酸残基212或213~716,简称PAC)[6]。
1.1 PA亚基氨基端的结构PAN含有1个由α螺旋和β片层构成的结构域,即7个α螺旋环绕着5个反向β片层构成的曲面,内部含有带负电的活性口袋,可能参与二价阳离子的结合。PAN属于PD-(D/E)XK超家族,内切酶活性位点可能位于P107D108X10E119K134[8-9]。由于结晶条件不同,Yuan等[8]的结构中,PAN与1个镁离子结合,结合位点由E80、D108及与H41、E119、L106、P107结合的3个水分子组成,而Dias等的结构中,PAN与2个锰离子结合,结合位点包括site1:E119,D108及两个水分子和site2:H41、D108、E119及I120[9]。PAN的三维结构及可能的金属结合位点的详细描述如Fig 1所示。
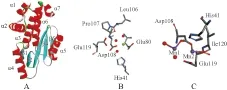
Fig 1 The three dimentional structure and mental binding sites of PAN
1.2 PA亚基羧基端的结构PAC通常和PB1亚基N端(PB1N)结合,两者共结晶的结构显示,PAC由13个α螺旋和9个β片层组成,包括两个结构域(domain 1和domain 2),domain 1由5个α螺旋(α1、α2、α3、α6、α7)环绕7个β片层(β1~β7)组成,domain 2由片层β8、β9及螺旋α4、α5、α8-α12组成,形状似一个开口的钳子,α8和α10组成开口的上沿,α11和α13组成开口的下沿。PB1N的15个氨基酸以N端朝里,C端朝外的方式与domain 2以氢键、疏水作用和范德华力结合。domain 2的钳状开口一直延伸,在domain 1上形成一道槽,该槽附近以碱性氨基酸为主,带大量的负电荷,可能与vRNA或cRNA的结合有关[10-11]。PAC的三维结构的详细描述见Fig 2。
2 PAN的内切酶活性
根据文献报道,PA亚基具有多种功能,如内切酶活性、蛋白水解酶活性、介导PB1亚基和RNA的相互作用等,下面主要对研究较为深入的内切酶活性做简要的概括。
2009年,Yuan和Dias首次确定RNA聚合酶的内切酶活性是存在PAN,并揭示PAN晶体结构。对酶活性中心进行分析,Dias等[9]通过同源序列比对的方式,获得的PAN活性中心的关键氨基酸残基有H41、D108、E119、K134;Yuan等[8]对PAN进行点突变,包括E80A、D108A、E119A、K134A,研究发现,带有这些点突变的RNA聚合酶能选择性抑制流感病毒mRNA的合成,而对cRNA和vRNA的合成没有影响,推测E80、D108、E119、K134可能是内切酶活性中心的关键氨基酸残基。
内切酶活性的发挥需要二价阳离子的辅助,目前研究者广泛接受两个锰离子结合机制。Yuan等[8]在0.1 mol·L-1MgCl2条件下结晶得到的PAN与1个镁离子结合,镁离子的结合位点是site2;Dias等[9]在2.5×10-3mol·L-1MnCl2和5×10-3mol·L-1MgCl2条件下得到的PAN与2个锰离子结合。Zhao等[12]获得PAN与单磷酸核苷酸的晶体结构中,镁离子位于site 2;Kowalinski等[13]得到PAN与单磷酸核苷酸的晶体结构中,活性中心存在2个锰离子,这种差异可能是由结晶环境不同造成。Crépin等[14]通过等温滴定法发现,PAN存在两个离子结合位点,这些位点对锰离子的亲和力是镁离子的500~600倍,同时PAN与锰离子的结合是放热过程,而与镁离子的结合是吸热过程,推测PAN倾向与锰离子结合。Noble等[15]报道,在不同的二价阳离子存在下,PA具有不同的构象,同时对底物的剪切模式也不同。此外,DouBis等[16]、Doan等[17]发现,在相同离子浓度下,RNP和PAN与锰离子结合后,内切酶活性和酶稳定性都明显高于与镁离子结合。Xiao等[18]应用分子动力学模拟技术发现,在没有底物的情况下,镁离子倾向与PAN的site1结合,而在底物RNA存在情况下,PAN倾向与2个镁离子结合,催化剪切反应[18]。因此,PAN结构中结合的金属离子的种类和数目与环境中的金属离子有关,虽然有实验表明,PAN与锰离子结合的亲和力较高,且结合后具有较高内切酶活性和稳定性,但是在镁离子浓度(毫摩尔级)远高于锰离子(微摩尔级)的生理环境中,作者认为镁离子可能是PAN的天然辅助因子,但是最终结论还有待于进一步研究。
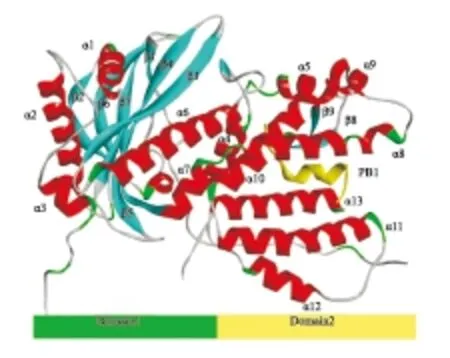
Fig 2 The three dimentional structure of PAC-PB1N complex
The three dimensional structure of PAC-PB1Ncomplex is in ribbon representation, and the yellow helix is PB1N, the rest PAC. The structure of PACis colored by different secondary structures: α-helices are red, β-sheets are cyan, and the linkers are slivery. The individual secondary structures are labelled. There are two domains in the structure, and the left part is domain 1, the right domain2(PDB ID 3CM8).
在底物选择性方面,PAN是广谱的核酸内切酶,对单链DNA和RNA都具有剪切作用,但对不同底物具有不同的剪切活性和剪切模式。Klumpp等[19]发现内切酶剪切单链DNA的活性比RNA低,推测可能是底物的不同结构化程度导致的。Dias等[9]研究了PAN对不同结构化程度单链RNA的剪切活性,结果显示PAN对高度结构化的RNA如tRNA和AluRNA,基本不表现剪切活性;对部分结构化的RNA如ph-RNA有一定的剪切活性,将RNA剪切成较大的片段;而对无结构的RNA如U-richRNA,表现出较强的剪切活性,可以将RNA剪切成较小的片段。Noble等[15]发现不同毒株的PA亚基的内切酶活性虽然存在明显的差异,但是对同一底物的剪切模式相同;而且同一毒株的PA亚基内切酶活性虽然比RNA聚合酶低,但是两者对同一底物的剪切模式也相同。Datta等[20]发现PA的内切酶活性具有序列特异性,倾向在鸟苷酸的3′端剪切,如果帽结构下游第10~13碱基处没有鸟苷酸,剪切发生在第12个碱基的3′端;如果帽结构下游第10~13碱基处有鸟苷酸,剪切就发生在鸟苷酸的3′端。
3 以PA亚基内切酶活性为靶点的抗流感药物研究进展
根据文献报道,针对PA亚基内切酶活性已开展大量研究工作,并取得一定成果。较为常用的内切酶抑制剂模型主要是体外帽依赖性转录酶活性检测和体外核酸水解活性检测,由于这两种方法需要电泳检测结果,不适用于高通量筛选。Baughman等[21]利用竞争性荧光极化方法建立内切酶抑制剂高通量筛选模型,通过检测极化光强度的变化,筛选能够与PAN特异性抑制剂竞争结合到PAN活性中心的化合物,作者利用该模型筛选得到具有内切酶抑制作用的4,5-二羟基嘧啶衍生物。张国防等[22]利用荧光共振能量转移方法建立内切酶抑制剂高通量筛选模型,通过检测荧光强度的变化,筛选能抑制内切酶剪切活性的化合物。本实验室基于内切酶对单链DNA的剪切原理,利用DNA荧光染色的方法,建立了内切酶抑制剂筛选模型,此模型简单、快速、稳定、经济,适用于化合物的高通量筛选,并利用此模型发现了一些具有活性内切酶抑制作用的化合物[23]。
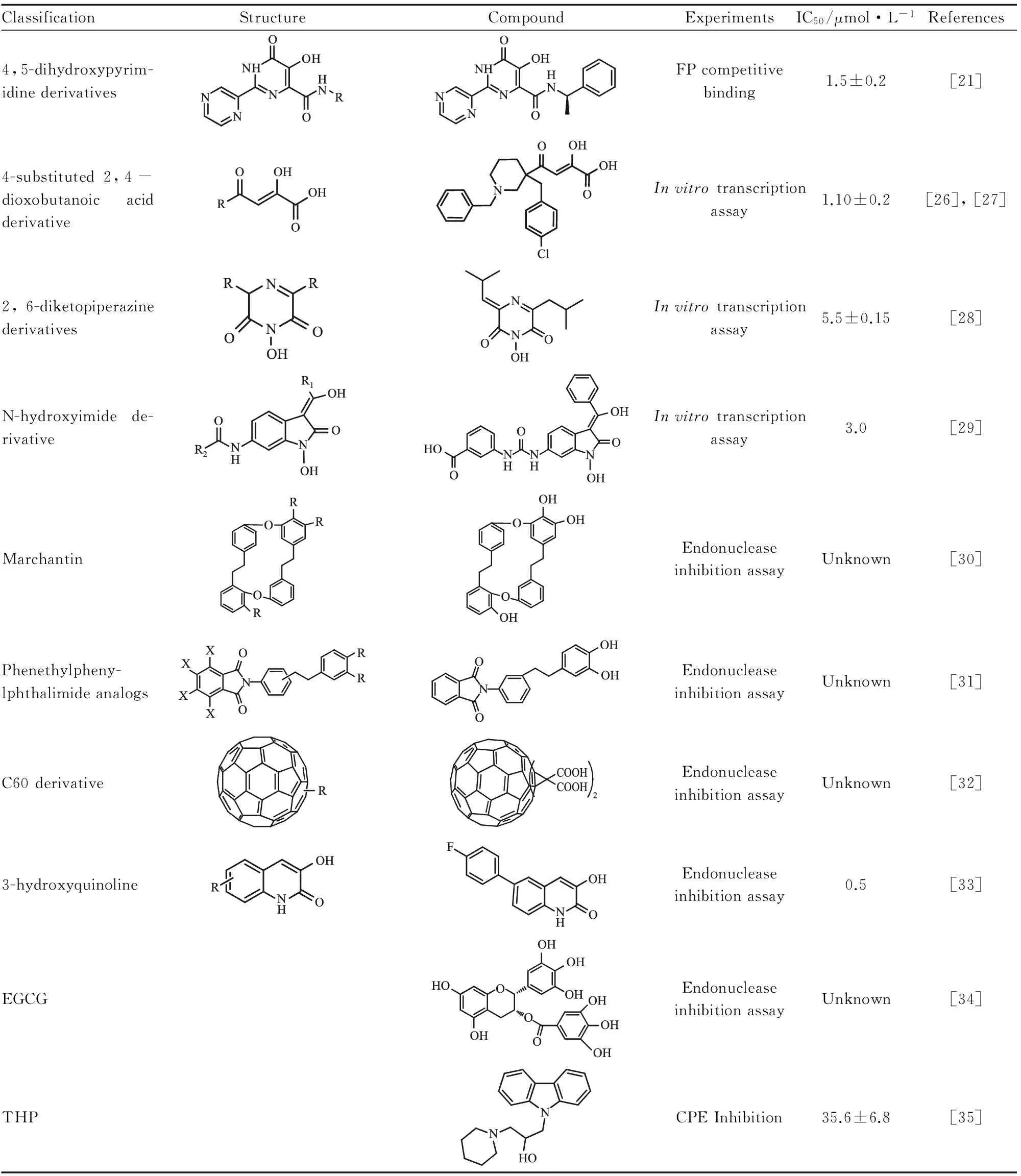
Tab 1 Classification of endonuclease inhibitors and their representative compounds
此外,虚拟筛选技术在内切酶抑制剂的筛选中也得到了很好的应用,Yan等[24]应用3D-QSAR建模、骨架迁移、分子对接等手段设计得到13个化合物,这些化合物能够与酶活性中心较好地结合,但缺乏具体实验验证。Chen等[25]通过建立药学团模型,对ChenBridge Express Pick 数据库进行筛选,获得了16个涉及5种结构的化合物,这些化合物具有较强的内切酶抑制作用(IC500.74×10-6~46×10-6mol·L-1),但细胞水平上对流感病毒的抑制作用参差不齐,有待于进一步优化。
基于内切酶抑制剂筛选模型、或虚拟筛选得到的、在细胞水平具有抗流感作用的内切酶抑制剂有很多,涉及多种结构,包括:4,5-二羟基嘧啶衍生物[21]、4-取代2,4-二氧丁酸衍生物[26-27],2,6-二酮吡嗪类天然产物flutimide[28]、N-羟基酰亚胺衍生物[29]、地钱素[30]、沙利度胺衍生物[31]、C60衍生物[32]、3-羟基喹啉类化合物[33],EGCG和EGC[34],THC-19[35]等,详细的结构信息见Tab 1。虽然这些化合物由于活性有限或毒性较大而未进入临床研究,结构生物学的发展为这些化合物的进一步优化、新化合物的设计提供重要信息。
DoBois等[16]和Kowalinski等[13]分别利用结构生物学获得多种内切酶抑制剂与H5N1、H1N1的PAN的晶体结构,发现化合母核上的羟基主要与酶活性中心的锰离子结合,而其他取代基则通过氢键或π键叠加,与酶活性中心中的疏水口袋相互作用,并指出内切酶抑制剂的强度可能与其结合的口袋数目有关,在优化内切酶抑制剂时应该尽可能通过增加取代基,增强化合物与酶活性中心疏水口袋的结合。Stevaert等[36]在DoBois和Kowalinski实验结果的基础上,进一步对参与L-742001结合的疏水口袋中及附近的氨基酸残基进行点突变,分析不同氨基酸残基在内切酶活性和抑制剂结合中的贡献,进而发现了一些同时参与内切酶活性和抑制剂结合的氨基酸残基(T20、T24、A37、T38、R84、V122、Y130),但是突变这些氨基酸残基后对内切酶活性和抑制剂活性影响不大,因此内切酶抑制剂优化时应该考虑加强抑制剂与这些氨基酸残基的作用,提高抑制剂活性的同时又可以防止耐药性的产生。根据DoBois和Kowalinski实验中获得的经验,即3个共平面的氧原子可以与酶活性中心的2个锰离子形成相互作用,Carceli等[37]设计了2-羟基苯甲酰胺母核,对其进行取代,分析不同化合物的内切酶抑制作用和其与内切酶活性中心的相互作用,最终确认DoBois和Kowalinski提出的理论,并建议2-羟基苯甲酰胺作为内切酶抑制剂的母核。这些结构信息将加快以PA亚基内切酶活性为靶点的抗流感药物的研究进程。
4 展望
流感病毒的基因组是由8个单负链RNA片段组成,在复制中极易发生突变和重组,导致耐药性的出现。在治疗流感时针对流感病毒生命周期的多个靶点联合给药,不仅可以提高抗流感的药效,而且可以减少耐药性的产生。然而,目前临床上可用于流感预防和治疗的药物主要是神经氨酸酶抑制剂,因此,针对不同靶点进行抗流感药物研发,有利于丰富抗流感药物,为抗流感联合用药提供足够的选择空间。
根据治疗慢性肝炎和艾滋病药物的启示[38],以聚合酶为靶点的药物,可以抑制病毒复制、转录,有效发挥抗病毒作用。RNA聚合酶PA亚基是流感病毒生命周期中关键蛋白,一方面其内切酶活性是病毒mRNA合成所必须的,并且在宿主细胞中不存在类似活性的酶;另一方面酶活性中心的关键氨基酸残基在3型流感病毒中高度保守、突变率低,耐药性出现的频率低,这些特点都决定PA亚基可能成为抗流感新靶点。针对此靶点的抑制剂,不仅能够有效抑制多种类型流感病毒转录过程,同时对宿主无毒,耐药性出现慢,能够成为高效、广谱、低毒抗流感药物。
[1] Hayden F. Developing new antiviral agents for influenza treatment: what does the future hold?[J].ClinInfectDis, 2009, 48(Supplement 1): S3-S13.
[2] Pizzorno A, Abed Y, Boivin G. Influenza drug resistance[C]//Seminars in respiratory and critical care medicine? Thieme Medical Publishers, 2011, 32(4): 409-22.
[3] Gutiérrez R A, Naughtin M J, Horm S V, et al. A (H5N1) virus evolution in South East Asia[J].Viruses, 2009, 1(3): 335-61.
[4] Boivin S, Cusack S, Ruigrok RW, et al. Influenza A virus polymerase: structural insights into replication and host adaptation mechanisms[J].JBiolChem, 2010, 285(37): 28411-7.
[5] Fodor E. The RNA polymerase of influenza A virus: mechanisms of viral transcription and replication[J].ActaVirol, 2012, 57(2): 113-22.
[6] Hara K, Schmidt F I, Crow M, et al. Amino acid residues in the N-terminal region of the PA subunit of influenza A virus RNA polymerase play a critical role in protein stability, endonuclease activity, cap binding, and virion RNA promoter binding[J].JVirol, 2006, 80(16): 7789-98.
[7] Zheng W, Olson J, Vakharia V, et al. The crystal structure and RNA-binding of an orthomyxovirus nucleoprotein[J].PlosPathogens, 2013, 9(9): e1003624.
[8] Yuan P, Bartlam M, Lou Z, et al. Crystal structure of an avian influenza polymerase PANreveals an endonuclease active site[J].Nature, 2009, 458(7240): 909-13.
[9] Dias A, Bouvier D, Crépin T, et al. The cap-snatching endonuclease of influenza virus polymerase resides in the PA subunit[J].Nature, 2009, 458(7240): 914-8.
[10] He X, Zhou J, Bartlam M, et al. Crystal structure of the polymerase PAC-PB1Ncomplex from an avian influenza H5N1 virus[J].Nature, 2008, 454(7208): 1123-6.
[11] Obayashi E, Yoshida H, Kawai F, et al. The structural basis for an essential subunit interaction in influenza virus RNA polymerase[J].Nature, 2008, 454(7208): 1127-31.
[12] Zhao C, Lou Z, Guo Y, et al. Nucleoside monophosphate complex structures of the endonuclease domain from the influenza virus polymerase PA subunit reveal the substrate binding site inside the catalytic center[J].JVirol, 2009, 83(18): 9024-30.
[13] Kowalinski E, Zubieta C, Wolkerstorfer A, et al. Structural analysis of specific metal chelating inhibitor binding to the endonuclease domain of influenza pH1N1 (2009) polymerase[J].PlosPathogens, 2012, 8(8): e1002831.
[14] Crépin T, Dias A, Palencia A, et al. Mutational and metal binding analysis of the endonuclease domain of the influenza virus polymerase PA subunit[J].JVirol, 2010, 84(18): 9096-104.
[15] Noble E, Cox A, Deval J, et al. Endonuclease substrate selectivity characterized with full-length PA of influenza A virus polymerase[J].Virology, 2012, 433(1): 27-34.
[16] DuBois R M, Slavish P J, Baughman B M, et al. Structural and biochemical basis for development of influenza virus inhibitors targeting the PA endonuclease[J].PlosPathogens, 2012, 8(8): e1002830.
[17] Doan L, Handa B, Roberts N A, et al. Metal ion catalysis of RNA cleavage by the influenza virus endonuclease[J].Biochemistry, 1999, 38(17): 5612-9.
[18] Xiao S, Klein M L, LeBard D N, et al. Magnesium-dependent RNA binding to the PA endonuclease domain of the avian influenza polymerase[J].JPhysicalChemB, 2014, 118(4): 873-89.
[19] Klumpp K, Doan L, Roberts N A, et al. RNA and DNA hydrolysis are catalyzed by the influenza virus endonuclease[J].JBiologicalChem, 2000, 275(9): 6181-8.
[20] Datta K, Wolkerstorfer A, Szolar O H, et al. Characterization of PA-N terminal domain of influenza A polymerase reveals sequence specific RNA cleavage[J].NucleicAcidsRes, 2013, 41(17): 8289-99.
[21] Baughman B M, Jake Slavish P, DuBois R M, et al. Identification of influenza endonuclease inhibitors using a novel fluorescence polarization assay[J].ACSChemBiol, 2012, 7(3): 526-34.
[22] 张国防, 李真真, 姚伟利, 等. 基于流感病毒 PAN蛋白的高通量药物筛选[J]. 生物技术通报, 2014, 2: 181-6.
[22] Zhang G F, Li Z Z, Yao W L, et al. High throughput drug screening on protein pan of influenza virus[J].BiotechnolBull, 2014, 2: 181-6.
[23] 周伟玲, 杨 帆, 祖 勉, 等. 流感病毒核酸内切酶抑制剂高通量筛选模型的建立[J]. 中国新药杂志, 2013, 22(10): 1137-42.
[23] Zhou W L, Yang F, Zu M, et al. Establishment of high throughput screening assay targeting the endonuclease activity of influenza virus PANprotein[J].ChinJNewDrugs, 2013, 22(10): 1137-42.
[24] Yan Z, Zhang L, Fu H, et al. Design of the influenza virus inhibitors targeting the PA endonuclease using 3D-QSAR modeling, side-chain hopping, and docking[J].BioorganicMedChemLett, 2014, 24(2): 539-47.
[25] Chen E, Swift R V, Alderson N, et al. Computation-guided discovery of influenza endonuclease inhibitors[J].ACSMedChemLet, 2013, 5(1): 61-4.
[26] Tomassini J, Selnick H, Davies M E, et al. Inhibition of cap (m7GpppXm)-dependent endonuclease of influenza virus by 4-substituted 2, 4-dioxobutanoic acid compounds[J].AntimicrobAgentsChemother, 1994, 38(12): 2827-37.
[27] Hastings J C, Selnick H, Wolanski B, et al. Anti-influenza virus activities of 4-substituted 2, 4-dioxobutanoic acid inhibitors[J].AntimicrobAgentsChemother, 1996, 40(5): 1304-7.
[28] Tomassini J E, Davies M E, Hastings J C, et al. A novel antiviral agent which inhibits the endonuclease of influenza viruses[J].AntimicrobAgentsChemother, 1996, 40(5): 1189-93.
[29] Parkes KEB, Ermert P, Fässler J, et al. Use of a pharmacophore model to discover a new class of influenza endonuclease inhibitors[J].JMedChem, 2003, 46(7): 1153-64.
[30] Iwai Y, Murakami K, Gomi Y, et al. Anti-influenza activity of marchantins, macrocyclic bisbibenzyls contained in liverworts[J].PlosOne, 2011, 6(5): e19825.
[31] Iwai Y, Takahashi H, Hatakeyama D, et al. Anti-influenza activity of phenethylphenylphthalimide analogs derived from thalid omide[J].BioorganicMedChem, 2010, 18(14): 5379-90.
[32] Shoji M, Takahashi E, Hatakeyama D, et al. Anti-influenza activity of C60 fullerene derivatives[J].PlosOne,2013, 8(6): e66337.
[33] Sagone H Y, Slavish P J, Baughman B M, et al. 3-Hydroxyquinolin-2(1H)-ones as inhibitors of influenza A endonuclease[J].ACSMedChemLett, 2013, 4(6):547-50.
[34] Kuzuhara T, Iwai Y, Takahashi H, et al. Green tea catechins inhibit the endonuclease activity of influenza A virus RNA polymerase[J].PlosCurr, 2009, 1:RRN1052.
[35] Yamada K, Koyama H, Hagiwara K, et al. Identification of a novel compound with antiviral activity against influenza A virus depending on PA subunit of viral RNA polymerase[J].MicrobeInfect, 2012, 14(9):740-7.
[36] Stevaert A, Dallocchio R, Dessì A, et al. Mutational analysis of the binding pockets of the diketo acid inhibitor L-742,001 in the influenza virus PA endonuclease[J].JVirol, 2013, 87(19): 10524-38.
[37] Carceli M, Rogolino D, Bacchi A, et al. Metal-chelating 2-hydroxyphenyl amide pharmacophore for inhibition of influenza virus endonuclease[J].MoleculPharma, 2014, 6(1):304-16.
[38] 邢世华,李晓波.清热解毒类中药抗病毒活性及作用机制研究进展[J].中国药理学通报, 2014, 30(4): 464-8.
[38] Xing S B, Li X B. Research progress of anti-virus activity and action mechanism of febrifuge and detoxifying herbs[J].ChinPharmacolBull, 2014, 30(4): 464-8.
PA subunit of RNA polymerase:a potential drug target for anti-influenza
LIAN Wen-wen1, LIU Ai-lin1,2,3, DU Guan-hua1,2,3
(1.InstituteofMateriaMedica,ChineseAcademyofMedicalSciencesandPekingUnionMedicalCollege,Beijing100050,China;2.BeijingKeyLaboratoryofDrugTargetResearchandDrugScreening,Beijing100050,China;3.StateKeyLaboratoryofBioactiveSubstanceandFunctionofNaturalMedicines,Beijing100050,China)
Influenza is a globally serious contanious desease. The main problems faced with anti-influenza agents are the drug resistance and low efficacy to highly pathogenic virus. RNA polymerase is acritical enzyme involved in replication and transcription of virus in the host cell, and the PA subunit, which provides primers for viral transcription via its endonuclease activity, is a promising anti-influenza target. This article mainly discusses the PA subunit, including its structures, functions, and progress in the development of anti-influenza agents, providing information for research on the PA subunit and anti-influenza agents targeting PA subunit.
infuenza virus; the amino terminal of PA subunit; the carboxyl terminal of PA subunit; endonuclease; metal binding site; high throughput screen model
时间:2015-3-3 11:08 网络出版地址:http://www.cnki.net/kcms/detail/34.1086.R.20150303.1108.001.html
2014-10-18,
2014-12-25
国家科技部“重大新药创制”科技重大专项(No 2014ZX09507003-002,2012ZX 09301002-2013HXW-11);国际合作项目(No 2011DF R31240)
连雯雯(1989-),女,博士生,研究方向:抗流感药物研究及新药发现,Tel:010-83157220,E-mail:lianwenwen1989@imm.ac.cn; 刘艾林(1968-),女,博士,博士生导师,研究方向:抗流感药物研究、计算药理学及新药发现,Tel:010-83150885,E-mail:liuailin@imm.ac.cn; 杜冠华(1956-),男,博士,博士生导师,研究方向:药理学及新药发现,Tel:010-63165184,E-mail:dugh@imm.ac.cn
10.3969/j.issn.1001-1978.2015.03.001
A
1001-1978(2015)03-0297-06
R-05;R345.57;R373.13;R977.3;R978.7