肾脏特异性microRNA的鉴定及功能预测
2015-06-08李丽民刘志红
孟 娇 李丽民 刘志红 曾 科,
肾脏特异性microRNA的鉴定及功能预测
孟 娇1李丽民2刘志红1曾 科1,2
目的:应用 Solexa 技术分析和筛选出肾脏特异性表达microRNA (miRNA),同时利用生物信息学技术预测特异表达 miRNA 的靶基因并对其功能进行初步分析,探讨特异表达miRNA在肾脏中的功能。 方法:选取8周龄C57/B6 雄性小鼠,取其全身11个组织器官,提取总RNA并从中分离小分子量RNA进行Solexa测序,筛选出相较其他组织器官,在肾脏中表达最为特异的miRNA;然后,利用实时荧光定量逆转录聚合酶链反应(qRT-PCR)技术对筛选出的肾脏特异性表达miRNA进一步验证;最后,通过生物信息学技术对其进行靶基因预测,并对交集的基因集合分别进行GO分析和KEGG 通路分析。 结果:Solexa测序结果显示在正常小鼠中miR-196a和miR-196b在肾脏最特异性高表达。qRT-PCR验证了miR-196a和miR-196b在小鼠11个组织器官中的表达分布情况,定量结果与测序结果相一致。TargetScan6.2和 PicTar两个软件预测到交集靶基因74个。GO分析显示miR-196a/b的预测靶基因集合显著富集在血小板源生长因子结合活性、转录调控活性、细胞外基质的结构组成等分子功能上并参与调控代谢、发育等生物学过程(P<0.01)。信号转导通路显著富集于促性腺激素释放激素信号通路、细胞外基质受体相互作用等多个信号通路和脑胶质瘤、前列腺癌等疾病通路中(P<0.05)。 结论:miR-196a/b在肾脏中特异性高表达,靶基因功能广泛,与细胞代谢、生长发育等密切相关。
肾组织 Solexa 测序 miR-196a miR-196b
MicroRNA (miRNA)是一类广泛存在于真核生物中的长约19~23个核苷酸的小分子单链非编码RNA,其通过对靶基因进行转录后水平的调控[1],从而参与生物个体发育、组织分化、细胞增殖和凋亡、能量代谢、免疫、神经内分泌、癌症等众多生命过程[2-6]。许多miRNAs 的表达都有一定的组织特异性,例如miR-122主要在肝脏中特异性表达[7,8],miR-143 为脂肪特异性 miRNA[9], miR-133在肌肉中特异性表达[10]。这些在组织中特异性高表达的miRNA,在组织器官功能中发挥着十分重要的调控作用。例如,肝特异性miR-122,在抑制肝肿瘤发生和调控脂质代谢的过程中发挥着十分重要的作用[7,11]。因此,明确动物组织中特异性高表达的miRNA并对其功能进行分析,对理解这些组织器官的生物学过程有十分重要的意义。但是,目前对于肾脏中特异性miRNA的表达情况还缺乏明确的分析。本研究通过Solexa测序分析和实时荧光定量逆转录聚合酶链反应(qRT-PCR)的方法筛选并鉴定出肾脏中特异性高表达的miRNAs,并对其靶基因及可能参与调控的生物学信号通路进行了生物信息学分析,为进一步研究miRNA在肾脏中的生物学功能提供理论基础。
对象和方法
研究对象 3只均为8周龄的C57/B6雄性小鼠,5%的戊巴比妥那麻醉后,进行心脏灌流,将小鼠全身血液灌出后,取其全身11处组织器官,包括大脑、小脑、心、肝、脾、肺、胃、胰腺、肾、小肠、大肠。将取出的组织立即投入液氮后,于-80℃保存。
总RNA 提取 按照 Trizol (Invitrogen, Gaithersburg, MD, USA)法提取所有组织的总 RNA,用NANODROP2000 紫外分光光度计(Nanodrop Nechnologies,Wilmington,DE)测定浓度后,用 Agilent BioAnalyzer 2100(Agilent Technologies,Palo Alto,CA)进行质量检测,总RNA于-80℃保存备用。
Solexa测序分析 将3个小鼠的相同组织器官的总 RNA进行混合,制备成11个小RNA测序文库的原始材料。Solexa测序在深圳华大基因科技有限公司完成。首先,采用 15%变性聚丙烯酰胺凝胶电泳技术(PAGE)从总 RNA 中分离小分子量(15~30 nt) RNA,纯化后对小RNA 连接 3′和 5′接头。然后运用SuperScriptⅡ反转录酶将连接有5′和3′接头的RNA进行反转录,合成 cDNA,并使用下列程序进行 PCR扩增:98℃预变性 30s; 98℃变性10s、60℃退火30s、72℃延伸 30s,17 个循环;72℃ 延伸10 min;4℃保存。然后进行文库检测、DNA的成簇扩增和上机测序。最后进行生物信息组装和分析。将测序获得的各组织器官的total reads (Solexa 测序所得的序列总数)进行去除低质量、去除污染、去除接头等处理,获得clean reads (分析处理后的干净序列),然后统计序列总数、种数和长度,最后获得全部有效序列用作后续分析。首先将有效序列比对到 GeneBank、Repeat sequence、Rfam数据库,去除mRNA、rRNA、tRNA、snoRNA、snRNA等非编码小RNAs。最后,将剩余序列与 miRBase 数据库 (Release 20.0)中已收录的哺乳动物中已知的miRNA的成熟序列或前体序列进行比较,从而鉴定样品中成熟 miRNA 的表达量。
qRT-PCR验证 肾脏特异性高表达miRNA的验证采用qRT-PCR的方法。总RNA 在AMV 逆转录酶(TaKaRa)和茎环逆转录引物(Applied Biosystems)的作用下逆转录成cDNA。10 μl逆转录体系:5×逆转录缓冲液 (TaKaRa) 2 μl,AMV逆转录酶 (TaKaRa) 0.5 μl,dNTPs (TaKaRa,10 mmol/L) 1 μl,茎环逆转录引物(Applied Biosystems)1 μl,RNA 2 μl (1 μg),DEPC水 3.5 μl。反应条件为:16 ℃/30 min,42℃/30 min,85℃/5 min。将获得的cDNA于-20 ℃ 中保存备用。qRT-PCR在ABI 7900 HT Fast 荧光定量PCR仪(Applied Biosystems)上进行。20 μl的定量PCR反应体系为:10×PCR缓冲液(TaKaRa)2 μl, dNTPs(TaKaRa,10 mmol/L) 0.4 μl,MgCl2 (TaKaRa,25 mmol/L)1.2 μl,Taq酶 (TaKaRa) 0.3 μl,TaqMan探针(Applied Biosystems)0.33 μl,水 14.77 μl, cDNA 1 μl。反应条件为:95℃/5 min, 95℃/15s,60℃/1 min进行40个循环。所有的PCR反应都有三个副孔。以U6 snRNA作为内参基因,相对表达量结果采用2-ΔCT法进行计算分析。
miRNAs靶基因预测及功能分析 利用 TargetScan 6.2(http://www.targetscan.org/)和 PicTar ( http://pictar.mdc-berlin.De)两种方法计算预测 miR-196a/b 的靶基因,将其交集进行后续的生物信息学分析。采用GO(http://www.geneontology.org /)数据库进行GO分析,并根据统计检验方法(P<0.01)筛选显著性差异的分类。采用 DAVID 数据库( http://david.abcc.ncifcrf.gov/)对 miR-196a/b进行 KEGG 通路分析。
统计学分析 不同组织器官表达量差异显著性检验采用 SPSS 19.0的独立样本的 T-test 法。GO注释的显著性分析是利用超几何检验计算显著性程度P值,以P<0.01为显著性阈值分别得到相对于背景具有统计意义的高频率注释。生物通路富集分析是通过Fisher Exact Test计算P值,以P<0.05为显著性阈值得到基因集合相对于背景具有统计意义的信号转导及疾病通路。
结 果
Solexa测序结果分析 首先对小鼠肾组织的测序结果分析后发现,在肾组织中表达量位于前20位的miRNAs依次为let-7b,miR-143,miR-196b,miR-21,miR-378,miR-29a,miR-27b,miR-140*,miR-148a,miR-146a,miR-30a,miR-200a,miR-148b,miR-872,miR-429,miR-196a,miR-30e,miR-151-3p,miR-200b,miR-30c-2*,其在肾脏miRNA的百分比如图1所示。这些miRNAs的表达量(拷贝数)都高于10 000,说明这些miRNAs在小鼠肾组织中都是表达量较高的miRNAs。为了筛选出在肾脏特异性高表达的miRNAs,我们检查了上述20个肾脏高表达的miRNAs在其他10个组织器官(心、肝、脾、肺、胃、大脑、小脑、胰腺、小肠、结肠)中的表达,发现在所测的11个组织器官中miR-196a和miR-196b大多在肾脏中出现,分别占61.69%和62.24%,其次是结肠,表达量分别占总量的33.26%和33.14%(图2A、B),并且测序结果表明miR-196b在肾脏中含量是miR-196a的11.26倍。为了保证测序和分析结果的可靠性,我们同时分析了目前被认为是肝脏特异性高表达的miR-122在各组织器官中表达情况,结果发现miR-122在肝脏中最为富集,占到了所测组织表达量的99.27%,而在其他组织器官中表达量都极低(图2C),这和已发表的实验结果具有一致性[7,8]。
qRT-PCR验证 对miR-196a/b在11个组织器官中表达水平进行了qRT-PCR验证,以U6为内参,分别计算出miR-196a和miR-196b在各组织器官中的相对表达量。如图3所示,和其他10个组织器官相比,miR-196a和miR-196b在肾脏中的表达量最高,并且miR-196b的含量高于miR-196a,这和Solexa的测序结果一致。
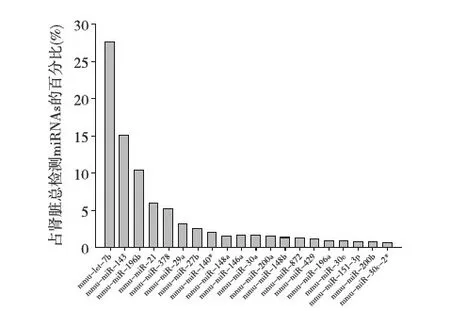
图1 肾脏中表达丰度位于前20位的microRNAs
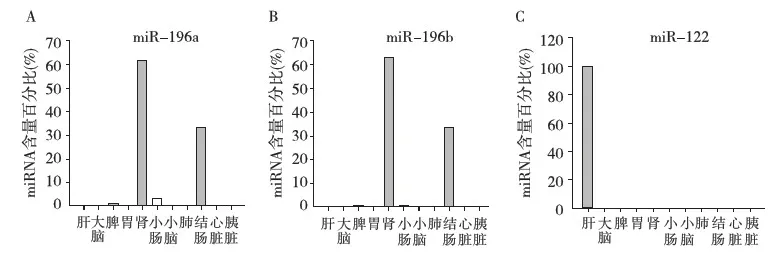
图2 miR-196a/b以及miR-122在小鼠全身所测各组织中的表达分布
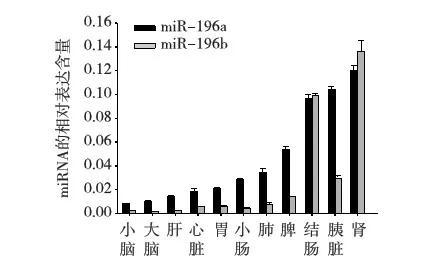
图3 qRT-PCR分析小鼠11个组织器官中miR-196a/b的相对表达量
miRNA196a/b的靶基因预测 miR-196a和miR-196b的序列仅有一个碱基之差,且其种子序列相同,在用软件预测靶基因时发现,其预测结果具有一致性,因此后续分析将miR-196a和miR-196b归为一类进行分析。TargetScan预测的靶基因为295个,PicTar为162个,将这两个数据库的计算结果取交集后共获得74个靶基因。
miR-196a/b预测靶基因的 GO分类富集分析 对基因集合中的74个靶基因 进行 GO 注释描述、GO 富集分析。通过GO注释描述得到63个基因的GO 细胞组分注释信息、65个基因的GO分子功能注释信息以及64个基因的GO生物学过程注释信息。进一步对这些基因的GO注释信息进行富集分析,结果显示miR-196a/b靶基因集合分别富集在血小板源生长因子结合活性、生长因子结合活性、转录调控活性及细胞外基质结构成分等分子功能上和骨骼系统形态发生/发育、代谢调控、发育进程等生物学过程中(P<0.01)(表1)。
miR-196a/b预测靶基因的生物通路富集分析 利用DAVID数据库对基因集合中的74个基因进行生物通路富集分析结果显示, 在KEGG代谢通路数据库中信号转导通路显著富集于胰岛素信号通路、黏着斑信号通路、细胞外基质受体相互作用信号通路、促性腺激素释放激素信号通路等信号通路和脑胶质瘤、前列腺癌等疾病通路中(P<0.05)(表2)。
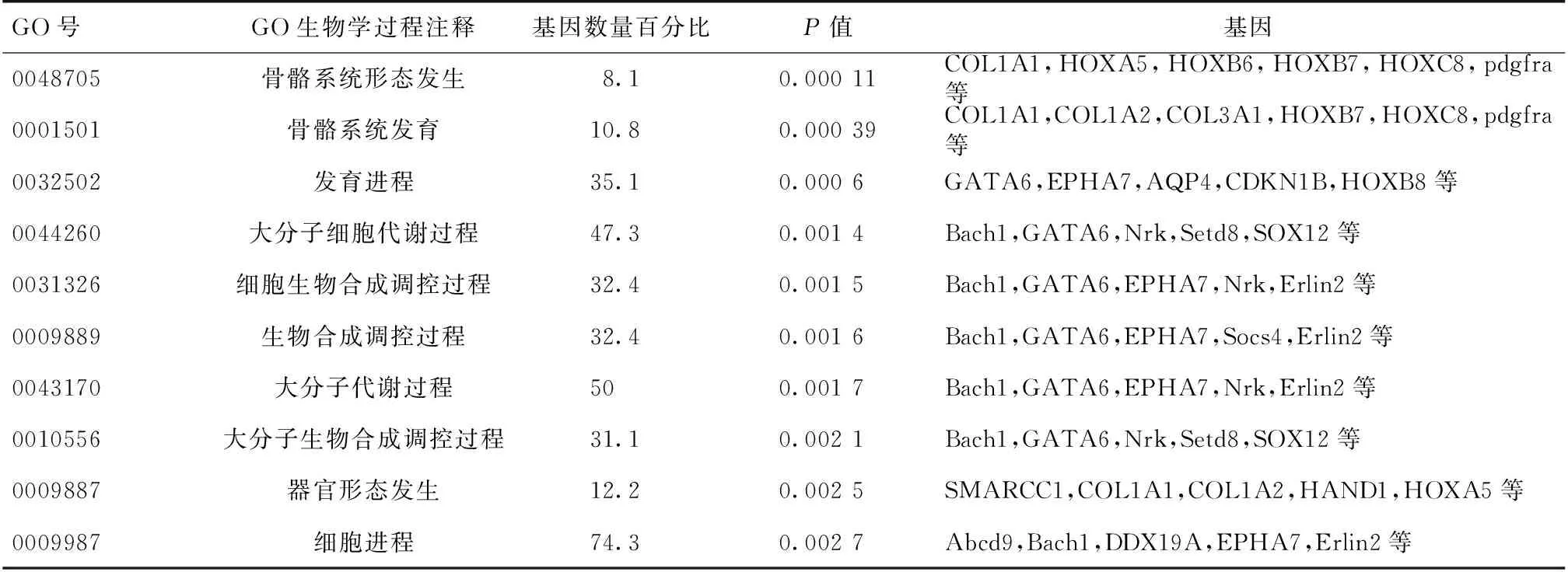
表1 miR-196a/b预测靶基因的GO生物学过程分类
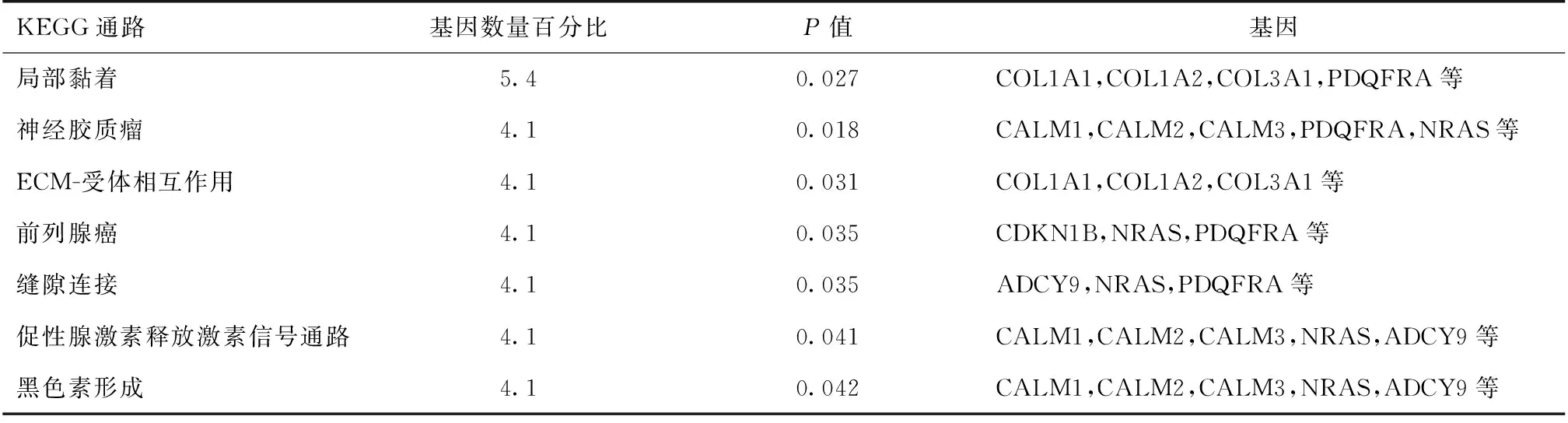
表2 miR-196a/b预测靶基因KEGG通路数据库富集分析结果
讨 论
随着对miRNA越来越广泛和深入的研究,miRNA在细胞分化、组织发育、疾病调控中的强大作用也越来越多的被人们所认识。miRNA在序列、结构、丰度和表达方式上具有多样性,在不同的组织及在组织的不同发育阶段,miRNA的表达水平不同。研究表明,某些miRNAs,如miRNA-122,miR-143等这些被认为是在组织中特异性表达的miRNAs,都在组织器官正常功能调节中发挥十分重要的作用。目前,在肾脏中,尚无明确的有关肾脏特异性高表达miRNA的报道,因此分析不同组织器官miRNA的表达情况,筛选出肾脏特异性高表达的miRNA,可以帮助我们更好的研究miRNAs在肾脏中的功能。
本研究运用Solexa高通量测序技术对小鼠11个组织器官中的小RNA进行了测序,获取miRNA表达谱,通过分析筛选出在肾脏中特异性高表达的两个miRNAs,分别为miR-196a和miR-196b,同时采用qRT-PCR的方法对测序结果进行了验证,结果与测序检测结果一致。随后,对这些miRNAs进行靶基因预测和功能分析,结果发现miR-196a/b参与多种细胞代谢和功能调控。
关于不同组织器官中miRNAs的表达分布情况已有不少研究。同时检测miRNAs表达水平的技术方法也有很多种,比如高通量测序、Northern分析、基因芯片分析和qRT-PCR等。本研究中采用的是Solexa高通量测序和qRT-PCR相结合的技术。Solexa作为第二代高通量测序技术具有高灵敏度、精确性及重复性[12]。相较于基因芯片是针对已知的数据库设计探针进行杂交,高通量测序的优势在于可直接对任何物种进行包括未知基因在内的全基因组表达谱分析,其发现能力和寻找新的信息的能力从本质上高于芯片技术[13]。同时,我们又结合具有高特异性和重复性的qRT-PCR探针法[14]对Solexa分析结果进行进一步验证,保证了肾脏特异性miRNA的筛选结果的可靠性。
和现有的有关肾脏miRNA的表达分布情况的研究相比较,我们的研究结果与其有一定的一致性,同时也有自己的独特性。Landgraf P 等[15]通过对小鼠、大鼠肾组织及人类足细胞中 的miRNA进行了广泛的克隆和测序,研究结果表明miR-196a/b、miR-10a、miR-200a和miR-335 在肾组织较富集。而我们的研究对哪一种miRNA是相较于其他miRNA在肾脏中特异性表达进行了更具体的分析。我们不仅筛选出在肾组织中比较富集的miRNAs,此外,还对这些miRNAs分别在肾脏中的富集程度以及这些miRNAs分别在所测小鼠11组织器官中表达分布情况进行了分析比较,从而筛选出在肾脏中最特异性表达的miR-196a/b。在实验结果的可靠性方面,我们不仅验证了已经公认的在肝脏中特异性表达的miR-122,同时还将miR-122表达分布情况作为阳性参照,对测序结果进行了qRT-PCR的验证,保证了实验结果的特异性及可靠性。但对miR-196a/b在肾脏组织和固有细胞中的表达分布情况,还需要我们用后续的实验进行分析验证。
miR-196a/b在物种之间具有高度的保守性,且在肾脏中如此特异性的高表达,这些提示我们miR-196a/b在肾脏中发挥着极其重要的调控作用。为了更好的研究miR-196a/b在肾脏中的功能作用,我们首先对其可能的调控的靶基因进行了预测。TargetScan 和PicTar预测靶基因预测的精确度较高,本实验同时采用这两种预测算法进行了miR-196a/b 靶基因的预测,然后取其交集,提高了预测结果的可靠性同时也减少了假阳性率[9,16,17]。但这种方法也使预测的靶基因数大大降低,对全面的研究miR-196a/b在肾脏中的功能可能带来影响。
本文通过对靶基因交集进行了GO功能注释、GO富集分析和KEGG信号转导通路富集分析,从多层次、多角度的探索miR-196a/b可能参与的功能调控。分析结果表明, 靶基因主要富集在代谢过程、 生物调控及发育等生物过程中。靶基因在发育和细胞生物合成等功能过程中的集中,表明其可能在机体发育和形态发生中起着重要调控作用,因此miR-196a/b对发育的调控作用也是不容忽视的。已有研究结果表明miRNA196a/b通过作用于HOX基因家族对胚胎发育起着十分重要的调控作用[18],这也为我们在研究肾脏发育方面提供了新的思路。在KEGG通路分析中我们发现细胞外基质受体相互作用为miR196a/b显著富集的信号通路之一,而Honda等[19]的研究也发现miR-196a可通过调控细胞外基质成分Ⅰ型胶原蛋白的表达参与硬皮病的发生。细胞外基质是肾脏组织的重要组成成分,这就提示我们miR-196a/b可能也会通过对肾脏基质成分的调节来参与一些肾脏疾病如肾纤维化疾病的发生发展。
miRNA对靶基因的调控作用具有多样性与复杂性。生物信息学技术以及已有的文献报道只是为我们提供可能性最大的参考信息和理论基础,对于miR-196a/b在肾脏中的具体功能研究还需通过实验进一步验证。
1 Pasquinelli AE.MicroRNAs and their targets:recognition,regulation and an emerging reciprocal relationship.Nat Rev Genet,2012,13(4):271-282.
2 Peláez N,Carthew RW.Biological robustness and the role of microRNAs:a network perspective.Curr Top Dev Biol,2012,99:237-255.
3 Kloosterman WP,Plasterk RH.The diverse functions of MicroRNAs in animal development and disease.Dev Cell,2006,11(4):441-450.
4 Ambros V.The functions of animal microRNAs.Nature,2004,431(7006):350-355.
5 Shivdasani RA.MicroRNAs:regulators of gene expression and cell differentiation.Blood,2006,108(12):3646-3653.
6 He L,Hannon GJ.MicroRNAs:small RNAs with a big role in gene regulation.Nat Rev Genet,2004,5 (7):522-531.
7 Jopling C.Liver-specific microRNA-122:Biogenesisand function.RNA Biol,2012,9(2):137-142.
8 Lagos-Quintana M,Rauhut R,Yalcin A,et al.Identification of tissue-specific microRNAs from mouse.Curr Biol,2002,12 (9):735-739.
9 Trakooljul N,Hicks JA,Liu HC.Identification of target genes and pathways associated with chicken microRNA miR-143.Anim Genet,2010,41 (4):357-364.
10 Yu H,Lu Y,Li Z,et al.microRNA-133:expression,function and therapeutic potential in muscle diseases and cancer.Curr Drug Targets,2014,15(9):817-828.
11 Esau C,Davis S,Murray SF,et al.miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting.Cell Metab,2006,3 (2):87-98.
12 Morozova O,Marra MA.Applications of next-generation sequencing technologies in functional genomics.Genomics,2008,92 (5):255-264.
13 ′t Hoen PA,Ariyurek Y,Thygesen HH,et al.Deep sequencing-based expression analysis shows major advances in robustness,resolution and inter-lab portability over five microarray platforms.Nucleic Acids Res,2008,36 (21):e141.
14 Duncan DD,Eshoo M,Esau C,et al.Absolute quantitation of microRNAs with a PCR-based assay.Anal Biochem,2006,359 (2):268-270.
15 Landgraf P,Rusu M,Sheridan R,et al.A mammalian microRNA expression atlas based on small RNA library sequencing.Cell,2007,129 (7):1401-1414.
16 Lewis BP,Shih IH,Jones-Rhoades MW,et al.Prediction of mammalian microRNA targets.Cell,2003,115 (7):787-798.
17 Sethupathy P,Megraw M,Hatzigeorgiou AG.A guide through present computational approaches for the identification of mammalian microRNA targets.Nat Methods,2006,3 (11):881-886.
18 Hornstein E,Mansfield JH,Yekta S,et al.The microRNA miR-196 acts upstream of Hoxb8 and Shh in limb development.Nature,2005,438 (7068):671-674.
19 Honda N,Jinnin M,Kajihara I,et al.TGF-beta-mediated downregulation of microRNA-196a contributes to the constitutive upregulated type I collagen expression in scleroderma dermal fibroblasts.J Immunol,2012,188 (7):3323-3331.
(本文编辑 人 正 一 讷)
Identification and function prediction of kidney-specific microRNAs
MENGJiao1,LILimin2,LIUZhihong1,ZENKe1,2
1NationalClinicalResearchCenterofKidneyDiseases,JinlingHosptital,NanjingUniversitySchoolofMedicine,Nanjing210016,China2JERC-MBB,StateKeyLaboratoryofPharmaceuticalBiotechnology,NanjingUniversitySchoolofLifeSciences
ZENKe(E-mail:kzen@nju.edu.cn);LIUZhihong(E-mail:liuzhihong@nju.edu.cn)
Objective:Solexa Sequencing was used to screen for kidney-specific microRNAs. The target genes of the identified miRNAs were analysed by bioinformatics to predict the biological functions of the miRNAs. Methodology:To screen for the miRNAs specifically expressed in kidney, leven tissues or organs from 8 weeks C57/B6 male mice were collected. Total RNA was extracted, followed by small RNA isolation. Solexa sequencing was performed to profile the small RNA samples. Real-time quantitative reverse transcription polymerase chain reaction (qRT-PCR) was used to validate the results from the sequencing. Bioinformatic analysis was performed to predict the target genes of the kidney specific miRNAs. The predicted target genes were subject to gene ontology (GO) and KEGG pathway analyses for function prediction. Results:Solexa sequencing results showed that miR-196a and miR-196b were the kidney specific miRNAs in normal mice. The distribution of miR-196a and miR-196b in 11 tissues or organs of mice were validated by q-PCR, and the results were consistent with those from sequencing. The number of overlapping predicted target genes by TargetScan6.2 and PicTar was 74. GO analysis showed that the genes involved in the regulation of platelet-derived growth factor binding, transcriptional activity and extracellular matrix structural constituent were enriched in the list of the 74 genes. The genes associated with several biological processes, including metabolic process and developmental process, were also enriched among the 74 genes (P<0.01). The pathway analysis showed that the genes involved in GnRH signaling, ECM-receptor interaction, and glioma and prostate cancer, were significantly enriched in the 74 genes (P<0.05). Conclusion:miR-196a/b specifically expressed in the kidney, and their functions are predicted to be closely related with cell metabolism, growth and development. Thus, our study may help understand the mechanism underlying kidney disease. Further studies are required to understand the role of miR-196a/b in the physiology and pathology of kidney.
kidney Solexa sequencing miR-196a miR-196b
国家重点基础研究发展计划(973计划)(2012CB517603)
1南京大学医学院附属金陵医院(南京军区南京总医院)肾脏科 硕士研究生(孟 娇);国家肾脏疾病临床医学研究中心 全军肾脏病研究所(南京,210016);2南京大学生命科学学院 医药生物技术国家重点实验室 江苏小核糖核酸工程研究中心
曾 科(E-mail:kzen@nju.edu.cn);刘志红(E-mail:liuzhihong@nju.edu.cn)
2015-01-19
ⓒ 2015年版权归《肾脏病与透析肾移植杂志》编辑部所有
