沤麻废水处理池微生物多样性分析及其Fosmid文库酶活初步筛选
2015-05-05俞勤丽董志扬王远亮
董 亮,俞勤丽,董志扬,王 丽,*,王远亮,*
(1.湖南农业大学食品科技学院,湖南长沙 410128;2.中国科学院微生物研究所,微生物资源前期开发国家重点实验室,北京 100101)
沤麻废水处理池微生物多样性分析及其Fosmid文库酶活初步筛选
董 亮1,俞勤丽2,董志扬2,王 丽2,*,王远亮1,*
(1.湖南农业大学食品科技学院,湖南长沙 410128;2.中国科学院微生物研究所,微生物资源前期开发国家重点实验室,北京 100101)
通过构建元基因组16S rRNA文库和Fosmid文库,对富含纤维素、半纤维素等大分子有机物的湖南沅江明星麻厂碱性沤麻废水处理池(pH9.35)微生物多样性进行了分析和新的工业用酶基因筛选,发现该碱性处理池原核微生物主要以细菌为主,包括厚壁菌门、变形菌门、拟杆菌门、绿菌门等四个细菌类群,其中变形菌门弯曲杆菌科硫磺单胞菌属细菌和腐败希瓦氏菌为优势菌群,其次为拟杆菌门和厚壁菌门。该环境古菌的生物量和丰度较少,以广古菌门马氏甲烷八叠球菌为优势类群。并且从该碱性池元基因组Fosmid文库中筛选出包括淀粉酶(36个)、脂肪酶(9个)、蛋白酶(4个)、纤维素酶(4个)在内的一系列食品和工业用酶活性克隆。为进一步开发和利用该碱性处理池微生物基因资源奠定了基础。
沤麻废水处理,原核微生物多样性,16S rRNA文库,碱性酶
极端微生物是最适合生活在极端环境中的微生物的总称,包括嗜热、嗜冷、嗜酸、嗜压、嗜金、抗辐射和极端厌氧等多种类型。嗜碱微生物是指那些最适生长pH为8.0以上,通常在pH9~10之间生长的微生物[1]。嗜碱微生物在许多自然和人工环境中都存在,天然环境如天然碱湖、碱性沙漠等,人工环境如水泥制造、印染、造纸、食品加工等工业过程排出的碱性废水和碱性废渣等。嗜碱细菌可以产生碱性胞外酶,具有高pH下稳定的特点,如碱性纤维素酶、果胶酶、蛋白酶、脂肪酶等,可用于洗涤剂添加剂、纺织、造纸、制革工业等。
亚麻是一种重要的韧皮纤维作物,其纤维具有耐用、卫生和附加值高等特点,是一种环保型的纤维原料,可加工成各种高档服装、装饰用品和军用、航空、消防、渔业、医疗等产业用布。亚麻纤维存在于亚麻原茎的韧皮部,为了制取亚麻中的可纺纤维,必须破坏纤维束与周围组织的连接程度,使得韧皮部与木质部易于分离,同时又较少破坏连接单纤维之间的胶质,这个过程被称为亚麻原茎脱胶。生物脱胶是目前采用的最广泛的方法,原料经过温水沤制[2-5],通过果胶分解微生物[6],把木质素和纤维素进行分离,同时麻茎中的各种有机物也扩散到水中,脱胶分离过程中产生高浓度有机废水。废水的处理往往采用厌氧生物处理法,污水中的有机物经多种微生物的共同作用,被最终转化为甲烷、二氧化碳、硫化氢和氨。在沤麻废水厌氧处理的过程中产酸是主导的反应,需要投加碳酸盐来控制酸碱度。从而会导致沤麻废水厌氧处理池进口的pH较高。
湖南沅江明星麻厂是具有几十年沤麻历史的企业,本研究自该企业沤麻废水处理池中采样,采集到pH9.35的碱性厌氧污泥,从中提取微生物总DNA,构建了16S rRNA基因组文库和元基因组Fosmid文库,分析了其中细菌和古菌多样性,并从元基因组Fosmid文库中筛选到纤维素酶、淀粉酶、脂肪酶、蛋白酶等具有工业应用价值的产酶活性克隆。
1 材料与方法
1.1 材料与仪器
2012年4月,样品采自湖南沅江明星麻厂沤麻废水处理池。样品pH为9.35。采集碱性厌氧沤麻废水处理池下层泥水混合物样品,置于无菌离心管,密封,运回实验室后-80 ℃保存。
土壤DNA提取试剂盒 MO BIO公司的UltraCleanTMMega Soil DNA Isolation Kit;Taq 酶、DNA纯化试剂盒 TIANGEN公司;T4连接酶 Promega公司;大肠杆菌EscherichiacoliDH5a 感受态细胞 北京全式金公司;酵母提取物、胰蛋白胨 Oxoid公司;其他生化试剂均为国产分析纯。
元基因组文库活性筛选培养基:基础LB培养基中加入相应底物:0.2%可溶性淀粉(淀粉酶筛选培养基);1.6%脱脂奶粉(蛋白酶筛选培养基);1%三丁酸甘油酯(脂肪酶筛选培养基);0.2% CMC-Na(纤维素酶筛选培养基);0.5%木聚糖(木聚糖酶筛选培养基);0.2%果胶(果胶酶筛选培养基)。木聚糖酶筛选培养基、蛋白酶筛选培养基8磅压力下20 min灭菌,其余培养基均15磅压力下高压蒸汽灭菌30 min。
元基因组文库活性筛选试剂:卢格氏碘液:22 g KI,11 g I2,溶于500 mL水中,得到5倍的储存液;DNS溶液:称取酒石酸钾钠182.0 g,溶于500 mL蒸馏水中,加热(不超过50 ℃),于热溶液中依次加入3,5-二硝基水杨酸6.3 g,NaOH 21.0 g,苯酚5.0 g,无水亚硫酸钠5.0 g,搅拌至溶解完全,冷却后用蒸馏水定容至1000 mL,过滤后避光保存;刚果红溶液:1 g刚果红溶于1 L水中,得到0.1%的刚果红染液。
1.2 样品总DNA的提取以及16S rRNA基因片段的PCR扩增
1.2.1 样品总DNA的提取 使用MO BIO公司的UltracleanTMMega Soil DNA Isolation Kit试剂盒提取碱性池元基因组DNA。
1.2.2 微生物16S rRNA基因片段的特异性扩增 细菌通用引物27F:5′-AGAGTTTGATCMTGGCTCAG-3′和1492R:5′-ACGGCTACCTTGTTACGACT-3′;古菌通用引物A571F:5′-GCYTAAAGSRICCGTAGC-3′和UA1204R:5′-TTMGGGGCATRCIKACCT-3′(M=C/A,R=A/g,Y=C/T,S=G/C)。PCR扩增程序为:95 ℃ 5 min;95 ℃ 1 min,50 ℃ 1 min,72 ℃ 1 min 30 s,30个循环;72 ℃ 10 min;4 ℃保存。PCR产物经1%琼脂糖凝胶电泳检测,并经DNA纯化试剂盒纯化。
1.3 16S rRNA基因克隆文库的构建
宋彬分析认为,目前新三板企业IPO持续升温。“新三板市场流动性差、估值低的局面未改变之前,挂牌企业转由IPO登陆A股的趋势将不会发生改变。”随着IPO的提速,新三板企业申请IPO的数量已出现了较大的增长,由于IPO审核标准趋严,新三板企业IPO通过率有所下降。
纯化后的PCR产物连接pGEM-T载体,转化大肠杆菌DH5α感受态细胞,涂布于含氨苄青霉素(50 mg/L)的IPTG/X-gal 平板上37 ℃培养过夜。挑取白斑菌落提取质粒,利用质粒酶切法进行筛选,对阳性克隆的插入片段进行测序。
1.4 基于16S rRNA基因片段的微生物多样性分析
使用DECIPHER http://decipher.cee.wisc.edu/FindChimeras.html分析并去除嵌合序列。将得到的有效序列用Sequence-Alingnment软件进行排序并获得相似性矩阵,相似性≥97%的序列被认为是相同的分类单元(OTU,operational taxonomic unit)并用其中一个序列作为代表[7],对每一个代表序列进行BLAST(http://blast. ncbi. nlm. nih.gov/Blast. cgi)相似性比对[8],根据比对结果选取参照序列,用Clustal X 2.1进行排序,Mega 4软件构建系统发育树(Jukes-Cantor参数模型计算进化距离,邻接法Neighbor-Joining构建系统进化树)。
1.5 碱性池元基因组Fosmid质粒文库的构建
根据试剂盒说明书进行文库的构建,采用DNA末端平滑化试剂盒对片段化的DNA片段进行凝胶电泳分析,切胶回收5~10 kb的片段,连接到pCC1FOS载体上,转化入感受态细胞E.coliDH10B中,涂布于含氯霉素的LB培养基平板,挑取克隆置于96孔板中,-20 ℃保存,用于后续文库的筛选。
1.6 水解酶的筛选
使用Qfill3分装含合适抗生素的新鲜LB培养基于96孔板,每孔150 μL。将保存冰箱的文库于4 ℃环境中解冻,使用全自动菌落复制点膜系统Qpix2将保存在384孔板的文库转移到96孔板,37 ℃、700 r/min过夜培养进行活化。通过影印方法复制到筛选培养基中,37 ℃倒置过夜培养。然后按照下列方法进行筛选:蛋白酶、脂肪酶筛选平板可以直接观察透明圈,淀粉酶筛选平板需经碘液染色后观察透明圈;纤维素酶和果胶酶筛选平板需经刚果红染色,1 mol/L NaCl脱色后观察透明圈;木聚糖酶阳性克隆筛选方法:在含0.5%木聚糖的液体LB培养基过夜培养,加入等体积的DNS溶液显色,阳性克隆中的木聚糖酶可水解培养基中木聚糖为还原糖,与DNS反应显色。
2 结果与分析
2.1 碱性处理池微生物元基因组DNA的提取与原核微生物16S rRNA基因扩增
10000 r/min离心采集样品,去除上清,剩泥土样,可得到病毒,真细菌,古细菌等微生物。使用MO BIO公司的UltraClean® Mega Soil DNA Isolation Kit对碱性污泥样品进行DNA的直接提取。从每克泥样中可提取约0.2~0.3 μg的DNA,电泳结果表明提取的DNA片段纯度较高、大于10kb。将DNA样品分别采用细菌和古菌16S rRNA基因通用引物进行PCR扩增,如图所示:细菌的扩增产物为1500bp左右的单一目的条带,古菌的扩增产物为650bp左右的单一条带,说明DNA样品纯度较好且PCR扩增的特异性良好,满足建库要求。湖南沅江明星麻厂沤麻废水处理池已闲置多年,其中的污泥有机质含量较高且长期处于厌氧碱性环境下,已经滋生出许多微生物。从基因组提取的结果来看,其中的生物量尚可,并且该土壤DNA提取试剂盒有效去除了其中的腐殖质和其他污染物,得到的DNA可以不经过后续的纯化直接用于微生物16S rRNA扩增。并且原位样品较高的pH(pH9.35)并未影响该试剂盒对总DNA的提取效率。此外,以相同量的起始模板进行16S rRNA扩增后,得到的细菌PCR产物的浓度明显比古菌PCR产物的浓度要高。表明该环境细菌的含量比古菌高。与许多碱性环境类似。

图1 沤麻处理池污泥样品总DNA电泳检测Fig.1 Total DNA of yuanjiang Alkaline pool注:泳道1:DNA Marker;泳道2、3、4:湖南沅江麻厂沤麻废水处理池碱性厌氧污泥元基因组总DNA。
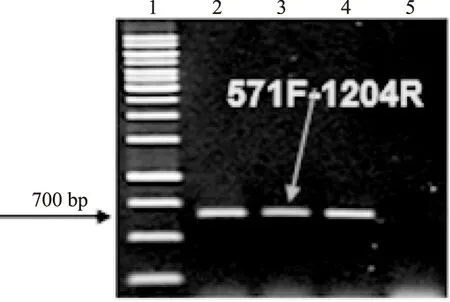
图2 古菌16S rRNA基因PCR扩增产物Fig.2 PCR amplification of 16S rRNA genes.注:泳道1:1kb DNA marker.;泳道2、3、4:湖南沅江麻厂沤麻废水处理池碱性厌氧污泥元基因组总DNA古菌16S rRNA PCR扩增产物;泳道5:阴性对照,以水为模板的古菌16S rRNA PCR扩增产物。
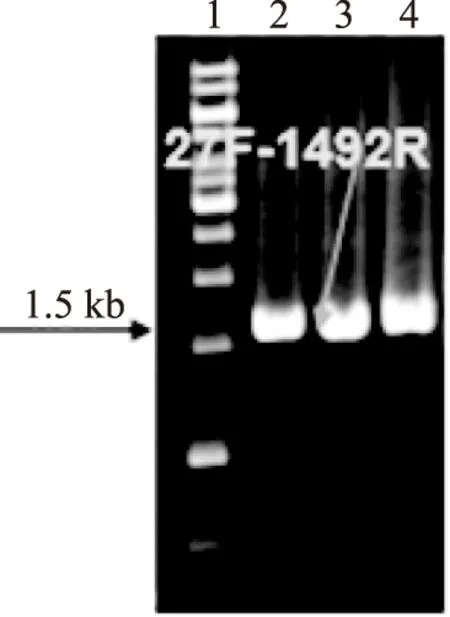
图3 细菌16S rRNA基因PCR扩增产物Fig.3 PCR amplification of 16S rRNA genes注:泳道1:1kb DNA marker;泳道2、3、4:湖南沅江麻厂欧麻废水处理池碱性厌氧污泥元基因组总DNA细菌16S rRNA PCR扩增产物。
纯化后的PCR扩增产物通过连接转化分别构建细菌和古菌16S rRNA基因克隆文库,从文库中各随机挑取96个阳性克隆测序并保存。得到细菌16S rRNA基因序列70条,古菌16S rRNA基因序列34条,将这104条序列运用DECIPHER http://decipher.cee.wisc.edu/FindChimeras.html程序进行嵌合体检验,发现其中14条为嵌合序列剔除,其余的90条用于后续分析。
2.3 基于16S rRNA基因序列的原核微生物多样性分析
通过序列之间的相似性比对发现,得到的63条细菌的序列可归属为13个不同的OTU,27条古菌序列可归属为4个不同的OTU。将代表13个细菌和4个古菌OTU的16S rRNA基因序列与GenBank数据库中的序列进行BLASTn相似性比对,结果见表1,表2。由表1可见,沤麻废水处理池碱性厌氧污泥的细菌优势克隆为BacA1(24条原始序列)和BacB1(11条原始序列)所代表微生物。其中与BacA1最相似的是耐碱硫磺单胞菌CampylobacteraceaebacteriumHTRB-L1(GQ863490),它们的相似性为98%;与BacB1最相似细菌为腐败希瓦式菌ShewanellaputrefaciensCN-32 strain(NR_074817),相似性也为98%。表明沅江碱性池的优势细菌大多为已知微生物,只含有少量的未培养细菌。并且大多数细菌都是碱性或者厌氧环境来源的,如盐碱湖、碱性污泥、地下水、油田等。古菌16S rRNA基因序列的相似性比对结果(表2)表明,该碱性处理池的优势克隆为产甲烷古菌,其中甲烷八叠球菌Methanosarcinamazei占据绝大部分(21条),另外还含有未培养古菌Methanomicrobiaarchaeon(AB742129)(1条)、甲烷螺菌属古菌Methanospirillumhungatei(AB517987.1)(1条)。
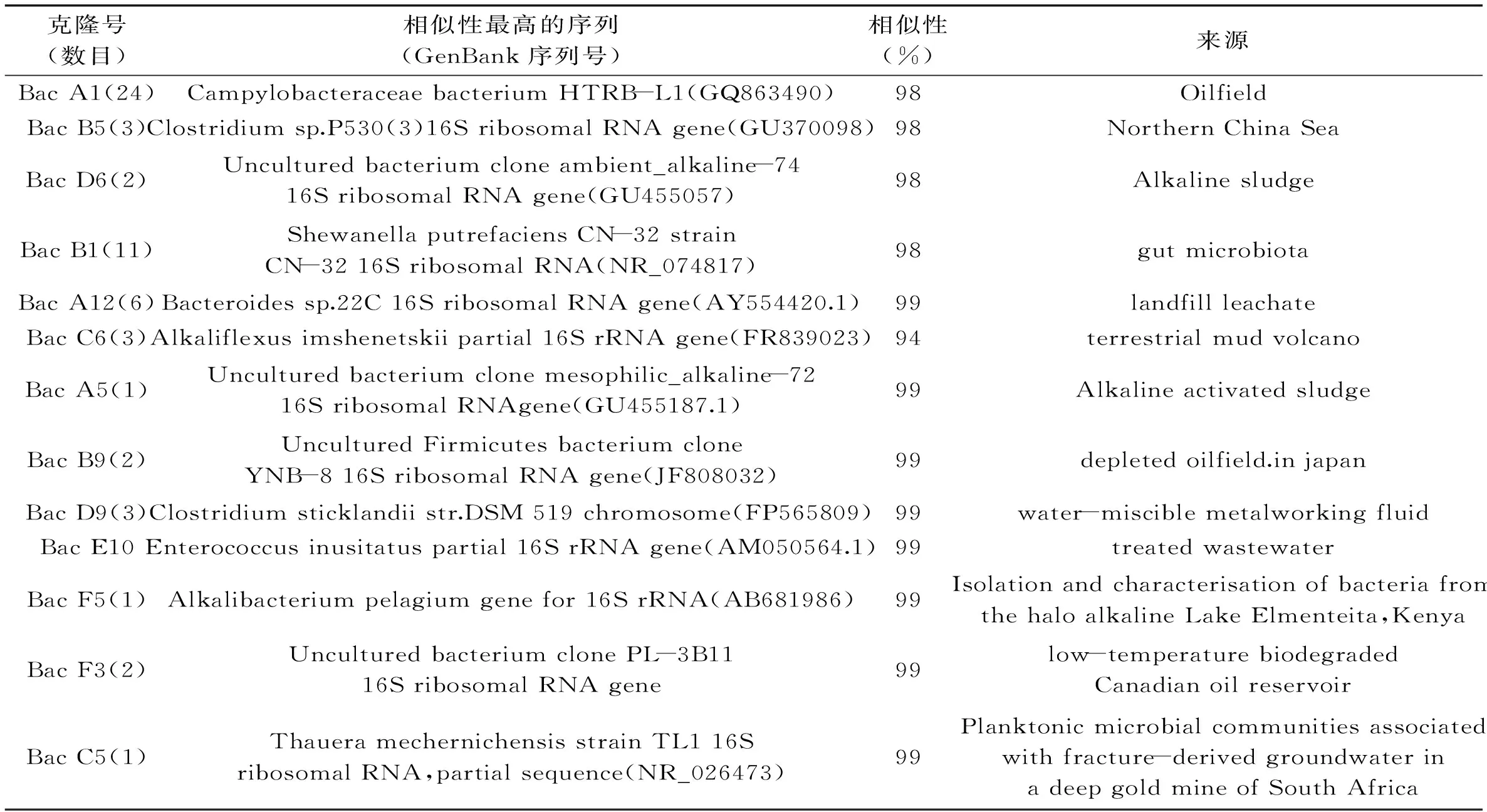
表1 沤麻废水处理池碱性厌氧污泥细菌16S rRNA基因序列BLASTn相似性分析

表2 沤麻废水处理池碱性厌氧污泥古菌16S rRNA基因序列BLASTn相似性分析
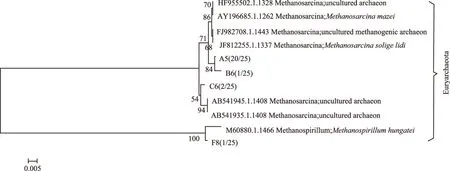
图4 基于16S rRNA基因序列的沤麻废水处理池碱性厌氧污泥古菌系统发育分析Fig.4 Phylogenetic analysis of archaea in Yuanjiang alkaline pool based on the 16S rRNA gene sequences
运用Mega4软件构建的湖南沅江明星麻厂碱性沤麻废水处理池原核微生物16S rRNA基因系统发育树见图4和图5。根据聚类树分析该碱性池微生物的类群归属可见:该碱性池的细菌多样性相对丰富,主要包括以下类群:变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)以及拟杆菌门(Bacteroidetes)的细菌以及广古菌门(Euryarchaeota)的古菌。其中细菌的优势菌种BacA1和BacB1与Sulfuropirillumalkalitolerans和Shewanellaputrefaciens的相似性分别为98%和98%。

图5 基于16S rRNA基因序列的沤麻废水处理池碱性厌氧污泥细菌系统发育分析Fig.5 Phylogenetic analysis of bacteria in Yuanjiang alkaline pool based on the 16S rRNA gene sequences.
2.4 碱性沤麻废水处理池元基因组Fosmid文库构建
本研究成功构建了该环境样品的元基因组Fosmid文库,平均插入片段20~30 ku,共包含19200个克隆。随机挑取27个克隆进行酶切鉴定,如图6所示。绝大部分都有插入片段。随机挑取20个阳性转化子,送测序,获得15条序列,序列分析结果表明,其中两株与人肠道微生物相似性较高,两株与瘤胃微生物相似性较高,一株与鸡肠道微生物相似性较高,一株与海洋细菌相似性较高,一株与人肠道来源的未培养微生物相似,其余8株为新的序列,初步鉴定多样性丰富,有利于后续功能基因的筛选。
2.5 Fosmid文库中水解酶的筛选
对湖南沅江明星麻厂沤麻废水处理池碱性厌氧污泥所构建的元基因组Fosmid文库进行功能酶基因筛选。总计筛选了7680个转化子,共筛选了6种酶基因,包括淀粉酶、纤维素酶、蛋白酶、脂肪酶、果胶酶和木聚糖酶。图7中,1号为淀粉酶活性筛选平板,淀粉酶活性克隆子产生的淀粉酶降解了周围的淀粉从而无法与碘液显色而出现了明显的透明圈;2号为纤维素酶活性筛选平板,纤维素酶活性克隆子产生的纤维素酶将周围的羧甲基纤维素钠分解产生单糖和低聚糖,0.1%刚果红染色,经过1 mol/L NaCl脱色后产生略带黄色的透明圈;3号为脂肪酶活性筛选平板,脂肪酶活性克隆子产生的脂肪酸与罗丹明B反应在紫外下发荧光;4号为木聚糖活性筛选培养基,在96孔板中每孔注入100 μL含0.5%的木聚糖,具有木聚糖酶活性的克隆子降解木聚糖产生还原糖,与等体积DNS在65 ℃条件下显色1 h后显色。初筛获得99个淀粉酶阳性克隆,24个脂肪酶阳性克隆,7个蛋白酶阳性克隆,4个木聚糖酶阳性克隆,11个纤维素酶阳性克隆,3个果胶酶阳性克隆。经过复筛,确定具有酶活性的阳性克隆包括:淀粉酶阳性克隆36个,脂肪酶阳性克隆9个,蛋白酶阳性克隆4个,纤维素酶阳性克隆4个。

表3 沤麻废水处理池元基因组Fosmid 文库大分子有机物降解阳性转化子

图6 元基因组Fosmid文库克隆酶切鉴定Fig.6 Restriction endonulease digestion of Fosmid library clones注:泳道1:λDNA HindⅢ酶切片段(23,9.6,6.6,2.3,2.0 kb);泳道2~27:元基因组Fosmid文库转化子Fosmid质粒HindⅢ酶切片段(pCC1Fos大小为8139 bp)脉冲场凝胶电泳检测。

图7 沤麻废水处理池元基因组Fosmid文库大分子有机物水解阳性转化子筛选Fig.7 Positive transformants with macromolecular organics hydrolysis activity screening in the Fosmid library of flax retting waste water microbes注:1.淀粉水解阳性转化子复筛(0.2%可溶性淀粉);2.纤维素水解阳性转化子复筛(0.2% CMC-Na);3.蛋白质水解阳性转化子复筛(1.6%脱脂奶粉);4.木聚糖水解阳性转化子初筛(0.5%木聚糖)。
3 结论
该沤麻废水处理池泥水混合物的pH达到了9.35,通过16S rRNA序列分析表明,其中包含变形菌门、厚壁菌门以及拟杆菌门的细菌以及广古菌门的古菌。
其中细菌的优势菌种BacA1和BacB1与Sulfuropirillumalkalitolerans和Shewanellaputrefaciens的相似性均为98%。另一个优势细菌BacB1与Shewanellaputrefaciens以及Shewanellahafniensis相似性均为98%。Sulfurospirillumalkalitolerans属于ε-变形菌门弯曲杆菌科的细菌,为严格厌氧细菌,可以利用甲酸盐、氢气、乳酸盐、丙酮酸盐和延胡索酸盐作为电子供体,硫代硫酸盐、硫、硝酸盐、亚硝酸盐、砷酸盐、延胡索酸盐为电子受体。它本身是个耐碱菌,可以在pH7.1~9.7下生长,最适pH为8.5,盐度范围为0.6~1.5 mol/L Na+,最适为0.6 mol/L。在pH9下的最高生长温度为41 ℃。在胞内以硝酸盐为电子受体的情况下可以氧化硫化物为硫元素[9]。另一个优势细菌BacB1与Shewanellaputrefaciens[10]以及Shewanellahafniensis[11]相似性均为98%。这两种细菌为兼性厌氧菌,耐冷产硫化氢细菌,在无氧的情况下能够利用十种以上的电子受体包括硝酸盐、亚硝酸盐、丙酮酸盐、硫代硫酸盐、硫元素、氧化三甲胺、四价锰以及三价铁。希瓦氏菌属的细菌在淡水、海水以及鱼类都能分离得到。优势古菌甲烷八叠球菌Methanosarcinamazei[12]为厌氧产甲烷古菌。可以利用H2/CO2、醋酸盐、甲醇、甲胺、三甲胺为碳源产生甲烷。在有机污染物厌氧发酵环境广泛存在,是厌氧沼气池中的重要功能菌群。而在pH9的环境中,该古菌的存在鲜有报道,是否在原位发挥功能需要进一步的验证。
对碱性池Fosmid文库进行活性酶基因的筛选,总计筛选了7680个克隆子,共筛选了6种酶:淀粉酶、纤维素酶、果胶酶、蛋白酶、脂肪酶、木聚糖酶。并对其做了复筛,最后的复筛结果为:有淀粉酶活性的36个(强阳性4个)、脂肪酶9个(强阳性2个)、蛋白酶4个(强阳性1个)、纤维素4个(强阳性1个)。表明该环境大分子有机质降解基因含量相对丰富,并且由于其独特的碱性环境,筛选到的新酶可能同时具有嗜碱或耐碱的特性,为后续的工业应用提供了良好的基因资源。目前正在对强阳性的克隆进行测序、拼接和注释,以期获得这些新酶基因。对于具有优良性质的酶基因可以进行后续的克隆表达以及酶学性质的研究,以期作为食品添加剂、饲料和工业用酶制剂等。
[1]Sarethy IP,Saxena Y,Kapoor A,et al. Alkaliphilic bacteria:applications in industrial biotechnology[J]. Journal of industrial microbiology & biotechnology,2011,38:769-90.
[2]Akin DE,Foulk JA,Dodd RB,et al. Enzyme-retting of flax and characterization of processed fibers[J]. Journal of biotechnology,2001,89:193-203.
[3]Valladares Juarez AG,Rost G,Heitmann U,et al. Development of a biotechnological process for the production of high quality linen fibers[J]. Bioprocess and Biosystems Engineering,2011,34:913-21.
[4]Di Candilo M,Bonatti PM,Guidetti C,et al. Effects of selected pectinolytic bacterial strains on water-retting of hemp and fibre properties[J]. Journal of Applied Microbiology,2010,108:194-203.
[5]Yadav S,Yadav PK,Yadav D,et al. Purification and characterization of pectin lyase produced by Aspergillus terricola and its application in retting of natural fibers[J]. Applied Biochemistry and Biotechnology,2009,159:270-83.
[6]Kashyap DR,Vohra PK,Chopra S,et al. Applications of pectinases in the commercial sector:a review[J]. Bioresource Technology,2001,77:215-27.
[7]McCaig AE,Glover LA,Prosser JI. Molecular analysis of bacterial community structure and diversity in unimproved and improved upland grass pastures[J]. Applied and Environmental Microbiology,1999,65:1721-30.
[8]Marteinsson VT,Hauksdottir S,Hobel CF,et al. Phylogenetic diversity analysis of subterranean hot springs in Iceland[J]. Applied and Environmental Microbiology,2001,67:4242-8.
[9]Sorokin DY,Tourova TP,Muyzer G. Isolation and characterization of two novel alkalitolerant sulfidogens from a Thiopaq bioreactor,Desulfonatronum alkalitolerans sp. nov.,and Sulfurospirillum alkalitolerans sp. nov[J]. Extremophiles:life under extreme conditions,2013,17:535-43.
[10]Venkateswaran K,Moser DP,Dollhopf ME,et al. Polyphasic taxonomy of the genus Shewanella and description of Shewanella oneidensis sp. nov[J]. International Journal of Systematic Bacteriology,1999,49 Pt 2:705-24.
[11]Satomi M,Vogel BF,Gram L,et al. Shewanella hafniensis sp. nov. and Shewanella morhuae sp. nov.,isolated from marine fish of the Baltic Sea[J]. International Journal of Systematic and Evolutionary Microbiology,2006,56:243-9.
[12]Simankova MV,Kotsyurbenko OR,Lueders T,et al. Isolation and characterization of new strains of methanogens from cold terrestrial habitats[J]. Systematic and Applied Microbiology,2003,26:312-8.
Microbial diversity analysis of alkaline flax retting wastewater and enzyme activity screen of Fosmid library clones
DONG Liang1,YU Qin-li2,DONG Zhi-yang2,WANG Li2,*,WANG Yuan-liang1,*
(1.Institute of Food Science and Technology,Hun an Agricultural University,Changsha 410128,China;2.State Kay Laboratory of Microbial Resources,Institute of Microbiology,Chinese Academy of Sciences,Beijing 100101,China)
The microbial samples from alkaline sludge of retting waste water(pH9.35)which were riched in cellulose and hemicelluloses were collected. DNA was isolated and 16S rRNA genes were amplified. The bacterias in the alkaline anaerobic sludge contained 13 OTUs belong to Proteobacteria,Firmicute and Bacteriodetes. The most abundant bacterias wereSulfuropirillumalkalitoleransandShewanellahafniensis. The archaeas in the alkaline anaerobic sludge contained 3 OTUs belong to Euryarchaeota. The most abundant one wasMethanosarcinamazei. Fosmid library was constructed and macromolecular organics hydrolysis activities include cellulose,hemicelluloses,starch,protein and lipid hydrolyze activities were screened. After two runs of screening,36 clones contain amylase,4 clones with cellulose hydrolysis activity,4 clones contain proteinase and 9 clones contain lipase were obtained. It is very helpful to further exploit and use these gene resources and enzymes in food industry from this alkaline envioronment.
flax retting wastewater;prokaryotic microbial diversity;16S rRNA library;alkaline enzymes
2015-03-20
董亮(1986-),男,硕士研究生,研究方向:生物化学与食品微生物学,E-mail:kunkunmumu2@163.com。
*通讯作者:王丽(1982-),女,博士,助理研究员,研究方向:生物化学与分子生物学,E-mail:wangli07@im.ac.cn。 王远亮(1977-),男,博士,教授,研究方向:食品微生物,E-mail:yuanliangw@gmail.com。
国家自然科学基金项目(青年基金31300007)。
TS201.3
A
1002-0306(2015)21-0167-07
10.13386/j.issn1002-0306.2015.21.026
