利用宏基因组技术发掘新木聚糖酶基因
2015-03-30王佳堃刘建新
罗 阳,何 波,王佳堃,刘建新
(浙江大学奶业科学研究所,动物分子营养学教育部重点实验室,浙江杭州310058)
利用宏基因组技术发掘新木聚糖酶基因
罗 阳,何 波,王佳堃*,刘建新
(浙江大学奶业科学研究所,动物分子营养学教育部重点实验室,浙江杭州310058)
木聚糖酶将日粮中的木聚糖降解成低聚糖。低聚糖及其厌氧发酵产物、短链脂肪酸可促进消化道发育,维持消化道微生物稳态。发掘新高效的木聚糖酶一直是酶工程研究的热点。宏基因组技术为此研究提供了保障。BAC和Fosmid宏基因组文库承载的信息量大,但阳性克隆率低,加大了分析工作量。木聚糖酶功能域由保守区和可变区嵌合构成,这为利用功能域基因研究木聚糖酶基因的多样性及其与微生物菌群的关系提供了条件。为此,本文在总结纯培养技术优缺点的基础上,重点归纳了BAC和Fosmid宏基因组文库及木聚糖酶功能域基因文库在发掘新木聚糖酶基因的应用。
木聚糖酶基因;宏基因组技术;纯培养技术
木聚糖酶是一类能将木聚糖水解为低聚木糖和木糖的复合酶,主要包括β-1,4内切木聚糖酶(endo-1,4-β-xylanase,E.C.3.2.1.8)、β-1,4外切木聚糖酶(β-1,4-D-extraxylanase,E.C 3.2.1.92)和β-木糖苷酶(β-xylosidase,E.C.3.2.1.37)等。依据底物特异性的不同,木聚糖酶可划分为糖苷水解酶(glycoside hydrolase,GH)5、7、8、10、11和43家族[1],其中以GH10和GH11为主。木聚糖酶已广泛应用于饲料、造纸、食品和生物能源工业。由于2个家族都具有内切功能,水解产物以低聚寡糖为主[2],因此近年来研究发现,木聚糖酶在促进肠道微生物发酵,维持消化道结构的稳态方面具有重要作用。为此,发掘新型木聚糖酶基因再度成为研究热点。
1 木聚糖酶基因的分布
木聚糖酶基因在自然界中分布广泛,存在于细菌、古细菌和真菌体内,主要以细菌门类为主,真菌次之,如芽孢杆菌(Bacillus)[3-4]、放线菌(Actinoplanes)[5]、链霉菌(Streptomyces)[6]等。含有该基因的微生物在土壤、水体环境(包括海洋、江河、沼泽等)、半纤维素分解液(生活垃圾、工业发酵液)以及有机体的消化道内均有分布,在自然界中扮演着“分解者”的角色。
动物日粮中的半纤维素成分不能被自身分泌的酶类降解,必须依靠微生物发酵产生的木聚糖酶水解成可吸收的成分[7]。发酵模式的差异直接影响到微生物的分布和功能。粗饲料是反刍动物最主要的饲料来源,各种纤维物质的降解是瘤胃中发生的最重要的消化过程。反刍动物之所以能对纤维物质具有利用优势,主要依赖于栖息在瘤胃内的大量具有纤维分解能力的微生物,因此瘤胃也成为纤维分解酶的主要发掘源。瘤胃内能够编码木聚糖酶基因的微生物主要来自放线菌门(Actinobacteria)[8-10]、拟杆菌门(Bacteroidetes)[11]、厚壁菌门(Firmicutes)[12]和纤维杆菌门(Fibrobacteres)[13]。其中放线菌门代表菌种有链霉菌(Streptomyces);拟杆菌门主要集中于瘤胃普雷沃氏菌属(Prevotella);厚壁菌门代表菌种有溶纤维丁酸弧菌(Butyrivibrio fibrisolvens)、黄色瘤胃球菌 (Ruminococcus flavefaciens)、 白色瘤胃球 菌(Ruminococcus albus)、溶纤维真细菌(Eubacterium cellulosolvens)、 反 刍 兽 真 细 菌 (Eubacterium ruminantium)等;产琥珀酸丝状杆菌(Fibrobacter succinogenes) 和 肠 道 丝 状 杆 菌 (Fibrobacter intestinalis)是纤维杆菌门的主要成员。随着高通量测序技术的发展,越来越多的瘤胃木聚糖酶基因将被筛选和定位。
2 木聚糖酶基因的发掘
目前针对木聚糖酶基因的研究方法很多,主要分为纯培养技术和宏基因组技术。2种方法均成功发掘了上千种木聚糖酶基因,但各具优势的同时,也各存局限。
2.1 纯培养技术 纯培养技术是从自然界中分离和研究微生物最传统和直接的方法。随着研究的深入,通过不断地改善营养条件和培养环境,微生物的可培养性得到了提升。常见的培养策略有加富培养、混合培养、稀释培养和模拟自然培养等。越来越多的微生物从菌群中分离开来,并用于科学研究。具体筛选流程:经刚果红或雷马素亮蓝R-D木聚糖(RBB)染色进行功能筛选,分离得到具有水解木聚糖功能的纯菌;提取基因组DNA并分析16sDNA序列,定位到微生物的种属;然后对基因组进行测序,找到编码木聚糖酶的完整ORF(开放阅读框),或者经过酶切(亚克隆文库)进一步缩小该编码基因的范围,克隆出完整ORF,进行异源表达或定向改造,目前,纯培养技术已经从曲霉属真菌(Aspergillus)[14-16]、链霉菌属(Streptomyces)[17]等微生物中成功发掘到新木聚糖酶基因。
虽然纯培养技术一直是研究微生物的基石,但其单一的营养成分和培养条件还是与生境中微生物的多样性和广泛性的现实相违悖,导致极端环境中的微生物无法在人为模拟的环境下复苏。例如,一些水生细菌离不开藻类分泌的生长因子和维生素;动植物病原菌对寄主活性物质的依赖等都是传统纯培养法难以提供的。传统微生物学培养技术评估不同生境微生物可培养性的分析结果显示,海水中微生物的可培养性约为0.001%~0.1%,淡水约为0.25%,土壤约为0.3%,活性污泥为1%~15%[18]。由此可见,纯培养技术无法完整地反映自然界中所有微生物的种群,成为限制其研究微生物木聚糖酶基因多样性的一个瓶颈。
2.2 宏基因组技术 宏基因组技术改变了传统纯培养的理念,以生境中微生物的总DNA为研究对象,将其克隆到合适的载体,转化到宿主菌中构建BAC或Fosmid基因组文库(图1)。在发掘新基因和生物分子的前提下,利用生物信息学的方法,揭示基因之间、微生物之间、微生物与环境之间相互作用的规律,为微生物的研究拓展新的思路与方法,并从群落结构水平上全面认识微生物的生态特征和功能[19]。因此,宏基因组技术打破了传统纯培养技术的壁垒,为生境中不可培养微生物的研究提供了可靠的技术保障。
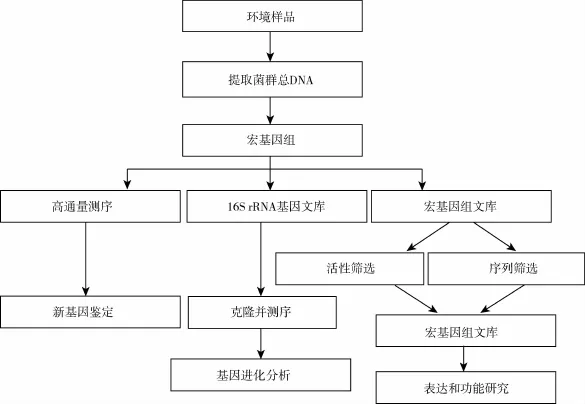
图 宏基因组学研究环境中新型基因流程示意图

表1 BAC文库和Fosmid文库中筛选的木聚糖酶基因
2.2.1 利用BAC和Fosmid宏基因组文库 宏基因组技术研究木聚糖酶基因的过程中,常构建的文库有BAC和Fosmid文库(表1)。其区别在于插入目的基因片段的大小、构建的载体和宿主等不同。BAC文库所用的载体为CopyControl pCC1BAC,插入的DNA平均片段为100 kb;Fosmid文库所用的载体为CopyControl pCC1FOSTM,插入的DNA平均片段为40 kb,因此BAC文库能够承载更多的微生物学信息,但Fosmid文库的构建更为简单,而且文库的信息量完全能满足木聚糖酶基因研究的需要,因此在该领域,Fosmid文库的应用更为广泛。
宏基因组文库给木聚糖酶基因的研究提供保障,但文库承载的信息量太大,库容量从400 Mb~10 G不等,而且阳性克隆率低,使得研究和分析工作量非常大。Xia等[20]构建了奶牛瘤胃宏基因组BAC文库,6 000多个克隆中筛选到了25个具有糖基水解酶的阳性克隆,阳性率为0.15%,包含GH5、GH8、GH9家族和纤维素酶的阳性克隆。Eun等[21]以猪粪便堆肥为研究对象,构建Fosmid文库,共获得12 380个克隆,其中有5个具有木聚糖酶活性的阳性克隆。Nimchua等[22]构建了白蚁肠道微生物的Fosmid文库,获得88 000个克隆,包括12个木聚糖酶和2个纤维素酶活性的阳性克隆。王佳堃等[23]以瘤胃微生物为研究对象,构建湖羊Fosmid文库,共获得12 704个克隆,经过刚果红染色,筛选到了18个具有水解木聚糖酶的阳性克隆,Yu等[24]利用同样的方法构建Fosmid文库,鸟枪法测序后获得3553个ORF,分析得到34个水解纤维素和52个编码木聚糖酶的克隆。
为了缩小新木聚糖酶基因的研究范围,需要对宏基因组文库中的阳性克隆建亚克隆文库。亚克隆分析是基于鸟枪法(Shotgun Strategy)构建外源DNA随机片段的重组质粒文库。把大片段的外源DNA通过合适的限制性核酸内切酶酶切或者采用机械性打断方法,整合成小片段DNA(2~8 kb)连接到合适的载体上,转化宿主菌中构建外源DNA随机片段重组质粒文库并筛选。通过这一过程的多次重复,得到木聚糖酶基因的阳性克隆,获得编码木聚糖酶的新基因。
2.2.2 木聚糖酶功能域基因文库 木聚糖酶功能域由保守区和可变区嵌合构成,这为利用功能域基因研究木聚糖基因的多样性及其和微生物菌群的关系提供了条件。GH10家族蛋白质一级结构的保守域由[W/Y]-D-W-D-V-[V/C/N]-N-E和[D/H]-[G/A/C]-[I/V/L]-G-[M/F/L/I]-Q-[S/G/M/C]-H[35]两部分组成,两个保守域之间嵌合了70个非保守氨基酸。GH11家族的蛋白质一级结构的两个保守域为S-Y-L-[C/S/ A]-[V/L]-Y-G-W和T-F-[V/L]-Q-[Y/F]-[W/F]-SV[36],保守域之间有55个非保守氨基酸。Rose等[37]针对功能域两端的保守区设计简并通用引物(X10-F和X10-R;X11-F和X11-R),Wang等[38-39]利用该引物构建功能域基因文库,发现天山冻土层微生物和山羊瘤胃微生物中富含木聚糖酶基因,并结合基因步移法(genome walking)扩增出全长ORF,在原核生物中表达后可产生较高的酶活性。
全长基因的扩增是依靠设计功能域远端引物、中间引物和近端引物,以及一个独立设计、退火温度较低的简并引物,进行3次热不对称巢式PCR(TAILPCR)反应使目的片段由非特异性向特异性转型,最终拼接得到全长的木聚糖酶基因。Luo等[40]、Zhou等[41-44]等利用保守区引物扩增功能域片段,结合基因步移法从鞘氨醇杆菌(Sphingobacterium sp.TN19)、链霉菌(Streptomyces sp.TN119)、耐酸嗜热真菌(Bispora sp) 中获得了新型木聚糖酶全长基因χyl11B、XynA119、XynA19、χynB19、χynB119、和XynGR40。
3 小 结
新基因的发掘,一直是木聚糖酶研究领域的一个方向,不断更新的生物技术为新木聚糖酶基因的研究提供了保障,尤其是二代测序技术的普及,使得木聚糖酶基因文库的研究更加快速、丰富和准确。而宏基因组技术在发掘更多新木聚糖酶基因的研究中发挥着不可替代的作用。因此,宏基因组技术与其它新的生物研究手段相结合,可以扩大木聚糖酶基因的研究范围,发掘到更多的新基因。
[1]Collins T,Gerday C,Feller G.Xylanases,xylanase families and extremophilic xylanases [J].FEMS Microbiol Rev,2005,29(1): 3-23.
[2] Sabini E,Wilson K S,Danielsen S,et al.Oligosaccharide binding to family 11 xylanases:both covalent intermediate and mutant product complexes display(2,5)B conformations at the active centre[J].Acta Crystallogr D,2001,57(9):1344-1347.
[3] Mamo G,Delgado O,Martinez A,et al.Cloning,sequence analysis,and expression of a gene encoding an endoxylanase from Bacillus halodurans S7[J].Mol Biotechnol,2006,33(2):149-159.
[4] Oakley A J,Heinrich T,Thompson C A,et al.Characterization of a family 11 xylanase from Bacillus subtillis B230 used for paper bleaching[J].Acta Crystallogr D,2003,59(Pt 4):627-636.
[5]Sriyapai T,Somyoonsap P,Matsui K,et al.Cloning of a thermostable xylanase from Actinomadura sp.S14 and its expression in Escherichia coli and Pichia pastoris[J].J Biosci Bioeng,2011,111 (5):528-536.
[6] Wang Y R,Zhang H L,He Y Z,et al.Characterization,gene cloning,and expression ofanovelxylanaseXYNB from Streptomyces olivaceoviridis A1[J].Aquaculture,2007,267(1-4):328-334.
[7] Flint H J,Bayer E A,Rincon M T,et al.Polysaccharide utilization by gut bacteria:potential for new insights from genomic analysis[J].Nat Revs Microbiol,2008,6(2):121-131.
[8] Kumar A,Gupta R,Shrivastava B,et al.Xylanase production from an alkalophilic actinomycete isolate Streptomyces sp.RCK-2010,its characterization and application in saccharification of second generation biomass[J].J Mol Catal B,2012,74(3-4):170-177.
[9] Li X,She Y,Sun B,et al.Purification and characterization of a cellulase-free,thermostable xylanase from Streptomyces rameus L2001 and its biobleaching effect on wheat straw pulp[J]. Biochem Eng J,2010,52(1):71-78.
[10]Liu Y,Deng Z,Tan H,et al.Characterization of cattle fecal Streptomyces strains converting cellulose and hemicelluloses into reducing sugars[J].Environ Sci Pollut Res Int,2014,21(9):6069-6075.
[11]Dodd D,Moon Y H,Swaminathan K,et al.Transcriptomic analyses of xylan degradation by Prevotella bryantii and insights into energy acquisition by xylanolytic bacteroidetes[J].J Biol Chem, 2010,285(39):30261-30273.
[12]Nyonyo T,Shinkai T,Mitsumori M.Improved culturability of cellulolytic rumen bacteria and phylogenetic diversity of culturable cellulolytic and xylanolytic bacteria newly isolated from the bovine rumen[J].FEMS Microbiol Ecol,2014,88(3):528-537.
[13]Ransom-Jones E,Jones D L,Mccarthy A J,et al.The Fibrobacteres:an important phylum ofcellulose-degrading bacteria[J].Microbial Ecol,2012,63(2):267-281.
[14]Hmida-Sayari A,Taktek S,Elghabbi F,et al.Biochemical characterization,cloning and molecular modeling of a detergent and organic solvent-stable family 11 xylanase from the newly isolated Aspergillus niger US368 strain[J].Process Biochem,2012, 47(12):1839-1847.
[15]Zhou C,Bai J,Deng S,et al.Cloning of a xylanase gene from Aspergillus usamii and its expression in Escherichia coli[J]. Bioresource Technol,2008,99(4):831-838.
[16]Chantasingh D,Pootanakit K,Champreda V,et al.Cloning, expression, and characterization of a xylanase 10 from Aspergillus terreus(BCC129)in Pichia pastoris[J].Protein Express PuriF,2006,46(1):143-149.
[17]Kim D Y,Han M K,Oh H W,et al.Catalytic properties of a GH10 endo-β-1,4-xylanase from Streptomyces thermocarboxydus HY-15 isolated from the gut of Eisenia fetida[J].J Mol Cataly B,2010,62(1):32-39.
[18]Amann R I,Ludwig W,Schleifer K H.Phylogenetic identification and in-situ detection of individual microbial-cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[19]Zengler K,Palsson B O.A road map for the development of community systems(CoSy)biology[J].Nat Rev Microbiol,2012, 10(5):366-372.
[20]Gong X,Gruninger R J,Qi M,et al.Cloning and identification of novel hydrolase genes from a dairy cow rumen metagenomic library and characterization of a cellulase gene[J].BMC Res Notes, 2012,5:566.
[21]Kwon E J,Jeong Y S,Kim Y H,et al.Construction of a metagenomic library from compost and screening of cellulaseand xylanase-positive clones[J].J Korean Soc Appl Bi,2010,53 (6):702-708.
[22]Nimchua T,Thongaram T,Uengwetwanit T,et al.Metagenomic analysis of novel lignocellulose-degrading enzymes from higher termite guts inhabiting microbes[J].J Microbiol Biotechnol,2012, 22(4):462-469.
[23]王佳堃,安培培,陈振明,等湖羊瘤胃微生物Fosmid文库的构建和分析[J].动物营养学报,2010,22(2):341-345.
[24]Wang L,Ayat H,Yu Z T,et al.Metagenomic insights into the carbohydrate-active Enzymes carried by the microorganisms adhering to Solid digesta in the rumen of cows[J].PloS One,2013, 8(9):1-11.
[25]Mientus M,Brady S,Angalov A,et al.The rmostable xylanase and β-glucanase derived from the metagenome ofthe avachinsky crater in kamchatka[J].Current Biotechnol,2013,2 (11):284-293.
[26]Jeong Y S,Na H B,Kim S K,et al.Characterization of xyn10J, a novel family 10 xylanase from a compost metagenomic library[J].Appl Biochem Biot,2012,166(5):1328-1339.
[27]Wang J,Sun Z,Zhou Y,et al.Screening of a xylanase clone from a fosmid library of rumen microbiota in Hu sheep[J]. Anim Biotechnol,2012,23(3):156-173.
[28]Sudo M,Sakka M,Kimura T, et al.Characterization of paenibacillus curdlanolyticus intracellular xylanase xyn10B encoded by the xyn10B gene[J].Biosci Biotech Bioch,2010,74 (11):2358-2360.
[29]Cheng F,Sheng J,Dong R,et al.Novel xylanase from a holstein cattle rumen metagenomic library and its application in xylooligosaccharide and ferulic Acid production from wheat straw[J].J Agric Food Chem,2012,60(51):12516-12524.
[30]王敏,陈富荣,张山,等.牦牛瘤胃元基因组文库中木聚糖酶基因的分析[J].微生物学报,2011,51(10):1364-1373.
[31]冯国栋,吕丹青,陈振明,等.一个新型湖羊瘤胃木聚糖酶基因的克隆和鉴定[J].科技通报,2010,26(5):345-349.
[32]Gong X,Gruniniger R J,Forster R J,et al.Biochemical analysis of a highly specific,pH stable xylanase gene identified from a bovine rumen-derived metagenomic library[J].Appl Microbiol Biot,2013,97(6):2423-2431.
[33]Han Q,Liu N,Robinson H,et al.Biochemical characterization and crystal structure of a GH10 xylanase from termite gut bacteria reveal a novel structural feature and significance of its bacterial Ig-like domain[J].Biotechnol Bioeng,2013,110(12): 3093-3103.
[34]Mai Z,Su H,Yang J,et al.Cloning and characterization of a novel GH44 family endoglucanase from mangrove soil metagenomic library[J].Biotechnol Lett,2014,36(8):1701-1709.
[35]Sunna A,Bergquist P L.A gene encoding a novel extremely thermostable 1,4-beta-xylanase isolated directly from an environmental DNA sample[J].Extremophiles:life under extreme conditions,2003,7(1):63-70.
[36]Morris D D,Gibbs M D,Chin C W,et al.Cloning of the xynB gene from Dictyoglomus thermophilum Rt46B.1 and action of the gene product on kraft pulp[J].App Environ Microb,1998,64(5): 1759-1765.
[37]Rose T.CODEHOP(COnsensus-DEgenerate Hybrid Oligonucleotide Primer)PCR primer design[J].Nucleic Acids Res,2003, 31(13):3763-3766.
[38]Wang G,Luo H,Meng K,et al.High genetic diversity and different distributions of glycosyl hydrolase family 10 and 11 xylanases in the goat rumen[J].PloS One,2011,6(2):1-10.
[39]Wang G,Wang Y,Yang P,et al.Molecular detection and diversity of xylanase genes in alpine tundra soil [J].Appl Microbiol Biotechnol,2010,87(4):1383-1393.
[40]Luo H,Wang Y,LiJ, etal.Cloning,expression and characterization of a novel acidic xylanase,XYL11B,from the acidophilic fungus Bispora sp.MEY-1[J].Enzyme Microb Tech, 2009,45(2):126-133.
[41]Zhou J,Huang H,Meng K,et al.Cloning of a new xylanase gene from Streptomyces sp.TN119 using a modified thermal asymmetric interlaced-PCR specific for GC-rich genes and biochemical characterization[J].Appl biochem Biot,2010,160(5): 1277-1292.
[42]Zhou J,Huang H,Meng K,et al.Molecular and biochemical characterization ofa novelxylanase from the symbiotic Sphingobacterium sp.TN19[J].Appl Microbiol Biot,2009,85(2): 323-333.
[43]ZhouJ,MengK,YangP,etal.Characterization ofa chromosomalsegmentshowingxylanolyticactivityfrom the symbiotic Sphingobacterium sp.TN19[J].World J Microbiol Biot, 2009,26(4):761-765.
[44]Zhou J,Shi P,Zhang R,et al.Symbiotic Streptomyces sp. TN119 GH 11 xylanase:a new pH-stable,protease-and SDS-resistant xylanase[J].J Industrial Microbiol Biot,2011,38(4):523-530.
Abstract:The oligosaccharides producted by xylanase from dietary hemicelluloses,and their microbial fermentation product,short-chain fatty acids,can promote growth of gut and maintain the balance of micro-environment.Therefore, development of new and efficient xylanase has been a hot research area in enzyme engineering.Metagenomic technology is a good tool for developing novel xylanase genes.There ismuchbiological information in the metagenomic library,such as BAC and fosmid,but thelow rate of positive cloningleads to a heavy analysis workload.The specific chimeric structure of full-lengthxylanase geneis consisted by conservative and variable domains.This provides opportunities to study diversity of xylanase genes and its relationship with microbiota.In this paper,we summarized the advantages and disadvantages of culture-dependent technology,and the application of BAC and fosmid metagenomic libraries and xylanase domains library in developing new xylanase gene.
Developing Novel Xylanase Genes with Metagenomic Technology
LUO Yang,HE Bo,WANG Jia-kun*,LIU Jian-xin
(The Research Institute of Dairy Science,ZhejiangUniversity,The Key Laboratory of Molecular Animal Nutrition, Ministry of Education,Zhejiang Hangzhou 310058,China)
xylanase;metagenomics;culture-dependent techniques
S816.7
A文献标识码:0258-7033(2015)11-0086-05
2014-10-23;
2014-12-15
科技部国际合作项目(2010DFA31040)
罗阳(1988-),男,土家族,湖南张家界人,硕士研究生,从事木聚糖酶基因开发的研究,E-mail:xinhelu509@163.com
*通讯作者:王佳堃,副教授,博士生导师,E-mail:jiakunwang@zju.edu.cn
