Opaque2转录因子对玉米 α-醇溶蛋白基因家族成员表达的影响
2015-03-26李国锋吕远大
李国锋, 葛 敏, 吕远大
(1.江苏省农业科学院六合基地,江苏 南京 210014;2.江苏省农业科学院农业生物技术研究所/江苏省农业生物学重点实验室,江苏 南京 210014)
玉米作为世界最大的种植作物之一,在国民经济中占据重要地位,全球对其需求量正在不断攀升。醇溶蛋白(Zein)是玉米最主要的储藏蛋白(占籽粒总蛋白的50%以上[1-2]),对玉米籽粒品质有重要影响。醇溶蛋白根据其溶解性、氨基酸组成以及其他物理性质可以分为α、β、γ和δ 4类,其中α-醇溶蛋白(α-zein)占总醇溶蛋白的60%,含量最为丰富。玉米α-醇溶蛋白富含谷氨酸、亮氨酸、脯氨酸和丙氨酸,但缺乏赖氨酸、色氨酸等几种人体和动物必需的氨基酸,且因聚集二硫键和糖基化的作用而难溶于水,在非反刍动物体内不易消化吸收,故其食用和营养价值不高[3-4]。Prasanna等[5]通过 SDS-PAGE电泳分析成熟籽粒中醇溶蛋白含量,发现几乎所有的优质蛋白玉米籽粒中醇溶蛋白含量较普通玉米显著较低,尤其是α-醇溶蛋白,因此降低α-醇溶蛋白含量一度成为提高玉米营养品质研究的重要目标之一。然而研究结果表明,过低的α-醇溶蛋白虽可提高胚乳蛋白质中赖氨酸和色氨酸的含量并大大改善玉米的营养品质,但同时引起玉米胚乳质地松软和产量无法提高等问题[6]。
α-醇溶蛋白由一个庞大的超级基因家族编码,根据序列相似性又分为4个基因亚族:z1A、z1B、z1C、z1D,其中z1A、z1B和z1D 3个基因亚族属于19 000 α-醇溶蛋白,z1C为22 000 α-醇溶蛋白[7-8]。Song等[6]利用BAC文库测序,发现在玉米B73自交系中19 000 α-醇溶蛋白基因家族组成了玉米基因组上最长的连续重复基因序列区域,19 000 α-醇溶蛋白基因家族中,z1A包含12个基因成员,z1B包含8个基因成员,z1D包含5个成员,它们分别位于3条染色体(第1、第4、第7条染色体)的4个不同基因组区域内。这25个基因成员DNA序列较为保守且基因转录方向基本一致(Head-to-tail orientation),但是其中只有12个基因具有完整的编码区域,另外13个基因由于突变等未知原因无法形成完整的转录本。Song等[8-9]利用BAC文库测序,基于比较基因组学分析,在 B73和 BSSS53自交系中,发现22 000 α-醇溶蛋白基因家族分别由15个和23个成员组成,它们位于第4号染色体的短臂上。z1C成员大致上是呈现串联阵列,基因转录方向完全一致。而在第4号染色体的短臂上距离着丝粒-20 cM另外一个区域内存在一个游离于串联阵列之外的z1C成员floury2。这些z1C成员在B73和BSSS53中分别只有7个和8个具有完整的编码区域。尽管α-醇溶蛋白超级基因家族的大小和复杂度在不同的自交系中有所不同,但是序列却高度保守。
Feng等[10]结合实时定量PCR和随机克隆测序的研究方法,对玉米自交系B73胚乳发育时期α-醇溶蛋白超基因家族(41个基因成员)的表达模式进行研究,结果表明该基因家族中仅18个基因表达,表达水平差异很大。该家族所有亚族(z1A、z1B、z1C、z1D)在授粉后10~34 d均表现出上下摆动不规则的表达模式,总体而言z1A具有最高表达量,然后表达量由高到低依次为z1C、z1D和z1B。亚族基因成员的表达模式也存在显著差异,但是,由于α-醇溶蛋白基因家族成员在编码序列存在高度的相似性,实时定量PCR对部分基因无法精确区分。二代测序技术如RNA-seq具有高敏感性和高通量等特征,可精确检测到基因家族中相似基因的不同转录本的单碱基差异。利用RNA-seq获得的高通量数据结合序列相似性比较和覆盖深度计算,可以对玉米α-醇溶蛋白基因家族成员基因表达进行高精度解析。
玉米中首个发现能调控α-醇溶蛋白的反式因子为Opaque2(O2),属于bZIP转录因子家族,O2基因在玉米胚乳中特异表达。O2突变体中α-醇溶蛋白基因的表达下调,导致籽粒中赖氨酸和色氨酸的含量增加,但也导致籽粒胚乳粉质呈现出不透明的表型[11-13]。基于上述原因该突变体广泛应用于优质蛋白玉米种质资源的创制研究,但是Opaque2对α-醇溶蛋白基因家族成员的表达是否均存在影响并没有得到很好的解析。
本研究拟根据α-醇溶蛋白基因家族成员的保守序列修订该基因家族在玉米B73染色体上的位置,并利用RNA-seq数据对玉米自交系B73胚乳中从授粉至成熟各时期编码α-醇溶蛋白的各个基因的表达模式进行分析,解析该基因家族成员的表达特性,并通过随机克隆Sanger测序法对结果进行验证,同时比较Opaque2野生型和突变型的编码α-醇溶蛋白基因家族成员的表达差异。
1 材料与方法
1.1 玉米α-醇溶蛋白基因的鉴定与分类
基与最新版(V3)玉米自交系B73参考基因组数据(http://www.maizegdb.org/),利用tBlastx方法以α-醇溶蛋白cDNAs(NCBI)为目标序列在玉米基因组中搜寻同源区域,并利用生物信息学方法获得同源区域对应基因的位置及核酸序列。然后利用ClustalX软件对玉米α-醇溶蛋白基因进行多重序列比对,并根据序列相似性进行人工校正分类。
1.2 玉米α-醇溶蛋白基因保守Motif分析
为了进一步解析α-醇溶蛋白基因的结构和保守性,我们利用MEME在线分析工具[14](http:// meme-suite.org/index.html)对本研究所鉴定的玉米α-醇溶蛋白基因的保守Motif进行分析。其中最大保守motif数目设置为10条,motif长度设置为6~50 bp。
1.3 玉米α-醇溶蛋白基因的表达谱分析及验证
为了进一步解析玉米α-醇溶蛋白基因表达模式,我们利用玉米自交系B73胚乳自授粉到成熟17个发育时期RNA-Seq数据[15],系统分析α-醇溶蛋白基因在玉米B73中的表达谱模式。RNA-Seq数据的处理及α-醇溶蛋白基因家族表达量FPKM值的获得均参照本实验室前期研究方法[16],最终利用MEV4.9软件绘制玉米 α-醇溶蛋白基因家族的Heatmap图,并分析表达模式。为验证本研究所利用RNA-Seq测序结果的可靠性,利用随机克隆Sanger测序法对结果进行了验证,比较2种测序方法中玉米B73授粉14 d后胚乳中α-醇溶蛋白基因相对表达量的一致性。
1.4 Opaque2对玉米α-醇溶蛋白基因表达的影响
利用 Li等[17]文章中野生型和突变体 O-paque2授粉后15 d籽粒的RNA-seq数据,并对其进行处理。最终整理出野生型和突变体中α-醇溶蛋白基因家族所有成员的相对表达量,比较分析转录因子Opaque2对玉米α-醇溶蛋白基因表达的影响。
2 结果与分析
2.1 玉米α-醇溶蛋白基因的确定及分类
利用tBlastx方法在玉米基因组中共比对搜索到39条α-醇溶蛋白基因序列。利用ClustalX软件对基因序列进行多重比对和人工校正,玉米α-醇溶蛋白基因分类结果见表1(参照前人研究结果对其命名),其中28条序列与目标序列相似度为100.00%,其余11条序列相似度高达97.75%以上,说明本研究比对结果可信度高。z1A亚族包含12个基因成员(Z19A1-1~Z19A1-9,Z19A2-1~Z19A2-3);z1B包含8个成员(Z19B1-1~Z19B1-6,Z19B2-1~Z19A2-2); z1D包含5个成员(Z19D1-1~Z19D1-3,Z19D2-1,Z19D3-1),3个亚族分别位于第4、第7和第1染色体上;z1C亚族14个成员位于第4染色体上,其中13个呈现串联阵列转录方向完全一致,仅AZS22-16游离于串联阵列之外,该基因为floury2。19 000 α-醇溶蛋白共25个(z1A、z1B和z1D),22 000 α-醇溶蛋白共14个(z1C)。α-醇溶蛋白超家族基因长度均值为872 bp,其中z1B亚族基因长度均值最长(1 507 bp),其余3个亚族(z1A、z1C和z1D)基因长度均值相近,约700 bp。39个α-醇溶蛋白基因家族中有30个可以对应到前人预测的基因ID上,9个基因还未预测出基因ID的表示为“-”。
2.2 玉米α-醇溶蛋白基因保守Motif分析
通过MEME在线分析工具对本研究所鉴定的39个α-醇溶蛋白基因的保守motif进行分析,结果如图1(图中α-醇溶蛋白基因家族成员按照比对E值大小排列)所示。图1显示,α-醇溶蛋白基因家族成员含有的保守motif数目和排列位置总体相似度高,尤其在各亚族成员间相似性非常高,说明该家族基因序列高度保守。10条motif长度为40~50 bp,其中motif 6含量最为丰富,在α-醇溶蛋白基因中存在2~4个重复,此外motif 9、motif 5和motif 1也存在重复。α-醇溶蛋白基因家族中AZS22-3、AZS22-11、AZS22-16、Z19D1-1、Z19A1-9基因所含motif少(1~4个),其余基因家族成员含有motif数目较为一致。

表1 玉米α-醇溶蛋白基因Table 1 α-zein gene family in maize

图1 玉米α-醇溶蛋白基因保守motif分析Fig.1 Conserved motif analyses of α-zein genes in maize
2.3 玉米籽粒发育时期α-醇溶蛋白基因表达谱分析
对玉米胚乳自授粉至成熟17个发育时期RNASeq数据进行预处理和统计分析后,获得了玉米α-醇溶蛋白基因对应表达量的FPKM值,根据FPKM值利用MEV4.9软件绘制出表达量的Heatmap图谱(图2)。玉米α-醇溶蛋白基因家族成员表达模式差异较大,Z19A1-9、Z19D1-1和AZS22-13这3个基因表达量最低,在图2中17个时期FPKM均值低于1,Z19A1-1、Z19A1-8、Z19B2-2、Z19D1-3、Z19D3-1和AZS22-11这6个基因表达量也很低,FPKM均值低于50,Z19A2-2表达量最高,FPKM均值为29 176。其中α-醇溶蛋白超家族4个亚族表达量 FPKM均值大小依次为 z1A (13 593)、z1B(4 910)、z1C(4 187)和z1D(2 589)。
玉米α-醇溶蛋白基因在胚乳中从授粉后10 d开始表达,授粉38 d后醇溶蛋白基因几乎不表达。α-醇溶蛋白z1A亚族约58%(7/12)的成员和z1B亚族约25%(2/8)的成员在授粉12~32 d后的胚乳中表达量持续较高,z1D亚族约40%(2/5)的成员在授粉后28 d的胚乳中出现最高表达量,z1C亚族约43%(6/14)的成员在授粉22~28 d后的胚乳中表现出最高的表达量。
为验证本研究所利用RNA-seq数据的可靠性,我们将α-醇溶蛋白基因家族成员在授粉14 d后Sanger测序与二代测序结果进行了比较。如表1所示,2种测序方法所得表达量数值非常一致,说明本研究分析所得α-醇溶蛋白基因的表达谱数据非常可信。

图2 玉米α-醇溶蛋白基因表达模式分析Fig.2 Expression patterns of α-zein genes in maize
2.4 Opaque2对玉米 α-醇溶蛋白基因表达的影响
根据野生型和突变体Opaque2授粉后15 d籽粒的RNA-seq数据,最终整理出野生型和突变体中α-醇溶蛋白基因相对表达量。如图3所示,突变体中z1C亚族成员相对表达量较野生型显著下调,z1A亚族成员表达量略有下调,但z1B和z1D亚族成员表达量没有显著的变化。以上结果说明Opaque2突变主要影响z1C基因亚族成员的表达,而对z1B和 z1D的表达影响不显著,转录因子Opaque2为z1C亚族基因表达所必须的反式作用因子。
3 讨论
本研究基于玉米v3版基因组数据共鉴定α-醇溶蛋白基因39个,其中19 000 α-醇溶蛋白家族成员25个,22 000 α-醇溶蛋白家族成员 14个。Song等[8]在玉米BSSS53中共鉴定22 000 α-醇溶蛋白23个,Song等[7]在玉米B73中鉴定19 000 α-醇溶蛋白25个,Song等[8]分别在玉米 B73和 BSSS53鉴定22 000 α-醇溶蛋白15个和23个,Xu等[18]报道玉米B73中19 000和22 000 α-醇溶蛋白数目分别为26个和15个,Feng等[9]在玉米B73中鉴定出19 000和22 000 α-醇溶蛋白数目分别为25个和16个。2011年Miclaus等[19]发现玉米B73中α-醇溶蛋白有41个(19 000和22 000 α-醇溶蛋白分别为26个和15个),BSSS53中 α-醇溶蛋白有 48个(19 000和22 000 α-醇溶蛋白分别25个和23个)。以上结果显示不同时期所鉴定的玉米α-醇溶蛋白基因数目有所不同,目前仍没有一致结果。本研究是在前人研究结果的基础上,利用NCBI数据库中所有α-醇溶蛋白基因的cDNA序列在玉米基因组中搜寻同源序列,并利用生物信息学方法定位到相关基因并对其位置进行修正,还得到对应基因的预测基因ID。总体而言,本研究所鉴定的玉米α-醇溶蛋白基因家族更为全面和准确,为进一步研究该基因家族提供了一定的依据。
α-醇溶蛋白基因家族各亚族成员在第1、第4和第7条染色体6个区域内分布,与Miclaus等[19]研究结果相似。通过对α-醇溶蛋白基因家族的保守motif分析发现该家族基因序列高度保守,某些motif存在重复现象。以上结果说明α-醇溶蛋白基因家族在进化上同源关系很近,存在串联重复现象。Feng等[10]发现α-醇溶蛋白基因家族41个成员中仅18个基因表达。本研究利用RNA-seq获得高通量数据,结合序列相似性比较和覆盖深度计算,发现玉米α-醇溶蛋白基因家族中大部分基因均表达(约30个),以上结果说明RNA-seq技术具有高敏感性,可精确检测到基因家族中相似基因不同转录本的单碱基差异。
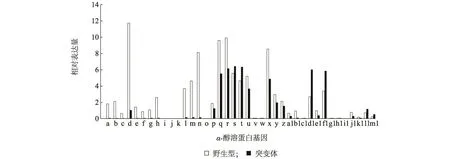
图3 Opaque 2对玉米α-醇溶蛋白基因表达的影响Fig.3 Effects of Opaque 2 on the expression of α-zein genes in maize
α-醇溶蛋白受顺式因子和反式因子协同调控。保守的顺式因子主要位于α-醇溶蛋白基因转录起始位点上游约300 bp处,这一保守区被称为胚乳盒。胚乳盒含有3类不同的保守序列模块:第1类是胚乳模块(EMs);第2类是GCN4结合位点类似模块(GLMs);第3类是醇溶谷蛋白盒(P盒)。P盒序列与EMs序列相同。P盒和GLMs可以介导α-醇溶蛋白基因的胚乳特异性表达。玉米中第一个发现调控α-醇溶蛋白的反式因子是一个bZIP类蛋白,称为Opaque2(O2),O2基因在玉米胚乳中特异表达,所编码的蛋白结合GLMs位点,正向调控玉米22 000 α-醇溶蛋白、b-32基因和cyPPDK1基因的表达[13]。Vicente-Carbajosa等[20]发现另一个调节α醇溶蛋白基因表达的反式因子PBF(Prolamin box binding factor),PBF属于植物Cys2-Cys2锌指结构DNA结合蛋白Dof成员,专化性结合在α-醇溶蛋白基因启动子的P盒区。同样,PBF蛋白质可以特异地与bZIP蛋白质相互作用,促进bZIP蛋白质与启动子中靶序列的结合。PBF和O2在玉米胚乳组织中以相同的方式表达,都是在玉米醇溶蛋白基因转录激活之前开始积累。O2与PBF的相互作用对于玉米醇溶蛋白基因的转录激活是非常必要的[21]。O2的自然突变或RNAi抑制可显著降低22 000 α-醇溶蛋白和其他α-醇溶蛋白基因的表达,导致胚乳粉质、产量及千粒质量降低,籽粒水分含量偏高,易破碎和易感染病虫害,因而使O2的自然突变无法直接用于育种[22-23]。目前,未见有关PBF的自然突变或RNAi抑制后导致玉米性状表现改变的报道,究其原因为PBF除了参与α-醇溶蛋白的调控,还可能涉及到种子发育的其他关键途径。本研究发现α-醇溶蛋白基因家族序列高度保守,但表达谱差异较大,可能与上述顺式和反式协同调控有关。本研究发现转录因子Opaque2主要调控z1C亚族(22 000 α-醇溶蛋白)的表达(与前人研究结果一致)[12],对z1A亚族的表达也有影响,但对z1B和z1D亚族的表达影响作用不明显。
[1] LARKINS B A,BRACKER C E,TSAI C Y.Storage protein synthesis in maize:isolation of zein-synthesizing polyribosomes[J].Plant Physiology,1976,57(5):740-745.
[2] 康美玲,田忠景,张倩倩.利用醇溶蛋白电泳图谱分析不同玉米品种的遗传多样性[J].江苏农业科学,2013,41(10):70-72.
[3] HARTINGS H,LAURIA M,LAZZARONI N,et al.The zea mays mutants opaque-2 and opaque-7 disclose extensive changes in endosperm metabolism as revealed by protein,amino acid,and transcriptome-wide analyses[J].BMC Genomics,2011,12:41.
[4] HOLDING D R,HUNTER B G,CHUNG T,et al.Genetic analysis of opaque2 modifier loci in quality protein maize[J].Theor Appl Genet,2008,117(2):157-170.
[5] PRASANNA B M,VASAL S K,KASSAHUN B,et al.Quality protein maize[J].Current Science-Bangalore,2001,81(10): 1308-1319.
[6] WU Y,MESSING J.Novel genetic selection system for quantitative trait loci of quality protein maize[J].Genetics,2011,188 (4):1019-1022.
[7] SONG R,MESSING J.Contiguous genomic DNA sequence comprising the 19-kD zein gene family from maize[J].Plant Physiology,2002,130(4):1626-1635.
[8] SONG R,LLACA V,LINTON E,et al.Sequence,regulation,and evolution of the maize 22-kD alpha zein gene family[J].Genome Research,2001,11(11):1817-1825.
[9] SONG R,MESSING J.Gene expression of a gene family in maize based on noncollinear haplotypes[J].Proc Natl Acad Sci USA,2003,100(15):9055-9060.
[10]FENG L,ZHU J,WANG G,et al.Expressional profiling study revealed unique expressional patterns and dramatic expressional divergence of maize alpha-zein super gene family[J].Plant MolBiol,2009,69(6):649-659.
[11]SCHMIDT R J,BURR F A,BURR B.Transposon tagging and molecular analysis of the maize regulatory locus opaque-2[J].Science,1987,238(4829):960-963.
[12]HARTINGS H,MADDALONI M,LAZZARONI N,et al.The O2 gene which regulates zein deposition in maize endosperm encodes a protein with structural homologies to transcriptional activators[J].EMBO J,1989,8(10):2795-2801.
[13]MULLER M,DUES G,BALCONI C,et al.Nitrogen and hormonal responsiveness of the 22 kDa alpha-zein and b-32 genes in maize endosperm is displayed in the absence of the transcriptional regulator Opaque-2[J].Plant Journal,1997,12(2):281-291.
[14]BAILEY T L,BODEN M,BUSKE F A,et al.MEME SUITE: tools for motif discovery and searching[J].Nucleic Acids Res,2009,37(Web Server issue):W202-W208.
[15]CHEN J,ZENG B,ZHANG M,et al.Dynamic transcriptome landscape of maize embryo and endosperm development[J].Plant Physiology,2014,166(1):252-264.
[16]葛 敏,吕远大,张体付,等.玉米YABBY基因家族的全基因组鉴定与分析[J].江苏农业学报,2014,30(6):1267-1272.
[17]LI C,QIAO Z,QI W,et al.Genome-wide characterization of cisacting DNA targets reveals the transcriptional regulatory framework of opaque2 in maize[J].Plant Cell,2015,27(3):532-545.
[18]XU J H,MESSING J.Organization of the prolamin gene family provides insight into the evolution of the maize genome and gene duplications in grass species[J].Proc Natl Acad Sci USA,2008,105(38):14330-14335.
[19]MICLAUS M,XU J H,MESSING J.Differential gene expression and epiregulation of alpha zein gene copies in maize haplotypes[J].PLoS Genetics,2011,7(6):e1002131.
[20]VICENTE-CARBAJOSA J,MOOSE S P,PARSONS R L,et al.A maize zinc-finger protein binds the prolamin box in zein gene promoters and interacts with the basic leucine zipper transcriptional activator Opaque2[J].Proc Natl Acad Sci USA,1997,94(14): 7685-7690.
[21]HWANG Y S,CICERI P,PARSONS R L,et al.The maize O2 and PBF proteins act additively to promote transcription from storage protein gene promoters in rice endosperm cells[J].Plant Cell Physiology,2004,45(10):1509-1518.
[22]WU Y,MESSING J.Rescue of a dominant mutant with RNA interference[J].Genetics,2010,186(4):1493-1496.
[23]WU Y,MESSING J.RNA interference-mediated change in protein body morphology and seed opacity through loss of different zein proteins[J].Plant Physiology,2010,153(1):337-347.
