施马伦贝格病毒病焦磷酸测序检测方法的建立
2015-03-22张永强吴晓东赵永刚王志亮
张永强,吴晓东,赵永刚,王志亮
(中国动物卫生与流行病学中心,青岛 266032)
施马伦贝格病毒病焦磷酸测序检测方法的建立
张永强,吴晓东,赵永刚,王志亮*
(中国动物卫生与流行病学中心,青岛 266032)
拟建立施马伦贝格病毒(SBV)S基因焦磷酸测序检测方法,用于施马伦贝格病毒病的快速检测和确诊。通过对公开发表的SBVS基因序列与布尼病毒科其他11种病毒S基因进行比对,找出不同病毒变异区域集中且变异区域两侧保守的序列片段,基因合成,体外转录,作为基因扩增模板。在特异性变异序列两端的保守区域设计扩增引物,进行RT-PCR扩增。在保守区域设计双向测序引物,对RT-PCR产物进行双向焦磷酸测序。通过比对测序结果,确定是否为SBV核酸序列;优化条件,确定检测方法的敏感性、特异性和重复性,建立SBVS基因焦磷酸测序检测方法。结果:建立的施马伦贝格病毒病焦磷酸测序检测方法扩增基因片段长度为166 bp;敏感性为可检测到104个拷贝;每一条测序引物可准确测出50 bp左右核酸序列,双向测序可准确测出100 bp左右核酸序列,具有较好特异性,完全满足施马伦贝格病毒病确诊要求;重复测序3次,均能准确测出100 bp左右核酸序列。本研究建立的施马伦贝格病毒病焦磷酸测序检测方法,可用于施马伦贝格病毒病的检测和确诊,整个检测过程可在1 d内完成,大大缩短了确诊时间。
焦磷酸测序;施马伦贝格病毒;S基因
施马伦贝格病毒病是由施马伦贝格病毒(Schmallenberg virus,SBV)引起的虫媒病毒性传染病,因最早于2011年11月在德国西部施马伦贝格小镇被发现而得名。SBV是有囊膜的单股负链RNA病毒,属于布尼亚病毒科(Bunyaviridae),正布尼亚病毒属(Orthobunyavirus)[1]。SBV主要感染绵羊、牛(包括野牛)和山羊等反刍动物,不感染人[2]。该病毒可经蠓等吸血昆虫叮咬而传播[3],成年牲畜感染后表现为发热、腹泻、乏力等症状,产奶量降低;而怀孕期动物感染后往往导致流产、早产和死产,新生幼畜发生关节弯曲积水性无脑综合征(arthrogryposis hydranencephaly syndrome, AHS),表现为畸形、大脑发育不全、脊柱弯曲、关节无法活动等病症[4-5]。该病自2011年发现以来,不断有新的疫情报告,现已蔓延至欧洲中部、北部大部分国家,引起了欧盟和OIE等国际组织的高度重视。
传统的病原学诊断技术包括病毒分离与鉴定、RT-PCR、实时PCR等方法,这些方法都具有较好的敏感性和特异性,在疫病的诊断上发挥了巨大的作用,但同时也具有自身的一些缺陷。病毒分离和鉴定,耗时长,对检测人员和检测设备要求高;RT-PCR、实时PCR可能出现假阳性,需要通过测序进行确诊,延长了确诊时间。
在分子生物学领域,最精确的诊断方法是基因测序。自1985年,焦磷酸测序技术(pyrosequencing technology,PSQ)[6]逐渐应用于病原的快速确诊,它通过核苷酸和模板结合后释放的焦磷酸引发酶的级联反应,促使荧光素发光而实现检测,其最大优点是可以实现高通量、快速,自该技术发明以来,已广泛应用于单核苷酸多态性分析、细菌、真菌和病毒的分型、突变位点的检测等。但其也有局限性,由于该项技术能测出的序列较短,要求目的序列变异区域相对集中,以便于测出完整变异区域;变异区域两侧需高度保守,以便于设计测序引物并确保可以与所有基因型结合。本研究针对SBVS基因,比对包括SBV在内的布尼病毒科12种病毒近千条序列,找出了符合上述要求的区域,建立SBVS基因的焦磷酸测序检测方法,为该病的快速确诊提供了技术储备。
1 材料与方法
1.1 主要试剂和仪器
Transcriptor One-Step RT-PCR Kit,T7 Transcription Kit购自Roche公司,胶回收试剂盒、DNA Marker购自大连宝生物有限公司、引物由大连宝生物有限公司合成,Pyro Gold SQA Reagents 1×96 Pyromark ID购自Biotage AB公司,PyroMark Q96 MD仪器购自QIAGEN。
1.2 引物设计
根据GenBank BLAST结果,与SBVS片段具有较高同源性的病毒有Simbu virus、Shamonda virus、Sathuperi virus、Sango virus、Sabo virus、Peaton virus、Douglas virus、Aino virus、Oropouche virus、Akabane virus和Rift valley fever virus。下载上述病毒所有S片段基因序列,应用Mega5.0软件进行比对,找出符合焦磷酸测序扩增和测序引物设计要求的区域,运用PSQ Assay Design软件设计SBV S扩增引物以及正向和反向测序引物,并在相应的扩增引物5′端标记生物素。详见表1。
表1 引物名称及序列
Table 1 Name and sequence of primers

焦磷酸测序引物TypeofPyrosequencing引物名Primer序列(5'→3')SequenceForwardPyrosequencingSBV-FFGCATATGTGKCATTTGASBV-FR(Biotin)Biotin-GGRAAATGGTTATTAACCSeq-FGAGTCTTCTTCCTYAAReversePyrosequencingSBV-RF(Biotin)Biotin-GCATATGTGKCATTTGASBV-RRGGRAAATGGTTATTAACCSeq-RTTRAGGAAGAAGACTC
1.3 模板制备
合成选取的SBVS基因片段,体外转录制备RNA,分装后-80 ℃冻存备用。
1.4 RT-PCR条件优化
以制备的RNA为模板,以不同退火温度进行RT-PCR反应,优化反应条件。RT-PCR产物一式两份,一份送交公司测序,一份用于焦磷酸测序反应。
1.5 焦磷酸测序反应
1.5.1 测序单链模板的制备 根据Gene Company Limited公司的焦磷酸测序反应操作说明制备测序单链模板。将45 μL RT-PCR单链模板分别加入到PSQ 96板的孔中,并加入测序引物,并在85 ℃的烘箱中放置2 min,取出冷却后就可进行焦磷酸测序。
1.5.2 测序反应 根据PSQTM 96MA System仪器的软件说明在试剂舱中分别加入的底物APS、dNTPs和酶混合物(DNA聚合酶、荧光素酶、ATP硫酸化酶和三磷酸腺苷双磷酸酶);然后将试剂舱和PSQ 96板放入PSQ TM 96 MD Sys tem仪器中,进行Pyrosequencing反应。
1.6 敏感性试验
分别用Roche、Invitrogen和TaKaRa公司生产的RT-PCR试剂盒,以10倍稀释的RNA为模板进行RT-PCR反应,确定最优反应试剂和敏感性。
1.7 特异性试验
优化测序反应,提高准确测出的碱基序列,使测出的序列长度满足待测样品是否为SBVS基因序列的确诊要求。
1.8 重复性试验
将扩增的RT-PCR产物分别进行3次焦磷酸测序,比较每次测得的序列结果,确定试验的重复性和稳定性。
2 结 果
2.1 RT-PCR反应
采用优化的RT-PCR反应条件以体外转录的
RNA为模板,用SBV扩增引物进行RT-PCR检测。结果显示,扩增出与目的条带大小相符的特异性条带,没有非特异性扩增产物,满足进行焦磷酸测序反应的需要(图1) 。
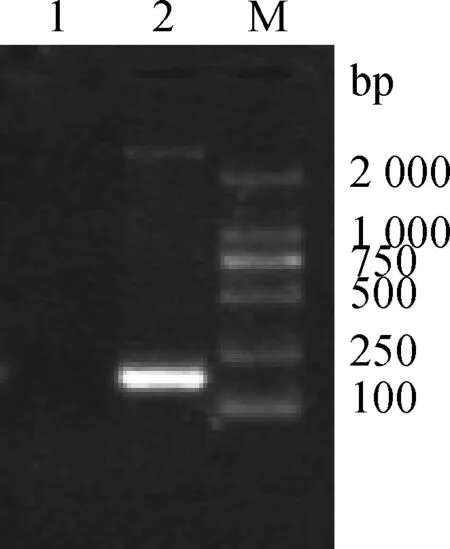
M.DL2000 DNA相对分子质量标准;1.SBV S 基因片段;2.阴性对照M.DL2000 DNA marker;1.SBV S gene segment;2.Negative control图1 SBV S基因RT-PCR扩增结果Fig.1 RT-PCR amplification of SBV S gene
2.2 焦磷酸测序反应
对SBVS基因片段焦磷酸测序反应正向和逆向测序均能准确测出50 bp左右,双向测序能准确测出100 bp左右,完全满足确诊需要。靶序列的焦磷酸测序结果与普通测序结果完全一致,符合率为100%,证明该试验具有很好的特异性(图2)。
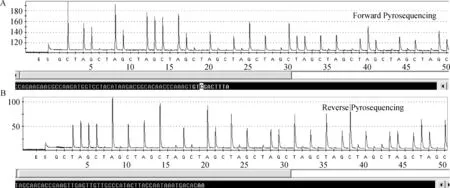
纵坐标:峰的高度(光信号强度)用来判断碱基的数目;横坐标:每个循环的碱基种类;正向测序结果显示,共测出58个碱基,其中48个碱基完全可信,另外10个碱基比较可信;反向测序结果显示,共测出51个碱基,其中49个完全可信,另外2个比较可信Y-axis is indicate the peak value of sequencing (optical signal strength) in order to identy the DNA base amount.X-axis is indicate the gene sequence.The forwarcl pyrosequencing result shows that 58 base were sequencing,48 base among them were confirmed correctly and the other 10 bases were relatively confirmed.The reverse pyrosequening result shows that 51 bases were sequencing,49 bases among them were confirmed correctly and the other 2 bases were relatively confirmed图2 焦磷酸测序技术检测SBV病毒S基因Fig.2 Detection molecular marker of S gene in SBV
2.3 敏感性试验结果
以合成基因制备的RNA为模板,10倍系列稀释后,分别用不同试剂盒进行RT-PCR反应。结果显示,Invitrogen公司RT-PCR试剂盒最为敏感,为102,经计算其最低检测出的拷贝数为104,其它两种试剂盒最低检测敏感性为103个拷贝。见图3。
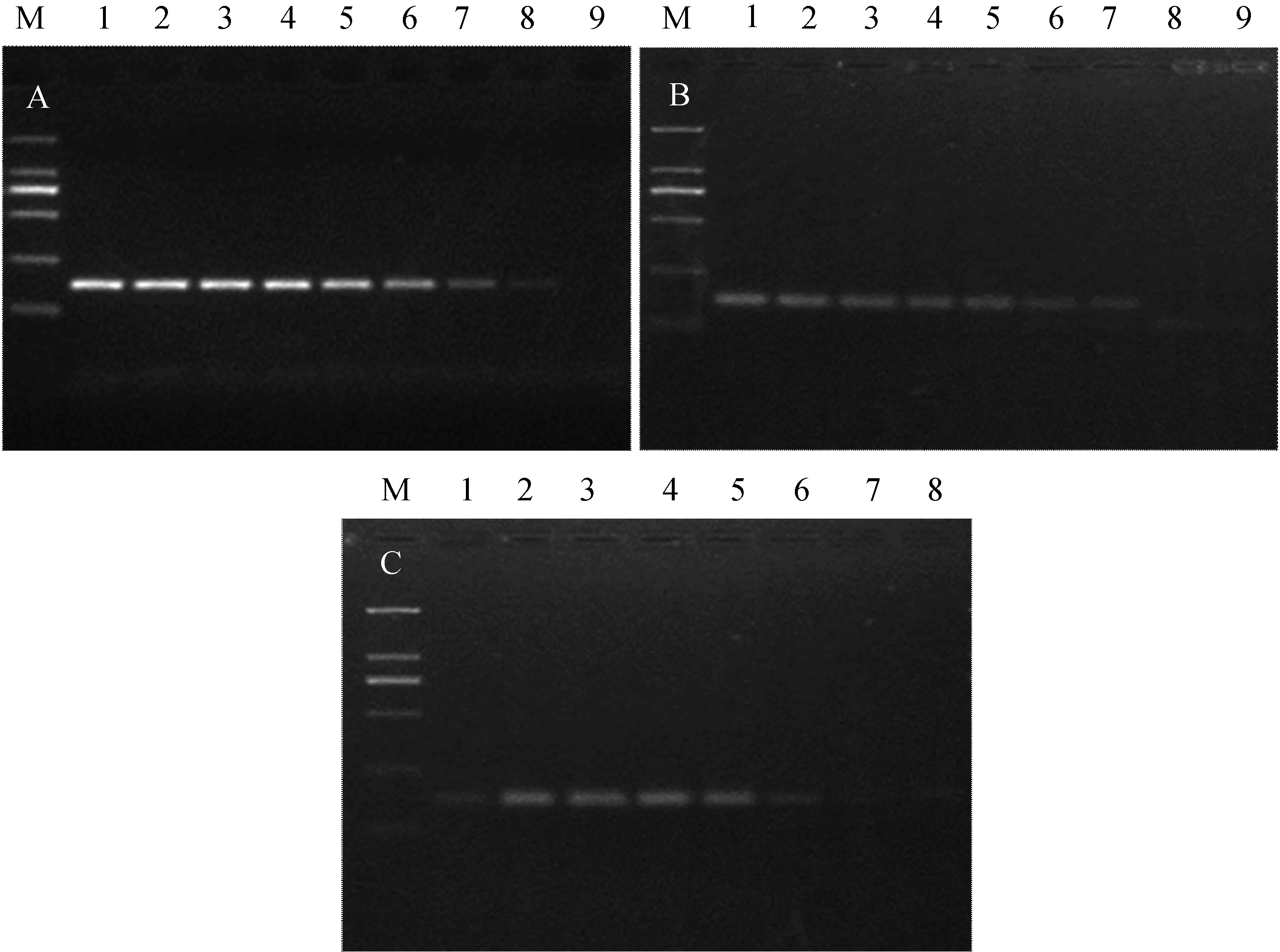
1~9分别为以109、108、107、106、105、104、103、102、101拷贝的RNA为模板;A.Invitrogen公司RT-PCR试剂盒;B.Roche公司RT-PCR试剂盒;C.TaKaRa公司RT-PCR试剂盒1-9 is 109,108,107,106,105,104,103,102,101 copies RNA templates.A.The results of RT-PCR kit made in Invitrogen Company;B.The results of RT-PCR kit made in Roche Company;C.The results of RT-PCR kit made in TaKaRa Company图3 敏感性试验结果Fig.3 Sensitivity of the method
2.4 重复性试验结果
对每个RT-PCR产物进行3次重复性检测,检测的结果均一致,证明该方法有很好的重复性与稳定性。
2.5 特异性试验结果
通常情况下,该测序反应准确和较准确测出的碱基数为20~30 bp。作者经优化测序反应后,提高了准确测出的碱基序列,使正向和逆向测序反应中准确和较准确测出的序列分别达到50 bp左右(图2),因此准确测出序列可达100 bp左右,完全满足证明待测样品是否为SBVS基因序列的确诊要求,证明该方法具有很好的特异性。
3 讨 论
SBV主要感染绵羊、牛(包括野牛)和山羊等反刍动物[7],主要经蠓等吸血昆虫叮咬传播[8],成年牲畜感染后表现为发热、腹泻、乏力等症状,产奶量降低;而怀孕期动物感染后往往导致流产、早产和死胎,新生幼畜发生AHS[9-10],与赤羽病具有相似的临床症状[11]。该病自2011年底发现以来,不断有新的疫情报告,截止目前已在二十几个国家发生[12]。中国目前尚无相关疫情,但随着国际交流的进一步深入,该病有传入的风险,因此急需建立必要的检测技术。
焦磷酸测序技术是1985年发展起来的一种能够通过对基因序列测定实现检测和确诊的新型检测技术,具有高通量、快速、敏感等特点[13],其基本原理是设计带有生物素标记的扩增引物,通过PCR反应生成含有生物素标记的目的基因片段,然后制备测序单链模板。将待测单链模板与测序引物杂交结合,放入PSQTM96MD System中进行测序反应。与普通RT-PCR和real-time RT-PCR等检测技术相比,该技术无需将PCR产物送交公司测序以便确诊,从而大大减少了病原确诊时间。同时,该项技术也有严格的应用要求,首先片段不能太长,最好在100~200 bp;其次该项技术能准确测出的序列较短,需要待测核酸的特异性变异区域比较集中,同时在该变异区域前面或后面的序列高度保守,以便于设计扩增和测序引物。
布尼病毒科的成员通常利用S基因建立核酸检测方法,如A.A.Chengula等建立了RVFS基因分子生物学检测方法并进行了应用[13],Y.Stram等建立了赤羽病和艾诺病毒的RT-PCR检测方法[14]。本研究通过对GenBank中发表的布尼病毒科十余种病毒千余条S基因序列的比对分析,找出S基因67—233区域符合焦磷酸测序设计要求,S片段67—80 bp、217—233 bp区域在布尼病毒科中为高度保守区域,可以设计上下游扩增引物,提高方法扩增环节的敏感性;119—134 bp区域高度保守,用于设计正向和逆向测序引物,而79—118和135—181 bp区域在布尼病毒中高度变异,测出该区域的核酸序列即可确定待测病原为何种病毒。
总之,本研究所建立的SBV焦磷酸测序方法准确性高、重复性和稳定性好、特异性强等优点,为SBV的快速检测和确诊提供了一种可行方法,为防止施马伦贝格病毒病传入中国奠定了防控基础。
[1] WALTER C T,BARR J N.Recent advances in the molecular and cellular biology of bunyaviruses [J].JGenVirol,2011,92(Pt 11):2467-2484.
[2] TARLINTON R,DALY J,DUNHAM S,et al.The challenge of Schmallenberg virus emergence in Europe [J].VetJ,2012,194(1):10-18.
[3] DE REGGE N,DEBLAUWE I,DE DEKEN R,et al.Detection of Schmallenberg virus in different Culicoides spp.by real-time RT-PCR [J].TransboundEmergDis,2012,59(6):471-475.
[4] HERDER V,WOHLSEIN P,PETERS M,et al.Salient lesions in domestic ruminants infected with the emerging so-called Schmallenberg virus in Germany [J].VetPathol,2012,49(4):588-591.
[5] DE REGGE N,VAN DEN BERG T,GEORGES L,et al.Diagnosis of Schmallenberg virus infection in malformed lambs and calves and first indications for virus clearance in the fetus [J].VetMicrobiol,2013,162(2-4):595-600.
[6] ELAHI E,RONAGHI M.Pyrosequencing:a tool for DNA sequencing analysis [J].MethodsMolBiol,2004,255:211-219.
[7] ELBERS A R,MEISWINKEL R,VAN WEEZEP E,et al.Schmallenberg virus in Culicoides spp.biting midges,the Netherlands,2011[J].EmergInfectDis,2013,19(1):106-109.
[8] MANSFIELD K L,LA ROCCA S A,KHATRI M,et al.Detection of Schmallenberg virus serum neutralising antibodies[J].JVirolMethods,2013,188(1-2):139-144.
[9] LOEFFEN W,QUAK S,DE BOER-LUIJTZE E,et al.Development of a virus neutralisation test to detect antibodies against Schmallenberg virus and serological results in suspect and infected herds [J/OL].ActaVetScand,2012,54:44.[2014-12-10].http://www.actavetscand.com/content/54//44.
[10] GARIGLIANY M M,BAYROU C,KLEIJNEN D,et al.Schmallenberg virus:a new Shamonda/Sathuperi-like virus on the rise in Europe[J].AntiviralRes,2012,95(2):82-87.
[11] HECHINGER S,WERNIKE K,BEER M.Evaluating the protective efficacy of a trivalent vaccine containing Akabane virus,Aino virus and Chuzan virus against Schmallenberg virus infection [J/OL].VetRes,2013,44:114.[2014-12-10].http://www.veterinaryresearch.org/content/44/1/114.
[12] OIE.OIE Immediate notification report(Schmallenberg-Virus).[EB/OL] http://www.oie.int/wahis_2/public/wahid.php/Reviewreport/Review?reportid=11514,2014-03-26.
[13] CHENGULA A A,KASANGA C J,MDEGELA R H,et al.Molecular detection of Rift Valley fever virus in serum samples from selected areas of Tanzania[J].TropAnimHealthProd,2014,46(4):629-634.
[14] STRAM Y,KUZNETZOVA L,GUINI M,et al.Detection and quantitation of akabane and aino viruses by multiplex real-time reverse-transcriptase PCR[J].JVirolMethods,2004,116(2):147-154.
(编辑 白永平)
Establishment of Detection and Identification Method for Schmallenberg Virus by Pyrosequencing
ZHANG Yong-qiang,WU Xiao-dong,ZHAO Yong-gang,WANG Zhi-liang*
(ChinaAnimalHealthandEpidemiologyCenter,Qingdao266032,China)
The aim of the present study was to establish a pyrosequencing method targetingSgene for detection and identification of schmallenberg virus (SBV).In alignment with published more than 10 virusesSgene sequences which belong to Bunyaviridae and are highly homogenous with SBV.Adaptive sequence for pyrosequencing was synthesized andinvitrotranscripted to cRNA as the RT-PCR templates.After RT-PCR reaction,the products were detected with forward and reverse pyrosequencing and identified by genomic alignment.Results showed that the RT-PCR products were 166 bp.The sensitivity of the method that the minimum copies could be detected was 104 copies.The specificity that sequencing length by pyrosequencing was around 100 bp which could make a definite diagnosis for SBV.The results presented here demonstrate that the detection and identification method for SBV by pyrosequencing was established.The method can be used in fast diagnosis for SBV.
pyrosequencing;schmallenberg virus;Sgene
10.11843/j.issn.0366-6964.2015.01.022
2014-05-26
现代农业产业技术体系(奶牛)(NYCYTX-0305)
张永强(1977- ),男,山东邹平人,博士,助理研究员,主要从事病毒分子生物学研究,Tel:0532-87839922,E-mail:zhangyq_l@163.com
*通信作者:王志亮(1966-),研究员,博士生导师,E-mail:zlwang111@163.com
S852.659.5
A
0366-6964(2015)01-0174-05
