不同毛色山羊皮肤组织Agouti基因剪接体类型研究
2015-03-22李祥龙周荣艳李兰会
张 天,李祥龙,周荣艳,李兰会
(1.河北科技师范学院,秦皇岛066600; 2.河北农业大学,保定 071000; 3.晋州市职教中心,晋州 052260)
不同毛色山羊皮肤组织Agouti基因剪接体类型研究
张 天1,2,3,李祥龙1,2*,周荣艳2,李兰会2
(1.河北科技师范学院,秦皇岛066600; 2.河北农业大学,保定 071000; 3.晋州市职教中心,晋州 052260)
本研究旨在探讨白色和黑色山羊皮肤组织Agouti基因所具有的不同剪接体类型。采用PCR扩增和5′RACE方法分别获得白色和黑色山羊皮肤组织Agouti基因部分基因组序列及mRNA 序列,并进行拼接比对。结果,分别获得了长度为27 793和27 858 bp的白色和黑色山羊Agouti基因序列,发现4段序列(88、180、81和79 bp)与绵羊mRNA序列相匹配,这些片段与5′RACE所检测到的外显子1可变剪接体长度对应,其中的180、81和79 bp为新发现片段。白色山羊检测到It、It′、IA、2C和IE 5种类型剪接体,黑色山羊仅检测到It和It′ 2种类型剪接体。剪接体之间和剪接体内部均存在长度变异。结果提示,IA、2C和IE可变剪接可能与山羊毛色形成有关。
山羊;Agouti基因;皮肤组织;剪接体类型
动物毛色主要通过肉眼来识别,是鉴定品种外貌的重要标志,它在识别品种纯度、亲缘关系,确定杂交组合、评价产品质量等方面有一定优势,是最能够引起人类关注的品种特征之一[1-2]。毛色对于许多动物来说是一项重要的生产及可利用的质量性状,是哺乳动物中第一个作为遗传学分析的表型性状[3]。Agouti基因编码周围黑素细胞真皮乳头细胞分泌的Agouti信号蛋白(Agouti signal protein,ASIP),这是一种旁分泌信号分子[4],此蛋白与黑素细胞刺激素α(α-melanocyte stimulating hormone,α-MSH)竞争性地结合黑素皮质素受体1(Melanocortin receptor1,MC1R),控制褐黑色素和真黑色素的生物合成,决定二者的数量与比例,从而形成不同类型的黑色素,产生不同的毛色[5-6]。另外,Agouti基因与其相关蛋白是糖尿病与肿瘤、肥胖等人类疾病的研究热点之一,Agouti基因位点的全面检测将为动物品种资源的保存与利用提供重要参考[7]。
控制山羊被毛颜色遗传的基因座主要有4个:野生型座位A(称为刺鼠毛色基因座位)、稀释毛色因子座位D、“上位白”座位I、白斑座位S,4个基因都位于常染色体上,它们之间不存在连锁关系[8-9]。毛色与经济性状关系不大,但与生长速度、瘦肉率及对疫病的抵抗力等生产性能有直接关系,Agouti基因与山羊毛色形成有很大关系,故Agouti基因对山羊生产性能方面也有影响[10]。山羊Agouti基因已被定位于13q22[11]。哺乳动物Agouti基因含有4个外显子,外显子2、3、4编码131~133个氨基酸,非翻译外显子1存在不同剪接体。动物皮肤组织中,Agouti基因非翻译外显子1所存在的不同剪接体类型可能与该基因的表达存在某种特定关系,进而影响毛色的变化。
本试验研究探讨了白色和黑色山羊皮肤组织Agouti基因5′非翻译区外显子1的剪接体变异类型,为探讨山羊毛色分子调控机制提供依据。
1 材料与方法
1.1 山羊Agouti基因序列的扩增
1.1.1 样品来源及引物设计 使用实验室-20 ℃冰箱保存的辽宁绒山羊(白色)和雷州山羊(黑色)血液样品各3个提取DNA,扩增山羊的Agouti基因序列。扩增所用引物见表1。
表1Agouti基因5′非翻译区PCR扩增测序引物
Table 1 Primers used for amplifying 5′UTR of goatAgoutigene

引物Primer序列(5′→3′)Sequence扩增长度/bpSize退火温度/℃Annealingtemperature延伸时间/sExtendingtime循环数CycleP1F:TGCTTGATTGGCTGTTCTTCR:TCACTGATGGCAATTCTTCC226160.014535P2F:GGCTGCTTCCATTTGCTGTAR:ACCTGGGTGGTTGGTGATAT267058.016035P3F:TAATGTTTCCCCTAATCCR:TGCTTTCTAAGAGTTTGTCG123856.07535P4F:CTCTTAGGGATAGGCAACACR:CCTCTTCCAAGGGATAGTTC289162.017035P5F:CTGGCTCTTGGTTGCTGTR:TGACTCTGGGTGGAGGTTA293556.917035P6F:GTGAGACTCTTCCTGCCTGTGR:GGTGGCGCAGACAGTAAAG352363.021535P7F:ATCAGCCCTGGGATTTCTTR:GCTTTATCACCCACCCTTCT235860.014535AGT1F:CGGGAGTAATCCAAGAGTCR:TGAGGGAAGAGGAAGCAG39355.06035AGT2F:CATCTGAGACCCAGCATAGCR:CTCACAAACCACCCTACCAT262362.716035AGT3F:AGAACCTGGCTACTATTGCTR:TTGAGAACTGCTGGTGAGAA129558.09035
F代表上游引物,R代表下游引物
The F and R represent forward and reverse primers,respectively
1.1.2 PCR反应及产物检测与回收 PCR反应总体积50 μL:灭菌水32 μL、10×LA PCR Buffer II(Mg2+Plus)5 μL、dNTP Mixture(各2.5 mmol·L-1)8 μL、模板DNA 2.5 μL、P1(20 μmol·L-1)1 μL、P2(20 μmol·L-1)1 μL、TaKaRa LATaq(5 U·μL-1)0.5 μL。
PCR反应条件:94 ℃ 预变性5 min;94 ℃ 变性30 s,退火30 s,72 ℃ 延伸(根据目的片段大小确定延伸时间),30~35个循环;72 ℃ 终延伸10 min,4 ℃终止保存。用1.0%~2.0%琼脂糖凝胶电泳检测,SanPrep柱式DNA胶回收试剂盒回收。
1.1.3 回收产物连接、转化、培养及阳性重组子鉴定 按照pEASY-T3 Cloning Kit说明书将回收的PCR产物通过TA克隆连接到pEASY-T3载体上,过夜培养。用灭菌牙签挑取白色且形状圆润的单菌落至含有3~5 mL 100 mg·L-1Amp+的LB 液体培养基中,37 ℃摇床振荡培养6~10 h,菌液PCR鉴定重组子。阳性克隆送北京中科希林生物科技有限责任公司测序。
1.2 山羊Agouti基因5′RACE
1.2.1 RNA提取、纯化及反转录 样品为12月龄左右的4只白色(B1、B2、B3和B4)和3只黑色(H1、H2和H3)太行山羊皮肤组织。按照TaKaRa RNAiso Plus说明书提取RNA,纯化后的RNA用NANODROP 2000超微量分光光度计测定浓度及纯度。经1.0%琼脂糖凝胶150 V,150 mA电泳10 min,凝胶成像系统观察、拍照并保存,将28S和18S条带的亮度和宽度比例均为2∶1的RNA按SMARTer 5′RACE cDNA Amplification Kit说明书反转录。RT产物保存于-20 ℃备用。
1.2.2 引物设计 以绵羊mRNA为模板,SMARTer 5′RACE cDNA Amplification Kit中10×Universal Primer A Mix(UPM)和Nested Universal Primer A(NUP,10 μmol·L-1)为上游引物,设计下游引物RT1、GSP和NGSP(表2)用作5′RACE试验的引物。
表2 5′RACE所用引物
Table 2 Primers for 5′RACE
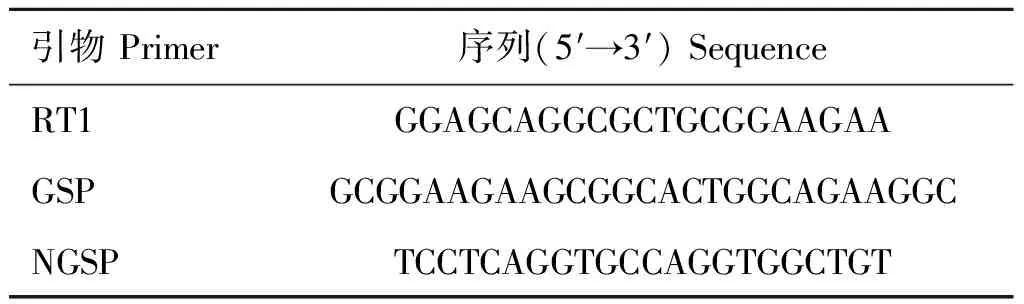
引物Primer序列(5′→3′)SequenceRT1GGAGCAGGCGCTGCGGAAGAAGSPGCGGAAGAAGCGGCACTGGCAGAAGGCNGSPTCCTCAGGTGCCAGGTGGCTGT
1.2.3 5′RACE反应条件及克隆测序 按照Advantage 2 PCR Kit说明书分别进行Outer PCR和Inner PCR,琼脂糖检测法检测PCR产物,高亮条带的产物均进行切胶回收。将回收的Inner PCR产物(B1、B4和H3)通过TA克隆连接到pMD19-T载体上,Inner PCR产物(B2、B3、H1和H2)通过TA克隆连接到pEASY-T3载体上,过夜培养。按上述过程进行阳性重组子鉴定和测序。
2 结 果
2.1 山羊Agouti基因序列分析
2.1.1 山羊Agouti基因序列扩增 引物P1~P7及AGT1~AGT3扩增产物用1.0%琼脂糖凝胶检测,10对引物扩增产物的大小与预期大小一致,均进行切胶回收,回收后的产物均进行连接和转化,菌液PCR检测、获取阳性克隆并测序,切胶回收后电泳结果如图1。
2.1.2Agouti基因结构分析 DNAMAN6.0软件分析后,利用CAP3 Sequence Assembly Program将P1~P7扩增获得的序列进行拼接,分别得到长度为16 161和16 226 bp的白色和黑色山羊拼接序列。将P1~P7拼接获得的序列再与12 847 bp(JN651404+EF587236)[12]的序列进行拼接,分别得到长度为27 793和27 858 bp的白色和黑色山羊Agouti基因拼接序列。引物AGT1、AGT2、AGT3扩增白色山羊获得的序列长度分别为364、2 625和1 296 bp;扩增黑色山羊获得的序列长度分别为367.2622和1 298 bp。
前期所得的长度为7 357 bp的JN651404包括Agouti基因外显子1(90 bp)和5′非翻译区部分序列(7 267 bp)[13]。利用BioEdit7.0以及Spidey软件与绵羊mRNA(NM_001134303.1)比对分析,本试验获得的白色和黑色山羊Agouti基因序列在外显子2之前除了88 bp已经预测外,又发现3段与绵羊mRNA相匹配序列,长度分别为180、81和79 bp(表3)。
2.2 山羊Agouti基因的5′RACE扩增
2.2.1 山羊Agouti基因反转录 山羊皮肤组织提取RNA后进行纯化,纯化后的RNA 28S 与18S 泳带亮度及宽度比例接近 2∶1,且 28S 整齐、清晰,无严重拖尾,表明 RNA 纯度和完整性较好。NANODROP 2000 超微量分光光度计测得样品 A260 nm/A280 nm为 1.8~2.0[14],说明纯化后的 RNA 质量较高,可以直接进行反转录。
7 只山羊 Outer PCR 扩增产物均无特异带,整个电泳条带呈现弥散状(即无法区分的抹带),故需将 Outer PCR 产物稀释后进行Inner PCR。Inner PCR 扩增产物经电泳检测:白色山羊 B1、B2、B3、B4 分别存在3、4、2、2条带,黑色山羊 H1、H2、H3 分别存在 3、2、1 条带,由于扩增过程中可能产生非特异性条带,故需将各条带均切胶回收通过连接、转化,菌液测序判定目的条带。切胶纯化结果如图2所示。
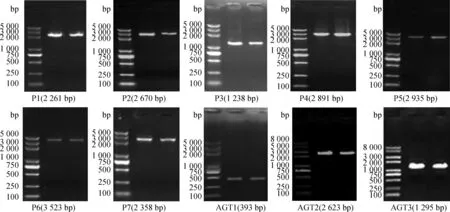
每个图中从左到右依次为Marker、白色山羊和黑色山羊的扩增条带From left to right are Marker,amplification products of white and black goats,respectively in each figure图1 山羊Agouti基因扩增Fig.1 Amplifying electrophoresis of goat Agouti gene
表3 白色和黑色山羊5′非翻译区分析
Table 3 Analysis results of 5′UTR in white and black goats
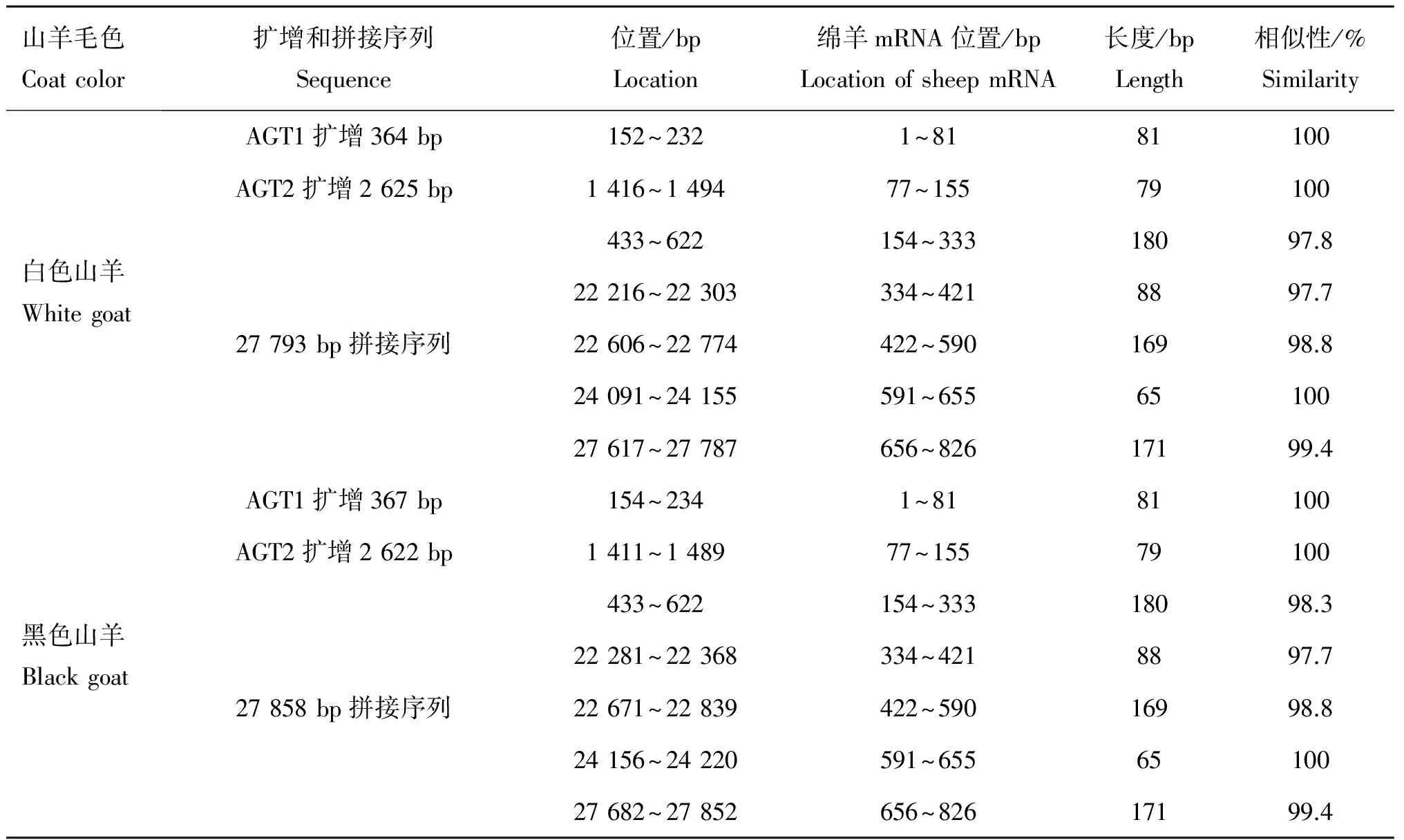
山羊毛色Coatcolor扩增和拼接序列Sequence位置/bpLocation绵羊mRNA位置/bpLocationofsheepmRNA长度/bpLength相似性/%Similarity白色山羊WhitegoatAGT1扩增364bp152~2321~8181100AGT2扩增2625bp1416~149477~15579100433~622154~33318097.822216~22303334~4218897.727793bp拼接序列22606~22774422~59016998.824091~24155591~6556510027617~27787656~82617199.4黑色山羊BlackgoatAGT1扩增367bp154~2341~8181100AGT2扩增2622bp1411~148977~15579100433~622154~33318098.322281~22368334~4218897.727858bp拼接序列22671~22839422~59016998.824156~24220591~6556510027682~27852656~82617199.4
2.2.2 山羊Agouti基因5′末端序列阳性克隆鉴定 山羊Agouti基因5′末端序列PCR扩增中出现的条带分别切胶回收后连接至载体。经菌液PCR检测B1、B2、B3、H1和H2均出现多种不同大小的片段,B4和H3出现1种片段结果(图3),将检测到的大小不同的片段均进行测序。测序得到的序列结果用BioEdit 7.0软件去除载体并找到上、下游引物,B1、B2、B3、B4、H1、H2和H3分别发现2、5、3、1、2、2和1种不同长度的序列可以与Agouti基因比对上。
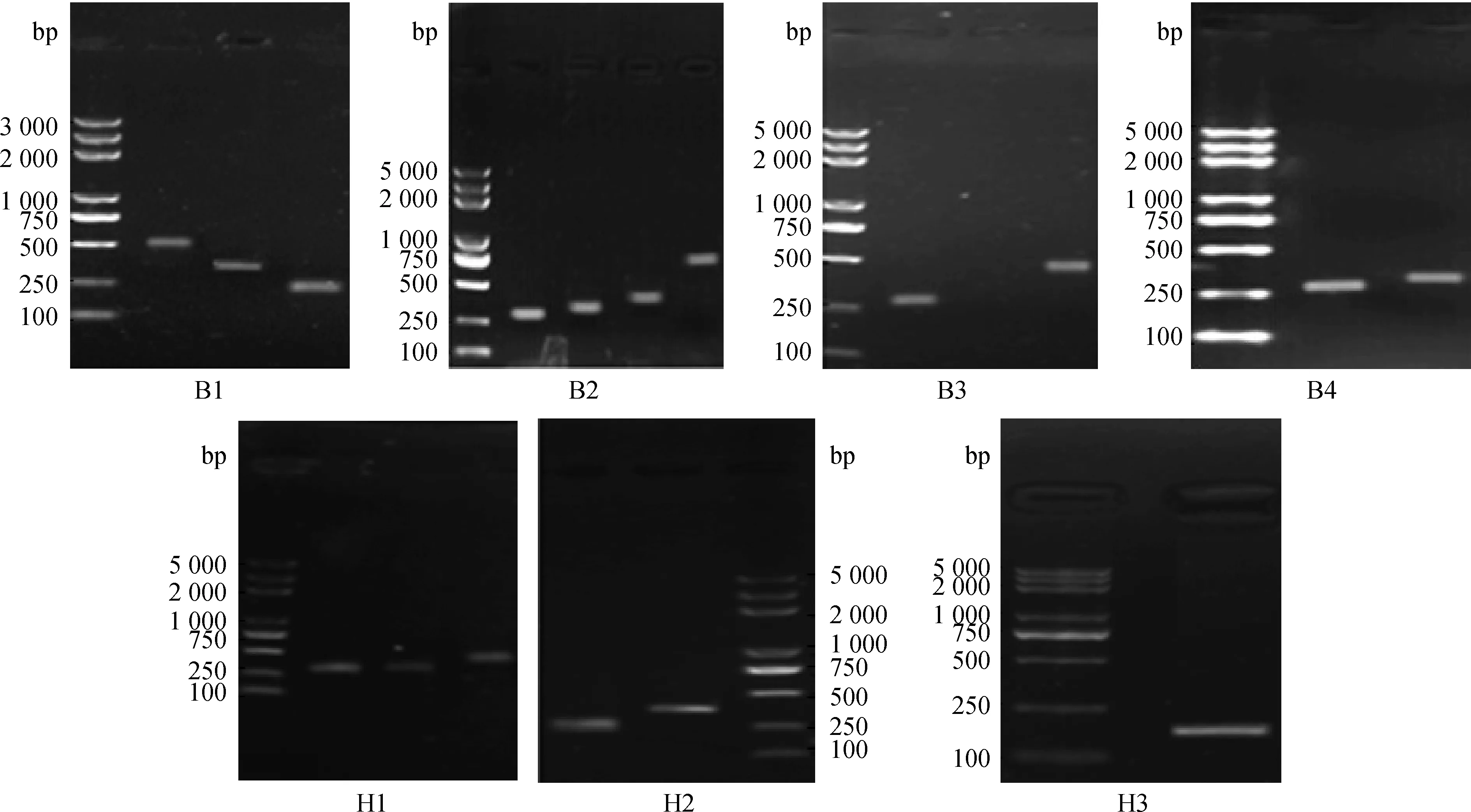
各图中从左到右依次为Marker、Inner PCR切胶回收条带From left to right are Marker,result of Inner PCR,respectively in each figure图2 Inner PCR切胶回收Fig.2 Results of Inner PCR

各图中从左到右依次为Marker、菌液PCR获得的不同大小片段From left to right are Marker,different fragments by bacterium PCR,respectively in each figure图3 山羊Agouti基因5′末端序列阳性克隆鉴定Fig.3 Clone of Agouti cDNA 5′ termination
2.2.3 山羊5′RACE序列分析
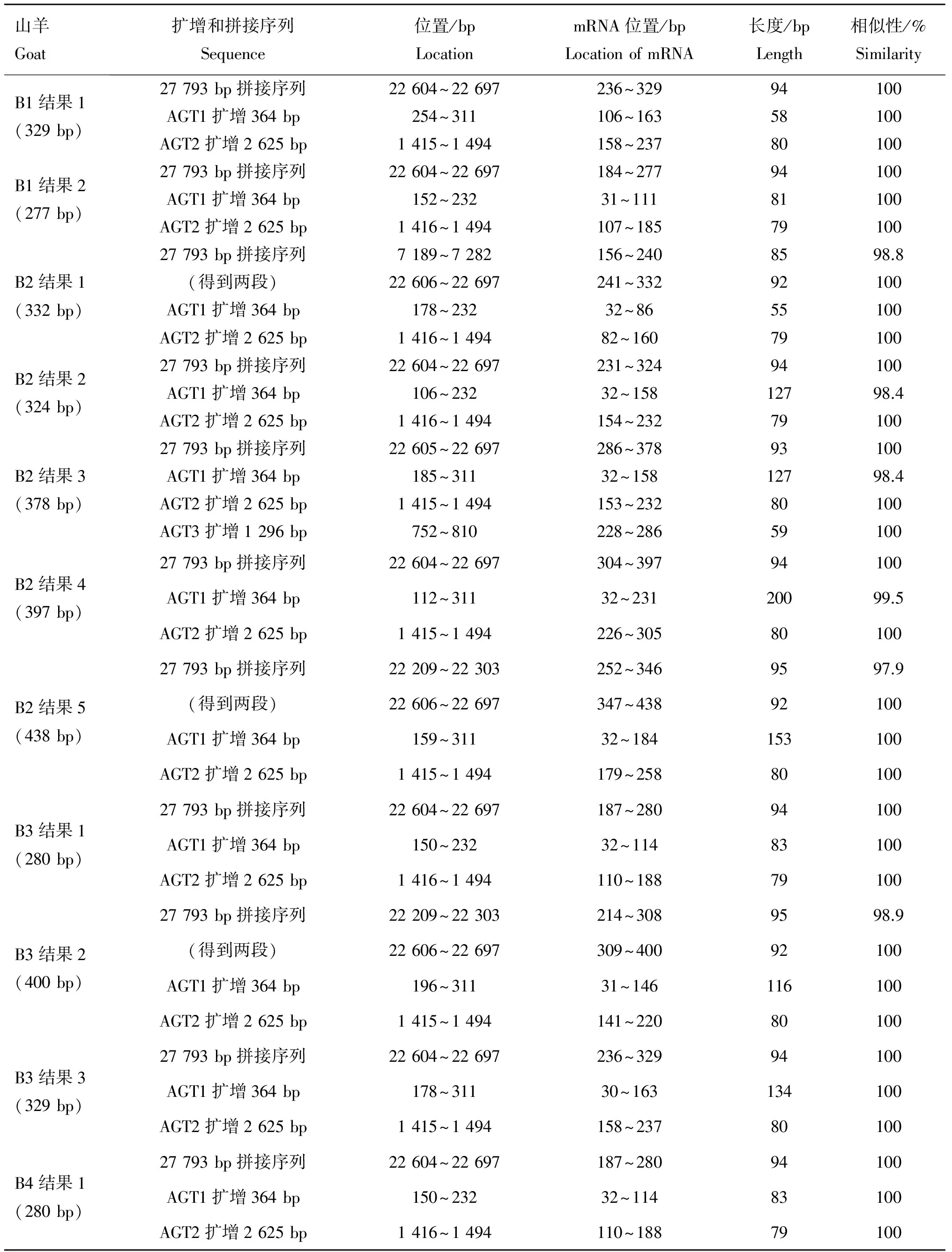
2.2.3.1 白色山羊外显子1剪接体类型:应用Spidey软件将B1、B2、B3和B4测序结果与获得的白色山羊Agouti基因序列比对分析,结果列于表4,分析得出4只白色山羊各种剪接体类型(图4),4只白色山羊中共检测到5种不同类型剪接体,同一类型的剪接体存在不同程度的长度变异。B1存在It和It′ 2种类型的剪接体,It包括两段,长度分别为58和81 bp。B2存在It、It′、IA、2C和IE 5种类型剪接体,2C在基因组上的位置包含在牛2C(DQ000238.1)剪接体在基因组位置之内,说明2C剪接体存在于B2中;最长的It为200 bp,其余It出现不同程度缺失;最长的It′为80 bp;IA、2C、IE长度分别为59、85、95 bp。B3存在It、It′和IE 3种类型的剪接体,最长的It为134 bp,其余It 出现不同程度缺失;最长的It′为80 bp;IE的长度为95 bp。B4存在It、It′ 2种类型的剪接体,长度分别为83和79 bp。将各个白色山羊所得的每条序列均用通过NCBI的Blast在线软件与山羊基因组序列(AJPT01136617.1)进行比较,每条序列与山羊13号染色体同一区域序列的相似性均在84% 以上,大多数相似性为100%,B1、B2、B3、B4 均包括部分Exon2和部分Exon1。综合4只白色山羊5′RACE结果得出,4只白色山羊Exon1都能检测两种不同的剪接体类型:It、It′,最长的It为200 bp,其余It序列出现不同程度缺失; It′一种长度为80 bp,另一种长度为79 bp。白色山羊B2还检测到IA、2C、IE 3种不同的剪接体类型,白色山羊B3也检测到IE剪接体的存在。

1~364,1~2 625,1~1 295,1~27 793代表Agouti基因序列;B1~B4为白色山羊剪接体类型及其在基因组上对应的位置和长度The 1-364,1-2 625,1-1 295 and 1-27 793 represent Agouti sequences;The B1-B4 represent the location and length of Agouti gene spliceosome in white goat图4 白色山羊剪接体类型Fig.4 Spliceosome types in white goat
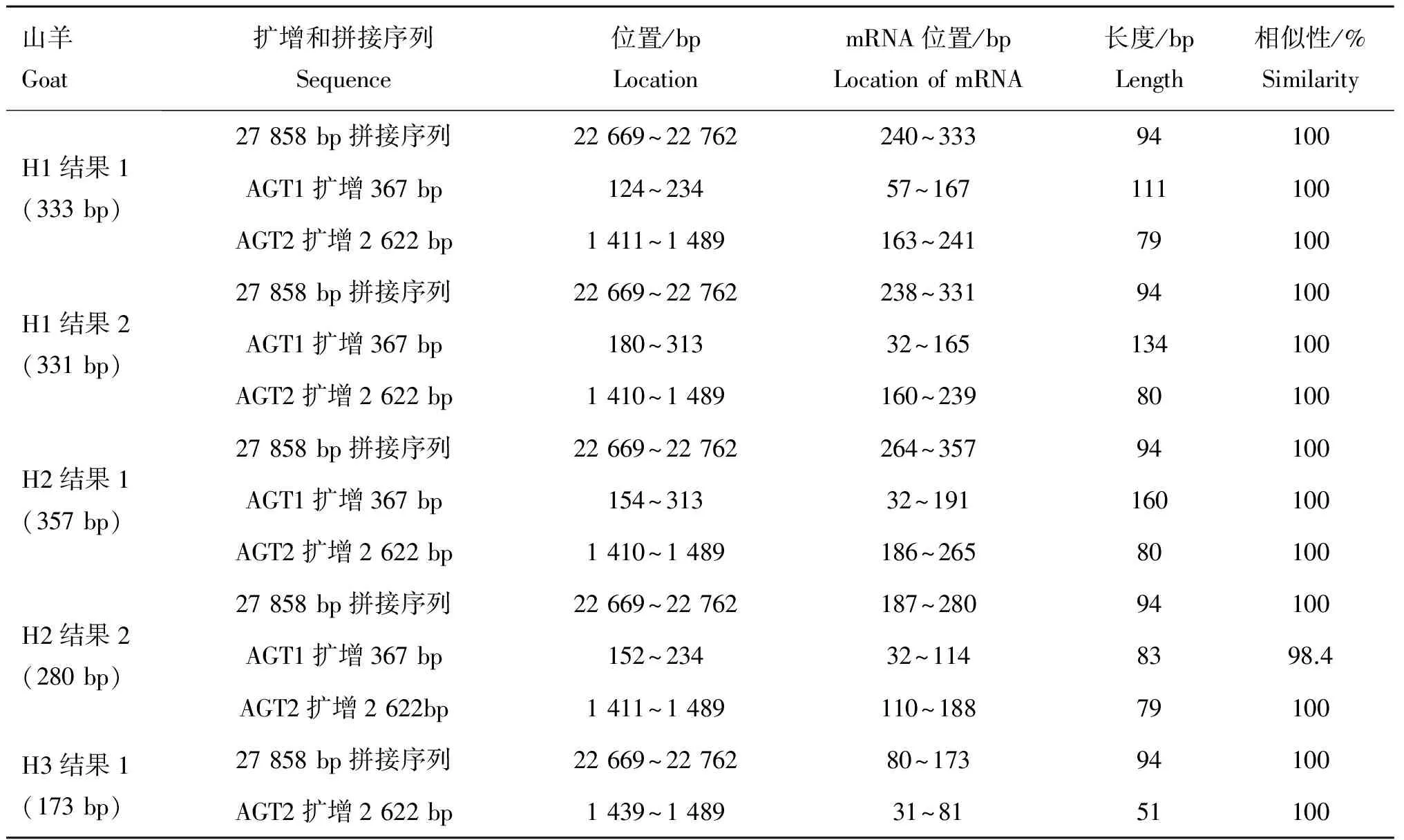
2.2.3.2 黑色山羊外显子1剪接体类型:应用Spidey软件将H1、H2和H3测序结果与获得的黑色山羊Agouti基因序列比对分析,结果列于表5,分析得出3只黑色山羊不同剪接体类型(图5),3只黑色山羊中仅检测到2种类型剪接体,即It和It′,同一种剪接体也存在不同程度的长度变异。H1存在It、It′ 2 种类型的剪接体,最长的It为134 bp;最长的It′ 为80 bp。H2 存在It、It′ 2种类型的剪接体,最长的It为160 bp;最长的It′ 为80 bp; H3只存在It′ 1种类型的剪接体,长度为51 bp。将各个黑色山羊所得的每条序列均用通过NCBI的Blast在线软件与山羊基因组序列(AJPT01136617.1)进行比较,每条序列与山羊13号染色体同一区域序列的相似性均在99% 以上,H1、H2、H3均包括部分Exon2和部分Exon1。综合3只黑色山羊5′RACE结果得出:黑色山羊Exon1检测到两种不同的剪接体类型:It和It′,最长的It为160 bp,其余It序列出现不同程度缺失;It′一种长度为80 bp,另一种长度为79 bp。
表4 白色山羊Spidey分析结果
Table 4 Spidey analysis of white goat B1-B4
山羊Goat扩增和拼接序列Sequence位置/bpLocationmRNA位置/bpLocationofmRNA长度/bpLength相似性/%SimilarityB1结果1(329bp)27793bp拼接序列22604~22697236~32994100AGT1扩增364bp254~311106~16358100AGT2扩增2625bp1415~1494158~23780100B1结果2(277bp)27793bp拼接序列22604~22697184~27794100AGT1扩增364bp152~23231~11181100AGT2扩增2625bp1416~1494107~18579100B2结果1(332bp)27793bp拼接序列7189~7282156~2408598.8(得到两段)22606~22697241~33292100AGT1扩增364bp178~23232~8655100AGT2扩增2625bp1416~149482~16079100B2结果2(324bp)27793bp拼接序列22604~22697231~32494100AGT1扩增364bp106~23232~15812798.4AGT2扩增2625bp1416~1494154~23279100B2结果3(378bp)27793bp拼接序列22605~22697286~37893100AGT1扩增364bp185~31132~15812798.4AGT2扩增2625bp1415~1494153~23280100AGT3扩增1296bp752~810228~28659100B2结果4(397bp)27793bp拼接序列22604~22697304~39794100AGT1扩增364bp112~31132~23120099.5AGT2扩增2625bp1415~1494226~30580100B2结果5(438bp)27793bp拼接序列22209~22303252~3469597.9(得到两段)22606~22697347~43892100AGT1扩增364bp159~31132~184153100AGT2扩增2625bp1415~1494179~25880100B3结果1(280bp)27793bp拼接序列22604~22697187~28094100AGT1扩增364bp150~23232~11483100AGT2扩增2625bp1416~1494110~18879100B3结果2(400bp)27793bp拼接序列22209~22303214~3089598.9(得到两段)22606~22697309~40092100AGT1扩增364bp196~31131~146116100AGT2扩增2625bp1415~1494141~22080100B3结果3(329bp)27793bp拼接序列22604~22697236~32994100AGT1扩增364bp178~31130~163134100AGT2扩增2625bp1415~1494158~23780100B4结果1(280bp)27793bp拼接序列22604~22697187~28094100AGT1扩增364bp150~23232~11483100AGT2扩增2625bp1416~1494110~18879100
表5 黑色山羊H1~H3 Spidey 分析
Table 5 Spidey results of black goat H1-H3
山羊Goat扩增和拼接序列Sequence位置/bpLocationmRNA位置/bpLocationofmRNA长度/bpLength相似性/%SimilarityH1结果1(333bp)27858bp拼接序列22669~22762240~33394100AGT1扩增367bp124~23457~167111100AGT2扩增2622bp1411~1489163~24179100H1结果2(331bp)27858bp拼接序列22669~22762238~33194100AGT1扩增367bp180~31332~165134100AGT2扩增2622bp1410~1489160~23980100H2结果1(357bp)27858bp拼接序列22669~22762264~35794100AGT1扩增367bp154~31332~191160100AGT2扩增2622bp1410~1489186~26580100H2结果2(280bp)27858bp拼接序列22669~22762187~28094100AGT1扩增367bp152~23432~1148398.4AGT2扩增2622bp1411~1489110~18879100H3结果1(173bp)27858bp拼接序列22669~2276280~17394100AGT2扩增2622bp1439~148931~8151100

1~367、1~2622、1~1298、1~27858代表Agouti基因序列;H1~H3部分为黑色山羊剪接体类型及其在基因组上对应的位置和长度The 1-367,1-2622,1-1298 and 1-27858 represent Agouti;The H1-H3 represent the location and length of Agouti gene spliceosome in black goat图5 黑色山羊剪接体类型Fig.5 Spliceosome types in black goat
3 讨 论
3.1 山羊Agouti基因5′非翻译区序列变异
山羊Agouti基因5′非翻译区外显子1存在不同类型剪接体,之前获得的12 847 bp(JN651404+EF587236)基因组序列不足以检测到更多的剪接体类型。研究发现,绵羊的白色是由于Agouti基因附近190 kb(包括AHCY基因和ICTH基因启动子)顺接重复引起的[15],而绵羊CH243-455O4 这条较长的序列跨越了两个重复,CH243-455O4 反向互补序列与12 847 bp这段序列相似性很高,故以绵羊CH243-455O4 反向互补序列为模板扩增12 847 bp 之前部分序列。另外,将绵羊CH243-455O4 反向互补序列与绵羊ItIt′IA(EU420026.1)、ItIt′(EU420024.1)比对,CH243-455O4的反向互补序列中包括53 571~53 651、57 572~57 650、64 870~64 928 bp的3段序列预测为外显子1的不同剪接体类型,故也扩增了CH243-455O4反向互补序列中包括该3段的序列。
获得的基因组序列与绵羊mRNA序列比对分析后,分别得到长度为88、180、81和79 bp的山羊Agouti基因5′非翻译区序列,推测山羊Agouti基因5′非翻译区可能包含4个或更多不同长度的非编码外显子,而5′ RACE所检测到的外显子1可变剪接体基本与此对应。
3.2 山羊外显子1剪接体变异
菌液测序得出的所有序列结果首先进行Blast分析,将非特异性扩增的序列去除,各个测序结果均与山羊基因组的相似性在98% 以上,只是结合的位置与长短略有差别,此部分结果与牛、绵羊中的报道一致[16-17]。对白、黑、棕色羊驼Agouti基因进行5′RACE检测,发现了3种不同长度的5′非翻译区序列,分别为136、142、196 bp;长度为196 bp的5′ 非翻译区序列与牛外显子1C的相似性高达85%[17]。本研究中白色山羊(B1、B2、B3),黑色山羊(H1、H2)分别获得2种或者大于2种5′ 非翻译区序列,其长度均不相同,与羊驼研究结果类似。5′RACE结果发现,白色山羊B2 存在5种类型剪接体,分别是It、It′、IA、2C、IE,白色山羊均存在It、It′ 2种类型,B3也检测到IE 剪接体的存在,每只山羊得出的剪接体的类型与数目不同,这与山羊个体本身也有关;黑色山羊H1、H2存在2种类型剪接体:It、It′,黑色山羊H3只存在一段很短的It′,可能与H3 RNA提取的完整性较差相关,或者H3本身就只有一种类型的剪接体。分析得出:白色、黑色山羊Agouti基因外显子1剪接体类型不同,不同的剪接形式可能产生不同毛色,IA、2C、IE可变剪接形式只在白色山羊中发现,推测:IA、2C、IE可变剪接体可能与山羊白色形成有关。黑色、白色山羊It、It′ 均存在不同程度的缺失,推测:可能是在反转录过程中由于使用的是特异性引物,包含It、It′ 的两段序列反转录后得到较多长度不同的片段,后续,需将5′RACE试验结果通过RT-PCR或qRT-PCR进行验证,进一步证实此部分结果的可靠性。根据白色、黑色山羊It、It′ 以及白色山羊IA在基因组上所处的位置推测:5′ RACE 所检测的4只白色山羊和3只黑色山羊均存在拷贝变异,而绵羊的白色是由于Agouti基因附近190 kb顺接重复的启动子引起位于下游的Agouti基因表达从而表现白色,黑色绵羊Agouti基因不表达,山羊所得结果与绵羊有差异,后续可以通过试验验证从而进一步确定拷贝是否确实存在。
4 结 论
4.1 扩增获得的基因序列与12 847 bp(JN651404+EF587236)序列拼接获得了长度分别为27 793和27 858 bp白色和黑色山羊Agouti基因序列。扩增获得CH243-455O4的反向互补序列中包括53 571~53 651、57 572~57 650和64 870~64 928 bp 3段序列,白色山羊获得的包含此3段序列长度分别为364、2 625和1 296 bp;黑色山羊获得的包含此3段序列的长度分别为367、2 622和1 298 bp。
4.2 获得了与绵羊mRNA序列相匹配的长度分别为88、180、81和79 bp的山羊Agouti基因5′非翻译区序列,其中的180、81和79 bp为新发现片段,推测山羊Agouti基因5′非翻译区可能包含4个或更多不同长度的非编码外显子,与5′RACE所检测到的外显子1可变剪接体长度有所对应。
4.3 白色山羊中检测到5种类型的外显子1剪接体,即It、It′、IA、2C和IE,黑色山羊仅检测到It和It′ 2种可变剪接体。剪接体之间和剪接体内部均存在长度变异。IA、2C和IE可变剪接体可能与山羊毛色形成有关。
[1] 潘红梅.KIT基因与荣昌猪毛色相关性研究[D].重庆:西南大学,2006. PAN H M.Study on the relationship betweenKITgene and coat color of Rongchang pig[D].Chongqing:Southwest University,2006.(in Chinese)
[2] 亏开兴,杨国荣,朱芳贤,等.BMY牛与婆罗门牛的毛色遗传[J].黄牛杂志,2005,4(31):19-21. QU K X,YANG G R,ZHU F X,et al.Coat color inheritance of BMY and Brahman cattle[J].JournalofYellowCattleScience,2005,4(31):19-21.(in Chinese)
[3] BYRD J C,BRESALIER R S.Mucins and mucin binding proteins in colorectal cancer[J].CancerMetastasisRev,2004,23(1-2):77-99.
[4] 何孟颉.家兔MITF基因同源性、多态性分析及不同时期表达水平研究[D].扬州:扬州大学,2012. HE M J.Homology,polymorphism analysis and expression levels in different stages ofMITFgene in rabbit[D].Yangzhou:Yangzhou University,2012.(in Chinese)
[5] 白小青,王金勇.猪毛色遗传的研究进展[J].动物科学与动物医学,2004,8(21):32-33. BAI X Q,WANG J Y.The advance of researches of genetics of coat color of pigs[J].AnimalScienceandVeterinaryMedicine,2004,8(21):32-33.(in Chinese)
[6] ARGESON A C,NELSON K K,SIRACUSA L D.Molecular basis of the pleiotropic phenotype of mice carrying the hypervariable yellow(Ahvy) mutation at the agouti locus[J].Genetics,1996,142(2):557-567.(in Chinese)
[7] 刘琳玲,邢秀梅,王桂武,等.Agouti基因研究进展[J].黑龙江畜牧兽医,2011,(8):37-38. LIU L L,XING X M,WANG G W,et al.Progress in the study ofAgoutigene[J].HeilongjiangAnimalScienceandVeterinaryMedicine,2011,(8):37-38.(in Chinese)
[8] SEARLE A G.Comparative genetics of coat color in mammals[M].London:Logos Press,1968.
[9] LUSH J L.Inheritance of horns wattles,and color in grade Toggenburg goats[J].JHered,1926,17:73-91.[10] ASDELL S A,SMITH A D B.Inheritance of color,beard,tassels and horns in the goat[J].JHered,1928,19(9):425-430.
[11] SCHIBLER L,VAIMAN D,OUSTRY A,et al.Comparative gene mapping:a fine-scale survey of chromosome rearrangements between ruminants and humans[J].GenomeRes,1998,8(9):901-915.
[12] TANG C J,ZHOU R Y,LI X L,et al.Variation of 423 G>T in theAgoutigene exon 4 in indigenous Chinese goat breeds[J].BiochemGenet,2008,46(11-12):770-780.
[13] 吴伟伟.山羊Agouti基因5′UTR变异及ASIP蛋白表达定位研究[D].保定:河北农业大学,2012. WU W W.Study on the variation ofAgoutigene 5′ UTR and ASIP location in goat[D].Baoding:Agricultural University of Hebei,2012.(in Chinese)
[14] 刘 佳,董常生,范瑞文,等.周期素依赖性蛋白激酶-5在不同毛色羊驼皮肤中的表达[J].畜牧兽医学报,2010,41(4):478-483. LIU J,DONG C S,FAN R W,et al.Expression of cyclin dependent kinase 5 in alpaca skin of different hair colors[J].ActaVeterinariaetZootechnicaSinica, 2010,41(4):478-483.(in Chinese)
[15] NORRIS B J,WHAN V A.A gene duplication affecting expression of the ovineASIPgene is responsible for white and black sheep[J].GenomeRes,2008,18(8):1282-1293.
[16] GIRARDOT M,GUIBERT S,LAFORET M P,et al.The insertion of a full lengthBostaurusLINE element is responsible for a transcriptional deregulation of the NormandeAgoutigene[J].PigmentCellRes,2006,19(4):346-355.
[17] CHANDRAMOHAN B,RENIERRI C,LA MANNA V,et al.The alpacaagoutigene:Genomic locus,transcripts and causative mutations of eumelanic and pheomelanic coat color[J].Gene,2013,521(2):303-310.
(编辑 郭云雁)
Study onAgoutiSpliceosome Types in Goat Skin with Different Coat Color
ZHANG Tian1,2,3,LI Xiang-long1,2*,ZHOU Rong-yan2,LI Lan-hui2
(1.HebeiNormalUniversityofScience&Technology,Qinhuangdao066600,China;2.AgriculturalUniversityofHebei,Baoding071000,China;3.JinzhouVocationalEducationCenter,Jinzhou052260,China)
The objective of this study was to investigate the variant types of goatAgoutigene spliceosome in goat skin with different coat color.The sequences and mRNA of goatAgoutigene were obtained and analyzed from goat skin with different coat color by PCR and 5′RACE methods,respectively.The 27 793 and 27 858 bp of partialAgoutigene sequences were obtained from white and black goat skin,respectively.Four fragments,88,180,81 and 79 bp,in which the 180,81 and 79 bp were first detected in goat,were matched with the sequences of sheep mRNA and corresponded with the length of exon 1 spliceosome obtained by 5′RACE.The 5 types(It,It′,IA,2C and IE) of spliceosome were detected in goat skin with white coat color,and only 2 types(It and It′) were found in goat skin with black coat color.There was length variation within and among spliceosomes.The result indicate that the IA,2C and IE might be related with the formation of goat coat color.
goat;Agoutigene;skin tissue;spliceosome types
10.11843/j.issn.0366-6964.2015.11.004
2014-12-31
国家自然科学基金(31172196);河北省高校创新团队领军人才培育计划(LJRC004);河北省应用基础研究计划重点基础研究项目(15962901D)
张 天(1987-),女,河北藁城人,硕士,主要从事动物遗传育种研究,E-mail:zhangtian19870417@163.com
*通信作者:李祥龙,博士,教授,博导,主要从事动物遗传育种研究,E-mail:Lixianglongcn@yahoo.com
S827;S813.3
A
0366-6964(2015)11-1934-10
