应用单细胞cDNA扩增技术鉴定循环肿瘤细胞
2015-03-20王玲兄李晶晶卜芳芳
王玲兄,潘 飞,李晶晶,卜芳芳
解放军总医院 肿瘤中心实验室,北京 100853
应用单细胞cDNA扩增技术鉴定循环肿瘤细胞
王玲兄,潘 飞,李晶晶,卜芳芳
解放军总医院 肿瘤中心实验室,北京 100853
目的建立单细胞cDNA扩增技术,实现在分子水平鉴定循环肿瘤细胞。方法建立结肠癌循环肿瘤细胞模型,通过免疫荧光染色和显微切割技术分离出单个血细胞和肿瘤细胞;裂解细胞后利用碱基修饰的P1(dT)24引物反转录出cDNA第一链,并给第一链cDNA加上PolyA尾后利用碱基修饰的P2(dT)24引物合成第二链cDNA,然后利用P1(dT)24和P2(dT)24引物扩增分别得到血细胞和肿瘤细胞的全长cDNA;最后通过PCR检测细胞特异的标记物CD45、EpCAM和CK19。结果改良后的单细胞cDNA扩增方法能够获得高质量的全长cDNA,并能显著增加PCR检测效率;利用扩增得到的cDNA通过PCR检测细胞表面标记物CD45、EpCAM和CK19的表达,能有效鉴定出外周血中混杂的肿瘤细胞。结论通过单细胞cDNA扩增技术在基因水平识别肿瘤细胞,建立了一种新的循环肿瘤细胞的鉴定方法。
单细胞cDNA扩增技术;循环肿瘤细胞;激光捕获显微切割;聚合酶链式反应
解析单细胞的行为对于深入认识肿瘤、神经系统疾病等尤为重要。由于单细胞RNA量少且稳定性差,相对于基因组DNA水平单细胞转录组RNA水平的检测技术尚不成熟。Brady和Iscove[1]最早建立了基于PCR技术对单个细胞cDNA指数扩增的方法,随后Eberwine等[2]建立了基于T7 RNA连接酶体外转录的cDNA的线性扩增的方法,成为单细胞转录组RNA检测的初步探索。2006年,Kurimoto等[3]改进了单细胞cDNA扩增方法,将定向的PCR扩增与线性扩增相结合,在此基础上研究者们不断对该技术进行改进,以求在高覆盖率和高准确性的前提下,使基因表达的代表性和再现性都有了明显的提高[4-6]。近来循环肿瘤细胞成为肿瘤研究的热点,肿瘤细胞经过血液循环播散至远隔器官是恶性肿瘤患者最主要的死亡原因[7]。循环肿瘤细胞的数量及表型的改变是研究恶性肿瘤生物学特征及肿瘤进展的窗口[8];更重要的是循环肿瘤细胞是肿瘤细胞异质性的典型表现,通过研究循环肿瘤细胞的生物学行为、遗传及表观遗传学改变对揭示肿瘤的转移及耐药机制非常重要[9-11]。由于循环肿瘤细胞量少、不易捕获,单细胞技术已广泛应用于循环肿瘤细胞的研究中[12-14]。本文中,我们以结肠癌循环肿瘤细胞为模型,通过显微切割技术捕获单个肿瘤细胞;并且成功建立单细胞cDNA扩增的技术,获得了高质量的循环肿瘤细胞的全长cDNA,最后利用PCR技术检测肿瘤细胞标记物,实现了在基因水平鉴定循环肿瘤细胞。
材料和方法
1 材料 固定透膜液(美国BD Cytofix/Cytoperm公司),显微切割PEN膜(德国Lecia公司),10×PCR bufferⅡ和MgCl2(美国Applied Biosystems公司),DTT、SuperScriptⅢ、dATP mix、RNase H、rTdT和细胞核复染剂DAPI(美国Invitrogen公司),Rnase inhibitor和PCR产物纯化试剂盒(德国Qiagen公司),Ribonuclease inhibitor、dNTP mix、10×ExonucleaseⅠ、10×ExTaq buffer和ExTaq Hot Start Version(大连Takara公司),DDW(美国Ambion公司),T4 gene 32 protein(瑞士Roche公司),抗EpCAM抗体、抗CD45单克隆抗体(美国BD公司)。
2 细胞培养 结肠癌肿瘤细胞系HCT116细胞用含10%胎牛血清的DMEM培养基,于37℃、5% CO2细胞培养箱中培养。每2 ~ 3 d传代1次,将培养的肿瘤细胞经胰酶消化后,取部分悬液备用。3 外周血循环肿瘤细胞模型建立 用ACD枸橼酸采血管采取正常献血者肘正中静脉清晨空腹外周静脉血2 ml。室温300×g离心10 min,弃血清,加入红细胞裂解液,室温避光孵育15 min后,200×g离心5 min,小心吸弃上清,重复裂解1次,去除外周血中的红细胞,计数剩余白细胞的数量,后按每107个白细胞加入1 000个肿瘤细胞的比例,向所得白细胞悬液中加入结肠癌细胞HCT116。所得混合细胞悬液室温300×g离心5 min,吸弃上清后加入200μl固定透膜液,4℃避光30 min,离心弃上清,缓冲液洗涤两次,10% BSA封闭15 min后,用PE/Cy5-anti-CD45和FITC-anti-EpCAM避光染色20 min,再用细胞核复染剂DAPI染核。将细胞稀释至浓度约为105/ml,滴加至显微切割专用的PEN膜50 mm平皿内备用[12]。
4 激光捕获显微切割获得单细胞 将细胞悬液均匀涂布于PEN膜上,置于显微切割仪(LMD6000,Lecia)的卡槽内,在其下方放置RNase-free 200μl PCR管用于收集切割下的细胞。PCR管盖正对玻片,管盖内加入4.5μl细胞裂解液(表1)。收集到的细胞如图1所示,扣紧管盖后最高转速离心,使细胞片落入细胞裂解液中,冰上保存。每个PCR管收集1个单细胞,全部细胞切割分离要在2 h内完成[15]。
5 单细胞全长cDNA扩增 首先,将上一步所得含单细胞的PCR管置于70℃金属浴90 s后冰浴1 min,充分裂解细胞,同时裂解液中的cDNA第一链引物P(dT)24与模板RNA变性并退火结合(图1)。然后加入0.3μl反转录预混液(表2),反转录合成cDNA第一链;反应条件:50℃,5 min;70℃,10 min;冰浴1 min以上。加入1μl外切酶预混液(表3),去除体系中游离的cDNA第一链引物;反应条件:37℃,30 min;80℃,25 min;冰浴1 min以上。加入6μl末端脱氧核苷酸转移酶(TdT)预混液(表4),在第一链cDNA 3’端加Poly A尾并去除体系中的RNA;反应条件:37℃,15 min;70℃,10 min;冰浴1min以上。将上一步所得样品分装到4个PCR管,每管3μl,加入19μl PCR预混液Ⅰ(表5)合成cDNA第二链;反应条件:95℃变性,3 min;50℃退火,2 min;72℃延伸,3 min;冰浴1 min以上;合成双链cDNA,模式图见图1。每管内加入19μl PCR混合物Ⅱ(表6),并加入1滴矿物油,扩增双链cDNA;反应条件:95℃变性,30 s;67℃退火,1 min;72℃延伸,3 min,延伸时间每个循环增加6 s;共24个循环。最后,将所得PCR产物(4个PCR管,共计164μl)混合,用PCR产物纯化试剂盒提纯产物,用50μl EB缓冲液进行洗脱。所得cDNA扩增产物于-80℃冰箱保存1年以上。第一链cDNA引物P1(dT)24序列:AT ATGGATCCGGCGCGCCGTCGACTTTTTTTTTTTTTTT TTTTTTTTT;第二链cDNA引物P2(dT)24序列:ATA TCTCGAGGGCGCGCCGGATCCTTTTTTTTTTTTTTT TTTTTTTTT。
6 细胞表面标记物的PCR扩增 对白细胞特异性标记物CD45和上皮型肿瘤细胞表面标记物EpCAM和CK19/CK19进行PCR检测。分别以0.4μl单细胞裂解液或单细胞cDNA扩增产物为模板,进行30个循环的扩增。扩增产物通过2%浓度琼脂糖凝胶电泳检测。β-actin引物序列,F:5'CTCCATCCTGGCCTCGCTGT3',R:5'GCTGTCAC CTTCACCGTTCC3';EpCAM引物序列,F:5'AATCG TCAATGCCAGTGTACTT3',R:5'TCTCATCGCAGT CAGGATCATAA3';CK19,F:5'ACCAAGTTTGAG ACGGAACAG3',R:5'CCCTCAGCGTACTGATTTC CT3';CD45引物序列,F:5'ACAGCCAGCACCTTT CCTAC3',R:5'GTGCAGGTAAGGCAGCAGA3'。

图 1 单细胞双链cDNA模式图Fig. 1 Schematic representation of double-stranded cDNA

表1 单细胞裂解液配方Tab. 1 Lysate formula of single cell

表2 反转录预混液配方Tab. 2 Mixture of reverse transcription system

表3 外切酶预混液配方Tab. 3 Mixture of ExoⅠ

表4 末端脱氧核苷酸转移酶预混液配方Tab. 4 Mixture of TdT

表5 PCR预混液Ⅰ配方Tab. 5 MixtureⅠof PCR

表6 PCR预混液Ⅱ配方Tab. 6 MixtureⅡof PCR
结 果
1 显微切割获得单细胞 免疫荧光染色鉴别出肿瘤细胞和白细胞,分别进行切割;其中绿色荧光标记而红色荧光缺失的细胞为肿瘤细胞,红色荧光标记而绿色荧光缺失的细胞为白细胞。见图2。
2 cDNA扩增成功 如图3A所示,将1 ~ 12号单细胞的cDNA扩增产物进行2%浓度琼脂糖凝胶电泳,其中10号和12号的扩增产物聚集在100 ~300 bp,认为是存在RNA的降解或引物二聚体的污染;其余cDNA扩增产物弥散范围在300 ~ 2 000 bp,均为合格扩增产物,且不含基因组DNA污染。
3 cDNA扩增增加PCR敏感性 分别以扩增前和扩增后的单细胞cDNA为模板,对β-actin基因进行PCR扩增,所得产物进行琼脂糖凝胶电泳,结果显示,以扩增后的cDNA为模板均能高效扩增出β-actin基因;但用未经过扩增的cDNA为模板进行扩增β-actin基因时,仅6号样本扩增效率高,其余样本扩增效率低或扩增失败;结果证实,经过cDNA扩增能够显著增加PCR的敏感性。见图3B。
4 细胞特异性基因扩增鉴定循环肿瘤细胞 按照免疫荧光染色的结果将样本分为白细胞组和肿瘤细胞组,选择了白细胞特异性标记基因CD45和上皮细胞表面标记基因EpCAM和CK19进行PCR检测,结果显示,白细胞组CD45均扩增成功,而EpCAM扩增失败(图4A);相反,肿瘤细胞组CD45扩增失败,而EpCAM和CK19扩增成功(图4B),结果证实通过细胞特异性基因扩增能够鉴定出外周血中的肿瘤细胞。
讨 论
本文旨在建立一种单细胞cDNA扩增技术,实现在单细胞表达谱水平鉴定和研究循环肿瘤细胞。循环肿瘤细胞的捕获技术有了突飞猛进的发展,然而关于循环肿瘤细胞的鉴定和后续研究技术尚不成熟[16-17]。
我们首先建立了结肠癌的外周血循环肿瘤细胞模型,结肠癌细胞HCT116是上皮来源的肿瘤细胞,同时表达上皮细胞的表面标记物EpCAM和CK19[18],而血源性细胞的表面标记物CD45表达为阴性,所以利用HCT116细胞建立外周血循环肿瘤细胞模型有利于进行后期研究。我们通过免疫荧光染色找到疑似肿瘤细胞和疑似白细胞,对这些细胞进行显微切割成单个细胞,然后对细胞进行裂解,以含RNA的裂解液为模板,通过用修饰的Oligo dT法进行单细胞cDNA扩增,得到高质量的全长cDNA;我们的扩增方法是在2007年Kurimoto等[4]的技术上改进的,一方面,我们利用裂解液为模板进行扩增,较先提取RNA后扩增的方法能够显著增加cDNA的得率;另一方面,我们选择两段非人类基因组序列的24个碱基序列修饰Oligo dT作为扩增引物,将细胞内含有PolyA尾的全部mRNA反转成两端具有特异序列的双链cDNA。再以两段修饰的Oligo dT作为引物,进行PCR以达到扩增cDNA的目的。这种通过对特异性序列引入的方法,大大提高了扩增的效率,也避免了扩增循环中引入过长Oligo dT的困扰。但是这种技术也存在一定缺陷,其仅能扩增mRNA 3'端的300 ~ 2 000 bp的序列。

图 2 显微切割前荧光染色鉴定细胞种类。 EpCAM阳性CD45阴性细胞为肿瘤细胞;EpCAM阴性CD45阳性细胞为白细胞Fig. 2 Cells were idetified by immunofluorescence staining and isolated by laser capture microdissection. Cells with positive EpCAM and negative CD45 showed as tumor cells, and cells with negative EpCAM and positive CD45 showed as white cells

图 3 A:单细胞cDNA扩增产物电泳 B:单细胞cDNA 为模板PCR扩增β-actin产物电泳Fig. 3 A: Electrophoresis of amplified products by single-cell cDNA; B: Electrophoresis of β-actin amplified by PCR with singlecell cDNA as sample
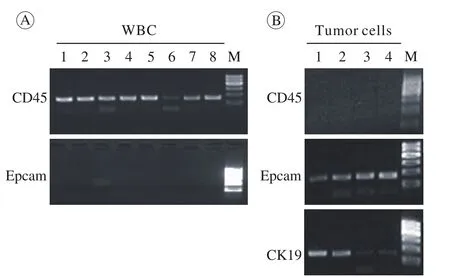
图 4 以扩增后的单细胞cDNA为模板PCR扩增CD45、EpCAM和CK19,白细胞为CD45+EpCAM-细胞(4A),肿瘤细胞为CD45-EpCAM+CK19+细胞(4B)Fig. 4 CD45, EpCAM and CK19 amplified by gene specific PCR with amplified single-cell cDNA as sample, the white cells were shown as CD45+EpCAM-(4A), and tumor cells were shown as CD45-EpCAM+CK19+(4B)
目前,单细胞cDNA扩增技术多被应用于单细胞表达谱芯片技术,而对于没有条件进行芯片研究的实验室,通过将单个肿瘤细胞极少量cDNA扩增,使其达到普通PCR可检测的浓度,进行特异基因的常规检测,可对肿瘤细胞的性质、来源、分化等进一步确认,还可对其EMT相关基因表达水平进行检测,实现更为准确地研究和评估肿瘤预后的目的。也可用于送检芯片前的预检测或对芯片结果的再验证,以节约实验成本,使单细胞PCR检测灵敏度大大提高。为今后研究单个肿瘤细胞提供了更敏感的检测方法。
1 Brady G, Iscove NN. Construction of cDNA libraries from single cells[J]. Methods Enzymol, 1993, 225: 611-623.
2 Eberwine J, Yeh H, Miyashiro K, et al. Analysis of gene expression in single live neurons[J]. Proc Natl Acad Sci U S A, 1992, 89(7):3010-3014.
3 Kurimoto K, Yabuta Y, Ohinata Y, et al. An improved single-cell cDNA amplification method for efficient high-density oligonucleotide microarray analysis[J]. Nucleic Acids Res, 2006, 34(5): e42.
4 Kurimoto K, Yabuta Y, Ohinata Y, et al. Global single-cell cDNA amplification to provide a template for representative high-density oligonucleotide microarray analysis[J]. Nat Protoc, 2007, 2(3):739-752.
5 Trimarchi JM, Stadler MB, Roska B, et al. Molecular heterogeneity of developing retinal ganglion and amacrine cells revealed through single cell gene expression profiling[J]. J Comp Neurol, 2007,502(6): 1047-1065.
6 Goetz JJ, Trimarchi JM. Single-cell profiling of developing and mature retinal neurons[J]. J Vis Exp, 2012, 19(62): pii:3824.
7 Plaks V, Koopman CD, Werb Z. Cancer. Circulating tumor cells[J]. Science, 2013, 341(6151):1186-1188.
8 Schwed Lustgarten DE, Thompson J, Yu G, et al. Use of circulating tumor cell technology (CELLSEARCH) for the diagnosis of malignant pleural effusions[J]. Ann Am Thorac Soc, 2013, 10(6): 582-589.
9 Powell AA, Talasaz AH, Zhang H, et al. Single cell profiling of circulating tumor cells: transcriptional heterogeneity and diversity from breast cancer cell lines[J]. PLoS One, 2012, 7(5):e33788.
10 Yu M, Ting DT, Stott SL, et al. RNA sequencing of pancreatic circulating tumour cells implicates WNT signalling in metastasis[J]. Nature, 2012, 487(748): 510-513.
11 Aceto N, Bardia A, Miyamoto DT, et al. Circulating tumor cell clusters are oligoclonal precursors of breast cancer metastasis[J]. Cell, 2014, 158(5): 1110-1122.
12 Ran R, Li L, Wang M, et al. Determination of EGFR mutations in single cells microdissected from enriched lung tumor cells in peripheral blood[J]. Anal Bioanal Chem, 2013, 405(23):7377-7382.
13 Chen CL, Mahalingam D, Osmulski P, et al. Single-cell analysis of circulating tumor cells identifies cumulative expression patterns of EMT-related genes in metastatic prostate cancer[J]. Prostate,2013, 73(8): 813-826.
14 Navin N, Kendall J, Troge J, et al. Tumour evolution inferred by single-cell sequencing[J]. Nature, 2011, 472(7341): 90-94.
15 Zhao L, Lu YT, Li F, et al. High-purity prostate circulating tumor cell isolation by a polymer nanofiber-embedded microchip for whole exome sequencing[J]. Adv Mater, 2013, 25(21): 2897-2902.
16 Maheswaran S, Haber DA. Ex vivo culture of CTCs: an emerging resource to guide cancer therapy[J]. Cancer Res, 2015, 75(12):2411-2415.
17 Deng Y, Zhang Y, Sun S, et al. An integrated microfluidic chip system for single-cell secretion profiling of rare circulating tumor cells[J]. Sci Rep, 2014, 4: 7499.
18 Zhou FQ, Qi YM, Xu H, et al. Expression of EpCAM and Wnt/ β-catenin in human colon cancer[J]. Genet Mol Res, 2015, 14(2):4485-4494.
Identification of circulating tumor cells by single-cell cDNA amplification techniques
WANG Lingxiong, PAN Fei, LI Jingjing, BU Fangfang
Cancer Center Lab, Division of Internal Medicine, Chinese PLA General Hospital, Beijing 100853, China
BU Fangfang, Email: bflily@163.com
ObjectiveTo identify circulating tumor cells by single-cell cDNA amplification techniques.MethodsIndividual tumor cells from blood cells were separated by immunofluorescence staining, and then the cells were lysed and total RNA was protected by the lysate. First strand cDNA was transcripted using modified primer P1(dT)24and then tailed with poly dA. Second-strand cDNA was synthesized using modified primer P2(dT)24. Then the double-strand cDNA was amplificated using primer P1(dT)24and P2(dT)24.At last, the amplificated cDNA was used to detect cell specific marker of CD45, EpCAM and CK19 by PCR.ResultsOur results showed that the high-quality full-length cDNA was produced by the single-cell cDNA amplification techniques which could improve the effect of PCR significantly. The expression of CD45, EpCAM and CK19 detected by cDNA amplification techniques combined with gene specific PCR could identify tumor cells from blood cells effectively.ConclusionSingle-cell cDNA amplification techniques can identify tumor cells from blood cells.
single-cell cDNA amplification techniques; circulating tumor cells; laser capture microdissection; polymerase chain reaction
R 730.43
A
2095-5227(2015)12-1222-05
10.3969/j.issn.2095-5227.2015.12.017
时间:2015-09-24 17:13:03
http://www.cnki.net/kcms/detail/11.3275.R.20150924.1713.008.html
2015-06-17
国家自然科学基金项目(81301781)
Supported by the National Natural Science Foundation of China (81301781)
王玲兄,女,本科,技师。研究方向:肿瘤分子靶向治疗的驱动基因研究。Email: lingxiongw@163.com
卜芳芳,女, 博士,助理研究员。Email: bflily@163.com
