不同牧草来源饲粮对奶牛瘤胃液中细菌群落结构多样性的影响
2015-03-14胡丽芳侯玉洁孙建勇赵国琦
徐 俊 胡丽芳 侯玉洁 孙建勇 赵国琦
(1.江西省农业科学院农产品质量安全与标准研究所,南昌 330200;2.扬州大学动物科学与技术学院,扬州 225009;3.新疆阿勒泰市第一牧场畜牧兽医站,阿勒泰 836500)
反刍动物瘤胃里面栖息着大量微生物,包括细菌、真菌和原虫等[1],纤维在瘤胃微生物作用下降解为挥发性脂肪酸为机体供能,而饲粮是影响瘤胃微生物组成和多样性的关键因素[2-3],研究不同饲粮组成对瘤胃细菌群落结构多样性的影响,对进一步研究瘤胃微生物对饲粮的降解和提高动物生产性能具有十分重要的意义[4]。Thoetkiattikul等[5]利用高通量测序技术研究了不同纤维和淀粉比例饲粮对奶牛瘤胃微生物特性的影响,发现瘤胃中主要的细菌包括拟杆菌门、硬壁菌门和变形菌门,且细菌丰富度与饲粮纤维含量密切相关。Tajima等[6]借助PCR技术和16S rDNA克隆文库测序的方法研究了干奶牛饲粮由高粗料变为高精料后瘤胃液中第0(干草9kg,精料3kg)、3(干草 0.5 kg,精料 12 kg)和 28 天(干草 0.5 kg,精料12 kg)的微生物区系变化,发现2种饲粮中优势菌群都是革兰氏阳性菌,且高精料饲粮中月形单胞菌属、解琥珀酸菌属和巨型球菌属为优势菌属。Mosoni等[7]发现高粗料饲粮条件下的优势菌属为丁酸弧菌属和普雷沃氏菌属,其中链球菌、乳酸杆菌、反刍兽新月形单胞菌和嗜淀粉瘤胃杆菌为优势菌。然而,前人研究多集中在饲粮精粗比和淀粉含量对瘤胃微生物的影响,很少考虑纤维来源和饲粮营养组分对瘤胃微生物的影响。因此,本研究选用我国牧场常用的4种牧草(苜蓿、燕麦草、羊草和稻草)为粗料来源,以玉米青贮为基础配制4组等能等氮且中性洗涤纤维(neutral detergernt fiber,NDF)和非纤维性碳水化合物(non-fiber carbohydrates,NFC)含量相同的饲粮,借助M iseq高通量测序技术研究不同牧草来源饲粮对泌乳奶牛瘤胃细菌群落结构多样性的影响。
1 材料与方法
1.1 试验动物、饲粮及设计
选用8头体重为(632±12)kg,泌乳天数为(135±16)d的泌乳中期荷斯坦奶牛,随机分为4组,采用4×4拉丁方设计,每组2头。分别以燕麦草、羊草、稻草和苜蓿为牧草来源,给奶牛饲喂以玉米青贮为基础、等能等氮且NDF和NFC含量相同的4种饲粮;每期21 d,前14 d为预试期。以全混合日粮(TMR)形式,日喂 3次(06:30、14:30、22:30)。日挤奶 3 次(07:00、15:00、23:00)。单栏栓系式饲养,自由饮水。试验饲粮组成及营养水平见表1。
1.2 瘤胃液样品采集与分析
每期试验的第19~20天通过口腔采集法采集晨饲后4 h(10:30)4组奶牛(每组各1头)瘤胃液样品50 m L[5],采用粪便基因组提取试剂盒(QIAGEN公司)提取瘤胃液总DNA,提取方法参照试剂盒说明书。瘤胃液样品分别编号为:燕麦草组(1、2、3 和 4)、羊草组(5、6、7 和 8)、稻草组(9、10、11 和 12)和苜蓿组(13、14、15 和 16)。根据细菌16S rDNA基因V3区的保守序列,设计通用引物 338F:5'-ACTCCTACGGGAGGCAGCAG-3',533R:5'-TTACCGCGGCTGCTGGCAC-3'。PCR扩增采用25μL反应体系,反应程序为:94℃预变性5 m in;94℃变性30 s,58℃退火30 s,72℃延伸30 s,进行21个循环;最后72℃延伸5 m in;结束时4℃保存。PCR扩增产物用1.5%琼脂糖凝胶电泳进行检测,然后使用Axygen DNA胶回收纯化试剂盒(Axy Prep DNA Gel Extration kit,APGX-500)对V3区扩增产物进行切胶回收纯化,经Biotek酶标仪对纯化好的PCR产物进行定量,将样品等量混匀后形成均一混合物。PCR混合物经质量控制后,采用标准的Illum ina TruSeq DNA文库制备流程构建Illum ina测序文库,最后按照Illum ina M iseq平台上机进行Barcoded Illum ina M iseq测序。
1.3 数据处理与分析
测序结束后,对原始序列数据进行质量控制得到有效序列,再对有效序列进行去杂处理,丢弃长度短于120 bp、含有模糊碱基,引物碱基含2个以上的错配信息、单个碱基重复数超过6个的序列,最终获得用于后续分析的优质序列。通过Qiime分析平台对优质序列进行生物信息学分析,根据序列相似度为97%的原则,将序列归为多个操作分类单元(operational taxonom ic unit,OTU)。然后对序列进行聚类分析,选取每个类最长序列为代表序列,采用RDP-classifier,以RDP数据库的序列为训练集,对OTU代表序列进行注释,得到每个OTU的分类学信息。通过软件Mothur(http://www.mothur.org)对生成的 OTU信息进行细菌群落多样性和丰富度分析。应用软件Cluster 3.0将所有样品在属水平上的分类信息进行聚类后作出Heatmap,同时对微生物群落进行UniFrac分析,从遗传距离上判断不同样本之间的距离远近,比较不同样品间的差异性。
1.4 统计分析
数据采用Excel 2007进行整理,结果采用SAS 8.2统计软件的PDIFF模块进行方差分析和显著性检验,以 P<0.05作为差异显著性判断标准。
2 结果与分析
2.1 细菌丰富度和多样性
通过Illum ina M iseq高通量测序后,本研究16个样品共产生有效序列824 422条,经质量控制后得到800 261条高质量序列,每个样品平均产生50 016条序列,序列平均长度为145 bp。由表2可知,苜蓿组和稻草组有效序列和优质序列数均显著高于燕麦草组和羊草组(P<0.05)。物种丰富度指数Chao指数的范围为6 125~7 847,且苜蓿>稻草>燕麦草>羊草;Ace指数的范围为8 026~10 169,且苜蓿>稻草>羊草>燕麦草,苜蓿组和稻草组均显著高于燕麦草组和羊草组(P<0.05)。物种多样性指数Simpson指数和Shannon指数组间无显著差异(P>0.05),这说明牧草来源对奶牛瘤胃液中的细菌多样性无显著影响。各牧草组文库覆盖率均在94%以上,这表明每个样品的测序量合理,能较好反映各组瘤胃液中细菌群落种类和结构的多样性。
2.2 细菌组成和群落结构
本试验采用RDP和Blast同源性序列对所有样品进行比对,共鉴定得到16个门,28个纲,42个目,64个科和89个属。在门水平上,各组中拟杆菌门、硬壁菌门、螺旋体门和纤维杆菌门均为优势菌门,尤其是拟杆菌门和硬壁菌门分别占序列总数的 39.37% ~45.62%和 22.53% ~25.16%。而变形菌门、TM 7、无壁菌门、疣微菌门、酸杆菌门和蓝细菌门序列含量都较低。
由表3可知,在门水平下,相对含量大于0.1%的门共有10个,试验得出的其余6个相对含量均低于0.1%,丰度很低。此外,各组中还有1%左右(0.93%~1.23%)的序列无法进行归类。统计结果表明:稻草组和苜蓿组中的拟杆菌门分别占测序总量的45.62%和44.37%,显著高于燕麦草组和羊草组(P<0.05),且燕麦草组最低(39.37%),然而硬壁菌门则是燕麦草组最高,占测序总量的25.16%,显著高于其他 3 组(P<0.05)。羊草组中纤维杆菌门、变形菌门和疣微菌门相对含量极显著高于其他3组(P<0.01)。燕麦草组中的酸杆菌门和蓝细菌门相对含量极显著高于羊草组、稻草组和苜蓿组(P<0.01)。各组中的TM 7相对含量无显著差异(P>0.05)。
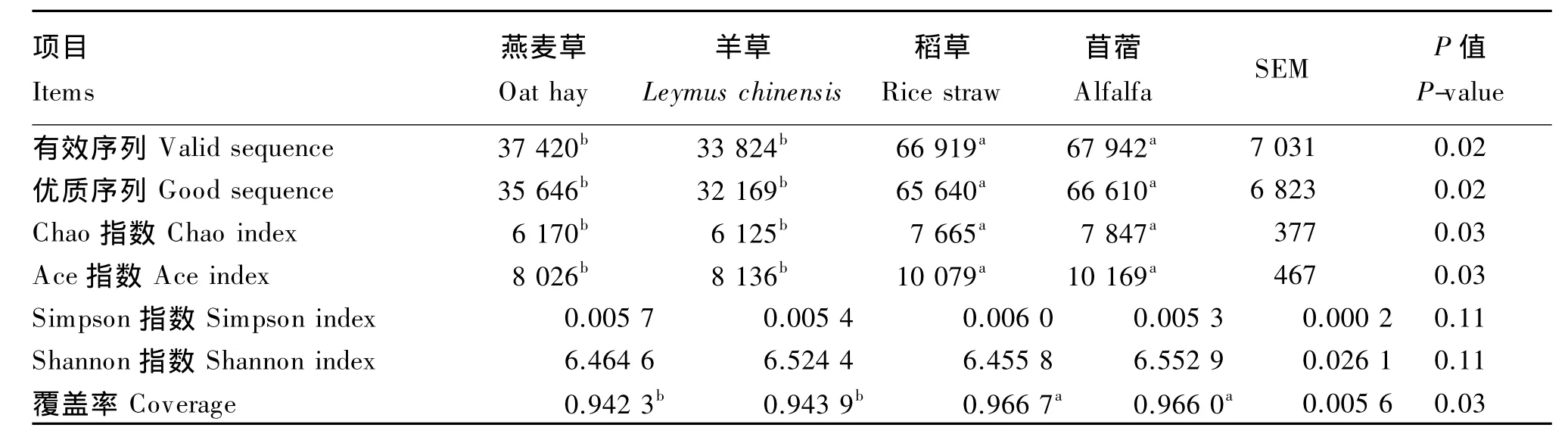
表2 97%相似性水平下物种丰富度和多样性指数Table 2 Richiness and diversity index of species at 97%sim ilarity level
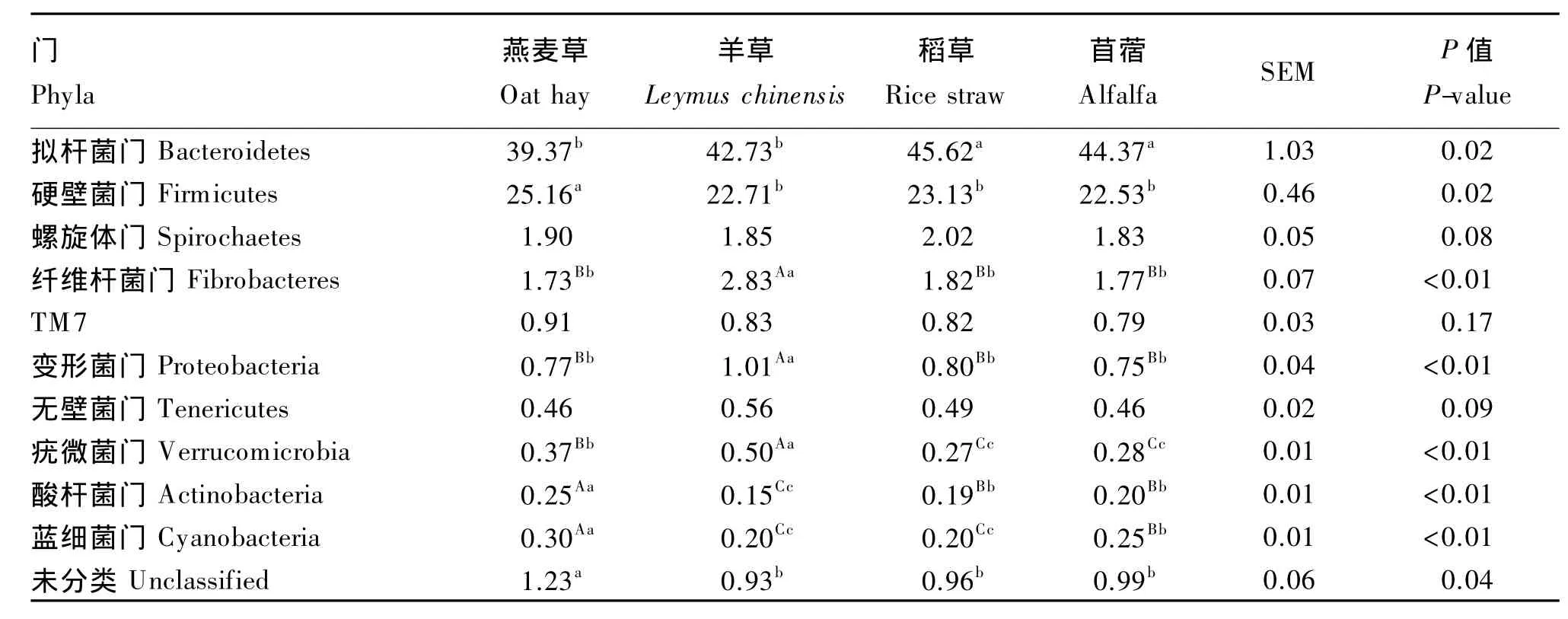
表3 相对含量大于0.1%(序列占测序总量比例)的菌门Table 3 Bacterial phyla w ith relative aboundunce above 0.1%(sequence percentage of total sequence amount) %
由表4可知,在属水平上,序列比对得到的89个属中共有13个菌属相对含量大于0.1%,瘤胃液中有很多相对丰度较低的菌属。在相对含量大于0.1%的菌属中,各组中均以普雷沃氏菌属、丁酸弧菌属、纤维杆菌属和密螺旋体属为优势菌属,且普雷沃氏菌属占测序总量的 19.10%(16.88%~22.84%)。各组间密螺旋体属、厌氧原体属、粪球菌属、毛螺旋菌属和新月形单胞菌属相对含量无显著差异(P>0.05)。稻草组普雷沃氏菌属极显著高于燕麦草组和羊草组(P<0.01),羊草组中纤维杆菌属极显著高于其他3组(P<0.01)。燕麦草组和羊草组丁酸弧菌属显著高于稻草组和苜蓿组(P<0.05),羊草组瘤胃球菌属显著高于其他3组(P<0.05),羊草组琥珀酸弧菌属显著高于燕麦草组和苜蓿组(P<0.05)。

表4 相对含量大于0.1%(序列占测序总量比例)的菌属Table 4 Bacterial genus w ith relative aboundunce above 0.1%(sequence percentage of total sequence amount) %
2.3 组间的相似性分析
将属水平分类信息聚类后得到Heatmap,16个样品分成2大簇,左边第1大簇7个样品(样品1、5、6、7、8、13 和 15),右边第 2 大簇 9 个样品(样品2、3、4、9、10、11、12、14 和 16),同一组样品如羊草(5、6、7、8)和稻草(9、10、11、12)可以很好地聚在一起,但不同组样品之间存在交叉现象(图1)。
基于UniFrac的加权主坐标分析其第1主成分和第 2主成分的贡献率分别为 37.46%和28.73%,除羊草组4个样品可以较好地和其他3组分开外,燕麦草组、稻草组和苜蓿组的遗传距离更接近(图2),该结果与Heatmap的结果相吻合。
我们利用瘤胃液细菌群落中共享和独自拥有的OTU的细菌丰度对4个不同组进行分析,在97%相似性水平下,4个组共产生9 329个OTU(图3),其中燕麦草组、羊草组、稻草组和苜蓿组中 OTU 数目分别为6 710、6 522、8 032 和8 212,4组共享4 120个OTU,占瘤胃液细菌总OTU数目的44.16%,各组独有的 OTU数目仅占 0.39%、0.37%、1.11%和 1.49%。
3 讨论
饲粮组成会影响瘤胃微生物区系,本研究发现稻草组和苜蓿组OTU数目高于燕麦草组和羊草组,在97%相似性水平下丰富度指数Chao指数和Ace指数显著高于燕麦草组和羊草组,这可能与它们优质序列数高有关,该结果与通过16S rRNA基因双末端焦磷酸测序技术研究不同纤维/淀粉饲粮对奶牛瘤胃微生物丰富度的结论相一致[5],给奶牛饲喂营养组分均衡的饲粮瘤胃微生物多样性比高纤维组或高淀粉组更好,然而,本试验各组饲粮营养组分相当,这可能是造成多样性指数Simpson指数和Shannon指数差异不显著的原因。
各组拟杆菌门和硬壁菌门均为优势菌门,不同饲喂组均未发现特有菌门,该结果与前人研究结果一致[5,8-9],拟杆菌门和硬壁菌门有时可高达序列总量的50%和43%[8],改变饲粮中纤维和淀粉比例也未改变它们的优势地位[5]。Thoetkiattikul等[5]发现在高纤维(88%)和低淀粉(2%)饲粮条件下,奶牛瘤胃中拟杆菌门和硬壁菌门菌门含量分别达到66.53%和24.98%,本研究中拟杆菌门数量低于上述结果可能与饲粮中纤维含量(50%)低有关。拟杆菌门是瘤胃中非纤维植物成分的主要降解者,该菌门中的普雷沃氏菌属在瘤胃中含量最高,有报道称普雷沃氏菌属可达瘤胃微生物总量的60%~70%[10],它拥有高活性的半纤维素分解菌[11],并对植物非纤维多糖和蛋白质的降解至关重要[12-13]。此外,普雷沃氏菌属还具有消化和利用淀粉、木聚糖和果胶的能力[14-15]。瘤胃中硬壁菌门含有大量分解纤维的菌属,如瘤胃球菌属、丁酸弧菌属、假丁酸弧菌属、颤螺旋菌属以及真杆菌属[14]。本试验稻草组中硬壁菌门和拟杆菌门之和稍高于其他3组,这可能与稻草组饲粮降解速率更慢需要更多微生物参与降解有关。
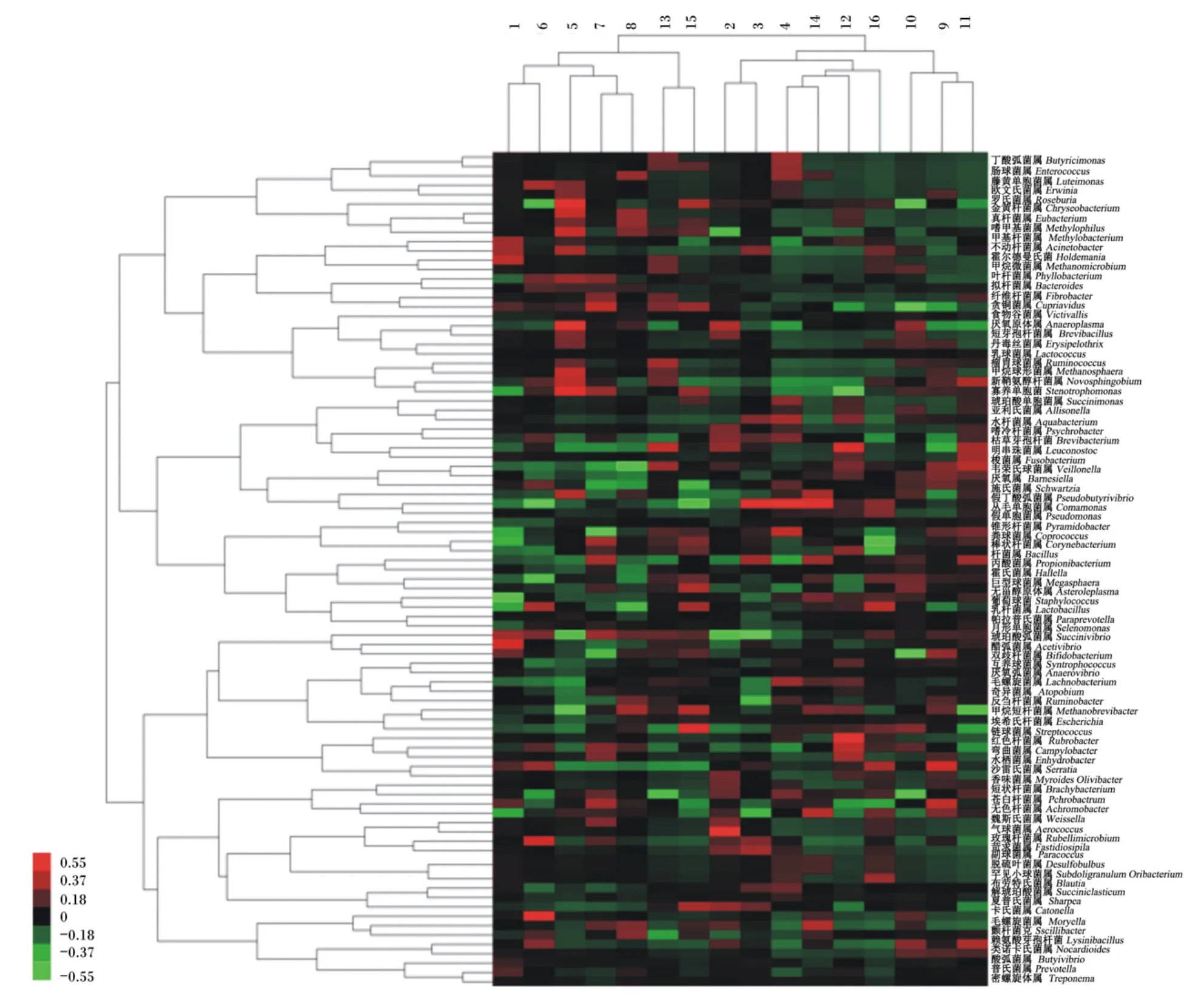
图1 属水平下16个瘤胃液样品的聚类分析Heatm apFig.1 Heatmap under the genus level of the 16 rumen fluid samples
山羊瘤胃中普雷沃氏菌属、分支杆菌属、琥珀酸菌属、新月形单胞菌属、丁酸弧菌属和瘤胃球菌属等为优势菌属[16],该结果与本研究的差异可能是试验动物和取样部位的不同造成的。Pitta等[2]研究了肉牛饲喂百慕大干草(34 d)和小麦(28 d)后瘤胃液相、固相和混合相的细菌组成,结果表明,百慕大干草组优势菌为普雷沃氏菌属和理研菌属,分别占细菌总数的33%和28%,但小麦组中普雷沃氏菌属高达56%。百慕大干草组中的坦纳菌属和解琥珀酸菌属占细菌总数的1.0%~2.5%,但小麦组上述菌属含量可上升到2%~8%。本研究中未发现存在理研菌属和坦纳菌属,且解琥珀酸菌属含量都低于0.5%,这可能与动物种类和饲粮中牧草来源和精料组成相关联。对细菌16S rRNA基因序列分析表明普雷沃氏菌属是瘤胃微生物中含量最多的菌属,可高达42%~60%[17],该菌属在瘤胃液相中含量更高[18-19],这与本研究中普雷沃氏菌属相对含量最高的结论相一致。
本研究瘤胃液中还发现了很多低丰度菌门。变形菌门中含有大量可以降解饲料的菌群[14]。螺旋体门在瘤胃细菌中占的比例也不高,研究表明螺旋体门可以有效降解纤维素、果胶和磷酸酯,利用可发酵碳水化合物形成挥发性脂肪酸,为动物机体提供能量[20],本研究中不同牧草来源对螺旋体门含量无显著影响。很多低丰度菌门可能存在某种特定功能,但还需要在不同饲粮试验中加以验证。本研究瘤胃液中某些菌门和菌属的相对含量会伴随饲粮的改变而发生变化,这与饲粮结构类型和不同牧草降解速率不同有关[5],尽管有研究表明,即使奶牛饲喂相同饲粮瘤胃中细菌群落结构也存在一定程度的差异,但本研究发现,在饲粮营养组分均衡的条件下,不同牧草来源饲粮中瘤胃微生物存在一定的相似性,各组并未出现特定菌门和菌属,且各组间细菌含量在数值上十分接近。与此同时,Heatmap和UniFrac主坐标分析的结果也表明不同组瘤胃微生物群落结构存在很高的相似性,且苜蓿组、燕麦草组和稻草组的细菌遗传距离也非常接近。Xu等[21]先前研究发现,奶牛在饲喂上述相同的饲粮条件下,不同牧草来源并不影响奶牛泌乳性能(奶产量、乳脂率和乳蛋白率)、瘤胃发酵和微生物蛋白的合成,且干物质、粗蛋白质和有机物消化率各组间也无显著差异,这可能与该研究瘤胃液中的微生物群落结构多样性存在一定的相似性密切有关,我们推测正是由于瘤胃中细菌群落结构的相似性,才使得不同牧草来源的饲粮对奶牛瘤胃发酵和营养物质消化率无显著影响,最终体现在对生产性能无不良作用。
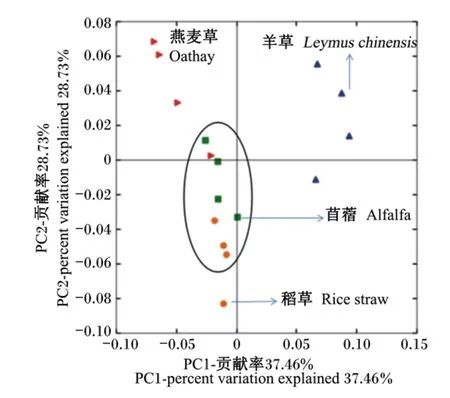
图2 97%相似性水平下16个瘤胃液样品菌群结构Unifrac加权主坐标分析图Fig.2 Principal coordinate analysis(PCoA)generated by a weighted UniFrac analysis at the 97%sim ilarity level of the 16 rumen fluid samples
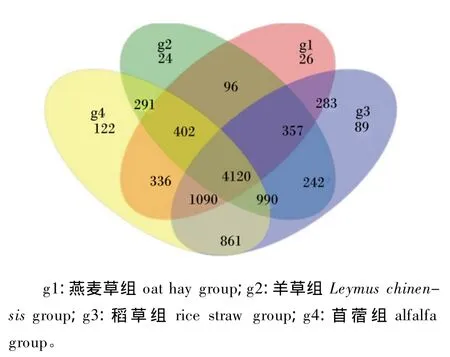
图3 不同组对瘤胃液中细菌群落共享和独有的OTU的Venn分析Fig.3 Venndiagram representation of the shared and exclusive OTUs among different groups.
4 结论
当饲粮等能等氮且NDF和NFC含量相同时,不同牧草饲喂组瘤胃液中的细菌群落结构多样性存在一定的相似性,结合前期研究的生产性能数据表明,今后可考虑使用不同牧草饲喂泌乳中期奶牛并对奶牛无不良影响。
[1] SUN X Z,HOSKIN S O,JOBLIN K N,et al.Forage cellwalls:rum inal degradation[J].Acta Agrestia Sinica,2007,15(4):386-393.
[2] PITTA D W,PINCHAK W E,DOWD S E,et al.Rumen bacterial diversity dynam ics associated w ith changing from bermudagrass hay to grazed w inter wheat diets[J].M icrobial Ecology,2010,59(3):511-522.
[3] BELANCHE A,DOREAU M,EDWARDS JE,et al.Shifts in the rumen m icrobiota due to the type of carbohydrate and level of protein ingested by dairy cattle are associated w ith changes in rumen fermentation[J].The Journal of Nutrition,2012,142(9):1684-1692.
[4] JONESR T,ROBESON M S,LAUBER C L,et al.A comprehensive survey of soil acidobacterial diversity using pyrosequencing and clone library analyses[J].The ISME Journal,2009,3(4):442-453.
[5] THOETKIATTIKUL H, MHUANTONG W,LAOTHANACHAREON T,et al.Comparative analysis ofm icrobial profiles in cow rumen fed w ith different dietary fiber by tagged 16S rRNA gene pyrosequencing[J].Current M icrobiology,2013,67(2):130-137.
[6] TAJIMA K,ARAIS,OGATA K,et al.Rumen bacterial community transition during adaptation to highgrain diet[J].Anaerobe,2000,6(5):273-284.
[7] MOSONIP,FONTY G,GOUET P.Competition between rum inal cellulolytic bacteria for adhesion to cellulose[J].Current M icrobiology,1997,35(1):44-47.
[8] JAM I E,M IZRAHI I.Composition and sim ilarity of bovine rumen m icrobiota across individual animals[J].PLoSOne,2012,7(3):e33306.
[9] DEMENEZES A B,LEW IS E,O’DONOVAN M,et al.M icrobiome analysis of dairy cow s fed pasture or totalm ixed ration diets[J].FEMSM icrobiology Ecology,2011,78(2):256-265.
[10] MEYER M,STENZEL U,HOFREITER M.Parallel tagged sequencing on the 454 platform[J].Nature Protocols,2008,3(2):267-278.
[11] MATSUIH,OGATA K,TAJIMA K,et al.Phenotypic characterization of polysaccharidases produced by four Prevotella type strains[J].Current M icrobiology,2000,41(1):45-49.
[12] KAMRA D N.Rumen m icrobial ecosystem[J].Current Science,2005,89(1):124-135.
[13] TAJIMA K,AM INOV R I,NAGAM INE T,et al.Rumen bacterial diversity as determ ined by sequence analysis of 16S rDNA libraries[J].FEMS M icrobiology Ecology,1999,29(2):159-169.
[14] EVANS N J,BROWN JM,MURRAY R D,et al.Characterization of novel bovine gastrointestinal tract Treponema isolates and comparison w ith bovine digital dermatitis treponemes[J].Applied and Environmental M icrobiology,2011,77(1):138-147.
[15] KOPECNY J,ZOREC M,MRÁZEK J,et al.Butyrivibrio hungatei sp.nov.and Pseudobutyrivibrio xylanivorans sp.nov.,butyrate-producing bacteria from the rumen[J].International Journal of Systematic and Evolutionary M icrobiology,2003,53(1):201-209.
[16] CUNHA IS,BARRETO C C,COSTA O Y A,et al.Bacteria and Archaea community structure in the rumen m icrobiome of goats(Capra hircus)from the sem iarid region of Brazil[J].Anaerobe,2011,17(3):118-124.
[17] STEVENSON D M,WEIMER P J.Dom inance of Prevotella and low abundance of classical rum inal bacterial species in the bovine rumen revealed by relative quantification real-time PCR[J].Applied M icrobiology and Biotechnology,2007,75(1):165-174.
[18] KOCHERGINSKAYA S A,AM INOV R I,WHITE B A.Analysis of the rumen bacterial diversity under two different diet conditions using denaturing gradient gel electrophoresis,random sequencing,and statistical ecology approaches[J].Anaerobe,2001,7(3):119-134.
[19] WHITFORD M F,FORSTER R J,BEARD C E,et al.Phylogenetic analysis of rumen bacteria by comparative sequence analysis of cloned 16S rRNA Genes[J].Anaerobe,1998,4(3):153-163.
[20] HESSM,SCZYRBA A,EGAN R,et al.Metagenom ic discovery of biomass-degrading genes and genomes from cow rumen[J].Science,2011,331(6016):463-467.
[21] XU J,HOU Y J,YANG H B,et al.effects of forage sources on rumen fermentation characteristics,performance,and m icrobial protein synthesis in m idlactation cows[J].Asian-australasian Journal of Animal Sciences,2014,27(5):667-673.
