鼻咽癌细胞MeDIP-Seq测序reads在染色体上分布差异的研究
2015-03-10林伟达王冬辉罗海丹苏箔金赵存友杨惠玲
林伟达 王冬辉 罗海丹,3 苏箔金 赵存友 杨惠玲★
鼻咽癌细胞MeDIP-Seq测序reads在染色体上分布差异的研究
林伟达1,2,3王冬辉1罗海丹1,3苏箔金1赵存友4杨惠玲1★
[摘要]目的探讨同源不同辐射抗拒鼻咽癌(nasopharyngeal carcinoma,NPC)细胞DNA甲基化差异,期望从表观遗传学角度阐述人鼻咽癌辐射抗拒的发生机制。方法采用MeDIP-Seq测序法对CNE2R/CNE2细胞进行测序,分析比较DNA甲基化水平差异区间的分布。结果CNE-2R和CNE-2细胞甲基化分布差异主要集中在第5、6、7、9、13、17号染色体上,其中第5、6、13、17号染色体上为甲基化上调区间的密集区域,第7、9号染色体上为甲基化下调的密集区域。结论同源不同辐射抗拒鼻咽癌细胞CNE-2R和CNE-2细胞的甲基化水平存在差异;差异甲基化水平可能与辐射抗拒相关。
[关键词]鼻咽癌;甲基化;辐射抗拒
作者单位:1.中山大学中山医学院病理生理学教研室,广东,广州510080 2.中山大学附属第六医院胃肠研究所,广东,广州510655 3.中山大学中山医学院临床医学五年制,广东,广州510080 4.南方医科大学基础医学院医学遗传学教研室,广东,广州510515
Research on different distributions of MeDIP-Seq reads in chromosomes of nasopharyngeal carcinoma cells
LIN Weida1,2,3, WANG Donghui1, LUO Haidan1,3, SU Bojin1, ZHAO Cunyou4, YANG Huiling1★
(1. Department of Pathophysiology, Zhongshan School of Medicine, Sun Yat-sen University, Guangzhou, Guangdong, China, 510080; 2. Gastrointestinal Institute, the Sixth Affiliated Hospital of Sun Yat-sen University, Guangzhou, Guangdong, China, 510655; 3. Clinical Medicine of Five-year Education, Zhongshan School of Medicine, Sun Yat-sen University, Guangzhou, Guangdong, China, 510080; 4. Department of Medical Genetics, School of Basic Medical Sciences, Southern Medical University, Guangzhou, Guangdong, China, 510515)
[ABSTRACT] Objective To expound the pathogenetic mechanism of radiation resistance of NPC from an epigenetics perspective by discussing the differences of the DNA methylation levels of nasopharyngeal carcinoma (NPC) cells in the same genetic background but different radiation resistances. Methods MeDIP-Seq was performed to sequence the CNE-2 and CNE-2R cells. The distribution of different intervals of DNA methylation level was analyzed to detect the association between the DNA methylation and radiation resistance.ResultsThe differences mostly centralized in 5, 6, 7, 9, 13 and 17 chromosomes, which were up-regulated in 5, 6, 13 and 17 chromosomes and down-regulated in 7 and 9 chromosomes, respectively. Conclusion The DNA methylation levels between NPC cells in the same genetic background but different radiation resistances(CNE-2 and CNE-2R cells) were quite different, indicating that DNA methylation may be associated with NPC radioresistance.
[KEY WORDS] Nasopharyngeal carcinoma; Methylation; Radiation-resistance
鼻咽癌(nasopharyngeal carcinoma,NPC)是源自于人鼻咽部上皮组织的恶性肿瘤,为我国最常见的头颈部恶性肿瘤之一[1]。目前放疗仍为NPC首选疗法,但由于放疗过程中辐射抗拒的产生,严重制约NPC病人5年生存率的提高[2]。DNA甲基化是研究最早且较深入的表观遗传学标志之一,其介导的表观遗传学调控在肿瘤发生、发展和肿瘤治疗中起了重要作用。已有研究报道DNA甲基化能够影响肿瘤的放化疗敏感性,如Kim等发现SERPINB5和S100A6基因CpG岛的高甲基化能降低基因的表达,从而增加非小细胞肺癌的辐射抗拒[3];Brieger等人发现利用5-氮杂胞苷使头颈部鳞癌细胞去甲基化,可以增加癌细胞的放疗敏感性[4]。另外也有研究提示NPC辐射抗拒与DNA甲基化有密切的关系[5-6]。本课题组已通过MeDIP Sequencing方法检测并比较CNE2R/CNE2细胞全基因组甲基化水平及甲基化区域的差异,结果表明不同NPC细胞之间总体甲基化水平及基因甲基化水平存在明显差异,考虑与其辐射抗拒相关。本研究拟进一步分析CNE-2R和CNE-2细胞甲基化区域在全基因组每条染色体上分布的差异,寻找出差异基因富集的区域,初步地从表观遗传学角度寻找甲基化与辐射抗拒的关系,为阐明NPC辐射抗拒机制、确立预测和逆转NPC辐射抗拒新标记物和靶点提供实验依据。
1 材料和方法
1.1实验材料及仪器
人低分化鼻咽癌细胞株CNE-2由中山大学肿瘤防治中心提供,CNE-2R细胞是本实验室在CNE2细胞基础上采用爬坡式放疗诱导生成的对放疗抵抗的细胞株。
Paired-End DNA Sample Prep Kit购自美国Illumina公司,Agnetic Methylated DNA Immunopre-Cipitation Kit购自比利时Diagenode公司。Covaris Sonication System超声波反应器为美国Covaris公司产品,PCR Thermal Cycler基因扩增仪为美国Applied Biosystems I公司产品,NanoDrop 1000分光光度计为美国Thermo Fisher Scientific公司产品,Agilent 2100生物分析仪为美国Agilent公司产品,Qubit核酸/蛋白定量系统为美国Invitrogen公司产品。
1.2方法
1.2.1细胞培养
胰酶消化对数生长期CNE-2R和CNE-2细胞,用含10%灭活牛胎儿血清、1×105U/L青霉素、1×105U/L链霉素的RPMI-1640培养液稀释细胞为2×108cells/L的细胞悬液,接种于10 mL的培养皿,每皿2×106,并置于37℃、5%CO2和饱和湿度的培养箱中培养。
1.2.2CNE-2R/CNE-2细胞基因组DNA的制备和提取
48 h后弃上清,用磷酸盐缓冲液(phosphate buffered saline,PBS)洗2次,每皿加1 mL Trizol使细胞裂解,转移至微量离心管中,加入200 μL PBS,吹打重悬。随后清除RNA,加入4 μL 100 mg/mL RNase A,旋涡混匀后室温(15℃~25℃)放置2 min;再加入20 μL蛋白酶K,旋涡混匀;加入200 μL样品裂解液B,旋涡混匀后70℃孵育10 min。加入200 μL无水乙醇,旋涡混匀后将所得混合物加入到DNA纯化柱内,8 000 r/min离心1 min。倒弃废液收集管内液体,加入500 μL洗涤液I,8 000 r/min离心1 min。倒弃废液收集管内液体。加入600 μL洗涤液II,14 000 r/min离心1 min。倒弃废液收集管内液体。进行以上离心步骤前需将纯化柱置于废液收集管上,倒弃废液后回收废液收集管。再14 000 r/min离心1 min,以去除残留的乙醇。将DNA纯化柱置于一洁净的1.5 mL离心管上,加入50 μL~200 μL洗脱液。室温放置1 min~3 min。14 000 r/min离心1 min,所得液体即为纯化得到的总DNA。洗脱液需要直接加至纯化柱管内柱面中央,使液体被纯化柱吸收。对提取的DNA采用1%的琼脂糖凝胶电泳进行检测。
1.2.3MeDIP-Seq测序
该部分实验由华大基因科技有限公司完成。采用Agnetic Methylated DNA Immunoprecipitation Kit对基因组DNA进行甲基化处理,利用超声波反应器将基因组DNA超声打断至100 bp~500 bp片段,DNA片段末端修复、3′端加A碱基,连接测序接头,双链DNA变性成单链DNA。采用5-mC抗体对基因组进行沉淀富集,实时荧光定量核酸扩增检测系统(real-time quantitative polymerase chain reaction detecting system,QPCR)对抗体富集结果进行验证。PCR扩增后行琼脂糖凝胶电泳,切割凝胶并纯化,进行DNA片段大小选择。切取220 bp~320bp的片段回收,并用Agilent2100 Analyzer定量,合格的文库用于上机测序。将MeDIP-Seq序列与参考基因组进行比对,每条read(一次测序中仪器读取的核苷酸长度)最多容许2个碱基的错配,可唯一比对的序列信息用于后续标准信息分析及个性化分析。
1.2.4统计reads在全基因组每条染色体上的分布及差异
reads不是基因在基因组中的组成,实际是一小段短的测序片段,是高通量测序仪产生的测序数据。应用华大基因工程自制软件将全基因组每条染色体等分成长度为10 kb的窗口,统计各个窗口上reads的分布并比较CNE-2R和CNE-2细胞的差异,同时找出各染色体上差异的密集区间。
2 结果
2.1 CNE-2R/CNE-2 MeDIP-Seq测序reads在全基因组每条染色体上的分布
以10 kb为窗口对基因组每条染色体进行扫描,计算每个窗口的reads覆盖深度,并对每个窗口的reads采用如下公式进行标准化:特定10 kb窗口中reads个数*1000000/唯一比对reads数,以代表CNE-2或CNE-2R细胞MeDIP-Seq测序reads在全基因组每条染色体上分布(图1,图2)。通过reads的高低观察每个窗口的甲基化水平,可获得CNE-2 或CNE-2R细胞各染色体甲基化区域的分布情况。

图1 CNE-2R细胞MeDIP-Seq测序reads在全基因组每条染色体上的分布Figure 1 The distribution of MeDIP-Seq reads in each chromosome in CNE-2R
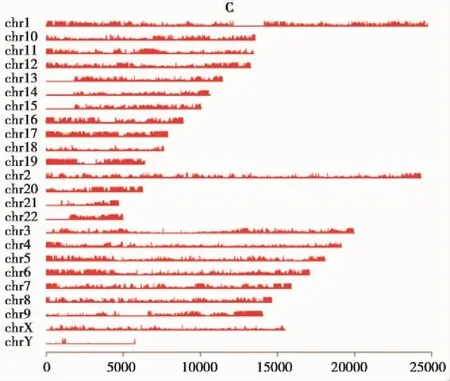
图2 CNE-2细胞MeDIP-Seq测序reads在全基因组每条染色体上的分布Figure 2 The distribution of MeDIP-Seq reads in each chromosome in CNE-2
2.2 CNE-2R和CNE-2 MeDIP-Seq测序reads在全基因组每条染色体上的分布的差异
统计每个窗口的reads数。归一化两样品的数据量后,对两样品中每窗口其中一个样品最少200 reads,两样品reads数差异2倍的reads数做卡方检验,以P<0.01作为筛选条件,挑出差异区间(表1)。这样的差异区间共有236个,其中,甲基化上调的差异区间有148个,甲基化下调的差异区间有88个。以每条染色体每10 kb窗口为横轴坐标,区间中甲基化上调r值=CNE-2样品reads数/CNE-2R样品reads,区间中甲基化下调r值=CNE-2R样品reads数/CNE-2样品reads(图3)。从表1和图3我们可以发现差异区间分布较为密集的区域位于第5、6、7、9、13、17号染色体上,其中第5、6、13、17号染色体上为甲基化上调区间的密集区域,第7、9号染色体上为甲基化下调的密集区域。
3 讨论
目前制约鼻咽癌预后的主要因素是辐射抗拒,故阐明NPC辐射抗拒产生的机制尤为迫切。表观遗传学是当前生命医学研究的热点之一,其机制主要包括DNA甲基化、组蛋白修饰及microRNA调控[7]。本研究通过MeDIP-Seq分析CNE-2R和CNE-2细胞的甲基化水平,观察测序reads在全基因组每条染色体上分布,并比较CNE-2R和CNE-2细胞的差异。结果发现,CNE-2R和CNE-2细胞总体甲基化水平有约30%的差异,提示DNA甲基化与NPC辐射抗拒相关(另文发表)。两细胞株间甲基化差异区间有236个,上调区间明显多于下调区间,提示产生辐射抗拒的CNE-2R细胞基因甲基化水平更高。另外,这些差异区间有集中分布的趋势,其中第5、6、13、17号染色体以上调为主,第7、9号染色体以下调为主。因此,我们推测CNE-2R和CNE-2细胞染色体甲基化水平存在的这种明显差异可能与NPC辐射抗拒的产生有关。

表1 差异区间的分布Table 1 The distribution of different intervals

图3 CNE-2R与CNE-2细胞MeDIP-Seq测序reads在全基因组染色体上分布位置的差异Figure 3 The different distribution of MeDIP-Seq reads in each chromosome between CNE-2R and CNE-2
大量的研究证实DNA甲基化在肿瘤发生发展中起着非常重要的作用[8-10]。头颈部鳞癌中抑癌基因因高甲基化而失活非常普遍,科学家们认为其更倾向于一个表观遗传学疾病[11-12]。另外也有研究提示NPC是一种CpG岛甲基化频率非常高的肿瘤[13-14]。肿瘤细胞甲基化程度与预测预后也有密切的关系[15],如Asuthkar等发现miRNA-211的低甲基化与MMP-9的高表达有关,是多形性成胶质细胞瘤对放化疗不敏感的一个重要原因[16];CHFR的甲基化程度可以作为判断胃癌对化疗敏感性的一个指标,MLH1的甲基化能导致卵巢癌、胃癌对放疗的敏感性降低[17-18];CTNNB1、TP53、CDKN2A、RB1、MEN1、ZNRF3、DAXX、TERT、MED12等多种基因的异常甲基化可以影响肾上腺皮质癌的预后[19-21]。但是DNA甲基化是否也能够用于预测NPC预后仍不清楚。另外,不同肿瘤甲基化水平差异也有集中分布的趋势,如子宫内膜癌中X染色体上就存在大范围的去甲基化,并伴随着XIST基因的低表达[22];Zhang提出3号染色体上RASSF1A、BLU、FHIT、CRBP1等抑癌基因的高甲基化与肝癌有密切的关系[23]。由于CNE-2R和CNE-2细胞的甲基化差异以差异区间为单位进行统计,难以体现具体基因的甲基化水平差异,故本课题组也将展开进一步研究,获得具体某些基因的甲基化水平差异并分析。
在本研究中,不同辐射抗拒的NPC细胞甲基化水平存在着明显的差异,表明除了缺失和突变之外甲基化状态同样是NPC辐射抗拒产生的重要原因之一,这也意味着NPC细胞甲基化状态对其预后有直接影响,尽管DNA甲基化和肿瘤辐射抗拒的研究已取得许多进展,但对于两者之间联系的影响仍是一个新发现,其对综合评价肿瘤的预后具有一定的意义。实验研究的最终目的是服务于临床,由于甲基化在NPC辐射抗拒产生中的重要作用,将其应用到肿瘤风险评估判断肿瘤复发情况、预测肿瘤疗效和预后、开发特异新靶点药物等方面具有很大的潜力。至于其确切作用及机制有待进一步探讨。
参考文献
[1]Xiao J, Tan Y, Pan Y, et al. A new cyclooxygenase-2 inhibitor, (1E, 4E)-1, 5-bis(2-bromophenyl)penta-1, 4-dien-3-one (GL63) suppresses cyclooxygenase-2 gene expression in human lung epithelial cancer cells: coupled mRNA stabilization and posttranscriptional inhibition[J]. Biol Pharm Bull, 2010, 33(7):1170-1175.
[2]Qin HD, Shugart YY, Bei JX, et al. Comprehensive pathway-based association study of DNA repair gene variants and the risk of nasopharyngeal carcinoma[J]. Cancer Res, 2011,71(8):3000-3008.
[3]Kim EH, Park AK, Dong SM, et al. Global analysis of CpG methylation reveals epigenetic control of the radiosensitivity in lung cancer cell lines[J]. Oncogene, 2010,29(33):4725-4731.
[4]Brieger J, Mann SA, Pongsapich W, et al. Pharmacological genome demethylation increases radiosensitivity of head and neck squamous carcinoma cells[J]. Int J Mol Med, 2012,29(3):505-509.
[5]Wang S, Zhang R, Claret FX, et al. Involvement of microRNA-24 and DNA methylation in resistance of nasopharyngeal carcinoma to ionizing radiation[J]. Mol Cancer Ther, 2014,13(12):3163-3174.
[6]Cai MY, Tong ZT, Zhu W, et al. H3K27me3 protein is a promising predictive biomarker of patients' survival and chemoradioresistance in human nasopharyngeal carcinoma[J]. Mol Med, 2011,17(11-12):1137-1145.
[7]Portela A, Esteller M. Epigenetic modifications and human disease[J]. Nat Biotechnol, 2010,28(10):1057-1068.
[8]Herman JG, Baylin SB. Gene silencing in cancer in association with promoter hypermethylation[J]. N Engl J Med, 2003,349(21):2042-2054.
[9]Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation[J]. Cell, 2011,144(5):646-674.
[10]Lee CJ, Evans J, Kim K, et al. Determining the effect of DNA methylation on gene expression in cancer cells[J]. Methods Mol Biol, 2014,1101:161-178.
[11]Laytragoon-Lewin N, Chen F, Castro J, et al. DNA content and methylation of p16, DAPK and RASSF1A gene in tumour and distant, normal mucosal tissue of head and neck squamous cell carcinoma patients[J]. Anticancer Res, 2010,30(11):4643-4648.
[12]Tong JH, Ng DC, Chau SL, et al. Putative tumour-suppressor gene DAB2 is frequently down regulated by promoter hypermethylation in nasopharyngeal carcinoma [J]. BMC Cancer, 2010,10:253.
[13]Tao Q, Chan AT. Nasopharyngeal carcinoma: molecular pathogenesis and therapeutic developments[J]. Expert Rev Mol Med, 2007,9(12):1-24.
[14]He D, Zeng Q, Ren G, et al. Protocadherin8 is a functional tumor suppressor frequently inactivated by promoter methylation in nasopharyngeal carcinoma[J]. Eur J Cancer Prev, 2012,21(6):569-575.
[15]Chan KC, Jiang P, Chan CW, et al. Noninvasive detection of cancer-associated genome-wide hypomethylation and copy number aberrations by plasma DNA bisulfite sequencing[J]. Proc Natl Acad Sci USA, 2013,110(47): 18761-18768.
[16]Asuthkar S, Velpula KK, Chetty C, et al. Epigenetic regulation of miRNA-211 by MMP-9 governs glioma cell apoptosis, chemosensitivity and radiosensitivity[J]. Oncotarget, 2012,3(11):1439-1454.
[17]Li Y, Yang Y, Lu Y, et al. Predictive value of CHFR and MLH1 methylation in human gastric cancer[J]. Gastric Cancer, 2014,18(2):280-287.
[18]Strathdee G, MacKean MJ, Illand M, et al. A role for methylation of the hMLH1 promoter in loss of hMLH1 expression and drug resistance in ovarian cancer[J]. Oncogene, 1999,18(14):2335-2341.
[19]Soon PS, Sidhu SB. Molecular basis of adrenocortical carcinomas [J]. Minerva Endocrinol, 2009,34 (2):137 -147.
[20]Wandoloski M, Bussey KJ, Demeure MJ. Adrenocortical cancer[J]. Surg Clin North Am, 2009,89(5):1255-1267.
[21]Assie G, Letouze E, Fassnacht M, et al. Integrated genomic characterization of adrenocortical carcinoma[J]. Nat Genet, 2014,46(6):607-612.
[22]Zhang B, Xing X, Li J, et al. Comparative DNA methylome analysis of endometrial carcinoma reveals complex and distinct deregulation of cancer promoters and enhancers[J]. BMC Genomics, 2014,15(1):868.
[23]Zhang X, Li HM, Liu Z, et al. Loss of heterozygosity and methylation of multiple tumor suppressor genes on chromosome 3 in hepatocellular carcinoma[J]. J Gastroenterol, 2013,48(1):132-143.
通讯作者:★杨惠玲,E-mail:yanghl@mail.sysu.edu.cn
基金项目:国家自然科学基金(81071837);广东省自然科学基金重点项目(92510089 01000005);2013年和2012年国家大学生创新训练计划项目(201310558074,201210558076);2013年广东省大学生创新训练项目(1055813171)
