翘嘴鳜(Siniperca chuatsi)转录组EST-SSR位点的信息分析及其多态性检测*
2015-03-08袁文成黄鹤忠李文龙张茂友王荣泉
袁文成 黄鹤忠, 李文龙 路 瑶 张茂友 王荣泉
(1. 苏州大学基础医学与生物科学学院 苏州大学水产研究所 苏州 215123; 2. 江苏省区域现代农业与环境保护协同创新中心 淮安 223300; 3. 苏州市水产技术推广站 苏州 215000; 4. 苏州市长漾生态科技发展有限公司 苏州 215200)
翘嘴鳜(Siniperca chuatsi)隶属于鲈形目(Perciformes)、暖鲈科(Percichthidae)、鳜属(Siniperca),俗称桂鱼、桂花鱼、胖鳜、季花鱼等, 是淡水底栖的肉食性鱼类。它具有体型大、生长快、味道鲜美等特点, 深受国内外消费者喜爱, 有“淡水石斑鱼”之称(张春光等, 1999), 是我国重要的名贵经济鱼类和养殖对象。近年来, 由于江河过度捕捞、水域生态环境恶化及养殖生产中近亲繁殖等原因, 导致自然种质资源衰退、养殖种质性状退化、鱼类疾病不断爆发等现状, 已影响到翘嘴鳜种质延续及其养殖业的正常发展(黄志坚, 1999; He et al, 2002)。因此, 迫切需要运用现代生物学手段开展翘嘴鳜种质资源保护和良种选育等方面的研究。
微卫星 DNA, 即简单重复序列(simple sequence repeats, SSR)或简单序列长度多态性(simple sequence length polymorphism, SSLP), 是指由 1—6个核苷酸组成的简单串联重复DNA序列, SSR标记主要包括基因组SSR和表达序列标签SSR(EST-SSR)。其中, 基因组SSR的种属特异性强、操作复杂、技术难度大,极大地限制了其发展和应用; 而 EST-SSR除具有传统基因组来源的 SSR标记优势以外, 还具有信息量大、保守性好、开发简单等优点, 可以为功能基因提供直接的分子标记, 已被广泛应用于遗传多样性评价、基因定位及分子标记辅助育种(MAS)等领域(Morgante et al, 1993; Powell et al, 1996)。Huang 等(2013)从64对微卫星引物中筛选出38对翘嘴鳜和斑鳜杂交 F1代的多态性微卫星引物并在翘嘴鳜和斑鳜之间进行了跨物种扩增, 但是微卫星位点是随机筛选, 获得的标记数量也相对较少。目前有关翘嘴鳜功能性 EST-SSR分子标记开发的研究鲜有报道。本课题组运用高通量 Illuminua测序技术获得了翘嘴鳜转录组数据, 开展了大规模转录组 EST-SSR标记的研究开发, 本文基于转录组数据重点对 EST-SSR标记的组成、分布、特征及多态性进行了分析和研究, 为翘嘴鳜功能性 EST-SSR分子标记的大量开发提供帮助, 从而可弥补当前翘嘴鳜种质保护和良种选育研究中遗传背景较模糊、分子标记数量少的局限性, 以推动翘嘴鳜种质资源保护、分子标记辅助育种和水产养殖业的健康发展。
1 材料与方法
1.1 材料来源
翘嘴鳜来自于江苏省海门市养殖场, 活体运回实验室, 挑选健康的Ⅰ、Ⅱ、Ⅲ龄翘嘴鳜各3尾(心、肝、脾、胃、肠、肾、肌肉及脑), 无菌水冲洗干净后作为测序材料, 随机挑选 30尾健康个体作为多态性验证的材料。
1.2 翘嘴鳜转录组数据来源及处理
翘嘴鳜转录组数据由本实验室之前采用Illumina高通量测序技术对翘嘴鳜各组织混合样本进行转录组测序获得, 总共得到 4G的数据。经过序列拼接得到51245条unigenes, 最大的isogene为 18278bp, 最小的为 651bp。用软件 SeqPrep (https://github.com/jstjohn/SeqPrep)和condetri_v 2.0.pl (http://code.google.com/p/condetri/downloads/detail?name=condetri_v2.0.pl)进行序列除杂, 筛除测序质量较低的序列(测序质量值小于 25nt), 接头序列, 含有 N值 (未知碱基)较多的序列; 低质量值及去接头后小于25nt的小片段。
1.3 翘嘴鳜SSR的筛选
利用软件 SSRFINDER (http://www.maizemap.org/bioinformatics/SSRFINDER/)对翘嘴鳜转录组中Unigene的cDNA序列数据进行SSR搜索, 筛选的标准为: 重复单元长度2—6 bp, 单核苷酸重复的次数在16次或 16次以上, 二核苷酸重复的次数在 6次或 6次以上, 三至六核苷酸重复的次数在5次或5次以上。
1.4 引物设计、PCR扩增及多态性检测
利用Primer 3.0总共设计出7335对引物, 随机选取100对经济性状相关EST-SSR微卫星引物, 由上海生工生物技术有限公司合成, 用于多态性验证。扩增均采用 20μL PCR 反应体系, 包括 2×ES TaqMIX 10μL, 上下游引物(10μmol/L)各1μL, 模板DNA 1μL,无菌水补足至 20μL, 然后在 PCR仪(TaKaRa)上进行扩增, 扩增程序为: 94°C预变性 2min, 26个循环(94°C变性30s, 50—60°C退火30s, 72°C延伸30s), 最后72°C延伸2min。PCR扩增产物用1%琼脂糖凝胶电泳检测, 特异的扩增产物采用 8%非变性聚丙烯酰胺凝胶电泳结合银染法显色进行检测。用Bio-rad凝胶自动成像系统拍照, Image lab软件分析所有电泳图片。
2 结果与分析
2.1 翘嘴鳜转录组中EST-SSR的分布及频率
测序组装和转录组拼接后共得到 51245条unigenes, 在 35285个unigenes中共检测到14094个SSR位点, 这意味着至少68.86%的unigene含有一个SSR位点, 微卫星分布频率和密度分别是 27.51%和184 per Mb。在这些微卫星位点中统计其重复类型,如表1所示, 翘嘴鳜转录组EST-SSR较为丰富, 一至六核苷酸重复类型都能看到, 但各类型 SSR出频率不同, 主要集中在一至三核苷酸重复类型上, 占总SSR的 98.52%。其中, 单核苷酸重复类型数量最多,占总SSR的53.67%, 其次是二和三核苷酸重复类型,分别占总SSR的30.62%和14.23%, 四、五和六核苷酸重复类型数量相对较少, 分别占 1.11%、0.28%和0.09%。所有重复类型中, 以重复12次以上的单核苷酸SSR最多, 占24.61%, 其次是10次和6次重复类型的SSR, 分别占22.33%和13.72%, 4次重复类型的SSR仅有0.34%。
翘嘴鳜转录组 EST-SSR位点的序列总长度达到195086 bp, SSR位点长度分布从10—25个碱基不等,平均长度13bp, 其中, 单核苷酸重复基元、二核苷酸重复基元、三核苷酸重复基元、四核苷酸重复基元、五核苷酸重复基元和六核苷酸重复基元的 SSR位点的碱基总长度分别是76385、54878、56568、6420、595和 240bp, 各类型 SSR位点的平均长度分别是12.4、15.6、14、17.5、20.5 和 24bp(表 1)。
2.2 翘嘴鳜转录组EST-SSR的特性
在翘嘴鳜14094个转录组EST-SSR位点中, 共观察到90种重复基元, 其中, 一、二、三、四、五及六核苷酸重复基元分别有4、6、19、33、28和10种。二和三核苷酸重复类型及出现频率由表2所示, 在二核苷酸重复类型中以 GT/AC出现次数最多, 占二核苷酸SSRs的32.12%; 其次是AC/GT, 占21.89%,而CG/CG的发生频率最低, 仅占0.02% (图1); 在三核苷酸重复类型中, 以AGG/CCT出现频率最多,占三核苷酸 SSRs的 6.36%; 其次是 AGC/GCT、CCT/AGG、AAG/CTT和 GCT/AGC四种类型重复基元, 占2.59%—3.59%; 其它重复基元类型的出现频率均较低。
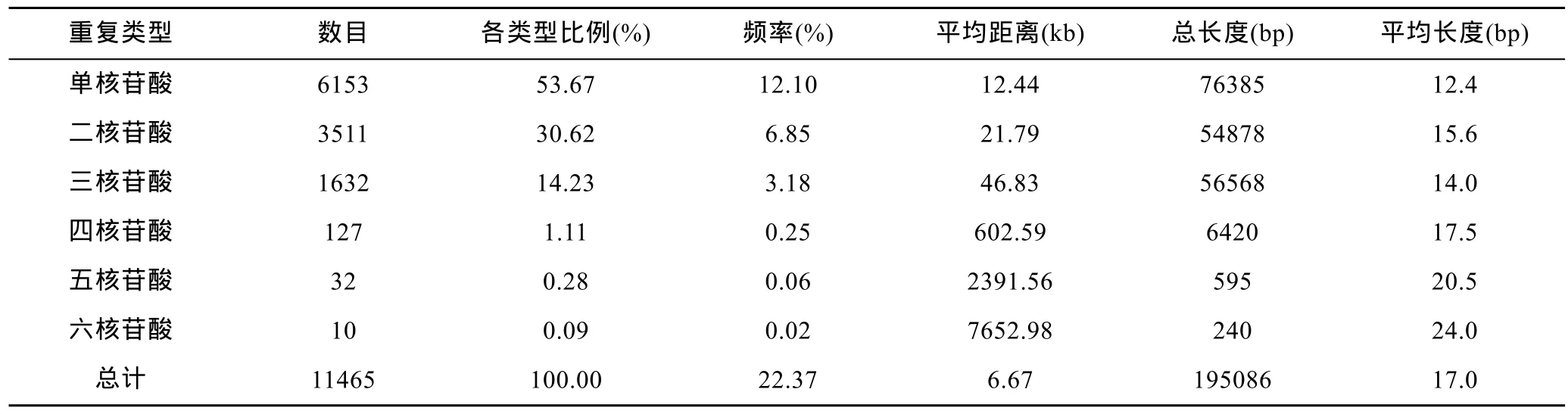
表1 EST- SSRs在翘嘴鳜转录组中的特征Tab. 1 Characterization of transcriptome EST-SSRs in S. chuatsi
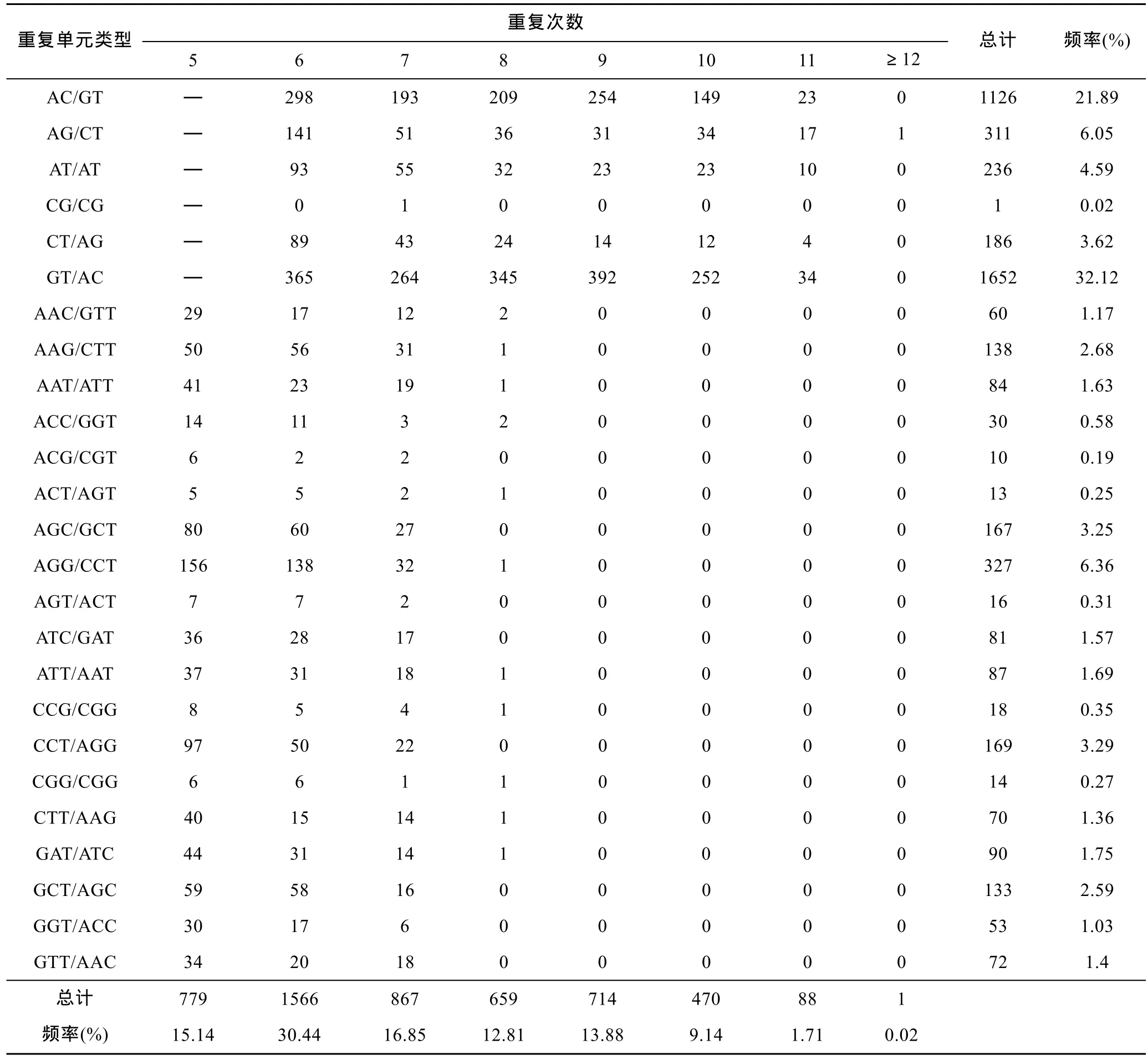
表2 翘嘴鳜转录组中EST-SSRs二碱基和三碱基重复基元的类型及频率Tab.2 Di- and tri-nucleotide transcriptome EST-SSRs repeat motifs and their frequency in S. chuatsi

图1 翘嘴鳜转录组二核苷酸SSRs不同重复基元的百分比Fig.1 Percentage of different motifs of transcriptome dinucleotide SSRs in S. chuatsi
从翘嘴鳜转录组 EST-SSRs位点的重复次数看,重复基元以重复 4次和 10次的频率较高, 分别有2665和 2559个, 占总 SSR的 18.91%和 18.16%; 其次为5、6、7、8、11和12次重复, 出现频率均在659—1566个之间。统计表明, 4—10次重复的SSR位点有9943个, 占总SSR的71%; 11—15次重复的位点有3194个, 占23%; 16—20次重复的位点有809个,占6%; 重复20次以上的位点有147个, 仅占总SSR的 1% (图 2) 。

图2 翘嘴鳜转录组EST-SSRs重复次数分布Fig.2 Distribution of the repeats frequency of transcriptome EST-SSRs in S. chuatsi
2.3 翘嘴鳜转录组EST-SSR的可用性评价
SSR分子标记的多态性是判断其可用性的主要依据, 根据Temnykh等(2001)的研究, SSR的长度是影响其多态性高低的重要因素, 当 SSR长度大于或等于 20bp时多态性较高, 长度在 12—20bp之间的SSR多态性中等, 而长度在 12bp以下时多态性潜能极低。本研究在翘嘴鳜转录组 EST-SSR筛选过程中已将12bp以下的SSR除去, 因此获得的翘嘴鳜转录组EST-SSR长度主要集中在12—45bp范围内, 其中长度在12—20bp的SSR有9478条, 占SSR总数的67.25%, 这类 SSR具有中等多态性; 而长度在 20bp以上的SSR有1290条, 占SSR总数的9.15%, 此类SSR具有较高多态性。此外, 在翘嘴鳜转录组数据统计中发现, 大多数都是低级基元 SSR, 例如, 二、三核苷酸有5143个, 占总SSR的44.85%。Dreisigacker等(2004)发现, 低级基元的 SSR多态性普遍比高级基元的高。由此可预见翘嘴鳜转录组具有较高的多态性潜能, 在基因功能研究开发方面具有较高的利用价值。
2.4 翘嘴鳜转录组EST-SSR引物扩增产物的多态性检测
随机从与经济性状相关的 EST-SSR中选取 100对引物, EST-SSR位点包括2—6核苷酸重复单元, 以随机挑选的30尾健康翘嘴鳜基因组DNA为模板分别对引物进行扩增并检测多态性。结果表明, 共有 81对引物成功获得扩增产物, 扩增效率为81%, 其中有22对引物扩增片段与预期产物片段大小不符。在100个微卫星座位中, 以二碱基、三碱基重复类型的扩增成功率较高。在81个微卫星座位中共有20个座位表现出多态性(表3), 多态性比率为24.7%, 61个座位表现为单态。20对多态性引物所在微卫星位点中, 有8个为二碱基重复, 其余为三碱基和四碱基重复。
3 讨论
转录组 EST-SSR分子序列大多数与已知和未知的功能基因连锁, 在生物进化中起到编码氨基酸和蛋白质的重要作用(张琼等, 2010)。本研究结果显示,翘嘴鳜转录组 EST-SSR位点的分布频率达 27.51%,大于灯盏花(6.99%)(陈茵等, 2014)、野三七(16.82%)(李翠婷等, 2014)、鲫鱼(8.13%)(杨曦, 2012)及中华鳖(22.3%)(Wang et al, 2013), 说明翘嘴鳜转录组EST-SSR数量很丰富。本研究发现, 翘嘴鳜转录组EST-SSRs中双碱基所占比例最大, 重复序列以(AC)n/(GT)n为主, 占总二核苷酸重复类型的32.12%。李偲等(2011)对草鱼头肾组织 EST序列的研究中发现, 二核苷酸重复位点最多, 且AC/GT占其中的50.3%; Wang等(2007)利用公共EST数据库进行草鱼新型 EST-SSR分子标记的开发中发现(AC)n/(GT)n的数量最多, 占双碱基重复类型的60.19%; 宋春妮等(2011)研究发现两碱基重复类型(AC)n在日本蟳(Charybdis japonica)基因组中含量也非常丰富。以上这些相似研究结果的原因有待进一步研究。

表3 20对多态性EST-SSRs引物的序列及重复类型Tab.3 Sequence and repeat types of 20 polymorphic EST-SSRs primers
大多数鱼类的EST-SSRs以二核苷酸和三核苷酸重复类型为主。本研究结果表明, 翘嘴鳜转录组EST-SSR重复基元以二核苷酸重复为主, 其次是三核苷酸重复, 分别占SSR总数的30.62%和14.23%, 这与朱滔等(2013)在翘嘴鳜 EST-SSR研究中的优势重复类型以二核苷酸为主、三核苷酸其次的结果相似,也与团头鲂(曾聪等, 2013)的研究结果相似, 但各物种的重复基元有所差异, 存在种属特异性。本研究结果显示, 翘嘴鳜转录组 EST-SSR双碱基重复类型中仅有一个(CG)n。已有报道显示, 在团头鲂(曾聪等,2013)中(CG)n只占总SSR的0.55%, 斑海豹(Gao et al,2013)为0.2%, 在日本沼虾(Jin et al, 2013)中未发现。而在拟南芥、杏树、桃树(Jung et al, 2005)、水稻、大豆(Gao et al, 2003)等植物中均未发现(CG)n重复基元,这种情况是否与生物进化有关, 有待进一步研究。本研究中没有发现(GA)n双碱基重复类型, 与匡刚桥等(2007)用FIASCO筛选的、基于翘嘴鳜DNA微卫星标记中(GA)n双碱基重复类型约占13%的研究结果有很大不同, 这可能是由于本研究是基于翘嘴鳜转录组 EST-SSR序列, 最终导致了碱基重复基序偏倚性的假象。
本研究中, 翘嘴鳜转录组 EST-SSR多态性比率为 24.7%, 较之于野生型灯盏花(52.78%)(陈茵等,2014)、野生型斑海豹(70%)(Gao et al, 2013)等的研究结果有所偏低。推测可能的原因主要有以下两方面:首先, 用于多态性验证的 30尾翘嘴鳜是经过人工选育的养殖群体; 其次, 实验选取的100个位点都来自于生长、发育、增殖和免疫等经济性状相关的基因。因而导致等位基因数量及遗传多样性有所降低, 符合遗传规律。今后还需要对野生翘嘴鳜群体进行研究,以便挖掘更多经济性状相关的 EST-SSR多态性基因位点。刘瑞成等(2013)利用磁珠富集法获得宽口光唇鱼的495个阳性克隆中, 只有163个克隆含有微卫星位点, 经过PCR扩增, 仅有11.4%的多态性位点, 较本研究获得的多态性位点比率低, 且这些位点的基因功能尚不得而知。因此, 采用转录组测序技术来开发与经济性状相关的多态性微卫星标记具有简便、快捷和高效的优势。
综上所述, 翘嘴鳜转录组 EST-SSR不但出现频率高, 而且类型丰富; 从多态性潜能的角度考虑, 这些SSR标记具有较高的可用性。因此, 本研究结果可为进一步开发新的翘嘴鳜功能基因 EST-SSR标记奠定基础, 对加速翘嘴鳜的资源开发利用, 丰富功能基因分子标记类型和遗传资源评价, 实现分子标记辅助育种和进行比较基因组学研究都具有重要的意义。
匡刚桥, 刘 臻, 鲁双庆等, 2007. FIASCO法筛选鳜鱼微卫星标记. 中国水产科学, 14(4): 608—614
朱 滔, 梁旭方, 彭敏燕等, 2013. 翘嘴鳜EST-SSR标记的开发及 3个群体遗传多态性分析. 暨南大学学报(自然科学与医学版), 34(3): 347—352
刘瑞成, 张富铁, 但胜国等, 2013. 宽口光唇鱼微卫星位点的筛选与特征分析. 四川动物, 32(2): 161—166
李 偲, 刘 航, 黄 容等, 2011. 草鱼Ⅰ型微卫星标记的发掘及其多态性检测. 水生生物学报, 35(4): 681—687
李翠婷, 张广辉, 马春花等, 2014. 野三七转录组中 SSR位点
信息分析及其多态性研究. 中草药, 45(10): 1468—1472杨 曦, 2012. 鲫鱼(Carassius auratus)表达序列标签资源的SSR构成与分布分析. 生物技术通报, 11: 140—143
宋春妮, 李 健, 刘 萍等, 2011. 日本蟳微卫星富集文库的建立与多态性标记的筛选. 水产学报, 35(1): 35—42
张 琼, 刘小林, 李喜莲等, 2010. EST-SSR分子标记在水生动物遗传研究中的应用. 水产科学, 29(5): 302—306
张春光, 赵亚辉, 1999. 我国鳜资源现状及其恢复和合理利用的途径. 生物学通报, 34(12): 9—11
陈 茵, 李翠婷, 姜倪皓等, 2014. 灯盏花转录组中SSR位点信息分析及其多态性研究. 中国中医药杂志, 39(7): 1220—1224
黄志坚, 何建国, 1999. 鳜鱼疾病的研究概况. 水产科技情报,26(6): 268—271
曾 聪, 高泽霞, 罗 伟等, 2013. 基于454 GS FLX高通量测序的团头鲂 ESTs中微卫星特征分析. 水生生物学报,37(5): 982—988
Dreisigacker S, Zhang P, Warburton M L et al, 2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different mega environments. Crop Science, 44(2): 381—388
Gao L, Tang J, Li H et al, 2003. Analysis of microsatellites in major crops assessed by computational and experimental approaches. Molecular Breeding, 12(3): 245—261
Gao X, Han J, Lu Z et al, 2013. De novo assembly and characterization of spotted seal Phoca largha transcriptome using Illumina paired-end sequencing. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 8(2): 103—110
He J G, Zeng K, Weng S P et al, 2002. Experimental transmission,pathogenicity and physical-chemical properties of infectious spleen and kidney necrosis virus (ISKNV). Aquaculture,204(1): 11—24
Huang W, Liang X, Qu C et al, 2013. Development and characterization of novel polymorphic microsatellite loci in Siniperca scherzeri Steindachner and Siniperca chuatsi(Basilewsky). Molecular Biology Reports, 40(2): 751—756 Jin S, Fu H, Zhou Q et al, 2013. Transcriptome analysis of androgenic gland for discovery of novel genes from the oriental river prawn, Macrobrachium nipponense, using Illumina Hiseq 2000. PloS One, 8(10): e76840
Jung S, Abbott A, Jesudurai C et al, 2005. Frequency, type,distribution and annotation of simple sequence repeats in Rosaceae ESTs. Functional & Integrative Genomics, 5(3):136—143
Morgante M, Olivieri A M, 1993. PCR-amplified microsatellites as markers in plant genetics. The Plant Journal, 3(1): 175—182
Powell W, Machray G C, Provan J, 1996. Polymorphism revealed by simple sequence repeats. Trends in Plant Science, 1(7):215—222
Temnykh S, DeClerck G, Lukashova A et al, 2001. Computational and experimental analysis of microsatellites in rice(Oryza sativa L.): frequency, length variation, transposon associations, and genetic marker potential. Genome Research,11(8): 1441—1452
Wang D, Liao X, Cheng L et al, 2007. Development of novel EST-SSR markers in common carp by data mining from public EST sequences. Aquaculture, 271(1): 558—574
Wang W, Li C, Ge C et al, 2013. De-novo characterization of the soft-shelled turtle Pelodiscus sinensis transcriptome using Illumina RNA-Seq technology. Journal of Zhejiang University Science B, 14(1): 58—67
