小鼠烧伤后时序芯片数据分析及免疫标志物的初步筛选
2015-02-24高艳彬卢志阳史鹏伟王桂芳温皇鼎
高艳彬, 卢志阳, 金 辉, 史鹏伟, 王桂芳, 温皇鼎, 杨 磊
(南方医科大学南方医院烧伤科,广东广州510515)
小鼠烧伤后时序芯片数据分析及免疫标志物的初步筛选
高艳彬1, 卢志阳1, 金 辉1, 史鹏伟1, 王桂芳1, 温皇鼎1, 杨 磊1
(南方医科大学南方医院烧伤科,广东广州510515)
目的:从基因转录水平探讨烧伤早期免疫系统功能变化情况,并筛选烧伤后免疫相关标志物,为临床诊疗提供新思路.方法:GEO数据库下载GSE7404数据集(小鼠,25%TBSA,3度),共获得32例基因表达谱数据.应用配对样本t检验和倍比法(fold-change,FC)筛选差异表达基因(P-value<0.01和|lgFC|>1).分别利用DAVID数据库和STRING数据库进行生物学过程功能富集分析及构建免疫相关蛋白互作网络.互作网络的模块及可视化分析应用Cytoscape软件进行,并用BINGO插件进行模块功能分析.结果:在烧伤后第1 d的基因表达谱的变化最显著,共1 825个差异基因被选出,其中上调基因658个,下调基因1 167个.功能富集分析显示刺激反应和免疫系统过程等生物学功能贯穿整个烧伤早期.蛋白互作网络分析表明LCK、CCR2、TLR2和MyD88等免疫相关基因可能在烧伤早期免疫功能变化过程中起着重要的作用.结论:利用生物信息学方法,分析烧伤后时序基因芯片数据,可以有效地揭示烧伤后免疫系统潜在的分子机制,可以为早期诊断和治疗靶点的筛选提供新的思路.
烧伤; 免疫系统过程; 功能富集分析; 免疫相关蛋白互作网络
烧伤,尤其是严重烧伤将引起机体免疫功能紊乱,免疫功能紊乱被认为是引发全身炎症反应综合征(SIRS)、多器官功能障碍(MODS)甚至死亡等的重要原因[1].烧伤后免疫功能的变化一直贯穿着烧伤治疗全程,不仅引起全身非特异性免疫功能的变化,引发大量炎症因子及细胞因子的释放,并能造成全身特异性免疫功能的改变,引起体液免疫失调,细胞免疫功能受损[2].
烧伤后免疫功能变化的过程是一个动态变化的过程,其病理生理学过程要受到烧伤面积,深度,年龄等多方面因素的影响,尽管烧伤后免疫功能的研究一直备受关注,但其确切的发生机制至今尚未明确.在2008年和2011年James A[3]和Wenzhong Xiao[4]等就分别利用基因芯片技术对小鼠和人创伤及烧伤后免疫细胞基因组的序贯性变化做出了初步探究,并发现了许多值得深思及颠覆性的信息,遗憾的是这些研究均着重于烧伤与创伤之间的对比,并无对烧伤后基因转录水平变化进行深入的研究与分析.本实验应用小鼠烧伤后免疫细胞时序基因芯片数据,对烧伤早期不同时间点的免疫相关基因进行生物信息学分析,以期从细胞转录水平了解烧伤后免疫功能变化情况,并筛选潜在的重要的生物学靶点.
1 材料和方法
在美国生物技术信息中心GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)下载GSE7404[3]数据集.GSE7404数据集(小鼠,25%TBSA,三度烧伤)为GPL1261平台Affymetrix Mouse Genome 430 2.0 Array基因芯片.从中选取烧伤相关芯片数据进行分析,总共有32个样本符合分析要求(包括16个烧伤和16个对照).根据芯片注释及处理情况进行分组:烧伤后2 h组及其对照组、烧伤后1 d组及其对照组,烧伤后3 d组及其对照组,烧伤后7 d组及其对照组.
1.1 统计方法
芯片质量评估、差异基因获取及统计分析应用开源统计软件R3.01进行(http://www.r-project.org).通过对原始数据进行RMA(robust multiarray average)背景矫正、四分位数法进行归一化处理,并进行汇总以获取表达水平数据[5-6].差异基因获取采用配对样本t检验和倍比法(fold change,FC),选出P-value<0.01和|lgFC|>1的基因作为差异表达基因进行下一步分析.
1.2 功能富集分析及原始蛋白互作网络的构建
应用DAVID[7](Database for Annotation,Visualization,and Integrated Discovery)(http://david.abcc.ncifcrf.gov/)网络数据库对所选出的差异基因进行Gene Ontology(GO)[8]功能富集分析.选取EASE(Expression Analysis Systemic Explorer)<0.05的生物学功能,并将差异表达基因根据功能富集结果进行功能分类.原始蛋白互作网络的构建由DAVID及STRING数据库进行.先将不同组的差异基因经DAVID基因转换工具(Gene ID Conversion Tool)进行基因ID转换;再将转换后的基因通过STRING9.1[9](functional protein association networks)数据库构建原始蛋白互作网络(Protein-protein interaction network)并选取中等可信度的互作关系(交互作用评分大于0.4)进入下一步分析.
1.3 免疫系统过程相关基因的选取及蛋白互作网络分析
将经过Gene Ontology(GO)功能富集分析所选取的免疫系统过程相关基因(GO:0002376 immune system process)映射到各组的原始蛋白互作网络中构建免疫相关基因的蛋白互作网络.免疫相关基因的蛋白互作网络分析及MCL网络模块分析采用生物图表可视化工具Cytoscape软件[10]进行,并应用BinGO2.44[11]对网络模块进行Gene Ontology功能富集分析.
2 结果
2.1 功能富集分析及免疫系统相关差异基因获取
共有2 725个差异基因符合P-value<0.01和|lgFC|>1,且表达量变化超过2倍标准(表1).差异基因最显著变化发生在烧伤后第1 d,有1 825个基因被选出,其中上调基因658个,下调基因1 167个.
小鼠烧伤后不同组间基因功能富集(Gene Ontology)情况如表2所示,其中以烧伤后1 d组的变化最显著,上调和下调差异基因同时富集在细胞过程,生物调节,代谢过程,免疫系统过程及刺激反应等生物学功能中.通过对不同组间基因功能富集情况进行分析发现:免疫系统过程及应对刺激反应等生物学功能一直贯穿烧伤早期,说明烧伤后免疫系统过程在烧伤后起着十分重要的作用.
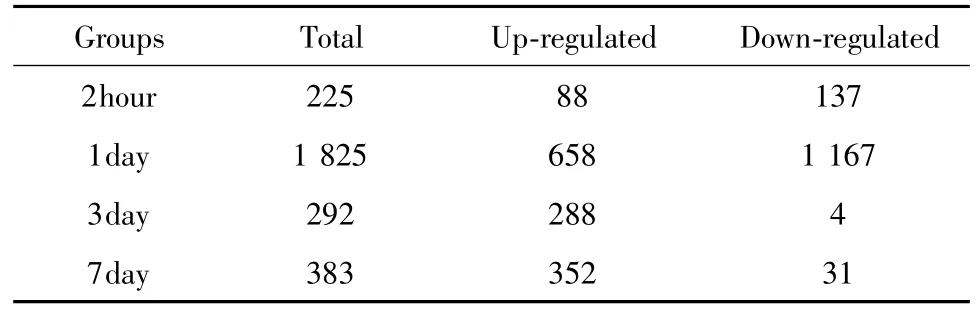
表1 烧伤早期不同时间点差异表达基因Table 1 Signature genes involved in time series stage of burn

表2 差异表达基因功能富集结果Table 2 Results of functional enrichment analysis for DEGs
2.2 免疫相关基因选取及蛋白网络分析
为了进一步了解烧伤早期免疫相关基因功能变化情况,我们将不同时间点的差异基因构建原始蛋白互作网络,并将免疫系统相关基因映射到网络中构建免疫相关基因的蛋白互作网络.同时应用Cytoscape软件对网络进行MCL网络模块化分析及应用BinGO2.44对模块进行Gene Ontology功能富集分析(如图2、表4).
以LCK-CD48为中心的网络模块a(图2-a)为烧伤后2h组免疫互作网络经MCL聚类分析所得,LCK、CD48、CD5,CD80,PTPN6等5个基因在网络模块a中拥有最大互作关系,功能富集分析显示,其在免疫系统过程正性调节中起到重要的作用.
在烧伤后1 d组中原始蛋白互作网络及免疫相关差异基因映射情况如图1所示.MCL富集分析显示Traf6,Lck,Cd19,Tlr2,Myd88,Cd79a,Cxcr5,Ccr2等28个差异基因在网络模块b-f中具有最高互作关系(如表3).功能富集分析显示他们在固有免疫反应,免疫系统正性调节,抗原处理和表达及防御反应中起到关键作用.
网络模块g为烧伤后3 d组MCL模块分析所得,CCR2,MYD88,TLR4,TLR2,在模块中处于中心位置,且拥有最大互作关系,且在炎症反应中有着重要作用.网络模块h-i为烧伤7 d组差异基因经MCL分析所得,Tlr2,MYD88,Gp49a,Clec4n,AF251705等 14个基因拥有最大互作,并在固有免疫反应中起着重要作用.
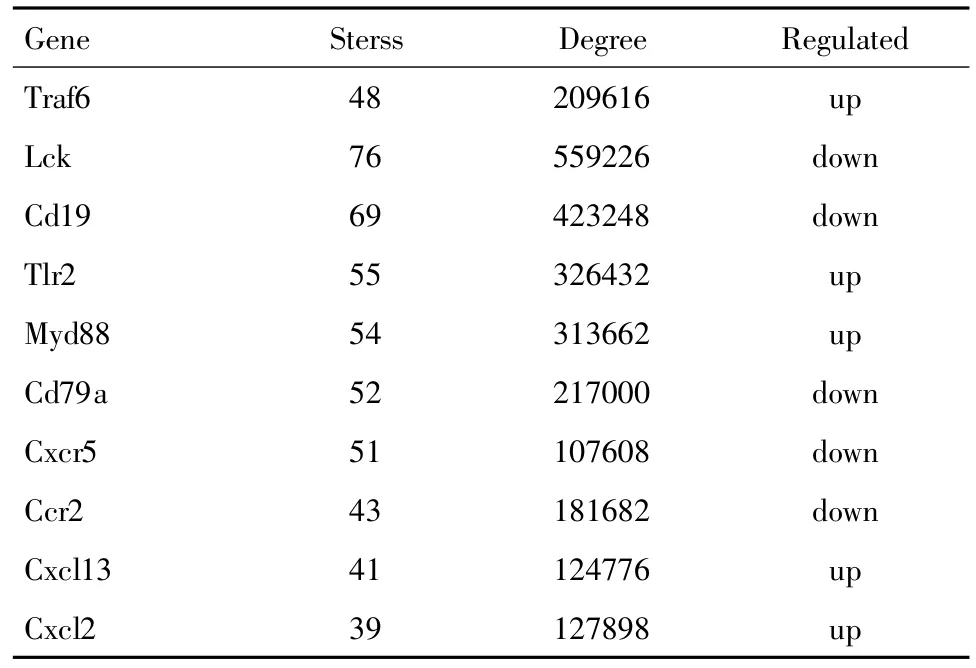
表3 烧伤后1 d组拥有最大互作关系的差异基因(前10)Table 3 The characteristics for the 10 genes in the network

图1 烧伤后第一天原始蛋白互作网络及免疫相关基因(150个)映射情况.红色为免疫系统过程上调基因,蓝色为免疫系统过程下调基因Fig.1 The interaction network for the 150 immune related DEGs in the 1day post burn injury.The red dots represent up-regulated gene and blue dots down-regulated gene.The edges represent interaction.

图2 经MCL聚类分析后各组网络模块.a为烧伤后2小时组网络模块,b-f为烧伤后1天组网络模块,g为烧伤后3天组网络模块,h-i为烧伤后7天组网络模块.红色为免疫系统进程上调基因,蓝色为免疫系统进程下调基因.Fig.2 Clusters a-i and the top eight subnetworks in PPI network in detail.Red nodes represent genes upregulated and blue nodes represent genes downregulated.Cluster a was the subnetworks for 2 hour group post injury;cluster b-f were the subnetworks for 1day group post injury;cluster g was the subnetworks for3day group post injury and cluster h-1 were the subnetworks for7day group post injury.

表4 MCL网络功能富集分析结果Table 4 The largest eight PPI subnetworks
3 讨论
基因芯片作为一种高效、大规模获取生物信息的技术,可以同时捕捉成千上万的基因的动态变化,现已经广泛地用于分子生物学研究中[12].本研究应用小鼠烧伤后免疫细胞时序芯片数据进行初步分析,通过选取免疫系统过程相关基因构建蛋白互作网络,筛选烧伤后免疫系统功能变化过程中可能起到关键作用的基因.通过分析我们发现Lck,Ccr2,TLR2和Myd88等基因可能在烧伤早期免疫功能紊乱发生、发展中起到关键性的作用.
功能富集分析显示Lck(淋巴细胞蛋白酪氨酸激酶)作为烧伤后2 h组和1 d组的下调基因在烧伤后免疫系统正性调节过程中起到重要的作用(P值分别为5.8948E-11,2.8087E-6).LCK基因编码所产生p56(LCK),是非受体酪氨酸蛋白激酶致癌基因家族成员之一,表达于所有的T细胞谱系细胞中[13].p56(LCK)蛋白锚定到细胞膜并与CD4/CD8共同受体的胞内结构域相互作用,并具有酪氨酸激酶活性[14].质膜上的Lck使得TCR复合中的信号模体磷酸化从而启动T细胞受体信号转导过程,然后磷酸化的信号模体为TCR复合招募磷酸化的ZAP-70 PTK分子[15].p56(LCK)是在绝大多数胸腺细胞发育过程中启动TCR信号转导的蛋白酪氨酸激酶[16],对成熟T细胞的选择和成熟中发挥核心作用[17].Lck可能在烧伤后T细胞所介导的免疫功能中起着重要的作用.
CCR2是单核细胞趋化蛋白-1(monocyte chemoattractant protein-1,MCP-1)的特异的受体.CCR2在炎症细胞表面普遍表达.趋化因子MCP-1与CCR2特异性结合后可趋化多种炎症细胞(单核、巨噬细胞、记忆和杀伤T淋巴细胞等)至炎症病灶而发挥免疫作用[18].实验表明CCR2缺乏可抑制MCP-1的基因表达,成纤维细胞积累和巨噬细胞浸润降低TNF-α和IFN-γ基因表达[19].同时研究发现CCR2信号调节细胞外基质蛋白的生产[20].CCR2分析显示CCR2在烧伤后防御反应及炎症反应中起着重要的作用,可能在烧伤后免疫细胞趋化作用及创面修复过程中起着中起着重要的作用.
TLR2和Myd88同时在烧伤后第1d组至第7 d组,功能富集分析显示其在固有免疫反应中起着重要的作用.其中,TLR2为Toll样受体(TLRs)家族成员,为Ⅰ型跨膜蛋白受体,广泛表达于先天免疫细胞中(如巨噬细胞和树突细胞),可以识别细菌和病毒及相关物质等外源性配体.Myd88(髓样分化因子88)作为衔接蛋白在机体固有免疫应答反应中能直接参与到Toll样受体信号转导通路中[21].TLR2是启动Myd88依赖性防御反应的重要的模式识别受体,免疫细胞胞膜上的TLR2受体可特异性识别革兰氏阳性和革兰氏阴性细菌的膜组件可以病原体,并启动Myd88依赖性防御反应,并通过IRAK1,IRAK2和TRAF6[22]等作用可以通过快速激动NF-κB和MAPK传导通路诱导产生促炎性细胞因子(TNFα,IL-1β,IL-6,IL-12)的产生引发前炎性反应并能上调共刺激分子(IL-8,RANTES,MIP-1α)的上调引发先天免疫的快速激活[23-24].TLR2和Myd88在烧伤后可以通过Toll样受体信号转导通路启动机体的固有免疫反应,使机体对入侵的病原体产生最适当的免疫应答,从而维持内环境的稳定.
在本研究中,我们应用生物信息学方法对小鼠烧伤后免疫细胞基因芯片数据进行了分析,并构建免疫系统相关基因的蛋白互作网络.通过互作网络分析我们发现LCK,CCR2,TLR2,Myd88等基因在烧伤后免疫系统功能变化过程中可能起到关键作用.由于分析结果基于基因转录水平数据所得,尽管我们应用更严格的分析方法和工具来确保分析数据和结果的可靠性,但是仍需要一个严谨的生物学实验,以验证分析结果是否适用于不同烧伤面积及深度.
[1] PARK M S,SALINAS J,WADE C E,et al.Combining early coagulation and inflammatory status improves prediction of mortality in burned and nonburned trauma patients[J].J Trauma,2008,64(2):S188-S194.
[2] 彭代智.我国烧伤免疫的研究[J].中华烧伤杂志,2008,24(5):390-392.
[3] LEDERER J A,BROWNSTEIN B H,LOPEZ M C,et al.Comparison of longitudinal leukocyte gene expression after burn injury or trauma-hemorrhage in mice[J].Physiol Genomics,2008,32(3):299-310.
[4] XIAO W,MINDRINOS M N,SEOK J,et al.A genomic storm in critically injured humans[J].J Exp Med,2011,208(13):2581-2590.
[5] IRIZARRY R A,HOBBSB,COLLIN F,et al.Exploration,normalization,and summaries of high density oligo-nucleotide array probe level data[J].Biostatistics,2003,4(2):249-264.
[6] GAO L,GAO H,ZHOU H,et al.Gene expression profiling analysis of the putamen for the investigation of compensatory mechanisms in Parkinson's disease[J].BMC Neurol,2013,13:181.
[7] HUANG D W,SHERMAN B T,LEMPICKIR A.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J].Nat Protoc,2009,4(1):44-57.
[8] ASHBURNER M,BALL C A,BLAKE JA,et al.Gene ontology:tool for the unification ofbiology.The Gene Ontology Consortium[J].Nat Genet,2000,25(1):25-29.
[9] FRANCESCHINI A,SZKLARCZYK D,FRANKILD S,et al.STRING v9.1:protein-protein interaction networks,with increased coverage and integration[J].Nucleic Acids Res,2013,41(Database issue):D808-D815.
[10]SHANNON P,MARKIEL A,OZIER O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J].Genome Res,2003,13(11):2498-2504.
[11]MAERE S,HEYMANS K,KUIPER M.BiNGO:a Cytoscape plugin to assess overrepresentation of gene ontology categories in biological networks[J].Bioinformatics,2005,21(16):3448-3449.
[12]ZHOU B,XU W,HERNDON D,et al.Analysis of factorial time-course microarrays with application to a clinical study of burn injury[J].Proc Natl Acad Sci U SA,2010,107(22):9923-9928.
[13]PAIGE L A,NADLER M J,HARRISON M L,et al.Reversible palmitoylation of the protein-tyrosine kinase p56lck[J].J Biol Chem,1993,268(12):8669-8674.
[14]VANLAETHEM F,TIKHONOVA A N,POBEZINSKY L A,et al.Lck availability during thymic selection determines the recognition specificity of the T cell repertoire[J].Cell,2013,154(6):1326-1341.
[15]GASCOIGNE N R,PALMER E.Signaling in thymic selection[J].Curr Opin Immunol,2011,23(2):207-212.
[16]PALACIOS E H,WEISS A.Function of the Src-family kinases,Lck and Fyn,in T-cell development and activation[J].Oncogene,2004,23(48):7990-8000.
[17]NERVIS,GUINAMARD R,DELAVAL B,et al.A rare mRNA variant of the human lymphocyte-specific protein tyrosine kinase LCK gene with intron B retention and exon 7 skipping encodes a putative protein with altered SH3-dependent molecular interactions[J].Gene,2005,359:18-25.
[18]XIA Y,ENTMAN M L,WANG Y.CCR2 regulates the uptake of bone marrow-derived fibroblasts in renal fibrosis[J].PLoS One,2013,8(10):e77493.
[19]HAUDEK SB,CHENG J,DU J,et al.Monocytic fibroblast precursors mediate fibrosis in angiotensin-Ⅱ-induced cardiac hypertrophy[J].J Mol Cell Cardiol,2010,49(3):499-507.
[20]XU J,LIN SC,CHEN J,et al.CCR2 mediates the uptake of bone marrow-derived fibroblast precursors in angiotensin Ⅱ-induced cardiac fibrosis[J].Am J Physiol Heart Circ Physiol,2011,301(2):H538-H547.
[21]VON BERNUTH H,PICARD C,JIN Z,et al.Pyogenic bacterial infections in humans with MyD88 deficiency[J].Science,2008,321(5889):691-696.
[22]BONNERT T P,GARKA K E,PARNET P,et al.The cloning and characterization of human MyD88:a member of an IL-1 receptor related family[J].FEBS Lett,1997,402(1):81-84.
[23]KAWAIT,AKIRA S.TLR signaling[J].Semin Immunol,2007,19(1):24-32.
[24]UEMATSU S,AKIRA S.Toll-like receptors and innate immunity[J].J Mol Med(Berl),2006,84(9):712-725.
[责任编辑:孙升云 王景周]
Screening of biomarkers for immune system process with post burn injury time-course gene expression profiling data in m ice
GAO Yanbin1, LU Zhiyang1, JIN Hui1, SHI Pengwei1, WANG Guifang1,WEN Huangding1, YANG Lei1
(Department of Burns,Nanfang hospital,Southern Medical University,Guangzhou 510515,China)
Aim:Our study aims to deepen the understandings about the mechanisms of immune system process post-burn injury via screening relevant biomarkers with time-course gene expression profiling data in mice.Methods:Microarray data set GSE7404 was downloaded from GEO database,including 16 burn injury(Mus musculus,25% TBSA,full-thickness)and 16 controls.Student’s t test and fold change method were employed to identified differentially expressed genes with P<0.01 and|lgFC|>1.Then,we used DAVID to perform functional enrichment analysis to uncover dysfunctional biological processes.Immune related Protein-Protein interaction(PPI)network was constructed by STRING and visualized in Cytoscape.Functional analysis of the hub protein was performed by BinGO.Results:The maximumchange of gene expression profile was found at 1-days post injury a total of 658 up-regulated genes and 1167down-regulated genes were identified.The response to stimulus and immune system process were captured by gene expression signature through all time points.Protein-protein interaction network and module analysis suggested that some immune related genes,such as Lck,Ccr2,TLR2 and Myd88 could be of great value for further investigation.Conclusion:It could be inferred that understanding the underlying molecular mechanism post burn injury,may provide novel insight for development of therapeutics strategy.
post burn injury; immune system process; functional enrichment analysis; Immune related Protein-Protein interaction network
R644
A
1000-9965(2015)03-0275-07
10.11778/j.jdxb.2015.03.016
2014-12-08
南方医科大学南方医院院长基金(2014B013)
高艳彬(1988-),男,研究方向:烧伤危重病救治
杨 磊,副教授.Tel:020-61641841;E-mail:yuanyang@fimmu.com
