基于线粒体16S rRNA基因序列的鳢属鱼类系统进化探讨
2015-02-22朱树人山东省淡水渔业研究院山东省淡水水产遗传育种重点实验室山东济南250017上海海洋大学农业部淡水水产种质资源重点实验室上海201306华东师范大学生命科学学院上海200241
朱树人(山东省淡水渔业研究院,山东省淡水水产遗传育种重点实验室,山东济南250017;上海海洋大学农业部淡水水产种质资源重点实验室,上海201306;华东师范大学生命科学学院,上海200241)
孟庆磊,孙玉旋,张延华,朱永安(山东省淡水渔业研究院,山东省淡水水产遗传育种重点实验室,山东济南250017)
基于线粒体16S rRNA基因序列的鳢属鱼类系统进化探讨
朱树人(山东省淡水渔业研究院,山东省淡水水产遗传育种重点实验室,山东济南250017;上海海洋大学农业部淡水水产种质资源重点实验室,上海201306;华东师范大学生命科学学院,上海200241)
孟庆磊,孙玉旋,张延华,朱永安(山东省淡水渔业研究院,山东省淡水水产遗传育种重点实验室,山东济南250017)
[摘要]对4种鳢属(Channa)鱼类共108个个体的16SrRNA基因进行PCR扩增,经比对校正得到572bp(C.striata)和573bp(C.argus,C.maculata,C.asiatica)的基因片段,共检测到10个单倍型,4种鳢所有单倍型之间共存在1个插入/缺失;此外有63个变异位点,其中简约信息位点60个,序列表现出明显的A偏倚;多态位点比例为11.2%;序列中转换多于颠换,转换/颠换之比为1.4。基于Kimu-ra双参数法,计算的种间遗传距离介于0.021[乌鳢(C.argus)与斑鳢(C.maculata)之间]到0.079[乌鳢(C.argus)与线鳢(C.striata)之间、斑鳢(C.maculata)与线鳢(C.striata)之间]。以非洲真副鳢(Parachannainsignis)为外群构建的NJ树和ML树中,线鳢位于进化树的基部,表明线鳢是最早分化出来的种;进化树显示乌鳢和斑鳢亲缘关系最近,表明2个种分化较晚。
[关键词]线粒体DNA;16SrRNA;鳢属;系统进化
鳢科鱼类隶属于鲈形目,仅包括鳢属(Channa)和副鳢属(Parachanna)2个属。鳢属有26个种,原产于亚洲,主要是东南亚;副鳢属有3个种,来自热带非洲[1,2]。因其肉质鲜美、营养价值高,而且具有生肌补血、敛疮生肌、促进伤口愈合等药用价值,富含人体所需的钙、磷、铁、锌等多种微量元素,深受广大消费者的喜爱[3~5]。前人对鳢科鱼类分类的研究偏重于形态比较方面[6],然而形态特征容易受环境影响,且随着更多的鳢科鱼类被发现[7~9],仅从形态特征对这些物种进行分类和亲缘关系分析,会有一定的偏差。
与核DNA相比,线粒体DNA具有结构简单、母系遗传、进化速度较快、几乎不发生重组等特点。因此作为一种重要的分子遗传标记,广泛应用于分子进化、物种鉴定、种群遗传结构和系统发生研究中[10~12]。16S基因是线粒体DNA很重要的基因片段,易扩增并且序列比较保守,因此常用于物种鉴定及种群遗传多样性分析,在鱼类研究中已经广泛得到应用[13~15]。本研究通过对中国常见的4种鳢科鱼类——乌鳢(C.argus)、斑鳢(C.maculata)、月鳢(C.asiatica)和线鳢(C.striata)的线粒体DNA 16S rRNA基因片段序列进行测定,对4种鳢科鱼类的亲缘关系进行分析,以期为鳢属鱼类的分子水平的系统学研究及种间杂交提供参考。
1材料与方法
1.1 材料
4种鳢属鱼类样品于2013年5~10月份采集。乌鳢样品采集于4个湖泊(鄱阳湖、洞庭湖、微山湖、太湖)和1个江域(钱塘江),斑鳢样品采自福建和广东,月鳢和线鳢样品都采自广东。每个群体采集12尾。样本均购自水上作业的渔民或附近的水产品市场,剪取少量胸鳍,保存于无水乙醇中,贴好标签置于4℃冰箱备用。
1.2 DNA提取及PCR扩增
实验前将胸鳍组织从酒精中取出,充分漂洗晾干并剪碎,采用标准的酚-氯仿法提取基因组DNA。提取的DNA取2μL用1.5%的琼脂糖凝胶电泳检测,并稀释到合适浓度用于PCR扩增。
用于扩增线粒体DNA的16S rRNA 基因片段的引物序列为:16SA 5′-CGC CTG TTT ATC AAA AAC AT-3′[16];16SB 5′-CCG GTT GAA CTC AGA TCA-3′[17],引物合成时前面加M13接头。
PCR反应体系为20μL,包括DNA 模板(100 ng/μL)1μL,2×Taq PCR MasterMix (天跟生化)10μL,(10μmol/L)和16SB(10μmol/L)各0.5μL,dd H2O 8μL。PCR扩增程序为:94℃预变性3 min,然后是35个循环,每个循环包括94℃变性30s,58℃退火30s,72℃延伸1min,最后72℃再延伸10min。
扩增产物经过1.5%的琼脂糖凝胶电泳分离后用EB染色并于凝胶成像仪上观察,都能扩增出明显单一亮度很亮的条带,符合预期中的目的条带,扩增产物送上海生工用M13F和M13R双向测序。
1.3 数据分析
利用Clustal X(1.83)[18]对序列进行对位排列,并结合人工校正。通过MEGA5.0软件[19,20]分析序列的碱基组成、变异位点,同时采用Kimura双参数法计算种间的遗传距离。设置非洲真副鳢(Parachannainsignis,AP006042)为外群参考,分别采用NJ法(Neighbor-joining)和ML法(Maximum Likehood)对序列数据进行分析,采用Kimura 2-parameter模型、Bootstrap置信值估算重复次数1000次,最后构建系统进化树。
2结果与分析
2.1 16S rRNA PCR 扩增及序列分析
经PCR扩增,分别得到了4种鳢的16S rRNA基因片段扩增产物,得到序列大小为573bp(乌鳢、斑鳢、月鳢)和572bp(线鳢)。在所测得的108个序列中,共检测到10个单倍型,已提交NCBI(KT358469-K358478)。其中,乌鳢4个单倍型(CA-H1,CA-H2,CA-H3,CA-H4);斑鳢2个单倍型(CM-H1,CM-H2);月鳢2个单倍型(CAS-H1,CAS-H2);线鳢2个单倍型(CS-H1,CS-H2)。4种鳢所有单倍型之间共存在1个插入/缺失,此外有63个变异位点(图1),其中简约信息位点60个,多态位点比例为11.2%。总体上看,序列中转换多于颠换,转换(si=16)/颠换(sv=11)之比为1.4,说明序列突变未达到饱和,各单倍型之间变异位点如图2所示。在所有单倍型中,碱基A(30.2%)占最多,其余T、C、G分别占21.2%、25.9%和22.7%,其中A+T含量(51.4%)高于C+G含量(48.6%)(表1)。

仅列出变异点;H1~H4:单倍型;CA:乌鳢;CM:斑鳢;CAS:月鳢;CS:线鳢图1 4种鳢基于线粒体16S rRNA基因的比对

图2 4种鳢基于16S部分基因序列的NJ树
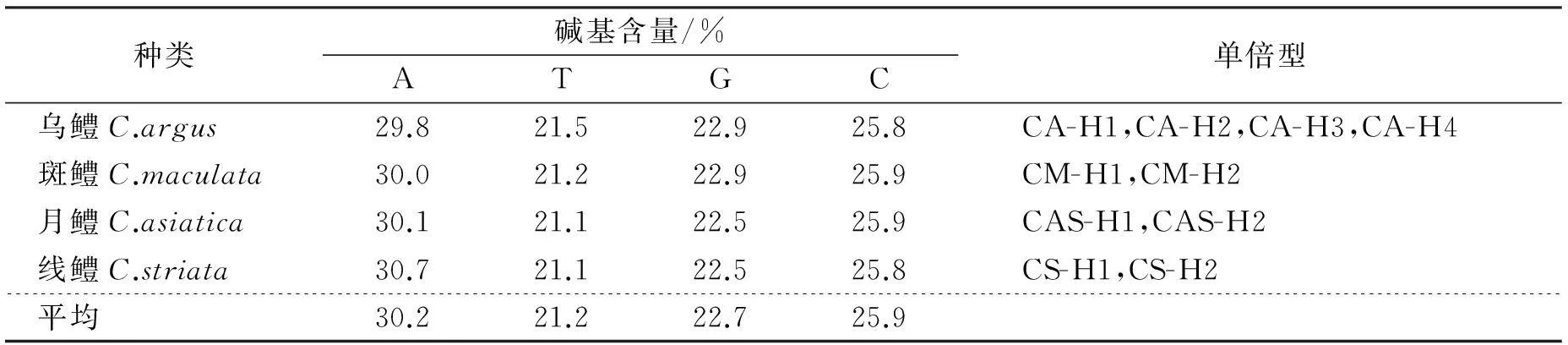
表1 4种鳢线粒体16S rRNA片段碱基组成及单倍型
基于Kimura双参数法,以转换+颠换、转换/颠换计算单倍型间的相对遗传距离(表2)。K2P模型下显示,4种鳢属鱼类种间差异(0.021~0.079)明显大于种内差异(0.002~0.004)。种间最小遗传距离位于乌鳢和斑鳢之间(0.021),种间最大遗传距离位于乌鳢与线鳢之间(0.079)、斑鳢与线鳢之间(0.079)。
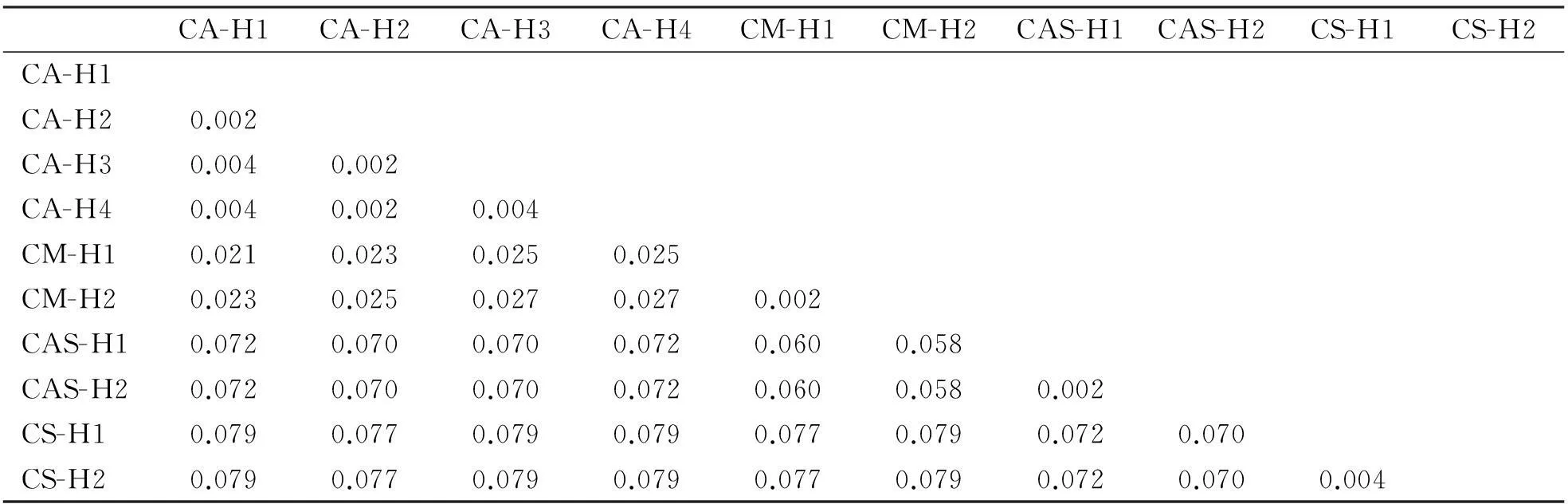
表2 4种鳢基于部分16S基因序列在K2P模型下的遗传距离
注:H1~H4、单倍型;CA、乌鳢;CM、斑鳢;CAS、月鳢;CS、线鳢
2.2 系统发育分析

图3 4种鳢基于16S部分基因序列的ML树
以非洲真副鳢为外群,采用MEGA5.0软件构建了NJ和ML进化树,结果分别见图2和图3。2种类型的系统进化树获得一致的拓扑结构,均支持乌鳢和斑鳢亲缘关系最近,然后和月鳢聚为一支,线鳢位于进化树的基部,是4种鳢属鱼类最早分化出来的种,最后与非洲真副鳢聚在一起。
3讨论
基于16S rRNA序列的保守性和存在的普遍性,目前普遍应用于鱼类的分类鉴定。本研究在扩增16S rRNA时,在合成引物时加了M13接头,与常用于COI部分基因扩增一样[21,22],测得的结果更准确。从108个样品中仅获得10个单倍型,每个物种的单倍型序列差异度最高为0.004,这跟每个群体取得的样本数量有关,同时证实了16S rRNA基因在这4种鳢中同样比较保守。研究中发现,4种碱基所占的百分含量在4种鳢16S rRNA扩增片段中相差不大;在所有单倍型中,碱基A(30.2%)占最多,A+T含量(51.4%)高于C+G含量(48.6%),这与报道的其他鲈形目鱼类16S rRNA基因的组成相似[23]。
NJ和ML构建进化树的方法不一样。NJ法运算速度快,但该算法每迭代运算一次只能够搜索最近邻居配对,对其他可能的配对不加以考虑,只能生成单一最优树,这样可能遗漏一些拓扑结构更合理的次优树;而ML法是考虑了所有可能的途径,能完全利用数据的系统发生信息[24]。在本研究中,不管采用NJ法还是ML法构建进化树,都显示4中鳢属鱼类中,首先聚在一起,乌鳢和斑鳢的亲缘关系最近,这也说明乌鳢和斑鳢是4种中国鳢属鱼类最晚从一个物种分化出来的,表明乌鳢和斑鳢杂交的可交配性较高。Herbert等学者提出物种鉴定差异度临界值为2%[25],本研究中乌鳢和斑鳢的种间遗传距离为0.021,微大于临界值。从理论上讲,有利于杂交鳢的形成。
本研究利用16S rRNA基因序列对4种鳢属鱼类进行了系统进化分析,从分子进化方面为4种鳢属种间鉴定提供了基础参考资料,也为种间杂交提供了依据。
[参考文献]
[1]Li X,Musikasinthorn P,Kumazawa Y.Molecular phylogenetic analyses of snakeheads (Perciformes:Channidae) using mitochondrial DNA sequences [J].Ichthyol Res,2006,53:148~159.
[2]Vishwanath W,Geetakymari KH.Diagnosis and interrelationships of fishes of the genusChannaScopoli (Teleostei:Channidae) of northeastern India[J].Threatened Taxa,2009,1:97~105.
[3]Qin J,Fast A.Food selection and growth of young snakeheadChannastriatus[J].Appl Ichthyol,2007,1:21~25.
[4]Wee K L.Snakeheads-their biology and culture [M].Wee K L.Recent advances in aquaculture,London & Canberra:Croom Helm,1982:180~213.
[5]Sampath K,Pandian T.Interactions of feeding frequency and density on food utilization in air-breathing murrel,Channastriatus[J].Proc Indian Acad Sci,1984,93(5):445~453.
[6]刘苏,朱新平,陈昆慈.斑鳢、乌鳢及其杂交种形态差异分析 [J].华中农业大学学报(自然科学版),2011,30(4):488~493.
[7]Musikasinthorn P.Channapanaw,a new channid fish from the Irrawaddy and Sittang River basins [J].Myanmar Ichthyol Res,1998,45:355~362.
[8]Musikasinthorn P.Channaaurantimaculata,a new channid fish from assam (Brahmaputra River basin),India,with designation of a neotype forC.amphibeus(McClelland,1845) [J].Ichthyol Res,47:27~37.
[9]Musikasinthorn P,Taki Y.Channa siamensis (Günther,1861),a junior synonym ofChannalucius(Cuvier in Cuvier and Valenciennes,1831) [J].Ichthyol Res,2001,48:319~324.
[10]Broughton R E,Milam J E,Roe B A.The complete sequence of the zebrafish (Daniorerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA [J].Genome Res,2001,11:1958~1967.
[11]Chang Y S,Huang F L,Lo T B.The complete nucleotide sequence and gene organization of carp (Cyprinuscarpio) mitochondrial genome [J].J Mol Evol,1994,38:138~155.
[12]Curole J P,Kocher T D.Mitogenomics:digging deeper with complete mitochondrial genomes [J].Trends Ecol Evol,1999,14:394~398.
[13]刘楚吾,徐田军,刘丽,等.笛鲷属 (Lutjanus) 鱼类线粒体 16S rRNA [J].海洋与湖沼,2009,40(5):563~571.
[14]毕潇潇,高天翔,肖永双,等.4 种鳕鱼线粒体 16S rRNA,COI 和 Cyt b 基因片段序列的比较研究 [J].南方水产科学,2009,5(3):46~52.
[15]郑文娟,朱世华,邹记兴,等.基于16S rRNA 部分序列探讨12种鲹科鱼类的分子系统进化关系 [J].水产学报,2009,32(6):46~52.
[16]Simons C,Frati F,Beckenbach A,etal.Evolution,weighting,and phylogenetic utility of mitochondrial gene sequences and a compilation of conserved polymerase chain reaction primers [J].Ann Entomol Soc Am,1994,87:651~701.
[17]Kambhampati S,Smith P.PCR primers for the amplification of four insect mitochondrial gene fragments [J].Insect Mol Biol,1995,4:233~236.
[18]Thompson J D,Gibson T J,Plewniak F,etal.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,5:4876~4882.
[19]Kimura M.A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences [J].J Mol Evol,1980,16:111~120.
[20]Tamura K,Peterson D,Peterson N,etal.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods [J].Mol Biol Evol,2011,28:2731~2739.
[21]Ward R D,Zemlak T S,Innes B H,etal.DNA barcoding Australia's fish species [J].Phil Trans R Soc B,2005,360:1847~1857.
[22]Ivanova N V,Zemlak T S,Hanner R H,etal.Universal primer cocktails for fish DNA barcoding [J].Mol Ecol Notes,2007,7:544~548.
[23]Marescalchi O.Karyotype and mitochondrial 16S gene characterizations in seven South American Cichlasomatini species (Perciformes,Cichlidae) [J].J Zool Syst Evol Res.2007,43(1):22~28.
[24]李涛,赖旭龙,钟扬.利用 DNA 序列构建系统树的方法 [J].遗传,2004,26(2):205~210.
[25]Hebert P D,Ratnasingham S,deWaard J R.Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species [J].Proceedings of Biological Sciences,2003,270 (Suppl):S96~99.

[引著格式]朱树人,孟庆磊,孙玉旋,等.基于线粒体16S rRNA基因序列的鳢属鱼类系统进化探讨[J].长江大学学报(自科版),2015,12(27):25~29.
[中图分类号]Q812;Q959.48
[文献标识码]A
[文章编号]16731409(2015)27002505
通信作者:
[作者简介]朱树人(1986-),男,博士,助理研究员,研究方向为水产动物遗传育种。朱永安,zhuyongan1965@163.com。
[基金项目]上海高校知识服务平台项目(ZF1206);山东省农业良种工程项目;国家大宗淡水鱼产业技术体系项目(CARS-46-37)。
[收稿日期]2015-08-15
