海洋来源真菌Aspergillus sp.SCSIO 81-F2抗菌活性次级代谢产物的研究
2015-01-11罗明和黄洪波李沉纹于彩平马俊英卢来春鞠建华
罗明和,黄洪波,李沉纹,于彩平,马俊英,卢来春*,鞠建华*
1 第三军医大学大坪医院野战外科研究所药剂科,重庆 400042;2中国科学院热带海洋生物资源与生态
重点实验室中国科学院海洋微生物中心广东省海洋药物重点实验室中国科学院南海海洋研究所,广州 510301
自上世纪50 年代从海洋来源的头孢霉菌(Cephalosporium)分离得到头孢菌素C 以来[1],海洋真菌天然产物研究越来越受到人们的关注。海洋所独具的低温、高压、高盐、寡营养等特殊的环境,为海洋真菌产生结构新颖、活性独特的代谢产物提供了可能。目前从海洋真菌的发酵产物中分离鉴定的新化合物已经超过1000 个,并且数量还在不断增加;其结构类型主要是聚酮类和生物碱,其次是萜类、肽类。人们从这些数量众多的海洋真菌次级代谢产物中筛选到有药用价值的、具有抗肿瘤、抗菌、抗病毒等活性的抗生素[2-4]。其中,Diketopiperazine halimide 最具有代表性,是由加州大学圣地亚哥分校的Fenical 研究小组发现的,它能抑制微管蛋白聚合,在此基础上研发的该化合物的结构类似物plinabulin(NPI-2358)正在进行治疗非小细胞肺癌的临床二期研究[5,6]。
本课题组在对南海海洋来源真菌活性次级代谢产物的持续研究中,发现了一些新的活性次级代谢产物:从海洋来源曲霉属真菌Aspergillus sp.SCSIO F063 分离获得了少见的氯代及溴代蒽醌[7],从虫草属真菌Acremonium persicinum SCSIO 115 中分离获得了具有细胞毒活性的环肽[8],从炭角属真菌Xylaria sp.SCSIO 156 中分离得到具有细胞毒活性的细胞松弛素[9],从青霉属真菌Penicillium sp.SOF 07中获得了有免疫酶抑制剂活性的霉酚酸类化合物[10]。最近,我们发现一株南海海洋来源的真菌SCSIO 81-F2 的发酵产物对枯草芽孢杆菌Bacillus subtilis ATCC6633、苏云金芽孢杆菌Bacillus thuringiensis ATCC10792、金黄色葡萄球菌Staphyloccocus aureus ATCC29213 和大肠杆菌 Escherichia coli ATCC25922 具有抑菌活性。随后,我们通过18S rDNA 序列分析并构建系统发育进化树,鉴定该菌株属于曲霉属真菌Aspergillus sp.。采用土豆培养基(PDB)发酵,利用抑菌活性追踪与色谱层析技术相结合的方法从其发酵产物中分离获得三个化合物,经ESI-MS、1H/13C NMR 及X-单晶衍射分析鉴定为含异腈基的抗生素xanthocillin X dimethylether(1)、xanthocillin X monomethylether(2)、6',8'-dimethoxy xanthocillin X dimethylether(3)。本文报道该菌株的分子生物学及由该菌株产生的异腈基类化合物的分离和鉴定。
1 材料与方法
1.1 仪器与材料
Bruker DRX 500 核磁共振仪(500/125 MHz,TMS 为内标);Bruker amazon SL 离子肼质谱仪;Oxford Gemini A Ultra X-单晶衍射仪;Varian ProStar 210 高效液相色谱仪(配PDA 检测器);YMC-pack ODS-A 色谱柱(250×10 mm,5 μm);HS-GF254硅胶薄层板(烟台江友硅胶开发有限公司);色谱纯乙腈(安徽时联公司),其它试剂均为国产分析纯。
海洋真菌SCSIO 81-F2 是从中国南海北部深海沉积土中分离纯化而来,菌种保存于中国科学院海洋微生物研究中心。
PDA 培养基:去皮马铃薯(200 g/L),切成小块,加水煮沸30 min,砂布过滤,取滤液加葡萄糖(20 g/L),海盐(30 g/L),2 % 琼脂粉,115 ℃灭菌30 min,冷却后备用。
PDB 培养基:PDA 培养基配方中不加琼脂粉,115 ℃灭菌30 min,冷却后备用。
1.2 实验方法
1.2.1 海洋真菌81-F2 的分子生物学鉴定
1.2.1.1 18S rDNA 序列PCR 扩增
用真菌18S 通用引物,ITS1:(5'-TCCGTAGGTGAACCTGCGG-3')和ITS4:(5'-TCCTCCGCTTATTGATATGC-3')[11],进行PCR 扩增。PCR 反应体系(20 μL):DNA 模板1 μL,10×Buffer 2 μL,dNTPs(2.5 mmol/L)1.6 μL,ITS1 primer(10 μmol/L)0.4 μL,ITS4 primer(10 μmol/L)0.4 μL,HiFi(5U/μL)0.2 μL,ddH2O 14.4 μL。PCR 反应条件为:94℃预变性4 min;94 ℃变性1 min,58 ℃退火45 s,72℃延伸40 s,30 个循环;72 ℃延伸10 min;4 ℃保温。
1.2.1.2 扩增产物的序列测定
用凝胶回收试剂盒(OMEGA)纯化PCR 产物,克隆至pCR2.1 载体(INVITROGEN)上,18S rDNA序列由美吉生物技术有限公司测定。将测得的18S rDNA 序列进行BLAST 分析(http://blast.ncbi.nlm.nih.gov/)。
1.2.1.3 系统发育树的构建
通过CLUSTAL X 软件对海洋真菌菌株SCSIO 81-F2 及真菌中最相似菌株的18S rDNA 序列进行比对,使用软件MEGA4.0 中的Neighbor-joining 法构建系统进化树[12,13]。
1.2.2 菌株的发酵培养
用250 mL 锥形瓶,每瓶加入50 mL PDB 培养基,从平板上接入海洋真菌81-F2 的菌丝体,于28℃、200 rpm 摇床上培养2 d 后分别转入含200 mL PDB 培养基的1 L 锥形瓶中,于28 ℃、200 rpm 培养8 d。
1.2.3 抗菌活性检测
取5 mg/mL 的样品提取物DMSO 溶液5 μL,加到直径为0.6 cm 无菌滤纸片上,置于生长有枯草芽孢杆菌Bacillus subtilis ATCC6633、苏云金芽孢杆菌Bacillus thuringiensis ATCC10792、金黄色葡萄球菌Staphyloccocus aureus ATCC29213 和大肠杆菌Escherichia coli ATCC25922 的LB 检测平板,37 ℃培养24 h,测量抑菌活性。
1.2.4 发酵产物的提取和分离
发酵产物(6 L)于4000 rpm 离心10 min,分离得到上清液和菌丝体。取上清液及菌丝体分别用乙酸乙酯和丙酮萃取得少量粗提物进行抑菌活性检测,结果显示菌丝体的萃取物有活性,上清液萃取物无活性。同时,HPLC 检测发现菌丝体的提取物有次级代谢产物色谱峰,而上清液中未见。于是弃去上清液,将菌丝体用丙酮萃取3 次,经减压浓缩后得菌丝体粗提物总浸膏6 g。采用正相柱层析,氯仿-甲醇体系梯度(100∶0,98∶2,96∶4,94∶6,92∶8,90∶10,8∶2,5∶5,V/V)洗脱,得到Fr.1~Fr.8 共8 个馏份,馏份Fr.1,Fr.2 有抑菌活性。对馏份Fr.1 用常压正相硅胶柱层析,乙酸乙酯-石油醚梯度(10∶90,20∶80,30∶70,40∶60,50∶50,V/V)洗脱,反复纯化3次,最后用二氯甲烷对较纯产物重结晶得到化合物1。Fr.2 采用正相柱层析,氯仿-甲醇梯度(100∶0,98∶2,96∶4,94∶6,92∶8,9∶1,V/V)洗脱,经HPLC 检测分析合并Fr.2-(2-5),再对Fr.2-(2-5)用半制备高效液相色谱(CH3CN/H2O 30%~100% 梯度洗脱20 min,流速2.5 mL/min)纯化,得到化合物2(tR=15.5 min)、3(tR=19.4 min)。
2 结果与讨论
2.1 菌株鉴定
18S rDNA 序列分析表明菌株SCSIO 81-F2 与曲霉菌属Aspergillus unguis DFFSCS009 及Aspergillus sp.SCSIOF063 菌株的18S rDNA 序列相似性较高,所以鉴定为Aspergillus sp.81-F2,其菌落形态见图1,系统发育树见图2。

图1 菌株SCSIO 81-F2 在PDA 培养基上的形态图Fig.1 Phenotype of strain SCSIO 81-F2 on PDA-medium plate

图2 基于18S rDNA 序列构建的SCSIO 81-F2 与其他真菌菌种NJ 分子系统进化树Fig.2 Neighbour-joining tree based on 18S rDNA sequences showing relationships between strain SCSIO 81-F2 and closely related members of fungi
2.2 结构鉴定
化合物1 黄色块状晶体,(+)ESI-MS 给出分子准离子峰m/z 317.1[M+H]+、339.0[M+Na]+及655.3[2M+Na]+,据此推测化合物1 的分子量为316。13C NMR 谱仅显示8 个C 信号,含一个低场区信号δC173.3,一个连氧芳碳信号δC161.1,五个芳碳信号,一个连氧脂肪碳信号δC55.4,提示该化合物存在对称结构。1H NMR 谱揭示4 处质子信号δH7.79、6.99、6.98、3.86,其积分比为2∶1∶2∶3,因此,推测其分子式为C20H16N2O2。进而推断13C NMR 谱中低场区碳信号δC173.3 可能为异腈基碳信号。再仔细分析1H NMR 谱信号,在低场区存在芳环的邻位偶合系统δH7.79(d,J=8.5 Hz)、6.98(d,J=8.5 Hz),推断分子中可能含有对位取代的苯环结构;δH6.99(s)为孤立烯键质子信号,说明分子中存在双键;δH3.86(s)结合δC55.4 碳信号,推断分子中存在甲氧基。经查阅文献[14],与报道的xanthocillin X dimethylether 的波谱数据一致,故化合物1 鉴定为xanthocillin X dimethylether。经培养获得了化合物1 的单晶,进一步运用X-单晶衍射验证了化合物1 的结构(图4)。
1H NMR(CDCl3,500 MHz)δ:7.79(4H,d,J=8.8 Hz,H-5,5';9,9'),6.99(2H,s,H-3,3'),6.98(4H,d,J=8.8 Hz,H-6,6';8,8'),3.86(6H,s,H-10,10');13C NMR(CDCl3,125 MHz)δ:173.3(C-1,1'),161.1(C-7,7'),131.7(C-5,5';9,9'),127.4(C-3,3'),124.8(C-4,4'),116.1(C-2,2'),114.4(C-6,6';8,8'),55.4(C-10,10')。

图3 化合物1 的X-单晶衍射结构图Fig.3 The X-ray structure of compound 1
化合物2 黄色粉末,(+)ESI-MS 给出分子准离子峰m/z 303.0[M+H]+、325.0[M+Na]+、655.3[2M+Na]+,推测化合物2 的分子量为302,较化合物1 少14 个质量数,推断其分子式为C19H14N2O2。化合物2 的1H NMR 谱在低场区有δH7.78与6.99(J=8.9 Hz),7.71 与6.92(J=8.7 Hz)两对典型的苯环邻位质子偶合系统,δH6.98 处有一个呈单峰的烯氢信号,δH3.89 存在一个甲氧基质子信号。13C NMR 给出15 个碳信号:低场区δC172.6,172.4 两个碳信号,两个连氧芳碳信号δC161.3 及159.5,10 个芳碳信号,以及一个连氧脂肪碳信号δC55.1。推断化合物2 存在两个对位取代苯环,两个双键,两个异腈基,以及一个甲氧基,与化合物1 比较,少了一个甲氧基。经查阅文献[15],与报道的波谱数据一致,故鉴定为Xanthocillin X monomethylether。
1H NMR(CD3OD,500 MHz)δ:7.78(2H,d,J=8.9 Hz,H-5,9),7.71(2H,d,J=8.7 Hz,H-5',9'),6.99(2H,d,J=8.9 Hz,H-6,8),6.98(2H,s,H-3,3'),6.92(2H,d,J=8.7 Hz,H-6',8'),3.89(3H,s,H-10);13C NMR(CD3OD,125 MHz)δ:172.6(C-1),172.4(C-1'),161.3(C-7),159.5(C-7'),132.2(C-5',9'),131.9(C-5,9),128.3(C-3'),127.5(C-3),125.1(C-4),123.9(C-4'),116.4(C-2),116.2(C-6',8'),115.5(C-2'),114.7(C-6,8),55.1(C-10)。
化合物3(+)ESI-MS 给出分子准离子峰m/z 399.2[M+Na]+及775.2[2M+Na]+,因此化合物3 的分子量为376,推断其分子式为C22H20N2O4。1H NMR 谱低场区给出苯环邻位偶合体系的特征信号δH7.81(d,J=8.5 Hz)、6.99(d,J=8.5 Hz),说明分子中存在对位取代苯环。1H NMR谱还给出呈单峰的烯氢信号δH7.08、7.06、7.00,积分比为2∶1∶1,提示分子中存在两个双键,以及一个1,3,4,5-四取代苯环;另外还有甲氧基质子信号δH3.93 和3.88,积分比为3∶1,推断分子中存在4 个甲氧基。13C NMR 谱低场区δC173.9 和δC173.4 对应两个异腈基碳,高场区在δC61.0、56.3、55.5 有三个甲氧基碳信号,其中56.3 处的信号显著偏高,为两个碳原子。经比对文献数据[16],发现与6',8'-dimethoxy xanthocillin X dimethylether 的波谱数据一致,因此,化合物3 为6',8'-dimethoxy xanthocillin X dimethylether。
1H NMR(CD3OD 500 MHz)δ:7.81(2H,d,J=8.5 Hz,H-5,9),7.08(2H,s,H-5',9'),7.06(1H,s,H-3),7.00(1H,s,H-3'),6.99(2H,d,J=8.5 Hz,H-6,8),3.88(3H,s,H-10),3.93(9H,s,H-10',11',12');13C NMR(CDCl3125 MHz)δ:173.9(C-1),173.4(C-1'),161.3(C-7),153.3(C-6',8'),140.0(C-7'),131.9(C-5,9),131.8(C-3'),131.4(C-3),128.2(C-4'),127.8(C-4),127.4(C-2'),124.7(C-2),114.5(C-6,8),107.3(C-5',9'),61.0(C-10'),56.3(C-11',12'),55.5(C-10)。
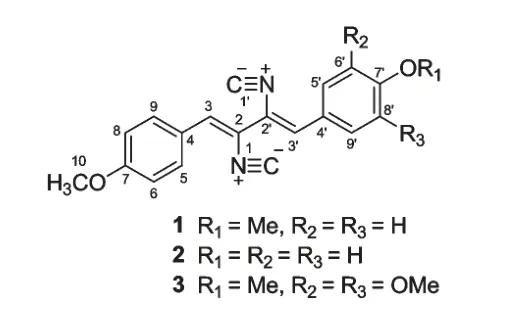
图4 化合物1、2、3 的化学结构式Fig.4 Chemical structures of compounds 1,2 and 3
2.3 讨论
Xanthocillin 最早于1950 从一株青霉Penicillium notatum Westling 的发酵产物中得到,并于1957年由Hagedorn 和Tonjes 确定其化学结构[17]。它们都带有一个异腈基,具有很强的生物活性,是一类广谱抗生素,能抑制大部分革兰氏阴性和阳性菌以及部分真菌如大肠杆菌、枯草芽胞杆菌、金黄色葡萄球菌、白色念珠菌、酵母菌等的生长,并能抑制耐青霉素和磺胺类药物的耐药菌的生长。但是由于它在哺乳动物的胃肠中吸收很差,所以只能做成软膏作为外用制剂使用。Xanthocillin 类化合物不具有溶血效应,但是在剂量过大时会有神经毒活性。报道最多的Xanthocillin X monomethylether 还能特异性的抑制花生四烯酸到前列腺素H2 的生物合成,它的衍生物对实体瘤也有很好的抑制活性,其成果已被日本的Hiroshi Kurihara 等申请专利保护[18,19]。另外有报道显示Xanthocillin 作为促血小板生成素模仿物,能选择性诱导c-Mpl-expressed UT-7 细胞的生长,极有可能开发成促血小板生成的新药物,Xanthocillin X monomethylether 同时还能诱导一些蛋白的磷酰化[16]。本研究从一株海洋来源的真菌中分离获得了Xanthocillin 类化合物,为该类化合物提供了新的来源,为其结构改造和药理药效研究提供了物质基础。
1 Pollegioni L,Rosini E,Molla G.Cephalosporin C acylase:dream and(/or)reality.Appl Microbiol Biotechnol,2013,97:2341-2355.
2 Huang Z(黄智),Wang FZ(王发左),Zhang S(张偲).Secondary metabolites of fungi from marine sediments.Nat Prod Res Dev(天然产物研究与开发),2013,25:129-139.
3 Zhu WM(朱伟明),Wang JF(王俊锋).A review on studies of secondary metabolites from marine fungi.Mycosystema(菌物学报),2011,30:218-228.
4 Rateb ME,Ebel R.Secondary metabolites of fungi from marine habitats.Nat Prod Rep,2011,28:290-344.
5 Fenical W,Jensen PR,Cheng XC.Halimide,a cytotoxic marine natural product,and derivatives thereof.US Pat,6069146,2000-05-30.
6 Kingston DG.Tubulin-interactive natural products as anticancer agents.J Nat Prod,2009,72:507-515.
7 Huang H,Wang F,Luo M,et al.Halogenated anthraquinones from the marine-derived fungus Aspergillus sp.SCSIO F063.JNat Prod,2012,75:1346-1352.
8 Chen Z,Song Y,Chen Y,et al.Cyclic heptapeptides,codyheptapeptides C-E,from marine-derived fungus Acremonium persicinum SCSIO 115.J Nat Prod,2012,75:1215-1219.
9 Chen Z,Huang H,Chen Y,et al.New cytochalasins from the marine-derived fungus Xylariasp.SCSIO 156.Helv Chim Acta,2011,94:1671-1676.
10 Chen Z,Zheng Z,Huang H,et al.Penicacids A-C,three new mycophenolic acid derivatives and immunosuppressive activities from the marine-derived fungus Penicillium.sp.SOF07.Bioorg Med Chem Lett,2011,22:3332-3335.
11 White TJ,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics,pp.315-322 in M.A.Innis,Gelfand DH,Sninisky JJ,White TJ(Eds):PCR Protocols:a Guide to Methods and Applications.Academic Press,San Diego(USA),1990.
12 Thompson JD,Gibson TJ,Plewniak F,et al.The ClustalX windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Res,1997,25:4876-4882.
13 Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0.Mol Biol Evol,2007,24:1596-1599.
14 Takatsuki A,Suzuki S,Ando K,et al.Kunio Ando et al.New antiviral antibiotics:xanthocillin mono and dimethylether and methoxy xanthocillin X dimethylether.Isolation and characterixation.J Antibiot,1968,21:671-675.
15 Ando K,Tamura G,Arima K.A new antibiotic,1-(phydroxyphenyl)-2,3-diiocyano-4-(p-methoxyphenyl)-butal,3-diene.II Elucidation of the structure.J Antibiot,1968,21:587-591.
16 Sakai R,Nakamura T,Nishino T,et al.Xanthocillins as thrombopoietin mimetic small molecules.Bioorg Med Chem,2005,13:6388-6393.
17 Hagedorn H,Tonjes H.Konstitutionsaufklarung von Xanthocillin,einem neuen Antibiotikum.Parmazie,1957,12:567-580.
18 Takatsuki A,Tamura G,Arima K.New antiviral antibiotics:xanthocillin X mono and dimethylether,and methoxy-xanthocillin X dimethylether.J Antibiot,1968,21:676-680.
19 Kurihara H,Watanabe H,Koyama M,et al.Xanthocillin monomethyl ether derivative and antitumor agent containing the same.US Patent,5210097,1993-05-11.
