HLA-DR4 分子与抗类风湿性免疫活性肽构效关系的研究进展
2015-01-09吕翠翠
吕翠翠,曹 慧,徐 斐
上海理工大学医疗器械与食品学院,上海 200093
类风湿性关节炎(rheumatoid arthritis,RA)是一种以关节滑膜炎为特征的慢性全身性自身免疫性疾病,发病率约占我国人口的0.35%~0.4%,其中致残率可达15%,然而目前对RA 尚没有一个较好的治疗方法。最近在研究人类RA 的过程中,一些学者报道了口服免疫活性肽产生免疫耐受可使RA 的发病过程受到抑制,其机制为T 细胞对特异性肽段的识别作用。而有效的免疫耐受的激发依赖于:抗原递呈细胞(Antigen Processing Cells,APCs)加工处理蛋白抗原成8~10 个氨基酸组成的抗原肽,并与其表面的HLA-DR4 分子形成HLA-DR4/肽复合物,供受体特异性T 细胞表面受体(TCR)所识别,进一步结合成TCR-HLA/DR4-抗原肽三分子复合体,T细胞被活化、增值并分泌各种免疫因子而启动免疫应答[1]。
目前一些食源性免疫活性肽已经被制备或合成,并应用于RA 的预防和治疗,但对其构效关系仍缺乏系统的研究。因此,本文从AntiJen 数据库或相关文献中查阅到和RA 有关的免疫活性肽,并对其与HLA-DR4 分子的构效关系进行了深入探讨。
1 与RA 相关的免疫活性肽
免疫活性肽是一类具有促进淋巴细胞分化成熟、转移免疫信息机及增强机体免疫机能等生物学功能的肽[2]。到目前为止,科研工作者已从乳蛋白、大豆蛋白、玉米蛋白、鱼贝类蛋白、胶原蛋白等蛋白酶解物中分离出多种具有免疫活性的肽段。然而,与RA 相关的免疫活性肽的开发却十分有限,不同来源的氨基酸组成及结构的免疫活性肽的IC50值差异也较大(见表1)。

表1 与RA 有关的免疫活性肽及活性值Table 1 Immunoactive peptides and the IC50values related to RA
2 HLA-DR4 分子的结构特征
HLA-DR4 分子由MHC II 基因群编码,由于其最先在白细胞表面被发现且含量较高,所以称为人类白细胞抗原(human leucocyte antigen,HLA)。有足够的证据发现和RA 有关的人类白细胞抗原主要为HLA-0401、0404、0405 等亚型,其中绝大部分为HLA-DR4* 0401[3,4]。Lanchbury[5-7]等通过研究不同地区RA 患病情况的结果表明HLA-DR4* 0401相对于其他等位基因,其致病的可能性更高,尤其是对严重关节炎患者,其92%~95% 的发病是由HLA-DR4* 0401 分子引起的。由于HLA-DR4 分子在人类免疫疾病中起重要作用,其结构已被广泛研究。发现其分子晶体结构是由α 链和β 链以非共价键连接的异源二聚体,α 链由α1 和α2 结构域构成,β 链由β1 和β2 结构域构成,α1 和β1 结构域各含有1 个α 螺旋和4 个β 片层,它们形成了抗原肽结合槽(peptide-binding groove),其中,α 螺旋为结合槽壁,β 片层为结合槽底[8](见图1)。该抗原结合槽的两端是开口的,所以抗原肽在结合槽内可以向两端伸展。抗原肽主要借助特定锚定残基(anchor residue)驻扎在HLA-DR4 分子槽底部深浅不同的“口袋”(Pocket)中,其中,最重要的6 个口袋分别是Pocket 1、2、4、6、7、9(命名为P1、P2、P4、P6、P7、P9)(见图2、图3)[9]。这些Pockets 富含多态性氨基酸,且某一等位基因编码的HLA-DR4 分子只能识别具有某一特定结构的一组抗原肽[10,11]。根据HLA-DR4 分子结构及CD4+T 细胞识别抗原肽序列的研究发现,HLA-DR4 分子的抗原结合槽一般可以容纳长度为13 个氨基酸残基的抗原肽,但能被CD4+T 细胞识别的抗原肽仅有9 个氨基酸残基,这9 个氨基酸序列称为核心结合序列(core binding sequence),它在抗原肽与MHC II 类分子的特异性结合中起关键作用[12-14]。

图1 HLA-DR4 的分子结构图[10]Fig.1 Structural and molecular characteristics of HLA-DR4

图2 HLA-DR4 分子的6 个主要抗原结合口袋[11]Fig.2 The peptide-binding groove of HLA-DR4
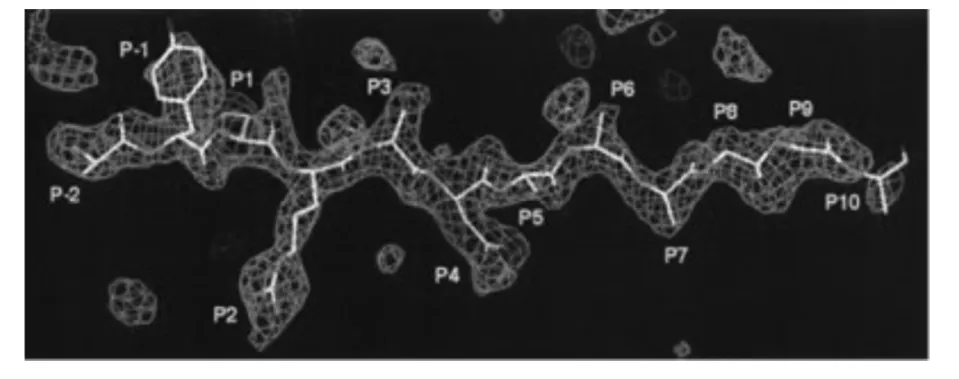
图3 HLA-DR4 分子的抗原肽结合口袋[9]Fig.3 Antigenic peptide binding pocket of HLA-DR4
3 抗RA 免疫活性肽与HLA-DR 分子的作用机理
目前,已有学者对抗RA 免疫活性肽与其功能之间的关系进行了研究,发现抗RA 免疫活性肽氨基酸残基的带电性质、大小及疏水性等性质直接影响其与HLA-DR4 分子的结合[15-17]。因而,对抗RA免疫活性肽的性质与HLA-DR4 X 射线衍射晶体结构之间的关系进行深入探讨,有助于更好的解释其构效关系。
根据与RA 相关的免疫活性肽的氨基酸组成可见(表1),其P-2 位置的氨基酸绝大部分为丙氨酸。Fugger[18]等研究证实P-2 位置的丙氨酸丢失可导致抗原肽与HLA-DR4 分子的结合能力损失6-7 倍,但对T 细胞的活化没有影响。Dessen[9]等通过X 晶体射线研究表明该位置侧链上的氢键对于TCR 与HLA-DR4 分子的结合起重要作用,这可能是由于侧链形成的氢键容易从肽-HLA-DR4 结合物中突出延伸出来,从而有利于其结合。由此可见,P-2 位置上的丙氨酸可能主要起到结合HLA-DR4 分子的作用。P-1 和P3 位置的氨基酸绝大部分为甘氨酸。Fugger[18]等研究发现P-1 位置甘氨酸丢失可导致抗原肽与HLA-DR4 分子的结合能力损失40 倍,并会影响T 细胞的活化。P3 位置甘氨酸的作用与之类似,即侧链形成的氢键从肽-HLA 结合物中突出延伸出来,使TCR 与HLA 分子结合的更紧密。Rosloniec[19]和Andersson[20]等研究还发现,在P3 位置用丙氨酸替代甘氨酸,可使抗原肽与HLA-DR4 分子的结合能力显著增强。由此说明,此位置氨基酸残基的立体特性与抗原肽的免疫活性密切相关,因而推测分子较小的甘氨酸残基更适合此位置。
置于Pocket 1 中的P 1 位置的氨基酸绝大部分为苯丙氨酸,其次为亮氨酸和缬氨酸。这三种氨基酸都是疏水性氨基酸,具有疏水性侧链。Andersson[20]等通过动物实验研究发现P1 的苯丙氨酸被带有芳香族侧链的氨基酸,如酪氨酸取代对T 细胞的活化仅有微弱的影响。但P1 的苯丙氨酸(F)丢失对T 细胞的活化有显著影响[18]。因为苯丙氨酸的疏水性侧链易与Pocket 1 内壁上的氨基酸(多为β 链上的Gly86)相互作用形成疏水内核,提高了抗原肽与HLA-DR4 分子的亲和力及形成复合物的稳定性,从而有利于抗原肽被MHC 分子递呈到抗原递呈细胞的表面[21]。HLA-DR4 分子的Pocket 1 为较深的疏水性口袋,主要由α 链上的残基Ile7、Ile31、Phe32、Phe34、Trp43 及β 链上的残基Val85、Gly86、Phe89 和Thr90 组成[12]。由此可见,P-1 位置氨基酸残基的亲水特征与抗原肽的免疫活性密切相关。
Hammer[22,23]等 研 究 发 现HLA-DR4 分 子 的Pocket 2 只接受带负电的氨基酸,但从搜集的抗RA免疫活性肽的组成来看,出现在Pocket 2 中的多为带正电的赖氨酸和精氨酸,这可能是由于HLA-DR4分子与抗原肽的结合不仅取决于氨基酸的带电性质,还与其分子大小密切相关。Dessen[9]通过X-射线衍射获得了HLA-DR4 的晶体结构,发现HLADR4 分子中的Pocket 2 比较深,只有长侧链的氨基酸残基才会被紧密的绑定到其结合槽中(见图3),从而有利于T 细胞受体识别。而赖氨酸和精氨酸的侧链都比较长,符合Pocket 2 的特点。此外,精氨酸的侧链可突出结合槽,使其更容易被T 细胞受体识别,而且精氨酸的胍基团所带正电荷可以与HLADR4 分子Pocket 2 中Thr-β77 的氧原子形成氢键,从而增强其结合力。Edward[24]等研究也发现,P1的苯丙氨酸和P2 的赖氨酸对抗原肽与HLA 分子的结合至关重要,如果取代了这两个位置的氨基酸,其结合力显著下降。
Pocket 4 是由β 链第70 位至74 位一段高度保守的五氨基酸序列,即QKRAA 构成的一个肽锚定区域[25,26]。由于Pocket 4 底部β71 位置上的赖氨酸(K)或精氨酸(R)带正电荷,因而推测其对抗原肽中带负电荷的氨基酸残基有较高的亲和力,而带正电荷的氨基酸残基则抑制其结合。这与表1 中P4位置绝大部分为谷氨酸,其次是天冬氨酸是一致的。Fugger[18]的研究表明带负电的谷氨酸的侧链伸向槽内,和带正电的Pocket 4 的肽结合槽的结合能力非常强,从而有利于T 细胞识别。同时,Dessen[9]等研究也发现,Pocket 4 适合侧链比较大的氨基酸。20 种天然氨基酸中符合既带负电又有较长侧链的的氨基酸只有谷氨酸和天冬氨酸两种,而谷氨酸的侧链比天冬氨酸的长,所以P4 的谷氨酸对于抗原肽与MHC 分子的结合性起非常重要的作用。
Li 等[27-29]通过单个氨基酸替换代实验和计算机模拟等方法证实P5 和P8 的氨基酸对HLA 分子与T 细胞受体的接触起重要作用。由表1 可见,这两个位置绝大多数的氨基酸分别是谷氨酰胺和赖氨酸,侧链都较长,而HLA-DR4 分子P5 和P8 处的口袋都不深(见图3),由此推测该位置氨基酸长侧链会突出肽-HLA-DR4 结合槽,从而有利于与TCR 相作用。Jardetzky[30]等通过晶体结构分析研究也发现T 细胞的活化需要抗原肽与HLA-DR4 分子在抗原呈递细胞表面相接触,有较长侧链的氨基酸的一部分侧链和TCR 相结合,而另一部分则包埋在HLADR4 分子的抗原结合槽口袋里。Fugger[18]等研究表明删除了该位置的赖氨酸,对T 细胞的刺激性显著降低。由此推测,P5 和P8 对HLA 分子与T 细胞受体的接触也起重要作用。
Dessen[9]和Hammer[22]等通过研究发现,在HLA-DR4分子的α链上有两个保守氨基酸,分别为Glu11 和Asp66,这两个氨基酸在空间上彼此靠近并形成氢键,从而阻止Pocket 6 与带电荷的氨基酸或长链氨基酸发生相互作用,因此Pocket 6 易于容纳小体积、不带电的或非长链的氨基酸残基。此外,由图3 也可看出,组成Pocket 6 的氨基酸形成了一个较浅的小口袋,这进一步说明此位置只适合侧链较短的氨基酸。
结合于Pocket 7 中的氨基酸绝大部分为脯氨酸(见表1)Fugger[18]等研究表明如果剔除P7 的脯氨酸,HLA-DR4 分子与抗原肽的结合力及对T 细胞的刺激作用都明显降低。Andersson[20]等研究也发现如果用丙氨酸替换P7 的脯氨酸会导致抗原肽与HLA-DR4 分子的结合能力降低5-10 倍。这可能是由于Pocket 7 比较浅但底部较宽(见图3),所以比较适合侧链短且大的氨基酸,而脯氨酸正好符合此特点,这与Dessen[9]等研究的脯氨酸适合浅口袋的Pocket 7 是一致的。
由图3 可看出,Pocket 9 非常浅,所以该位置只适合侧链比较小的氨基酸。Dessen[9]等研究也表明分子很小的甘氨酸适合Pocket 9。Andersson[20]等的研究表明用带极性的氨基酸替换P9 的甘氨酸后抗原肽与HLA-DR4 分子的结合能力增强5~10 倍,这有可能是因为Pocket 9 中HLA-DR4 分子的β57天冬氨酸可以和α76 精氨酸形成盐桥,从而易于结合带极性侧链的氨基酸,比如亮氨酸、异亮氨酸、缬氨酸、丝氨酸等。
抗原结合槽一般容纳13 个氨基酸的抗原肽,但能被T 细胞识别的抗原肽仅有9 个氨基酸,所以P10 和P11 的氨基酸对整个分子的结合力做出的贡献非常小,由图3 可见,这两个位置的口袋非常浅,而与之结合的氨基酸侧链又比较长,所以侧链与结合槽的作用不一定紧密,该位置所提供的分子结合力很有限。Andersson[20]等的研究表明用丙氨酸替换P10 的谷氨酸后抗原肽与HLA-DR4 分子的结合能力增加,这证明了以上假说是成立的。
4 结论
本研究对抗类风湿性免疫活性肽与HLA-DR4分子的结合机制进行了深入探讨。抗类风湿性免疫活性肽起主要免疫活性作用的9 个氨基酸核心结合序列为P1-P9,核心结合序列两端的氨基酸对肽的结合性的影响很小。作为主要锚点的P1 位疏水性氨基酸和次要锚点的P6 位极性小分子可包埋在HLA-DR4 分子的结合槽中,因而对免疫活性肽与HLA-DR4 分子的结合尤为重要。而P2 位的带正电荷氨基酸,P3 位的极性小分子,P4 位带负,长侧链的氨基酸以及P5 位带长侧链氨基酸主要负责其与HLA-DR4 分子的绑定,在这几个位置的氨基酸侧链会突出肽-HLA 分子结合槽,从而有利于TCR 的识别。P7、P8 和P9 位的口袋较浅,因而其与抗原肽氨基酸残基的结合力相对较弱。
1 Song Z(宋哲).Application of PLS method in the T cell epitopes prediction.Dalian:Dalian University of Technology(大连理工大学),PhD.2008.
2 Chen L (陈路),et al.Bioactive peptide (or oligopeptide)feed additive:Research and applications.Acta Zoonutri Sin(动物营养学报),2004,16(2):12-14.
3 Gonzalez Gay MA,et al.Influence of human leukocyte antigen-DRB1 on the susceptibility and severity of rheumatoid arthritis.Semin Arthritis Rheu,2002,31:355-360.
4 Goronzy JJ,et al.Prognostic markers of radiographic progression in early rheumatoid arthritis.Arthritis Rheum,2004,50:43-54.
5 Lanchbury JS,et al.Strong primary selection for the Dw4 subtype of DR4 accounts for the HLA-DQw7 association with Felty's syndrome.Hum Immunol,1991,32:56-64.
6 Nepom GT,et al.MHC Class-II Molecules and Autoimmunity.Annu Rev Immunol,1991,9:493-525.
7 Ranges GE,et al.Prevention of type II collagen-induced arthritis by in vivo treatment with anti-L3T4.J Exp Med,1985,162:1105-1110.
8 Yu CY(于畅宇),et al.Prediction of the cell epitopes based on ISC-SVR method.J Chem (化学学报),2013,71:670-678.
9 Dessen A,et al.X-ray crystal structure of HLA-DR4 (DRA* 0101,DRB1* 0401)complexed with a peptide from human collagen II.Immunity,1997,7:473-481.
10 Ferrante A,et al.Cooperativity of hydrophobic anchor interactions:evidence for epitope selection by MHC class II as a folding process.J Immunol,2007,178:7181-7189.
11 Agudelo WA,et al.Quantum chemical analysis of MHC-peptide interactions for vaccine design.Mini-Rev Med Chem,2010,10:746-758.
12 Madden DR.The three-dimensional structure of peptide-MHC complexes.Annu Rev Immunol,1995,13:587-622.
13 Jardetzky TS,et al.Crystallographic analysis of endogenous peptides associated with HLA-DR1 suggests a common,polyproline II-like conformation for bound peptides.Proc Natl Acad Sci USA,1996,93:734-738.
14 Zhu Y,et al.Crystal structure of MHC class II I-Ab in complex with a human CLIP peptide:prediction of an I-Ab peptide-binding motif.J Mol Biol,2003,326:1157-1174.
15 Hammer J,et al.Precise prediction of major histocompatibility complex class II-peptide interaction based on peptide side chain scanning.J Exp Med,1994,180:2353-2358.
16 Marshall KW,et al.Prediction of peptide affinity to HLA DRB1* 0401.J Immunol,1995,154:5927-5933.
17 Southwood S,et al.Several common HLA-DR types share largely overlapping peptide binding repertoires.J Immunol,1998,160:3363-3373.
18 Fugger L,et al.Specificity of an HLA-DRBl* 0401-restricted T cell response to type II collagen.Eur J Immunol,1996,26:928-933.
19 Rosloniec EF,et al.HLA-DR1(DRB1* 0101)and DR4(DRB1* 0401)use the same anchor residues for binding an immunol dominant peptide derived from human type II collagen.J Immunol,2002,168:253-259.
20 Andersson EC,et al.Definition of MHC and T cell receptor contacts in the HLA-DR4 restricted immunodominant epitope in type II collagen and characterization of collagen-induced arthritis in HLA-DR4 and human CD4 transgenic mice.Proc Natl Acad Sci USA,1998,95:7574-7579.
21 Falk K,et al.Allele-specific motifs revealed by sequencing of self-peptides elated from MHC molecules.Nature,1991,351:290-296.
22 Hammer J,et al.Peptide binding specificity of HLA-DR4 molecules:correlation with rheumatoid arthritis association.J Exp Med,1995,181:1847-1855.
23 Friede T,et al.Natural ligand motifs of closely related HLADR4 molecules predict features of rheumatoid arthritis associated peptides.Hum Immunol,1996,1316:85-101.
24 Edward F,et al.HLA-DR1 (DRB1~* 0101)and DR4(DRB1~* 0401)use the same anchor residues for binding and immunodominant peptide derived from human type II collegen.J Immunol,2002,168:253-259.
25 Jiang Z(蒋真),et al.Review on the research and development of HLA-DRB1 alleles on RA.Chin J Rheumatol (中华风湿病学杂志),2005,9:690-693.
26 du Montcel ST,et al.New classification of HLA-DRB1 alleles supports the shared epitope hypothesis of rheumatoid arthritis susceptibility.Arthritis Rheum,2005,52:1063-1068.
27 Li R,et al.Altered collagen II peptides inhibited T-cell activation in rheumatoid arthritis.Clin Immunol,2006,118:317-323.
28 Zhou Q,et al.Inhibition of T-cell activation with HLA-DR1/DR4 restricted non-T-cell stimulation peptides.Hum Immunol,2003,64:857-865.
29 Myers LK,et al.Peptide-induced suppression of collagen-induced arthritis in HLA-DR1 transgenic mice.Arthritis Rheum,2002,46:3369-3377.
30 Jardetzky TS,et al.Crystallographic analysis of endogenous peptides associated with HLA-DR1 suggests a common polyproline II-like confirmation of bound peptides.Proc Natl Acad Sci USA,1996,93:734-738.
