CRISPR/Cas9基因组编辑技术及其在动物基因组定点修饰中的应用
2015-01-03周金伟徐绮嫔姚婧余树民曹随忠四川农业大学动物医学院成都630中国科学院动物研究所计划生育生殖生物学国家重点实验室北京000
周金伟,徐绮嫔,姚婧,余树民,曹随忠. 四川农业大学动物医学院,成都 630;. 中国科学院动物研究所,计划生育生殖生物学国家重点实验室,北京 000
CRISPR/Cas9基因组编辑技术及其在动物基因组定点修饰中的应用
周金伟1,徐绮嫔1,姚婧2,余树民1,曹随忠1
1. 四川农业大学动物医学院,成都 611130;2. 中国科学院动物研究所,计划生育生殖生物学国家重点实验室,北京 100101
CRISPR/Cas系统是细菌和古生菌中抵抗外源病毒或质粒入侵的获得性免疫系统,利用 CRISPR RNAs (crRNAs)引导Cas核酸酶沉默入侵的核酸。通过分子生物学改造使Ⅱ型CRISPR/Cas系统成为一种高效的基因组定点修饰技术,并且比锌指核酸酶(Zinc-finger nucleases, ZFNs)和 TALE核酸酶(Transcription activator like effector nucleases, TALENs)结构更简单,更容易设计和应用。文章主要介绍了CRISPR/Cas9系统成为高效基因组定点修饰技术的发展历程、Ⅱ型CRISPR/Cas的工作原理和改造过程以及在动物基因组定点修饰的应用,剖析了该技术存在的问题和现有改进方案,并与成功案例相结合展望了 CRISPR/Cas9系统的应用前景,以期为动物性状改良和人类疾病动物模型的创立提供新思路。
CRISPR/Cas9;基因组编辑技术;人类疾病动物模型;基因组定点修饰
Keywords:CRISPR/Cas9; genome editing technique; animal models of human disease; site-directed genome modification
传统的动物性状改良是通过转基因技术将外源基因插入到受体基因组中,使外源基因整合在染色体基因组上改造基因功能,使其表达优良性状,并稳定地传给下一代。然而转基因技术存在如下缺点:如外源基因随机整合与效率低下,表达可控性差,转基因动物存在生物安全风险,并且不易获得表型良好且稳定遗传的后代。因此,动物的性状改良和人类疾病动物模型的创制迫切需要发展动物基因组定点修饰的精细基因编辑技术。
基因组定点编辑技术是实现动物品种改良和构建人类疾病动物模型的重要研究工具,与传统的转基因技术相比,基因编辑技术不依赖于胚胎干细胞,能够应用于更多物种,并且具有效率高、定向修饰精确、所需时间短以及得到的突变可以稳定遗传等优点。近几年,序列特异性的核酸酶发展迅速,并且这些核酸酶具有基因组靶向修饰的能力。其中锌指核酸酶[1](Zinc-finger nucleases, ZFNs)和类转录激活因子效应物核酸酶[2,3](Transcription activator like effector nucleases, TALENs)都是由序列特异DNA结合结构域与DNA核酸内切酶Fok Ⅰ的切割结构域组成。这些核酸酶能够在特定的基因组位置造成DNA双链断裂(Double strand break, DSB),激活细胞内固有的同源重组(Homologous-directed repair, HDR)或非同源末端连接(Non-homologous end joining, NHEJ)对断裂的DNA进行修复,实现基因组的定点修饰,并在一些物种(如小鼠[4]、斑马鱼[5]、牛[6]、猪[7])中成功实现了基因组定点修饰。最近,在基因组定点修饰方面有了突破性的进展,科学家发现了一种新的基因组定点编辑技术——Clustered regularly interspaced short palindromic repeats (CRISPR)-associated nuclease 9 (CRISPR/Cas9)系统[8]。CRISPR/Cas9系统是利用RNA引导核酸酶对基因组DNA进行定点修饰,现已广泛地应用于小鼠[9]、斑马鱼[10~12]、猪[13,14]、羊[15]等多个物种,暗示CRISPR/ Cas9基因组编辑技术能够摆脱动物无干细胞系的限制,并且可在任何动物中实现定向、精准的基因修饰。
1 CRISPR/Cas系统
CRISPR系统是原核生物中一种抵抗外源基因侵入的获得性免疫系统[16],在细菌和古生菌中CRISPR系统整合侵入宿主的噬菌体或者质粒DNA的片段到 CRISPR位点,然后通过相应的 CRISPR RNAs (crRNAs)引导Cas核酸内切酶损坏外源DNA序列,抵抗病毒或者噬菌体的入侵[17]。
1.1 CRISPR/Cas发展历程
1987年,日本学者首次在大肠杆菌(E. coli)基因组中发现一连串短的空间间隔重复序列[18],2002年科学家才将其命名为成簇的规律间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats, CRISPR)[19],现有测序结果显示40%的细菌基因组中含有CRISPR位点。2005年,科学家们发现 CRISPR中的许多间隔序列来源于质粒和病毒,CRISPR的基因座能够被转录,并且Cas基因编码的蛋白具有核酸酶和解旋酶结构域,因此推测CRISPR/ Cas是利用无义的 RNAs标记外源核酸序列的防御系统[20]。2008年,CRISPR/Cas被证明具有免疫能力,在E. coli中成熟的crRNAs引导Cas蛋白形成复合体,抑制病毒的增殖[21]。2012年,Jinek等[8]首次利用Ⅱ型的CRISPR/Cas系统实现了目的DNA特定位点双链断裂,为CRISPR/Cas系统用于基因组定点编辑奠定了基础。
1.2 CRISPR/Cas的结构
CRISPR是一种特殊的DNA重复序列,由高度保守的重复序列(Repeats)与间隔序列(Spacers)交叉排列组成(图1),在CRISPR前段有一些保守的Cas蛋白基因,这些Cas基因在CRISPR的防御体系中起着关键作用,并在Cas基因附近有一段帮助crRNAs成熟的转录活化RNA(Trans-activating crRNA, tracrRNA)。
CRISPR基因座之间都有一个重复序列隔开,转录这些基因座形成前体crRNA,然后通过处理加工形成成熟的crRNA,crRNA中的间隔序列能够引导效应核酸酶复合体切割与间隔序列互补配对的双链DNA。细菌固有的RNA引导免疫系统在执行免疫过程中包括3个不同的步骤:(1)获得外源DNA;(2)合成成熟crRNA,并形成RNAs-Cas核酸酶蛋白复合体;(3)通过crRNA识别外源DNA并利用Cas核酸酶损坏外源DNA[22]。
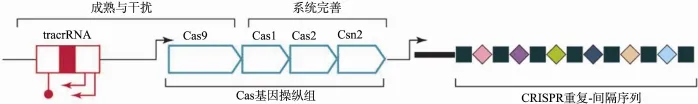
图1 CRISPR结构示意图(参考文献[23]并修改)
1.3 Ⅱ型 CRISPR/Cas系统的工作原理和 CRISPR/ Cas9基因组编辑技术的形成
在CRISPR/Cas系统的3种类型(Type Ⅰ、TypeⅡ和Type Ⅲ)中,Type Ⅱ系统具有简单、高效以及功能多样化等优点,是用于基因组修饰技术最合适的选择。Ⅱ型系统通过crRNA的引导利用单个Cas9核酸酶充分切割目的DNA的靶位点,并且能够对基因组的任何位置进行切割。
Ⅱ型CRISPR/Cas系统由间隔重复CRISPR基因座转录出来的pre-crRNA、多功能的Cas9核酸酶和tracrRNA组成,其中tracrRNA促进pre-crRNA的成熟,以及形成具有切割活性的crRNA-tracrRNA-Cas9三元复合体。Ⅱ型CRISPR/Cas系统对外源基因的干扰需要多个步骤:首先,tracrRNA与pre-crRNA的重复序列结合;第二,内源RNase Ⅲ切割pre-crRNA/ tracrRNAs复合体,切除5′末端的间隔序列,形成成熟的crRNA,并与tracrRNA形成被称为向导RNA (Single guide RNA, sgRNA)的嵌合RNA分子;第三,每一个成熟的sgRNA能够引导Cas9蛋白定位到双链 DNA的靶位点上并在原型间隔毗邻序列(Protospacer adjacent motif, PAM)的上游3~8 bp位置对结合的序列进行切割(图2A)。Cas9的特异性靶点依赖于原型间隔序列3′端PAM序列,不同的Cas9突变体在基因组中拥有不同的PAM序列,来源于酿脓链球菌(Streptococcus pyogenes)的 Cas9识别一个 5′-NGG-3′的PAM,而来源于嗜热链球菌(Streptococcus thermophilus)和脑膜炎奈瑟氏菌(Neisseria meningitidis)的 Cas9分别识别 5′-NNAGAAW-3′的 PAM 和5′-NNNNGATT-3′的PAM[24]。Cas9拥有两个核酸酶结构域,分别是蛋白中间的HNH结构域和蛋白氨基酸末端附近的RuvC-like结构域,其中HNH切割与crRNA互补的链,切割位点位于PAM序列上游3 bp,RuvC-like切割非互补链,切割位点位于PAM序列上游3~8 bp,从而造成双链的断裂[8]。CRISPR/ Cas9产生的 DSB能够激活细胞和生物体自身修复机制,产生两种不同的修复途径NHEJ和HDR修复(图 2B)。NHEJ能够高效地引入不同长度的插入或者缺失突变,导致转录阅读框编码序列,转录激活相关的启动子和增强子的损坏;而HDR的修复通常会引入特定位点的突变或者在外源供体 DNA模板存在时对靶位点进行可预测的插入[25]。
2012年Jinek等[8]在对Ⅱ型CRISPR/Cas系统的基因沉默机制进行研究时发现,crRNA的原型间隔序列和基因组中原型间隔毗邻序列决定了 Cas9在基因组中的靶位点,并且只有当crRNA和tracrRNA同时存在形成一种双链二级结构时 Cas9才能对质粒DNA或者短的双链DNA进行切割,因此Jinek等将crRNA的3’末端与tracrRNA的5’末端相连,使crRNA: tracrRNA成为单一转录的融合RNA,此融合RNA具有crRNA与tracrRNA互补配对的发卡结构从而模仿野生型crRNA: tracrRNA的成对RNA结构引导Cas9对DNA特定位点进行切割。在融合RNA的验证实验中发现tracrRNA 3’末端的长度将影响Cas9对DNA靶位点的识别效率,tracrRNA 3’末端的长度比crRNA:tracrRNA配对区域长5~12个碱基时Cas9对DNA靶位点的识别效率最高。通过改变crRNA中的原型间隔序列成功实现了Cas9在GFP基因中多个位点的打靶,开启了CRISPR/Cas9系统进行基因组编辑的序幕。
1.4 CRISPR/Cas9打靶区域和sgRNA打靶位点的选择
对于sgRNA来说,选择什么样的启动子来表达这一段RNAs可能会限制靶位点的选择。RNA聚合酶Ⅲ依赖的U6或者T7启动子在对RNA进行转录时分别需要在RNA的5′端多一个G或者GG。因此这些 sgRNA的表达就会形成 GN16-19NGG或者GGN15-18NGG的gRNA,导致在随机的基因组DNA中每32 bp或者128 bp才会出现一个靶位点。为了检测Cas9造成靶位点的双链断裂,需要在NGG的5′上游3 bp处设计一个用于限制性片段长度分析的酶切位点。一种策略就是减少靶位点设计的限制条件,比如放弃RNAs 5′端的第一个或者前两个碱基(此种方式将会使sgRNA在这些位置与基因组出现错配)。另外一种策略就是先设计出sgRNAs 5′端无G或者GG序列,然后在进行引物合成时sgRNA 5′端加入G或者GG,因此转录出的gRNA会比设计的长1~2 bp。以上两种方法都能合成有活性的sgRNA,但是在基因修饰中会使Cas9核酸酶有不同的效率[26]。
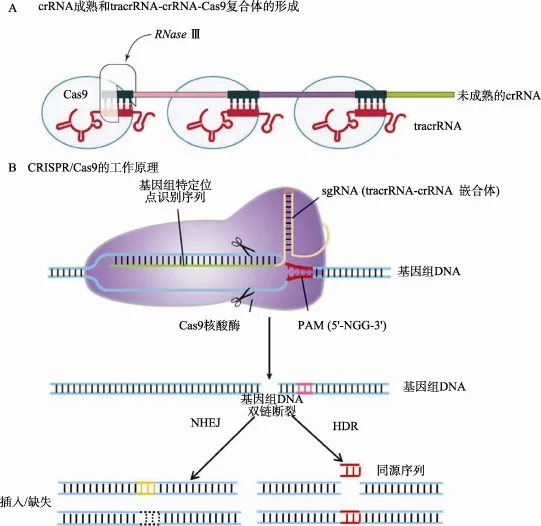
图2 Ⅱ型CRISPR/Cas系统的成熟与工作原理(参考文献[23]并修改)
CRISPR/Cas9对基因组 DNA的特异切割需要两个主要的因素:一个是 20 bp的引导序列,另一个是存在于sgRNA结合位点3′末端的PAM序列。sgRNA中20 bp的序列能够和基因组的DNA靶位点互补配对,也能够和基因组中成千上万的其他序列进行不完美的配对(脱靶)。研究表明,Cas9的活性和切割特异性由靠近PAM序列的8~12 bp决定,并且PAM末端的部分错配不会影响Cas9的活性,因此必须重视CRISPR/Cas9的脱靶效应[27]。
针对sgRNA的设计策略,在设计基因组靶位点的引导序列时,研究者需要对引导序列的特异性和潜在脱靶位点进行评估。对于靶位点的选择,研究者可以使用一些在线设计工具,如:ZiFiT Targeter software (http://zifit.partners.org)[28]和麻省理工学院张锋实验室建立的CRISPR设计工具(http://crispr.mit.edu/)[29]。其中张锋实验室的在线设计工具能够预测在基因组中的潜在脱靶位点,并根据脱靶位点错配碱基数位置和分布进行脱靶评分(分数反映脱靶的可能性),并且能够给每一个设计的 gRNA进行评分(分数高的表示脱靶效率低)。通过软件预测,选择脱靶位点少并且靶序列在外显子上的靶位点。在实验中还应该对5个潜在脱靶序列进行检测,评估靶序列的脱靶效应。
2 CRISPR/Cas9在基因组编辑中的应用
2013年,Cong等[30]利用Jinek改造的CRISPR /Cas9系统最早在人类细胞和小鼠细胞中成功实现了基因组的定点编辑,开启了基因组编辑的新时代。之所以称其开启了基因组编辑的新时代,是因为CRISPR/Cas9系统一方面同样具有ZFNs和TALENs的基因编辑可遗传以及修饰多样性等优点,另一方面还具有其独特优势:(1) CRISPR/Cas9的靶点在基因组中拥有非常高的分布频率,在基因组中几乎每8 bp就有一个靶点;(2) 可以同时对多个靶点进行基因操作;(3) 实验周期短,设计简单,节约大量的时间和经济成本。
到目前为止,CRISPR/Cas9被广泛地应用于多个物种和多种细胞类型,并且获得了高效的基因修饰结果(表1)。
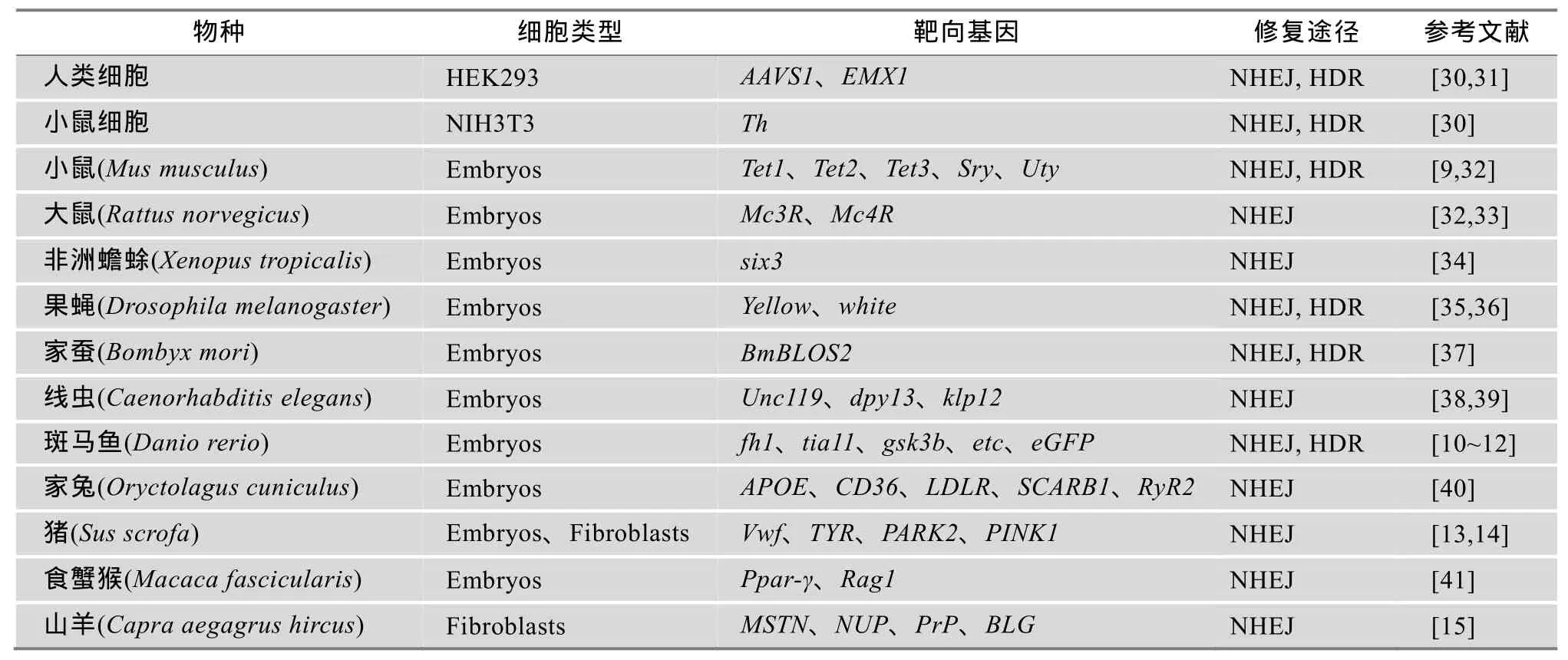
表1 通过CRISPR/Cas9基因定点编辑的细胞类型和生物体
Cong等[30]构建了两种类型的Ⅱ型 CRISPR/ Cas9系统,证明缩短的sgRNA(Truncated gRNA, trusgRNA)能够提高CRISPR/Cas9系统的特异性;PAM序列3′ 端碱基错配不会影响剪切效率;并且把多个引导序列集合在一个gRNA上能够引导Cas9实现基因组多位点的同时编辑,显示了CRISPR/Cas9系统操作简单、应用范围广的特点。Thomas等[12]利用CRISPR/Cas9系统,在斑马鱼胚胎中通过显微注射的方法成功实现了 Gal4定点编辑,并且在注射sgRNA和Cas9 mRNA的同时加入了与目的Gal4突变位点同源的含有eGFP的供体DNA序列,成功实现了 eGFP的插入,效率达到了 22%。Yin等[42]利用CRISPR/Cas9系统在斑马鱼眼中实现了ascl1a基因的条件性敲除,对斑马鱼光感受器的再生机制研究提供了新的方向。Wang等[9]在小鼠胚胎中同时注射体外转录的Cas9 mRNA和多基因sgRNAs实现了(Tet1、Tet2、Tet3、Sry和Uty)多基因同时定点突变。Wu等[43]利用CRISPR/Cas9对一只Crygc显性突变的小鼠进行基因治疗使其获得了健康的后代,共同注射Cas9 mRNA和靶向Crygc基因的sgRNA到Crygc显性突变合子中,利用外源的寡聚核苷酸或者内源的WT等位基因作为供体通过同源重组修复方式对 Crygc基因进行修复,获得了能够生育并且能将修正后的 Crygc基因传给下一代的健康小鼠,此项成果为CRISPR/Cas9系统用于遗传疾病的基因治疗提供了依据。Li等[44]利用多个sgRNAs共同作用于人和老鼠中,成功实现了DNA调控元件和基因簇的倒位和串联,并且能够高效正确的倒位和串联几十bp甚至几百kb的DNA片段;在人类细胞中4 个sgRNAs的共同作用实现了Pcdh基因簇的成功倒位和串联;在小鼠中利用成对的 sgRNAs实现了基因组任意片段大小的删除和染色体倒位,突变效率与两个sgRNAs在染色体中的间隔长度有关。
虽然CRISPR/Cas9系统被改造为基因编辑工具仅两年多时间,但是CRISPR/Cas9技术用于动物品种改良和人类疾病动物模型的创制已经有了一些可喜的成果。例如,南京大学模式动物研究所黄行许教授课题组首次利用CRISPR/Cas9技术获得了内源Ppar-γ、Rag1基因突变的灵长类动物食蟹猴(Macaca fascicularis)[41];Zhou等[14]利用 CRISPR/Cas9技术与体细胞核移植(Somatic cell nuclear transfer, SCNT)的有机结合,获得了TYR、PARK2、PINK1突变猪;Ni等[15]获得了MSTN、NUP、PrP与BLG基因突变的山羊;Sato等[45]获得了 α-1,3-半乳糖苷酶(α-1, 3-galactosyltransferase,α-GalT)双等位基因缺失的猪胎儿成纤维细胞,为人类异种器官移植研究提供了帮助;Chen等[46]利用 CRISPR/Cas9系统对猕猴(Macaca mulatta)的肌萎缩蛋白基因(Dystrophin gene)进行了嵌合打靶突变,其突变效率达到了87%,人类临床上杜氏肌萎缩症疾病(Duchenne muscular dystrophy, DMD)患者都存在肌萎缩蛋白基因突变,该研究发现肌萎缩蛋白基因缺失的猕猴出现肌营养蛋白明显减少以及肌肉萎缩的表型与人类早期 DMD临床表型相似。这些成果为利用CRISPR/Cas9系统对动物进行基因修饰、品种培育和人类疾病动物模型创制提供了重要的参考依据。
Hai等[13]和 Zhou等[14]以猪为研究对象,应用CRISPR/Cas9系统建立了基因修饰的动物疾病模型,将对人类疾病和家畜疫病的发病机理和治疗方法的研究起到推动作用。猪是我国最重要的家畜品种之一,同时也是生物医学研究中重要的模式动物。小型猪在心血管、胃肠道等多项生理特征上比啮齿类更接近人类,其器官大小与功能也与人十分接近,而且其产仔量高易繁殖。基于此,对猪进行精细的基因组修饰,从而建立用于研究猪的疾病或人类疾病的模型,对于畜牧生产和生物医学研究都具有重要价值。赖良学研究组利用CRISPR/Cas9系统获得TYR敲除的猪胎儿成纤维细胞(Pig fetal fibroblasts, PFF),然后通过SCNT 得到成活的TYR敲除猪,TYR敲除导致猪存在典型的白化表型[14];周琪领导的研究团队直接向广西巴马小型猪的受精卵中注射定点切割vWF的Cas9 mRNA和sgRNA,高效地获得了双等位基因突变的广西巴马小型猪,人类临床上Ⅰ型和Ⅲ型血管性血友病患者都存在vWF的突变,该研究还发现这些vWF敲除猪具有严重的凝血功能障碍,与血管性血友病患者的临床表型相似[13]。最近本研究团队还应用CRISPR/Cas9技术与显微注射技术获得小眼畸形相关转录因子(Microphthalmia-associated transcription factor, MITF)双等位基因缺失的广西巴马小型猪,其体内突变效率达到了100%。
2015年CIRSPR/Cas9在致病基因筛选方面有了新的突破。Chen等[47]将含有Cas9-GFP表达质粒的原癌细胞通过小鼠全基因组 CRISPR敲除引导文库处理,并将处理后的细胞皮下注射到小鼠体内,5周后发现这些原癌细胞转移到肺部,通过分离和测序分析肺部癌症细胞,发现 Nf2、Pten、Trim72、Cdkn2a、Fga、Cryba4、miR-152和miR-345等基因与肿瘤进化和转移有关。这项研究使得利用CIRSPR/Cas9在体内鉴别癌症和其他复杂疾病中的重要基因迈出了关键一步。
3 CRISPR/Cas9基因编辑技术的发展趋势
CRISPR/Cas9基因编辑技术的发展趋势可分为两个方面:(1)CRISPR/Cas9基因编辑技术自身的完善;(2)CRISPR/Cas9基因编辑技术新的生物学功能开发。
3.1 CRISPR/Cas9系统的自我完善
CRISPR/Cas9系统靶位点只有20 bp左右,并且在基因组中每8 bp就存在一个靶位点,因此能够灵活地实行基因定点编辑,可以将多个引导序列串联在一起引导 Cas9实现基因组多位点或多基因的同时编辑。此外,CRISPR/Cas9系统对不同物种和不同基因进行编辑时其效率有较大的差异,因此CRISPR/Cas9系统的稳定性还有待于更全面地探索和研究,进而提高该系统的高效性和普遍适用性。CRISPR/Cas9系统的特异性是由 PAM前 20 bp的RNA与DNA互补配对决定的,在基因组中存在很多与这 20 bp序列不完全匹配的位点,因此理论上CRISPR/Cas9系统脱靶的概率会较高。Cong等[30]在人类细胞中检测CRISPR/Cas9系统的特异性,发现RNA-DNA互补配对时存在5个碱基错配,该系统仍会对此位点进行非特异性切割。目前,已有科研人员对CRISPR/Cas9系统脱靶效应进行了相应研究。Fu等[48]在细胞中使用17~18 bp缩短的sgRNA能够降低脱靶效率,并且直接转染合成的sgRNA比转染质粒有更高的特异性;Jienk等[8]对sgRNA研究表明,延伸sgRNA的3′端能增加CRISPR/Cas9系统在目的基因位点的突变效率,降低脱靶效率。
3.2 CRISPR/Cas9新的生物学功能开发
目前CRISPR/Cas9的应用主要集中在基因组修饰,而CRISPR/Cas9还能开发出更多的生物学功能。由于RNAs粘附在sgRNA末端不会影响sgRNA绑定 Cas9,因此将功能蛋白与无核酸酶活性的Cas9(dCas9)融合,从而实现功能蛋白与 DNA序列的结合。因此,Cas9携带一些融合蛋白或融合RNA到双链DNA序列,可以调控基因的表达和染色体结构。例如:利用调控复合体与染色体的精细交互作用调控基因的转录,可以利用sgRNA将dCas9作为假的转录因子引入到基因的关键位点,可能会影响转录因子与染色体的结合;此外,利用 dCas9融合蛋白和sgRNA将未知功能的单个因子绑定到基因组的任何位置,将会为基因功能和细胞信号通路的研究提供新的策略[49]。
首先,CRISPR/Cas9能够实现根据科研人员的期望直接上调内源基因的转录水平。在之前研究中科研人员利用TALE效应将转录激活效应因子VP64结合到基因的启动子区域能够促进转录[50]。科研人员利用CRISPR/Cas9在基因组中的定位功能,构建了dCas9-VP64融合蛋白,以及sgRNA绑定的MS2-VP64蛋白,这两种重组蛋白需要多个Cas9-sgRNA活性因子的协同作用才能够明显的提高目的基因的转录水平。Polstein 等[51]将光诱导异二聚体蛋白CRY2融合到VP64的转录激活区域,CIBN融合到dCas9的C端和N端,并将这两种重构蛋白和靶向人类IL1RN启动子的4个sgRNA同时转染到HEK293T细胞,发现在蓝光的刺激下细胞IL1RN的表达量提高了10~100倍。Konermann等[52]将转录因子与dCas9融合或sgRNA绑定,将相应转录激活因子进行组合筛选出sgRNA-dCas9-VP64与MS2-p65-HSF1协同作用的组合方式,此种组合将 TINCR、HOTTIP、PCAT、LINC00925、LINC00514和LINC00028基因的表达量提升10~100倍。利用此种方法,建立了70 290个sgRNAs的文库,将CRISPR元件引入大量体外培养的黑色素瘤细胞,不同细胞对应靶标不同基因的引导RNA,然后用PLX-4720处理这些细胞,最终确定了 13个特殊的基因可以使癌细胞对PLX-4720产生抗药性。一般而言,只有当多个基因同时进行复杂调控时,才能够导致疾病或严重生物学效应的发生。因此,为了详尽地了解疾病或生物学效应的基因调控网络,需要利用基因分库和基因剂量进行实验,以便确定是哪些基因共同决定了某些性状。在未来的研究中,能够对各种不同的效应结构域进行多重靶向和多重调控的CRISPR/Cas9系统将会成为了解基因调控网络的万能工具。
其次,利用ZFN效应蛋白和TALE效应蛋白招募转录抑制结构域能够有效地抑制内源基因的转录。目前,对 CRSPRi的研究也有了新的成果。Ji 等[53]构建了dCas9与相应sgRNA共表达的质粒,使大肠杆菌中mRFP的表达量下降了330倍;Gilbert 等[54]将 dCas9与转录抑制因子 KRAB(Krüppel associated box)结合组成dCas9-KRAB融合蛋白,并用慢病毒转染使其在HeLa细胞中持续表达,在转铁蛋白受体CD71和趋化因子受体CXCR4的转录起始位点设计了相应的 sgRNAs,将其转染到稳定表达dCas9-KRAB融合蛋白的 HeLa细胞中,CD71和CXCR4表达量下降 60%~80%,并且干扰效率受到sgRNA与靶位点结合效率的影响;Choudhary等[55]构建了一种优化的 dCas9,其在结核分支杆菌(Mycobacterium tuberculosis)中的表达量是普通dCas9的80倍,在sgRNAs的靶向引导下使分支杆菌中engA、ftsZ、clpP2、groEL1、groES、clpC1和yidC的表达量下降80%,证明CRISPRi具有多基因同时沉默的能力。此外,CRISPR/Cas9在转录抑制方面的作用还可以用于真核生物抗病毒研究,通过阻止外源入侵病毒基因的转录,将与某些病毒基因特定的sgRNA和Cas9阻遏物的编码序列整合在生物体中,能够获得免疫DNA病毒的生物体。此种构想可用于培育出免疫任何 DNA病毒的优势农作物或者家畜。
再次,Maeder等[56]将TALE与Tet1的羟化酶催化域融合,使用此种TALE-Tet1融合蛋白使HEK293 和HeLa细胞中RHOXF2(一种只在雄性生殖细胞中表达的印迹基因)基因的去甲基化程度提高了15%,并且在HEK293和HeLa细胞中检测到了高水平的RHOXF2 基因mRNA。虽然文献中还未见CRISPR/Cas9的染色体修饰功能,但是其在表观遗传修饰方面仍然具有广阔的发展空间。原则上,Cas9能够精确地招募染色质-重构复合体(Chromatin-remodeling complexes),包括 Swi-Snf、组蛋白乙酰化酶(Histone acetylases)、组蛋白去乙酰化酶(Histone deacetylases)、甲基化酶(Methylases)和去甲基化酶(Demethylases)、激酶和磷酸化酶,以及 DNA甲基化酶(Methylases)和去甲基化酶(Demethylases)。如果这些构想能够成功实现,科研人员将有能力研究生物体的表观遗传调控,并且能延长任何基因的表达期限[49]。
此外,CRISPR/Cas9还可以用于调控基因组的结构,通过构建Cas9重组酶,引导更加精确的基因修饰,将用于人类基因治疗或者靶向药物的研制。
4 结 语
综上所述,CRISPR/Cas9基因编辑技术是一种强大的基因工程工具,将会在基因功能解析、人类基因治疗、靶向药物研制、人类疾病动物模型的创制以及家畜育种中大放异彩,有望加速猪、牛、羊等重要家畜性状改良与分子定向育种。更令人振奋的是,CRISPR/Cas9具有设计简单、耗时短、节约成本、实验操作性强等优势,必将成为普通生物学实验室的常用技术。因此,有理由相信该技术将对生物学领域的发展起到深远影响。虽然关于
CRISPR/Cas9系统存在的部分问题有待于进一步解决与发展,但是随着科研人员对CRISPR/Cas9系统研究的不断深入使其稳定性与广泛应用性的进一步完善,CRISPR/Cas9系统将更好的帮助科研人员研究基因的功能,探索基因组的奥秘。
[1] Bibikova M, Beumer K, Trautman JK, Carroll D. Enhancing gene targeting with designed zinc finger nucleases. Science, 2003, 300(5620): 764.
[2] Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, Lahaye T, Nickstadt A, Bonas U. Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 2009, 326(5959): 1509-1512.
[3] 周金伟, 王灵慧, 申义君, 余树民, 曹随忠. 类转录激活因子效应物核酸酶(TALENs)的构建及其在基因组定点修饰中的应用. 中国细胞生物学学报, 2013, 35(11): 1672-1680.
[4] Tesson L, Usal C, Ménoret S, Leung E, Niles BJ, Remy S, Santiago Y, Vincent AI, Meng XD, Zhang L, Gregory PD, Anegon I, Cost GJ. Knockout rats generated by embryo microinjection of TALENs. Nat Biotechnol, 2011, 29(8): 695-696.
[5] 沈延, 黄鹏, 张博. TALEN构建与斑马鱼基因组定点突变的实验方法与流程. 遗传, 2013, 35(4): 533-544.
[6] Carlson DF, Tan WF, Lillico SG, Stverakova D, Proudfoot C, Christian M, Voytas DF, Long CR, Whitelaw CBA, Fahrenkrug SC. Efficient TALEN-mediated gene knockout in livestock. Proc Natl Acad Sci USA, 2012, 109(43): 17382-17387.
[7] Whyte JJ, Zhao JG, Wells KD, Samuel MS, Whitworth KM, Walters EM, Laughlin MH, Prather RS. Gene targeting with zinc finger nucleases to produce cloned eGFP knockout pigs. Mol Reprod Dev, 2011, 78(1): 2.
[8] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821.
[9] Wang HY, Yang H, Shivalila CS, Dawlaty MM, Cheng AW, Zhang F, Jaenisch R. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 2013, 153(4): 910-918.
[10] Jao LE, Wente SR, Chen WB. Efficient multiplex biallelic zebrafish genome editing using a CRISPR nuclease system. Proc Natl Acad Sci USA, 2013, 110(34): 13904-13909.
[11] Chang NN, Sun CH, Gao L, Zhu D, Xu XF, Zhu XJ, Xiong JW, Xi JJ. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465-472.
[12] Auer TO, Duroure K, De Cian A, Concordet J-P, Del Bene F. Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair. Genome Res, 2014, 24(1): 142-153.
[13] Hai T, Teng F, Guo RF, Li W, Zhou Q. One-step generation of knockout pigs by zygote injection of CRISPR/Cas system. Cell Res, 2014, 24(3): 372-375.
[14] Zhou XQ, Xin JG, Fan NN, Zou QJ, Huang J, Ouyang Z, Zhao Y, Zhao BT, Liu ZM, Lai SS, Yi XL, Guo L, Esteban MA, Zeng YZ, Yang HQ, Lai LX. Generation of CRISPR/ Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer. Cell Mol Life Sci, 2015, 72(6): 1175-1184.
[15] Ni W, Qiao J, Hu SW, Zhao XX, Regouski M, Yang M, Polejaeva IA, Chen CF. Efficient gene knockout in goats using CRISPR/Cas9 system. PLoS One, 2014, 9(9): e106718. [16] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea. Science, 2010, 327(5962): 167-170.
[17] Terns MP, Terns RM. CRISPR-based adaptive immune systems. Curr Opin Microbiol, 2011, 14(3): 321-327.
[18] Ishino Y, Shinagawa H, Makino K, Amemura M, Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol, 1987, 169: 5429-5433.
[19] Coffey A, Ross RP. Bacteriophage-resistance systems in dairy starter strains: molecular analysis to application. Antonie van Leeuwenhoek, 2002, 82(1-4): 303-321.
[20] Bolotin A, Quinquis B, Sorokin A, Ehrlich SD. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology, 2005, 151(8): 2551-2561.
[21] Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV. A putative RNA-interference-based immune system in prokaryotes: Computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action. Biol Direct, 2006, 1: 7.
[22] Mashimo T. Gene targeting technologies in rats: zinc finger nucleases, transcription activator-like effector nucleases, and clustered regularly interspaced short palindromic repeats. Dev Growth Differ, 2014, 56(1): 46-52.
[23] Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science, 2014, 346(6213): 1258096.
[24] Hou ZG, Zhang Y, Propson NE, Howden SE, Chu LF, Sontheimer EJ, Thomson JA. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proc Natl Acad Sci USA, 2013, 110(39): 15644-15649.
[25] Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol, 2014, 32(4): 347-355.
[26] Ran FA, Hsu PD, Lin C-Y, Gootenberg JS, Konermann S, Trevino AE, Scott DA, Inoue A, Matoba S, Zhang Y, Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 2013, 154(6): 1380-1389.
[27] Wu XB, Scott DA, Kriz AJ, Chiu AC, Hsu PD, Dadon DB, Cheng AW, Trevino AE, Konermann S, Chen SD, Jaenisch R, Zhang F, Sharp PA. Genome-wide binding of the CRISPR endonuclease Cas9 in mammalian cells. Nat Biotechnol, 2014, 32(7): 670-676.
[28] Hwang WY, Fu YF, Reyon D, Maeder ML, Tsai SQ, Sander JD, Peterson RT, Yeh J-RJ, Joung JK. Efficient genome editing in zebrafish using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(3): 227-229.
[29] Mali P, Yang LH, Esvelt KM, Aach J, Guell M, DiCarlo JE, Norville JE, Church GM. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826.
[30] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823.
[31] Shen B, Zhang J, Wu HY, Wang JY, Ma K, Li Z, Zhang XG, Zhang PM, Huang XX. Generation of gene-modified mice via Cas9/RNA-mediated gene targeting. Cell Res, 2013, 23(5): 720-723.
[32] Li W, Teng F, Li TD, Zhou Q. Simultaneous generation and germline transmission of multiple gene mutations in rat using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(8): 684-686.
[33] Li DL, Qiu ZW, Shao YJ, Chen YT, Guan YT, Liu MZ, Li YM, Gao N, Wang LR, Lu XL, Zhao YX, Liu MY. Heritable gene targeting in the mouse and rat using a CRISPRCas system. Nat Biotechnol, 2013, 31(8): 681-683.
[34] Nakayama T, Fish MB, Fisher M, Oomen-Hajagos J, Thomsen GH, Grainger RM. Simple and efficient CRISPR/ Cas9-mediated targeted mutagenesis in Xenopus tropicalis. Genesis, 2013, 51(12): 835-843.
[35] Gratz SJ, Cummings AM, Nguyen JN, Hamm DC, Donohue LK, Harrison MM, Wildonger J, O'Connor-Giles KM. Genome engineering of Drosophila with the CRISPR RNA-guided Cas9 nuclease. Genetics, 2013, 194(4): 1029-1035.
[36] Bassett AR, Tibbit C, Ponting CP, Liu JL. Highly efficient targeted mutagenesis of Drosophila with the CRISPR/ Cas9 system. Cell Rep, 2013, 4(1): 220-228.
[37] Wang YQ, Li ZQ, Xu J, Zeng BS, Ling L, You L, Chen YZ, Huang YP, Tan AJ. The CRISPR/Cas system mediates efficient genome engineering in Bombyx mori. Cell Res, 2013, 23(12): 1414-1416.
[38] Friedland AE, Tzur YB, Esvelt KM, Colaiácovo MP, Church GM, Calarco JA. Heritable genome editing in C. elegans via a CRISPR/Cas9 system. Nat Methods, 2013, 10(8): 741-743.
[39] Waaijers S, Portegijs V, Kerver J, Lemmens BBLG, Tijsterman M, van den Heuvel S, Boxem M. CRISPR/ Cas9-targeted mutagenesis in Caenorhabditis elegans. Genetics, 2013, 195(3): 1187-1191.
[40] Yang DS, Xu J, Zhu TQ, Fan JL, Lai LX, Zhang JF, Chen YE. Effective gene targeting in rabbits using RNA-guided Cas9 nucleases. J Mol Cell Biol, 2014, 6(1): 97-99.
[41] Niu YY, Shen B, Cui YQ, Chen YC, Wang JY, Wang L,Kang Y, Zhao XY, Si W, Li W, Xiang AP, Zhou JK, Guo XJ, Bi Y, Si CY, Hu B, Dong GY, Wang H, Zhou ZM, Li TQ, Tan T, Pu XQ, Wang F, Ji SH, Zhou Q, Huang XX, Ji WZ, Sha JH. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 2014, 156(4): 836-843.
[42] Yin LL, Maddison LA, Li MY, Kara N, LaFave MC, Varshney GK, Burgess SM, Patton JG, Chen WB. Multiplex conditional mutagenesis using transgenic expression of Cas9 and sgRNAs. Genetics, 2015, doi: 10.1534/genetics. 115.176917 [Epub ahead of print].
[43] Wu YX, Liang D, Wang YH, Bai MZ, Tang W, Bao SM, Yan ZQ, Li DS, Li JS. Correction of a genetic disease in mouse via use of CRISPR-Cas9. Cell Stem Cell, 2013, 13(6): 659-662.
[44] Li JH, Shou J, Guo Y, Tang YX, Wu YH, Jia ZL, Zhai YN, Chen ZF, Xu Q, Wu Q. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol, 2015, doi: 10.1093/jmcb/mjv016 [Epub ahead of print].
[45] Sato M, Miyoshi K, Nagao Y, Nishi Y, Ohtsuka M, Nakamura S, Sakurai T, Watanabe S. The combinational use of CRISPR/Cas9-based gene editing and targeted toxin technology enables efficient biallelic knockout of the α-1, 3-galactosyltransferase gene in porcine embryonic fibroblasts. Xenotransplantation, 2014, 21(3): 291-300.
[46] Chen YC, Zheng YH, Kang Y, Yang WL, Niu YY, Guo XY, Tu ZC, Si CY, Wang H, Xing RX, Pu XQ, Yang S-H, Li SH, Ji WZ, Li XJ. Functional disruption of the dystrophin gene in rhesus monkey using CRISPR/Cas9. Hum Mol Genet, 2015, 24(13): 3764-3774.
[47] Chen SD, Sanjana NE, Zheng KJ, Shalem O, Lee K, Shi X, Scott DA, Song J, Pan JQ, Weissleder R, Lee H, Zhang F, Sharp PA. Genome-wide CRISPR screen in a mouse model of tumor growth and metastasis. Cell, 2015, 160(6): 1246-1260.
[48] Fu YF, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using trun-cated guide RNAs. Nat Biotechnol, 2014, 32(3): 279-284.
[49] Mali P, Esvelt KM, Church GM. Cas9 as a versatile tool for engineering biology. Nat Methods, 2013, 10(10): 957-963.
[50] Zhang F, Cong L, Lodato S, Kosuri S, Church GM, Arlotta P. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nat Biotechnol, 2011, 29(2): 149-153.
[51] Polstein LR, Gersbach CA. A light-inducible crispr-cas9 system for control of endogenous gene activation. Nat Chem Biol, 2015, 11(3): 198-200.
[52] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 2015, 517(7536): 583-588.
[53] Ji WY, Lee D, Wong E, Dadlani P, Dinh D, Huang V, Kearns K, Teng S, Chen SS, Haliburton J, Heimberg G, Heineike B, Ramasubramanian A, Stevens T, Helmke KJ, Zepeda V, Qi LS, Lim WA. Specific gene repression by CRISPRi system transferred through bacterial conjugation. ACS Synth Biol, 2014, 3(12):929-931.
[54] Gilbert LA, Larson MH, Morsut L, Liu ZR, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451.
[55] Choudhary E, Thakur P, Pareek M, Agarwal N. Gene silencing by CRISPR interference in mycobacteria. Nat Commun, 2015, 6: 6267.
[56] Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Bernstein BE, Costello JF, Wilkinson MF, Joung JK. Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins. Nat Biotechnol, 2013, 31(12): 1137-1142.
(责任编委: 高彩霞)
CRISPR/Cas9 genome editing technique and its application in site-directed genome modification of animals
Jinwei Zhou1, Qipin Xu1, Jing Yao2, Shumin Yu1, Suizhong Cao1
1. College of Veterinary Medicine, Sichuan Agricultural University, Chengdu 611130, China; 2. State Key Laboratory of Reproductive Biology, Institute of Zoology, Chinese Academy of Sciences, Beijing 100101, China
CRISPR/Cas system, which uses CRISPR RNAs (crRNAs) to guide Cas nuclease to silence invading nucleic acids, is self-defense system against exogenous virus or plasmid in bacteria and archaea. Through molecular modification, the typeⅡCRISPR/Cas system has become a highly efficient site-directed genome editing technique, which is simpler than zinc-finger nucleases (ZFNs) and transcription activator like effector nucleases (TALENs) and easier to be designed and applied. In this review, we summarize the evolutionary history of CRISPR/Cas9 system, the working principle and modification process of type Ⅱ CRISPR/Cas and its application in animal genome modification. We also analyze the existing problems and improvement program of the CRISPR/Cas9 system as well as its application prospect combined with successful cases, which may provide innovative perspectives on improving animal traits and establishing animal models of human diseases.
2015-02-03;
2015-04-22
国家自然科学基金项目(编号:31272400,31172379)和四川农业大学学科建设双支计划项目(编号:3570806)资助
周金伟,硕士,专业方向:大动物遗传与修饰。E-mail: zhoujinwei08@126.com
曹随忠,教授,研究方向:兽医产科学。E-mail: suizhongcao@126.com
10.16288/j.yczz.15-066
时间:2015-6-17 15:14:43
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150617.1514.001.html
