植物基因组编辑及衍生技术最新研究进展
2015-01-03单奇伟高彩霞
单奇伟,高彩霞
中国科学院遗传与发育生物学研究所,植物细胞与染色体工程国家重点实验室,北京 100101
特别推荐
植物基因组编辑及衍生技术最新研究进展
单奇伟,高彩霞
中国科学院遗传与发育生物学研究所,植物细胞与染色体工程国家重点实验室,北京 100101

高彩霞课题组主要从事重要农作物基因组定向编辑技术方法的研究与应用、农作物遗传转化技术体系的建立与应用,以及小麦、玉米、水稻等重要基因的功能解析及基因组编辑定向设计分子育种研究。他们率先利用CRISPR/Cas系统实现水稻和小麦的基因组编辑,并利用该技术首创抗白粉病小麦新材料和改良水稻的香味品质,近期他们将该CRISPR切割系统引入植物,在植物中建立了DNA病毒防御体系。相关成果以通讯作者身份发表在Nature Biotechnology、Nature Plants、Nature Protocols、PLoS Biology、Molecular Plant、Plant Biotechnology Journal和Journal of Genomics and Genetics等期刊上。
基因组编辑技术已经在多个模式植物、动物以及其他生物中得到成功应用。基因组编辑是利用序列特异核酸酶(Sequence-specific nucleases, SSNs)在基因组特定位点产生DNA双链断裂(Double-strand breaks, DSBs),从而激活细胞自身修复机制——非同源末端连接(Non-homologous end joining, NHEJ)或同源重组(Homologous recombination, HR),实现基因敲除、染色体重组以及基因定点插入或替换等。锌指核酸酶(Zinc finger nuclease, ZFN)、TALEN(Transcription activator-like effector nuclease)和CRISPR/Cas9(Clustered regularly interspaced short palindromic repeats/CRISPR-associated 9)系统是最主要的3类SSNs。ZFN和TALEN是利用蛋白与DNA结合方式靶向特定的基因组位点,而最新的CIRISPR/Cas9系统则是利用更简单的核苷酸互补配对方式结合在基因组靶位点,其构建简单、效率更高效,因而促进了基因组编辑在植物中的广泛应用。利用基因组编辑技术除了实现植物基因定点突变外,还可以将SSNs的DNA结合域与其他功能蛋白融合,实现基因的靶向激活、抑制和表观调控等衍生技术。本文从基因组编辑技术的原理与优势、SSNs组成及构建方法、基因组编辑及衍生技术在植物中应用、优化SSNs突变效率和减少脱靶突变方法等方面进行了系统介绍,并对未来需要迫切解决的一些问题进行了分析和展望。
基因组编辑;ZFN;TALEN;CRISPR/Cas9;同源重组;脱靶突变
随着生命科学研究进入基因组时代,越来越多物种的基因组完成测序,解读与改造基因组的功能就显得非常紧迫。以基因组编辑(Genome editing)为基础的反向遗传学技术是基因组改造与基因功能研究必不可少的手段之一[1]。基因组编辑技术是一项可以与分子克隆、PCR等技术相媲美的技术突破,虽然出现仅仅10余年,但已显著地促进了生物学研究的迅猛发展,应用前景广阔。以序列特异核酸酶(Sequence-specific nucleases, SSNs)为工具的基因组编辑技术已在全世界掀起了研究热潮。2012年Science将其列入年度10大科学进展,以TALEN为代表的SSNs被誉为“基因组巡航导弹”。2013年Science再次将SSNs技术的新星CRISPR/Cas9列入年度十大科学进展。2014年Nature Methods将基因组编辑技术评为过去 10年间对生物学研究最有影响力的10项研究方法之一。
1 基因组编辑技术的原理及优势
1.1 基因组编辑技术的原理及DNA断裂修复机制
近几年,生物学家们巧妙地利用蛋白质结构与功能领域的研究成果,将特异识别与结合DNA的蛋白质结构域和核酸内切酶结构域融合,创造出能够按照人们意愿特异切割DNA的SSNs,并藉此实现了对基因组特定位点的靶向修饰,即基因组编辑。SSNs主要包括3种类型:锌指核酸酶(Zinc finger nuclease, ZFN)、类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease, TALEN)和成簇的规律间隔的短回文重复序列及其相关系统(Clustered regularly interspaced short palindromic repeats/CRISPR-associated 9, CRISPR/Cas9 system)(图1,A~C)。它们的共同特征是能够作为核酸内切酶切割特定的DNA序列,创造DNA双链断裂(Double-strand breaks, DSBs)。在真核生物中,DSBs的修复机制是高度保守的,主要包括两种途径:非同源末端连接(Nonhomologous end joining, NHEJ)和同源重组(Homologous recombination, HR)。通过NHEJ方式,断裂的染色体会重新连接,但往往是不精确的,断裂位置会产生少量核苷酸的插入或删除,从而产生基因敲除突变体;通过 HR方式,在引入同源序列的情况下,以同源序列为模板进行合成修复,从而产生精确的定点替换或者插入突变体[2](图2)。在这两种途径中,NHEJ方式占绝对主导,可以发生在几乎所有类型的细胞以及不同的细胞周期中(G1、S和G2期);然而,HR发生频率很低,主要发生在S和G2期[3]。

图1 3种序列特异核酸酶(SSNs)模式图
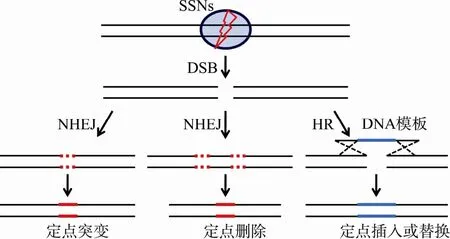
图2 DNA双链断裂(DSBs)修复途径
1.1.1 NHEJ途径
根据具体修复方式和参与修复的蛋白因子,可以将NHEJ分为两类:一类是Classical NHEJ (cNHEJ);另一类是Alternative NHEJ (aNHEJ)[4]。这两种机制在所有真核生物中是高度保守的。通过cNHEJ途径,SSN诱导的DSBs末端首先被具有环状结构的Ku蛋白异源二聚体(Ku70、Ku80)结合以防止 DNA断裂末端进一步降解,最后在特定的DNA连接酶Ⅳ的作用下,将两个开放的末端重新连接(图3A)。在连接前可能经过末端加工过程,因而产生几个核苷酸的插入或删除 (Indel),但多数情况下不经过加工过程而直接恢复为原始序列。从基因组编辑的角度看,经过末端加工产生少量核苷酸插入或删除是更有意义的。如果DSBs发生在编码基因的ORF区,插入或删除非 3整数倍的核苷酸,可能造成移码突变使基因功能完全丧失。
如果cNHEJ途径被抑制或DSBs两侧含有几个或十几个核苷酸的微同源序列,则可能通过aNHEJ途径修复。研究表明,DSBs末端分别发生5′至3′方向的DNA切除,释放出可局部互补配对的单链末端,微同源序列互补配对,再经过末端处理和重新连接修复 DSBs缺口。修复后微同源序列恰好位于DSBs的结合点。由于发生了核苷酸的删除,造成遗传信息丢失,因此在基因组编辑过程中aNHEJ修复方式很容易产生突变(图 3B)。研究表明,这两种NHEJ途径在细胞中会相互竞争。与野生型相比,拟南芥ku80突变体Indel突变效率增加2.6倍,并且DSBs位置核苷酸降解长度也有所增加[5]。

图 3 两种非同源末端连接(NHEJ)修复途径方式(参考文献[3]修改绘制)
1.1.2 HR途径
根据发生方式的不同,HR可以分为两类:单链退火(Single-strand annealing, SSA)和合成依赖式链退火(Synthesis-dependent strand annealing, SDSA)。DSBs产生后,在这两种途径下DNA断裂末端都会发生5’至3’方向的DNA切除,形成3’单链末端。SSA途径类似aNHEJ途径,DSBs两端各有一段同源序列,同源序列区域直接退火形成互补双链,再经过末端加工和连接修复DSBs(图4A)。在基因组串联重复区域,SSA是主要的DSBs修复方式[6]。

图4 两种同源重组(HR)修复途径方式(参考文献[3]修改绘制)
SDSA途径是依赖DNA合成的修复过程,基因组编辑过程中同源重组通常是指这种方式。DSB经过5’至3’方向的DNA切除产生的一个3’单链末端入侵同源供体DNA模板,形成D-loop环状结构,再利用同源DNA的互补链作为模板进行DNA合成修复,当延伸至可以与DSB另一个单链末端互补配对位置时,脱离 D-loop结构,两个单链DSB末端退火形成双链,完成修复过程(图 4B)。SDSA途径最终结果就是完成从同源DNA至DSB遗传信息的转换过程。SDSA途径发生频率非常低,相同条件下只有SSA方式的10%~20%[7]。
1.2 基因组编辑技术优势
当前遗传改良或基因修饰途径存在许多缺陷。例如:传统杂交育种法周期长,需要多个世代,耗时费力,且受物种间生殖隔离限制和不良基因连锁的影响;物理或化学诱变法虽然可以在基因组上随机产生大量突变位点,但突变位点鉴定十分困难;传统基因打靶方法效率极低(通常仅10-6~10-5),并且只限于少数物种如酵母(Saccharomyces cerevisiae)、小鼠(Mus musculus)等;RNAi方法下调基因表达不够彻底,其后代的基因沉默效果减弱甚至完全消失,不能稳定遗传。相比于上述方法,基因组编辑技术优势非常明显:(1)所有SSNs都可以通过设计DNA结合蛋白(或导向 RNA)使其靶向编辑基因组的任意位点,精确性非常高;(2)原理简单易懂,而且技术操作简便、成本相对低廉,原则上适用于任意物种;(3)利用SSNs定点突变目的基因具有非常高的效率,通常从几个或十几个转化株系中就能筛选到符合要求的基因突变材料,对于活性高的SSNs在T0代就可以得到纯合突变体。目前,基因组编辑技术已成功应用于酵母、线虫(Caenorhabditis elegans)、果蝇(Drosophila melanogaster)、斑马鱼(Danio rerio)、小鼠、人类(Homo sapiens)细胞、拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)、大豆(Glycine max)、水稻(Oryza sativa)、小麦(Triticum aestivum)、大麦(Hordeum vulgare)和玉米(Zea mays)等多种模式生物及经济作物中[8]。
2 SSNs组成及构建方法
2.1 ZFNs组成及构建方法
ZFNs是通过基因工程方法将锌指蛋白DNA结合域(ZFA)和核酸内切酶FokⅠ的切割结构域融合而成,DNA结合域通常由3~6个Cys2His2类型的锌指单元串联而成[9]。每个锌指单元含有 1个 α螺旋和2个β折叠结构,并且螯合1个锌原子。1个锌指单元能特异识别DNA单链上3个连续的核苷酸;由多个锌指单元串联形成的ZFA结构域则可识别更长的靶序列,同时增加了DNA靶向修饰的特异性。当两个ZFN单体按照一定的距离和方向同各自的目标位点特异结合,两个 FokⅠ切割结构域恰好可形成二聚体的活性形式,在两个结合位点的间隔区(Spacer,通常为5~7 bp)切割DNA (图1A)。
理论上每个锌指单元识别 3个核苷酸,1个包含 64个锌指的文库就可以识别所有的串联三联体核苷酸。在应用中发现单个锌指的识别特性在不同的串联锌指单元ZFA中差异非常大,是由于相邻锌指造成的上下文背景起了重要影响。当前主要有 4种设计方案,包括:模块组装方案(Modular assembly MA)[10]、基于文库筛选的OPEN方案[11]、Sangamo公司私有的双锌指模块组装方案(Two-finger modules)[12]和上下文依赖组装方案(CoDA)[13]。MA方案虽然简便,但完全不考虑上下文背景作用,其成功率最低。另外3种方案都考虑了上下文的背景影响。当前有几种程序可以辅助设计ZFN靶位点,具体见表1。
2.2 TALEN组成及构建方法
类转录激活因子效应物(Transcription activator-like effector, TALE)是黄单胞杆菌属(Xanthomonas)植物病原菌通过Ⅲ型分泌系统分泌到宿主细胞中的一种毒性蛋白,可以识别植物特定基因启动子序列,启动感病基因表达。TALE核酸酶(TALENs)就是利用TALE的DNA结合域和FokⅠ的切割结构域合成的人工核酸酶[14](图1B)。TALE的DNA结合域包含1个由数量不等的重复单元串联组成的重复序列结构,这些重复单元通常由33~35个高度保守的氨基酸组成,第12和13位氨基酸可变,被称为重复可变双残基(Repeat variable diresidue, RVD)。每个RVD与核苷酸A、T、C、G存在简单对应的关系,即NI识别A、HD识别C、NG识别T和NN识别G[15,16]。TALE蛋白以这种“一个重复单位一个核苷酸”的对应方式特异识别并结合DNA。2012年,TALE蛋白晶体结构被解析出来,结构显示 TALE的重复单元组成Helix-loop-helix的结构围绕DNA双螺旋主沟呈右手螺旋状排列,结构还显示RVD的两个残基中第一位氨基酸稳定RVD loop作用,第二位氨基酸与碱基特异识别相关[17,18],这些信息为改造 TALE蛋白提供了结构基础。

表1 SSNs资源及在线程序
由于TALE蛋白DNA结合域的高度串联重复特性,使TALE表达载体的合成和搭建具有一定困难。科研人员开发了多种方法,包括简单直接的模块组装法[19]、Golden Gate组装方法[20]、高通量合成TALE的固相组装[21]和不依赖连接的克隆方法[22]等。Golden Gate组装方法利用IIS型限制性内切酶切割位点位于识别序列外部的特性,各个TALE重复单元质粒经酶切后产生不同的 4nt粘性末端,具有兼容粘性末端的 DNA片段可按照正确顺序连接成完整的多RVD模块。表1汇总了当前各种TALEN辅助设计程序。
2.3 CRISPR/Cas系统组成及构建方法
CRISPR/Cas系统是在细菌的天然免疫系统内发现的,广泛存在于细菌及古生菌中,主要功能是抵抗入侵的病毒及外源 DNA。CRISPR/Cas系统由CRISPR序列与Cas基因家族组成,其中CRISPR由一系列高度保守的重复序列(Repeat)与间隔序列(Spacer)相间排列组成。在CRISPR序列附近存在高度保守的 CRISPR相关基因(CRISPR-associated gene, Cas gene),这些基因编码的蛋白具有核酸酶功能,可以对DNA序列进行特异性切割[23,24]。
根据Cas基因核心元件序列的不同,CRISPR/Cas可以分为Ⅰ型、Ⅱ型和Ⅲ型系统[23]。这3类系统又可以根据其编码Cas蛋白而分为更多的亚类。Ⅰ型和Ⅲ型CRISPR/Cas免疫系统需要多个Cas蛋白形成的复合体切割DNA双链,而Ⅱ型只需要1个Cas9蛋白。Cas9蛋白包含氨基端的RuvC-like结构域及位于蛋白中间位置的HNH核酸酶结构域,HNH核酸酶结构域切割与单导向RNA (Single guide RNA, sgRNA)互补配对的模板链,RuvC-like结构域对另1条链进行切割。切割位点位于原型间隔序列毗邻基序(Protospacer adjacent motif, PAM)上游3nt处。自2012年起,人们优化了Ⅱ型CRISPR/Cas系统,利用Cas9蛋白和sgRNA构成简单的sgRNA/ Cas9系统[25, 26],使其能够在真核生物中发挥类似 ZFN 和TALEN那样靶向切割DNA的作用(图1C)。Cas9蛋白与sgRNA结合形成RNA-蛋白质复合体,共同完成识别并切割DNA靶序列的功能。其中,Cas9蛋白作为核酸酶切割双链DNA,而sgRNA则通过碱基互补配对决定靶序列的特异性。
2014年,链球菌(Streptococcus pyogenes)和放线菌(Actinomyces naeslundii) Cas9蛋白的三维晶体结构被解析出来[27,28]。结果发现,Cas9家族的成员具有相同的核心结构,这一结构可以裂开两瓣形成钳状,一瓣负责目标识别,另一瓣具有核酸酶活性能切断DNA。两瓣中间有1个带正电的沟槽,可以容纳sgRNA:DNA异源双链分子。目标识别瓣对于结合sgRNA和DNA是必须的,而核酸酶瓣包含HNH 和 RuvC核酸酶结构域,它们所处位置恰好可以分别切开一条DNA链。Cas9蛋白单独存在时处于非活性状态,但与sgRNA结合后,它的三维结构会经历剧烈的改变,允许Cas9与目标DNA结合。这一结构可以帮助改良Cas9核酸酶,设计不影响其功能的小Cas9变体,使其更适合基础研究和基因工程。目前已积累了大量的CRISPR靶位点设计网络程序,详见表1。
2.4 ZFN、TALEN和CRISPR/Cas9系统的比较及选择
3种SSNs技术相比较,在效率、特异性及设计上各有不同。由于锌指单元同其靶序列的对应性并不特异,ZFN表现出较明显的脱靶效应(Off-target effect),并且获得有效的 ZFN相当困难,这两点严重妨碍了该技术的广泛应用。TALEN和 CRISPR/ Cas9系统相互补充,各具优点:TALEN优势是特异性高,脱靶效应较低,但TALEN蛋白较大,并且序列重复性强,表达载体构建较为繁琐;CRISPR/Cas9系统在设计和构建上更为简单,突变不同的靶位点时仅需重新设计、合成与靶序列互补的sgRNA,而不需要更换Cas9核酸酶,为CRISPR/Cas9系统应用提供了极大便利。由于sgRNA与靶位点通过核苷酸配对相互识别,个别核苷酸位点改变并不会对该系统的突变活性造成显著影响,因此,CRISPR/Cas9系统的特异性较TALEN稍差。目前ZFN基本上被TALEN和CRISPR/Cas9替代。TALEN的优势是特异性高,脱靶效应较低;而CRISPR/Cas9系统的优势则是使用简便、成本低(表2)。
在动物或人类的基因治疗等应用中,SSNs脱靶效应可能造成致命后果。然而在植物中脱靶效应不是主要制约因素,可以通过全基因组测序检测是否存在脱靶,或者通过与亲本多次回交的方法去除脱靶突变。另据报道,CRISPR/Cas9系统在植物中没有严重的脱靶突变[29,30],而且目前已有多种方法可以在一定程度上减少 CRISPR脱靶,提高靶向特异性。因此,从简便、效率和多基因编辑角度考虑,3 种SSNs中首选CRISPR/Cas9,TALEN次之。在基因组编辑以外的其他领域,TALE效应因子相对更容易与其他功能结构域融合,行使靶向激活、抑制和表观修饰等功能;将无核酸酶活性的 dCas9(dead Cas9)与功能蛋白融合,也能赋予CRISPR系统多种功能,但sgRNA由RNA聚合酶Ⅲ转录,目前没有可精确调控的sgRNA启动子,因此,从可扩展性及精细调控方面TALE略胜一筹[31]。
3 基因组编辑技术在植物生物学及分子育种研究中的应用
3.1 基因敲除
基因敲除是基因组编辑最简单的应用形式,只需要SSNs在靶位点制造1个DSB断裂。利用SSNs基因敲除的植物种类括拟南芥、烟草、矮牵牛(Petunia hybrida)、水稻、大豆、玉米和小麦等(表3)。

表2 ZFNs、TALENs和CRISPR/Cas9系统比较

表3 植物基因组编辑汇总

续表
Zhang等[32]用 OPEN(Oligomerized pool engineering)方法设计ZFN敲除拟南芥ADH1和TT4基因,并利用雌激素诱导启动子表达,T1代分别有7% 和 16%的植物含有体细胞突变,突变能够稳定传递到后代,并获得20%纯合突变植物。纯合adh1突变体具有预期丙烯醇抗性,而tt4突变体种皮因缺失花青素而呈现黄色。
Li等[33]利用 TALEN方法突变水稻蔗糖外排转运基因OsSWEET14启动子区域,破坏细菌性病原菌分泌的效应蛋白在基因组上的结合位点,从而提高水稻白叶枯病抗性。Shan等[34]对水稻甜菜碱乙醛脱氢酶基因(OsBADH2)的编码区设计 TALEN,badh2纯和突变体稻米2AP含量显著增加,提高了稻米的香味品质。Haun等[35]利用TALEN方法同时敲除大豆脂肪脱氢酶 2基因家族两个成员 FAD2-1A和FAD2-1B,纯合突变体(aabb)油酸含量从20%提高到80%,并同时降低对人体健康有害的亚油酸含量(从 50%到 4%),因此改善了大豆油的品质。Wang 等[36,37]利用TALEN方法同时敲除MLO基因在小麦A、B和D基因组上的3个拷贝,获得对白粉病具有广谱和持久抗性的纯合小麦突变体tamlo-aabbdd。
CRISPR/Cas9系统具有简捷和高效特性,目前已在多个植物中得到应用,如拟南芥、烟草、水稻、玉米、高粱、小麦、甜橙(Citrus sinensis)和番茄(Solanum lycopersicum)等(表3)。在拟南芥和烟草原生质体中,NHEJ突变效率分别达到5.6%和38.5%[38]。此外,利用农杆菌侵染方法稳定转化拟南芥,T1植物中有 26%~84%突变效率[39]。Shan等[40]利用水稻偏爱密码子优化Cas9核酸酶基因,并采用水稻小核RNA的U3启动子和小麦U6启动子转录sgRNA,定点敲除水稻PDS和小麦MLO等基因,在T0代就获得纯合基因敲除水稻突变体,突变效率达到10%。Xie等[41]在水稻原生质体中检测到3.5%~10.6%突变效率,植物中突变效率为 3%~8%。Miao等[42]分别敲除水稻叶绿素A加氧酶基因CAO1和控制分蘖夹角的LAZY1基因,T1代纯合cao1和lazy1突变体分别呈现叶片叶绿素含量降低和分蘖夹角增大表型。
3.2 基因插入和定点替换
一般情况下,基因插入或定点替换都可以通过HR方式实现,转化SSNs同时引入一个供体DNA载体或片段,供体DNA包含了待插入或替换的基因或核苷酸序列并在其两侧分别含有足够长的同源DNA(同源臂)。基因定点插入所用的供体DNA通常为双链环状载体或双链线性DNA;此外还可以使用单链寡核苷酸DNA(ssDNA),ssDNA的设计和合成比构建双链 DNA载体更简单方便[43]。由于 NHEJ效率相比HR效率高很多,因此,利用NHEJ途径定点插入基因或Tag标签成为一种替代方法[44]。植物中基因插入和定点替换的成功例子见表3。
Voytas实验室最早尝试用 HR方式精确编辑植物基因组,他们首先在烟草中整合了功能缺失的gus:nptII筛选基因,该基因上含有ZFN(Zif268)识别序列。再把ZFN和供体DNA转入含有gus:nptII基因的烟草原生质体中,从卡那霉素抗性克隆中筛选到有10%发生同源重组[45]。Townsend等[46]利用ZFN技术,通过 HR途径分别定点替换烟草乙酰乳酸合成酶基因(SuRA、SuRB)的3个关键核苷酸位点,得到抗除草剂烟草,基因打靶效率在0.2%~4%之间。Shukla等[47]在玉米中用ZFN定点突变肌醇六磷酸生物合成最后一步的关键酶基因IPK1,同时插入抗除草剂基因 PAT,玉米的肌醇六磷酸含量减少,营养品质得到改良,且减少了对环境的有机磷污染,基因打靶效率在大部分实验中高达10%以上。
Zhang等[48]在烟草原生质体中转化TALEN和供体DNA,有30%细胞发生ALS基因突变,另外高达14%烟草原生质体细胞ALS基因位点整合了YFP报告基因;另一组不加筛选剂实验中,烟草ALS基因关键核苷酸替换效率达到4%。Wang等[36]在小麦原生质体细胞MLO基因位点通过NHEJ途径整合不含启动子的 GFP报告基因,流式细胞仪检测到 6.5%细胞有GFP表达,测序结果证明GFP按照正确读码框整合在MLO位点。在另一项研究中,科研人员将 His-tag和 Myc-tag按照 NHEJ方式整合在小麦MLO位点,效率达到1.4%和2.6%,并且按照孟德尔分离分离比例传递到T1代。
利用CRISPR/Cas9系统,Shan等[38]和Li等[40]分别在水稻和烟草的原生质体PDS基因上通过HR途径插入1个或多个限制性内切酶位点,并利用酶切和测序方法验证了 HR效率。Fauser等[49]在拟南芥中利用DGU.US和IU.GUS两个GUS报告基因系统,证明Cas9核酸酶和Cas9切口酶都能有效诱导HR,且Cas9切口酶效率更高。
3.3 染色体重组及多基因敲除
多个 SSNs同时导入细胞可以实现染色体片段删除、倒位、易位等染色体重组突变和多基因同步敲除突变。染色体重组和多基因敲除是反向遗传学和现代生物技术的重要工具,基因大片段删除技术对于研究非编码RNA、基因调控序列和冗余基因功能具有重要作用;利用多基因敲除技术研究复杂性状或数量性状,能够在较短时间获得多基因同步敲除突变体,加快研究进程。Qi等[50]利用农杆菌转化法同时转化两对ZFNs,在拟南芥中同时删除了含有8个抗病基因长度为55 kb的RPP4基因簇,体细胞中突变效率在 1%左右;在另一组实验中,Qi等获得了长约为9 Mb染色体片段删除,同时检测到染色体倒位和片段重复的现象。借助于 TALEN技术,Christian等[51]和 Shan等[52]分别在拟南芥和水稻中实现4.4 kb和1.3 kb基因片段删除。CRISPR/Cas9系统由于构建简单,因此在染色体重组和多基因编辑领域更有优势,Zhou等[53]利用CRISPR系统在水稻中实现了115~245 kb大片段删除,其中包含了2~3个不同的基因簇,进一步表明SSN在染色体大片段删除中的有效性。
多基因同步敲除技术在植物中也有报道,Shan 等[34]通过基因枪法同时导入3对TALENs,分别靶向控制水稻产量和品质性状基因 DEP1、CKX2和BADH2,在T0代获得一系列单基因、两基因及三基因共同敲除株系,其中三基因突变效率达到1.9%(4/207)。Xing等[54]构建了一套适合进行植物多基因编辑的CRISPR/Cas9载体系统,在拟南芥的T1代植物中实现三基因同步敲除。最近,Xie等[55]利用内源的转运RNA(Transfer RNA,tRNA)加工系统提高了CRISPR/Cas9进行多基因编辑的能力,他们将多个tRNA-sgRNA结构串联排列,从构建的一个多顺反子 tRNA-sgRNA基因(PTG)可以转录并加工成多个sgRNA。利用这一策略在水稻中实现多达8个位点同时突变,并且个别位点效率高达100%。而且由于 tRNA及其加工系统在所有生物中都是非常保守的,这一策略有望在其他物种的多基因编辑中广泛应用。
3.4 SSNs与其他生物技术相结合的创新研究成果
SSNs技术与其他先进生物技术相结合,已经产生了很多意想不到的巨大突破。例如,CRISPR/Cas9系统的简便和低成本使全基因组水平的定向编辑(Genome-wide editing)变为现实,通过构建靶向几千个甚至上万个基因的大规模sgRNA文库,再结合功能性筛选平台和深度测序技术,以高通量方式研究疾病相关的基因,这一技术还有望研究更广泛的生物学问题[56~58]。华盛顿大学的研究团队将 CRISPR/ Cas9系统与饱和基因组编辑和深度测序技术相结合,检测大量单个核苷酸位点突变带来的功能性影响,这一研究可能推动对一些顺式调控元件和反式作用因子进行高分辨率的功能解析,并大大提高人们解读未知基因变异的能力[59]。华人科学家简悦威院士将CRISPR/Cas9与piggyBac转座子结合,修改引起β-地中海贫血病的 HBB基因,在诱导多能干细胞(iPSC)中实现无痕基因校正[60]。另外,SSNs及其他衍生的转录组、表观基因组靶向修饰工具在合成生物学领域也已发挥重要作用[61,62]。
4 SSNs的相关衍生技术及应用
除基因组编辑技术以外,DNA结合蛋白ZFA或TALE可以与多种功能蛋白融合而行使其他靶向修饰作用;同理,无DNA切割活性的dCas9 (dead Cas9)也可以与其他功能蛋白融合,借助sgRNA靶向特定位置进行基因修饰。目前,已报道与 ZFA、TALE 或dCas9融合的功能蛋白包括VP64、KRAB和TET1等,它们负责转录激活、转录抑制、表观调控和靶向RNA编辑等。这些SSNs相关衍生技术为生物学研究提供了更多的遗传学工具(图5)。

图5 SSN衍生应用技术
4.1 靶向基因激活
Sanchez等[63]将编码3个串联的锌指蛋白Zif268与孢疹病毒的转录激活结构域 VP16或 VP64(即4×VP16)基因融合,产生融合蛋白 ZF-VP16或ZF-VP64。拟南芥瞬时表达实验和稳定转化实验中,融合蛋白都可以激活报告基因LUC和GFP的转录。Morbitzer等[64]利用 AvrBS3的 TALE骨架并将其RVD替换,靶向番茄BS4、拟南芥EGL3和拟南芥KNAT1启动子上特定位点,所有dTALE都能特异性地激活 3个基因。Liu等[65]在烟草实验中利用农杆菌注射方法瞬时表达TALE-VP64或TALE-VP16融合蛋白,靶向35S启动子的TATA-Box旁侧序列,可以显著增强橙色荧光蛋白报告基因(pporRFP)的表达。实验结果还表明,靶向激活作用具有累加效应,即多个不同的TALE融合蛋白共同使用比单个TALE融合蛋白的激活作用更强。此外,利用TALE融合蛋白激活转基因烟草中的AtPAP1基因,烟草叶片注射后合成大量花青素因而呈现深紫色,进一步证明人工合成 TALE融合蛋白可以用于植物内源基因的靶向激活。Baazim[66]采用 CRISPR/dCas9系统在烟草中进行定点激活实验,通过在dCas9的C-末端融合激活结构域EDLL或TAD (TAL effector激活结构域),农杆菌注射烟草叶片后对烟草叶盘进行染色或半定量PCR实验,表明GUS基因或PDS基因可以被激活。上述靶向激活实验的效率通常较低,为进一步提高效率,Tanenbaum等[67]开发了一种名为“SunTag”的蛋白标签信号放大系统,可以高效率定向激活靶标基因。这一系统通过在 dCas9末端融合能与单链抗体 scFv特异性结合的串联多肽链GCN4,从而将多个VP64激活结构域募集至靶标基因启动子区域,对人类细胞中低表达丰度基因CXCR4,转录效果增强10~50倍;对高表达基因的激活效果也能达到3倍以上。另外,Konermann等[68]
重新设计CRISPR-Cas9激活复合体,采用sgRNA2.0、NLS-dCas9-VP64和MS2-p65-HSF1蛋白复合体这个优化组合能大幅度提高转录激活效率,在人类细胞中实现同时激活十个内源基因,并能够上调长基因间非编码RNA(lincRNA)的转录水平;此外,还构建靶向人类全基因组水平的70 290个sgRNA文库,进行大规模 Gain-of-function(GOF)遗传筛选抗BRAF蛋白抑制剂的细胞克隆。
4.2 靶向基因抑制
Mahfouz等[69]利用 dHAX3的 TALE骨架与SRDX转录抑制结构域构建嵌合蛋白 dHax3.SRDX转化拟南芥,该融合蛋白能够靶向RD29A启动子区域,报告基因 RD29A::LUC和拟南芥内源基因RD29A的转录都受到明显抑制。Baazim[66]在dCas9蛋白的 3′-末端融合 SRDX转录抑制结构域,构建dCas9-SRDX融合蛋白,在sgRNA引导下,半定量PCR实验表明可以靶向性抑制烟草PDS基因。在人类细胞及动物研究领域,Zhang等[70]构建了一套基于TALE-KRAB的转录抑制载体系统,并利用不同的荧光蛋白进行标记,可以同时抑制小鼠的多个内源基因,例如同时转入靶向两个基因 c.Kit和 PU.1 的3个TALE-KRAB载体,结合FACS荧光分选方法和qPCR鉴定,表明小鼠MEFs细胞两个基因的表达水平分别降低 50%和 80%。Gilbert等[71]利用CRISPR系统首先在人类细胞和酵母中实现靶基因表达的精确调控,通过将无切割活性的 dCas9与转录抑制结构域KRAB融合,可以显著沉默多个内源基因的表达。RNA测序表明CRISPRi介导的转录抑制具有很高的靶向特异性。最近,加州大学的科研人员设计了一种CRISPR的引导RNA,在上面加入了效应蛋白的招募位点,即支架RNA(scRNA)系统。scRNA同时定义了靶位点和调控功能,能同时对不同的基因进行激活或抑制。这种支架RNA可以用来设计多基因转录程序,在激活一些基因的同时抑制另一些基因。利用这种方法,成功在酵母中重新编程了一个复杂的代谢通路。研究还显示,scRNA在哺乳动物和酵母中均能有效发挥调控功能[72]。
4.3 靶向表观修饰
对表观基因组的定向修饰是另外一个重要研究方向。基因组的表观修饰包括DNA和组蛋白的甲基化、去甲基化、乙酰化和去乙酰基化等,在基因的表达调控方面发挥重要作用。目前在植物领域还没有靶向表观修饰研究的报道,只有少数几篇文章是在人类细胞、哺乳动物和细菌中的研究。Meister等[73]将天然存在的异源二聚体甲基转移酶 M.EcoHK31I的两个亚基分别与识别9 bp长的锌指蛋白融合,构建锌指甲基转移酶,可以特异性地对大肠杆菌靶位点进行DNA甲基化修饰,而不会造成非靶位点的甲基化。最近有两个实验室报道利用 TALE融合蛋白修改哺乳动物内源基因的表观状态。Mendenhall等[74]利用TALE与组蛋白去甲基化酶LSD1融合构建的TALE-LSD1蛋白研究增强子上的组蛋白印记对转录调控的作用,表明融合蛋白可以有效地去除靶向增强子位点的染色质修饰,而不会影响对照区域;他们还发现通过融合蛋白失活增强子往往导致下调临近基因的表达,从而确定了增强子的靶向基因。Konermann等[75]将TALE与光诱导系统结合构建了LITE系统,在串联mSin3结构域(SID4X)的作用下,靶标基因Grm2的转录水平下降,同时Grm2基因启动子区域 H3K9乙酰化水平降低两倍,表明转录作用被抑制。此外,Maeder等[76]开发了一种可在人类细胞中有效定点去甲基化的方法,通过采用TALE-TET1羟化酶催化结构域融合蛋白,可以改变启动子上关键CpG区域的甲基化状态,进而显著增加人类内源基因的表达水平。
4.4 靶向RNA编辑
RNAi是真核生物中一个最重要的RNA水平的转录调控机制。此前,CRISPR/Cas9系统被认为只能切割双链DNA,不能靶向RNA,然而最近研究发现CRISPR/Cas9系统也可以用来对RNA进行靶向编辑。O’Connell等[77]研究表明,当提供合适的含PAM序列的寡核苷酸(PAMer)DNA时,Cas9核酸酶可以特异性地结合并切断与sgRNA互补配对的单链RNA (ssRNA),PAMer DNA的作用类似于PAM序列,它能够激活Cas9核酸酶活性从而切断DNA或RNA,研究还表明这一技术可以用来从细胞中分离特定的內源mRNA转录本。最近研究发现,有些古菌的Ⅲ型CRISPR系统能够切割RNA。例如,Zebec 等[78]发现嗜热古菌 Sulfolobus solfataricus能利用CRISPR系统特异性降解入侵病毒的mRNA,qPCR实验表明超过40%的mRNA可以被降解,利用纯化的 CMR复合体在体外实验中证实了切割相同的mRNA底物。另外,通过重新设计crRNA证明这一系统可以靶向其他基因,靶标基因的mRNA和蛋白质水平降低50%左右。此外,Tamulaitis等[79]和Staals 等[80]发现 Streptococcus thermophilus和 Thermus thermophilus中的Ⅲ-A型 CRISPR系统也能特异性降解RNA。
植物中广泛存在一类称为PPR的蛋白家族,包括许多序列特异性RNA结合蛋白,它们参与了细胞器中RNA代谢的许多方面,包括RNA稳定性、加工、编辑和翻译等过程。PPR蛋白由2~30个串联的PPR重复单元组成,每个重复单元包含35个氨基酸,它可以特异性地识别一个核糖核苷酸,2~3个特定的氨基酸决定了识别特异性,这与TALE蛋白识别DNA方式非常类似。因此,PPR有可能经过人工设计并作为特异性的RNA结合蛋白用于靶向RNA编辑。Yagi等[81]在其综述中系统介绍了PPR蛋白的研究进展情况。
5 优化SSNs突变效率及减少脱靶效应的方法
5.1 提高SSNs切割效率及突变活性的方法
SSNs突变活性的高低决定了能否获得预期的定点修饰个体及获得突变的频率。目前有以下几种提高SSNs活性的方法:
第一、在细胞或生物体内高水平表达SSNs可以提高突变效率,但同时也可能会提高脱靶突变。例如,Ramakrishna等[82]发现在培养人类细胞时加入蛋白酶抑制剂可抑制 ZFNs蛋白降解,进而显著增加ZFNs诱导的突变效率;在 30℃培养哺乳动物细胞比 37℃下 SSNs的表达水平更高,可以提高 ZFNs 和TALENs的突变效率[12];采用合适的强启动子表达SSNs也可以在一定程度上提高突变效率。
第二、通过同时表达核酸外切酶提高突变效率。Certo等[83]报道转化SSNs同时共同转化DNA断裂末端加工核酸外切酶(Exo1、Trex2等),可以避免DSBs通过cNHEJ途径完好修复,因而提高aNHEJ途径的突变效率。
第三、TALE蛋白骨架结构对突变效率影响很大研究报道适当截短TALE蛋白N端和C端可提高其在人类细胞、烟草和家畜中的活性。有实验表明截短TALE蛋白N端和C端(特别是C端)可以提高蛋白的稳定性,并且能帮助蛋白正确折叠[48,83]。而且,由于TALEN蛋白较大,截短后还有利于载体构建及遗传转化。
第四、TALE蛋白对靶位点的表观状态比较敏感特别是 DNA的甲基化修饰(5mC)会明显抑制TALEN的切割活性。研究表明采用两种RVD(N*或NG; *为第13位缺失)代替HD识别5mC甲基化的DNA位点,可以提高TALE蛋白靶向识别能力[85,86]。另外,在设计TALEN靶向位点时可以尽量避开富含CG区域以减少DNA甲基化带来的影响。
第五、对于CRISPR/Cas9系统,通过优化sgRNA设计可以提高该系统的成功率和突变效率。Doench 等[87]利用抗体着色和流式细胞仪分析方法系统比较了不同 sgRNAs在基因敲除实验中的活性,发现一系列能增强 sgRNA活性的序列特征。具体而言,CGGT往往比典型的NGG序列更好。通过研究1841 个sgRNA中活性较高的sgRNA,他们得出了评分规则,并编写了在线设计高活性sgRNA的程序sgRNA Designer(表1)。另外,Ren等[88]发现紧邻PAM序列上游的6个核苷酸的GC含量与sgRNA诱导的突变效率有显著的正相关性,即 GC含量越高,突变效率越高。
5.2 同源重组机制与提高定点插入或替换效率
提高 HR效率是基因组编辑研究最重要也是最迫切任务之一。在DSBs修复的两种机制中,NHEJ是占绝对主导的,而 HR只发生在特定细胞类型和细胞周期中。
首先通过DSB修复机制的研究,使天平向HR方向倾斜。Ku和Lig4蛋白是NHEJ修复中必不可少的,在拟南芥中的实验表明,ku70或lig4突变体背景下HR与NHEJ修复比例显著提高[89],但很多物种lig4突变体是致死的,因此开发一种专门针对Lig4蛋白的小分子抑制剂实现瞬时消减其活性,对于提高HR介导的基因定点插入或替换有重要价值[8]。SMC6参与姐妹染色单体间的同源重组过程,有趣的是,拟南芥smc6b背景下NHEJ和HR的效率分别提高了6~8倍和3~4倍,但具体机制需进一步阐释[89]。在人类细胞实验中,利用只切开 DNA一条单链的ZFN、TALEN和Cas9切口酶(Nickase),能提高 HR/NHEJ比例[26,90]。DNA单链缺刻不经过NHEJ修复,在靶位点的indel突变减少,但可以提高HR效率。所有真核生物HR修复的起始步骤都是在Exo1等核酸外切酶作用下按照5′→3′方向降解DSB末端,3′单链寡核苷酸DNA末端能够结合Rad51多聚体蛋白并搜寻同源序列。在水稻中,表达SSNs同时超表达OsExo1和(或)OsRecQI4核酸外切酶,能够促进HR和SSA效率[91];在人类细胞和大鼠中,超表达Exo1或另一种核酸外切酶Trex2提高了NHEJ介导的基因敲除效率,但对HR是否有影响并未研究[83]。对DNA修复机制特别是HR修复过程的研究将有助于人们采用适当方法提高基因组编辑中定点插入或替换的效率。在细胞周期的不同阶段,DSB修复方式不同,研究表明HR主要发生在晚S期和G2期;而NHEJ可以发生在所有的细胞周期。因此,细胞周期在很大程度上影响了两种DSB修复方式的选择。Lin等[92]采用了一种化学诱导的方法首先将培养的人类细胞同步化至S期,在此时期将体外组装的 Cas9-sgRNA核糖核蛋白复合体RNP转染HEK293T细胞,与非同步化细胞相比,HR效率提高38%左右。
其次,通过优化供体DNA模板(数量、长度、类型、导入方式等)提高 HR效率是另一条途径。Baltes等[93]利用双生病毒(Geminivirus)载体表达SSNs和DNA模板,已在烟草中表明提高了HR效率。双生病毒是造成很多植物病害的小分子(3 kb左右)单链DNA病毒,如菜豆黄矮病毒(BeYDV)、小麦矮缩病毒(WDV)等。改造的病毒载体保留了复制子(Replicon)元件,并用SSNs和DNA模板代替致病和病毒运动基因,病毒载体在植物细胞内能够大量复制,因此提高 SSNs和 DNA模板数量,与普通T-DNA载体相比,HR效率提高1~2个数量级。此外,Replicon复制起始蛋白的多效性作用进一步提高了基因打靶效率。DNA模板同源臂的长度也影响HR效率,双链DNA模板的同源臂长度一般为1~4 kb,即在DSB两侧等分成大约0.5~2 kb。Byrne等[94]在人类诱导多能干细胞 iPSC中系统研究了基因打靶载体设计对 HR效率的影响,研究表明在无任何辅助筛选条件下,长度为2.7 kb的THY1基因的纯合替换效率最高可以达到 11%。最优化的基因打靶载体参数为大约2 kb同源臂长度,而且环状的载体比线性化载体效率要高。然而,Shin等[95]利用TALEN技术在斑马鱼中进行的 GFP报告基因定点插入实验表明,较长的同源臂和线性化的供体DNA载体更有利于HR,同时还发现,在较短的一侧同源臂的内部切断供体DNA载体获得的HR效率最高,定点插入事件的传代效率能达到 10%以上,具体机制尚不清楚。最近发现较短基因序列的精确修饰,可以采用单链寡核苷酸DNA作为模板,同源臂的长度甚至可以仅为 40nt,即两侧各有 20nt[43]。Puchta实验室采用一种称为植物体内基因打靶的策略提高同源重组效率[96],首先在植物基因组上稳定整合SSNs及供体DNA模板,SSNs表达后同时切开基因组靶位点及供体DNA两侧的识别位点,释放出线性供体DNA,发生HR修复。传统基因打靶实验中DNA模板由T-DNA载体提供,而植物体内基因打靶在所有细胞及植物发育周期中都能发生,因此 HR发生频率可能更高。
另外,NHEJ是DSB的主要修复方式,多个研究报道在人类细胞、斑马鱼、拟南芥、烟草和小麦中,利用没有同源臂的双链或单链DNA模板,成功实现基因替换或定点插入[36,44],有些实验同时利用抗生素筛选基因(HPT或 PURO)或荧光报告基因(GFP)的作用,进一步提高靶向编辑效率。尽管NHEJ方式不能保证与 HR一样的精确,基因片段可能被反向插入或者在 DSBs连接处产生核苷酸的插入或缺失,但NHEJ的效率要远高于HR,因此在精确性要求不高情况下,开启了基因打靶的一种新方法。
5.3 减少脱靶效应
脱靶(Off-target)是指SSNs在其靶位点以外的其他位置切割并产生突变,脱靶突变可能会造成非常严重后果。目前,减少或避免由SSNs引起的脱靶效应的方法有以下几种:
第一、最重要的是在设计靶位点时选择特异性较好的位点,由于在全基因组上没有和其相似性高的序列而可以最大限度减少脱靶。全外显子组和全基因组测序分析表明靶向特异性位点的 ZFNs、TALENs和CRISPR/Cas9系统不会引起细胞中明显的脱靶突变[97,98]。目前已有一些TALEN和CRISPR/ Cas9靶位点设计及脱靶位点预测工具,可以帮助使用者选择高活性、高特异性的靶向位点,例如 The ZFN-Site, PROGNOS Tool和CasOT等(表1)。
第二、FokⅠ核酸酶需要形成二聚体才能切断DNA,而使用ZFN和TALEN技术时可能会出现同型二聚体切割造成的脱靶,即两个 ZFN-L,或两个TALEN-R形成的二聚体。通过优化FokⅠ切割结构域,使其只有形成异型二聚体,如ZFN-L/ZFN-R或TALEN-L/TALEN-R时,二聚化的FokⅠ才具有切割活性,从而可以避免同型二聚体导致的脱靶突变[99]。
第三、对于CRISPR/Cas9系统,据报道可以使用截短的sgRNA(即17、18或19nt导向序列),将脱靶效应降低几个数量级,且不影响靶位点处的突变效率[100]。sgRNA截短后可能使 RNA-DNA复合体对核苷酸错配更加敏感,因而提高靶向特异性。但对于ZFNs或TALENs,适当延长识别序列长度才能提高其靶向特异性[101]。此外,据报道TALE蛋白C末端特定位点的3个或7个氨基酸(精氨酸或赖氨酸)替换成谷氨酰胺可以将人类细胞中的脱靶效应降低十倍左右,且不影响靶位点切割活性[101]。
第四、SSNs的脱靶效应可以通过优化核酸酶的表达水平来降低。高浓度的SSNs可以增加脱靶频率例如,使用重组蛋白或体外转录的mRNA直接转化细胞,由于其可以在细胞内快速降解,因此可以减少脱靶频率[102,103]。另外,采用诱导型启动子或在特定组织中表达的启动子转录SSNs,这种时空特异性表达SSNs方式也能减少脱靶效应。
第五、利用两个sgRNA引导Cas9切口酶或Cas9-FokⅠ融合蛋白,在互补双链DNA上分别形成两个单链DNA断裂,当两个sgRNA靶位点距离较近且方向正确时,可以形成双链 DNA断裂。由于两个sgRNA具有加倍的识别序列长度,因此可以极大降低脱靶突变,特异性提高50~1500倍,并且靶位点处基因敲除效率与野生型Cas9核酸酶相当[90,104]。
6 基因组编辑在植物分子育种应用中的机遇及技术展望
第一代转基因技术自20世纪80年代诞生至今已有30年时间,很多转基因作物如玉米、大豆、油菜和棉花在世界各地都被种植,但转基因作物的生物安全性问题一直备受关注。基因组编辑技术的出现为植物分子育种应用提供了前所未有的新机遇。由于基因组编辑具有精确靶向基因组特定位点的特性,首先将SSN导入植物细胞,实现靶标基因进行定点修饰后,再从后代中筛选只有目标基因突变而不含有 SSN表达载体的株系(图 6)。根据其修饰的方式主要分为三类:基因敲除、少数核苷酸精确替换和基因定点插入。前两类修饰的结果与自然突变或人工诱变非常相似,只有少数核苷酸的改变,无外源DNA导入;而第三类存在外源DNA导入。国际上对基因组编辑技术创制的植物新品种是否属于转基因尚无定论,目前欧盟对GMO 调控比较严格,只要有外源 DNA 转入细胞就视为转基因,而不论最终产品中是否还有外源DNA存在;美国USDA 根据最终产品属性进行评价,用ZFN和TALEN方式产生的前两类基因修饰不受转基因调控[105],利用CRISPR/Cas9 技术产生的作物是否如此目前还没有相关规定。迄今为止,美国USDA已认可至少3项利用基因组编辑技术创制的植物产品不属于转基因范畴:一项是通过ZFN技术创制的低肌醇六磷酸的玉米品系;另一项是通过TALEN技术创制的耐冷藏低丙烯酰胺的马铃薯;还有一项是通过TALEN技术创制的高油酸的大豆。2014年3月,加拿大也通过了其第一个基因组编辑油菜品种,该抗除草剂油菜品种是由美国Cibus Global公司研发,预计在2016年能够上市销售[106]。目前我国对于基因组编辑植物是否属于转基因尚无相关规定,因此,亟待相关部门出台政策规定以正确引导基因组编辑研究。

图6 基因组编辑技术应用于植物分子育种
自从序列特异核酸酶发明以来,经过短短的十几年时间,基因组编辑及相关领域已经取得了革命性变化,特别是CRISPR/Cas9技术的出现,极大加快了植物功能基因组学以及分子育种的研究步伐。但目前也存在一些亟待解决的问题:首先是精确基因定点编辑的能力,即通过 HR途径实现定点插入或替换的效率。不同物种和细胞类型中 HR频率差异很大,而且不同文章中报道的基因打靶载体设计以及提高效率方法也不尽相同。在植物中如何提高效率还需要进行深入系统的探索,建立一套适合植物的体系。本文 5.2部分中论述的方法多在动物体系中得到验证,在植物体内是否有效还需要实验证明。其次,在提高基因编辑效率的同时,还要避免产生脱靶突变,特别是在利用 CRISPR系统时。本文5.3部分多种方法可以提高基因组编辑的特异性,比如利用Cas9切口酶等,这些方法应该同样能够用于植物基因组编辑研究。另外Cas9蛋白晶体结构研究成果能够帮助设计出具有高切割活性和高特异性的小Cas9核酸酶变异体。第三,多基因编辑技术在植物中目前已能实现3~4个基因同时敲除,更多个基因(10个或以上)同时敲除尚无报道。CRISPR/Cas9系统无疑是多基因编辑最好的选择,但可能的困难包括特定物种中只有有限的 RNA聚合酶Ⅲ启动子可以利用,因此需要鉴定多个能高效转录sgRNA的自身或近缘物种中的启动子,或者采用其他系统同时表达多个 sgRNA,例如 Xie等开发的多顺反子tRNA-sgRNA系统利用了tRNA的自身加工机制可以将一个转录本加工成多个独立的sgRNA,大大提高了植物多基因编辑的能力[55];另外一个困难是缺少简易且高通量的多基因编辑载体构建方法。第四,在人类细胞系中利用全基因组水平 CRISPR文库进行功能基因的遗传筛选已经比较成熟,未来几年中在植物(或植物细胞)中建立大规模 CRISPR遗传筛选平台对于加快功能基因鉴定和基因组学研究都有重要意义。困难在于植物中缺乏合适的细胞培养及高效转化体系。第五,基因组编辑的衍生技术如靶向激活、抑制、表观修饰以及基因组呈像等在植物中研究很少,特别是定点表观调控和基因组呈像尚属空白,未来在这一领域还应该有更多的原创研究,建立适合不同植物的靶向基因表达调控等研究工具
[1] Voytas DF. Plant genome engineering with sequencespecific nucleases. Annu Rev Plant Biol, 2013, 64(1): 327-350.
[2] Symington LS, Gautier J. Double-strand break end resection and repair pathway choice. Annu Rev Genet, 2011, 45(1): 247-271.
[3] Puchta H, Fauser F. Synthetic nucleases for genome engineering in plants: prospects for a bright future. Plant J, 2013, 78(5): 727-741.
[4] Mladenov E, Iliakis G. Induction and repair of DNAdouble strand breaks: The increasing spectrum of non-homologous end joining pathways. Mutat Res Fund Mol Mech Mut, 2011, 711(1-2): 61-72.
[5] Osakabe K, Osakabe Y, Toki S. Site-directed mutagenesis in Arabidopsis using custom-designed zinc finger nucleases. Proc Natl Acad Sci USA, 2010, 107(26): 12034-12039.
[6] Siebert R, Puchta H. Efficient repair of genomic double-strand breaks by homologous recombination between directly repeated sequences in the plant genome. Plant Cell, 2002, 14(5): 1121-1131.
[7] Orel N, Kyryk A, Puchta H. Different pathways of homologous recombination are used for the repair of double-strand breaks within tandemly arranged sequences in the plant genome. Plant J, 2003, 35(5): 604-612.
[8] Carroll D. Genome engineering with targetable nucleases. Annu Rev Biochem, 2014, 83(1): 409-439.
[9] Kim YG, Cha J, Chandrasegaran S. Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci USA, 1996, 93(3): 1156-1160.
[10] Kim JS, Lee HJ, Carroll D. Genome editing with modularly assembled zinc-finger nucleases. Nat Methods, 2010, 7(2): 91.
[11] Maeder ML, Thibodeau-Beganny S, Osiak A, Wright DA, Anthony RM, Eichtinger M, Jiang T, Foley JE, Winfrey RJ, Townsend JA, Unger-Wallace E, Sander JD, Müller-Lerch F, Fu FL, Pearlberg J, Göbel C, Dassie JP, Pruett-Miller SM, Porteus MH, Sgroi DC, Iafrate AJ, Dobbs D, McCray PB Jr, Cathomen T, Voytas DF, Joung JK. Rapid “open-source“ engineering of customized zinc-finger nucleases for highly efficient gene modification. Mol Cell, 2008, 31(2): 294-301.
[12] Doyon Y, Choi VM, Xia DF, Vo TD, Gregory PD, Holmes MC. Transient cold shock enhances zinc-finger nuclease-mediated gene disruption. Nat Methods, 2010, 7(6): 459-460.
[13] Sander JD, Dahlborg EJ, Goodwin MJ, Cade L, Zhang F, Cifuentes D, Curtin SJ, Blackburn JS, Thibodeau-Beganny S, Qi Y, Pierick CJ, Hoffman E, Maeder ML, Khayter C, Reyon D, Dobbs D, Langenau DM, Stupar RM, Giraldez AJ, Voytas DF, Peterson RT, Yeh JRJ, Joung JK. Selection-free zinc-finger-nuclease engineering by context-dependent assembly (CoDA). Nat Methods, 2010, 8(1): 67-69.
[14] 沈延, 肖安, 黄鹏, 王唯晔, 朱作言, 张博. 类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术. 遗传, 2013, 35(4): 395-409.
[15] Boch J, Scholze H, Schornack S, Landgraf A, Hahn S, Kay S, Lahaye T, Nickstadt A, Bonas U. Breaking the code of DNA binding specificity of TAL-type III effectors. Science, 2009, 326(5959): 1509-1512.
[16] Moscou MJ, Bogdanove AJ. A simple cipher governs DNA recognition by TAL effectors. Science, 2009, 326(5959): 1501.
[17] Deng D, Yan CY, Pan XJ, Mahfouz M, Wang JW, Zhu JK, Shi YG, Yan N. Structural basis for sequence-specific recognition of DNA by TAL effectors. Science, 2012, 335(6069): 720-723.
[18] Mak ANS, Bradley P, Cernadas RA, Bogdanove AJ, Stoddard BL. The crystal structure of TAL effector PthXo1 bound to its DNA target. Science, 2012, 335(6069): 716-719.
[19] Li T, Huang S, Zhao XF, Wright DA, Carpenter S, Spalding MH, Weeks DP, Yang B. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic Acids Res, 2011, 39(14): 6315-6325.
[20] Cermak T, Doyle EL, Christian M, Wang L, Zhang Y, Schmidt C, Baller JA, Somia NV, Bogdanove AJ, Voytas DF. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res, 2011, 39(12): e82.
[21] Reyon D, Tsai SQ, Khayter C, Foden JA, Sander JD, Joung JK. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol, 2012, 30(5): 460-465.
[22] Schmid-Burgk JL, Schmidt T, Kaiser V, Höning K, Hornung V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat Biotechnol, 2013, 31(1): 76-81.
[23] Sorek R, Lawrence CM, Wiedenheft B. CRISPR-mediated adaptive immune systems in bacteria and archaea. Annu Rev Biochem, 2013, 82(1): 237-266.
[24] 李君, 张毅, 陈坤玲, 单奇伟, 王延鹏, 梁振, 高彩霞. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术.遗传, 2013, 35(11): 1265-1273.
[25] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821.
[26] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823.
[27] Jinek M, Jiang FG, Taylor DW, Sternberg SH, Kaya E, Ma EB, Anders C, Hauer M, Zhou KH, Lin S, Kaplan M,Iavarone AT, Charpentier E, Nogales E, Doudna JA. Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. Science, 2014, 343(6176): 1247997.
[28] Nishimasu H, Ran FA, Hsu PD, Konermann S, Shehata SI, Dohmae N, Ishitani R, Zhang F, Nureki O. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell, 2014, 156(5): 935-949.
[29] Zhang H, Zhang JS, Wei PL, Zhang BT, Gou F, Feng ZY, Mao YF, Yang L, Zhang H, Xu NF, Zhu JK. The CRISPR/Cas9 system produces specific and homozygous targeted gene editing in rice in one generation. Plant Biotechnol J, 2014, 12(6): 797-807.
[30] Feng ZY, Mao YF, Xu NF, Zhang BT, Wei PL, Yang DL, Wang Z, Zhang ZJ, Zheng R, Yang L, Zeng L, Liu XD, Zhu JK. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis. Proc Natl Acad Sci USA, 2014, 111(12): 4632-4637.
[31] Bogdanove AJ. Principles and applications of TAL effectors for plant physiology and metabolism. Curr Opin Plant Biol, 2014, 19: 99-104.
[32] Zhang F, Maeder ML, Unger-Wallace E, Hoshaw JP, Reyon D, Christian M, Li XH, Pierick CJ, Dobbs D, Peterson T, Joung JK, Voytas DF. High frequency targeted mutagenesis in Arabidopsis thaliana using zinc finger nucleases. Proc Natl Acad Sci USA, 2010, 107(26): 12028-12033.
[33] Li T, Liu B, Spalding MH, Weeks DP, Yang B. High-efficiency TALEN-based gene editing produces disease-resistant rice. Nat Biotechnol, 2012, 30(5): 390-392.
[34] Shan QW, Zhang Y, Chen KL, Zhang K, Gao CX. Creation of fragrant rice by targeted knockout of the OsBADH2 gene using TALEN technology. Plant Biotechnol J, 2015, 13(6): 791-800.
[35] Haun W, Coffman A, Clasen BM, Demorest ZL, Lowy A, Ray E, Retterath A, Stoddard T, Juillerat A, Cedrone F, Mathis L, Voytas DF, Zhang F. Improved soybean oil quality by targeted mutagenesis of the fatty acid desaturase 2 gene family. Plant Biotechnol J, 2014, 12(7): 934-940.
[36] Wang YP, Cheng X, Shan QW, Zhang Y, Liu JX, Gao CX, Qiu JL. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nat Biotechnol, 2014, 32(9): 947-951.
[37] 王延鹏, 程曦, 高彩霞, 邱金龙. 利用基因组编辑技术创制抗白粉病小麦. 遗传, 2014, 36(8): 848.
[38] Li JF, Norville JE, Aach J, McCormack M, Zhang DD, Bush J, Church GM, Sheen J. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9. Nat Biotechnol, 2013, 31(8): 688-691.
[39] Feng ZY, Zhang BT, Ding WN, Liu XD, Yang DL, Wei PL, Cao FQ, Zhu SH, Zhang F, Mao YF, Zhu JK. Efficient genome editing in plants using a CRISPR/Cas system. Cell Res, 2013, 23(10): 1229-1232.
[40] Shan QW, Wang YP, Li J, Zhang Y, Chen KL, Liang Z, Zhang K, Liu JX, Xi JJ, Qiu JL, Gao CX. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686-688.
[41] Xie KB, Yang YN. RNA-guided genome editing in plants using a CRISPR-Cas system. Mol Plant, 2013, 6(6): 1975-1983.
[42] Miao J, Guo DS, Zhang JZ, Huang QP, Qin GJ, Zhang X, Wan JM, Gu HY, Qu LJ. Targeted mutagenesis in rice using CRISPR-Cas system. Cell Res, 2013, 23(10): 1233-1236.
[43] Bedell VM, Wang Y, Campbell JM, Poshusta TL, Starker CG, Krug II RG, Tan WF, Penheiter SG, Ma AC, Leung AYH, Fahrenkrug SC, Carlson DF, Voytas DF, Clark KJ, Essner JJ, Ekker SC. In vivo genome editing using a high-efficiency TALEN system. Nature, 2012, 491(7422): 114-118.
[44] Maresca M, Lin VG, Guo N, Yang Y. Obligate ligationgated recombination (ObLiGaRe): custom-designed nuclease-mediated targeted integration through nonhomologous end joining. Genome Res, 2013, 23(3): 539-546. [45] Wright DA, Townsend JA, Winfrey RJ Jr, Irwin PA, Rajagopal J, Lonosky PM, Hall BD, Jondle MD, Voytas DF. High-frequency homologous recombination in plants mediated by zinc-finger nucleases. Plant J, 2005, 44(4): 693-705.
[46] Townsend JA, Wright DA, Winfrey RJ, Fu FL, Maeder ML, Joung JK, Voytas DF. High-frequency modification of plant genes using engineered zinc-finger nucleases. Nature, 2009, 459(7245): 442-445.
[47] Shukla VK, Doyon Y, Miller JC, DeKelver RC, Moehle EA, Worden SE, Mitchell JC, Arnold NL, Gopalan S, Meng X, Choi VM, Rock JM, Wu YY, Katibah GE, Zhifang G, McCaskill D, Simpson MA, Blakeslee B, Greenwalt SA, Butler HJ, Hinkley SJ, Zhang L, Rebar EJ, Gregory PD, Urnov FD. Precise genome modification in the crop species Zea mays using zinc-finger nucleases. Nature, 2009, 459(7245): 437-441.
[48] Zhang Y, Zhang F, Li XH, Baller JA, Qi YP, Starker CG,Bogdanove AJ, Voytas DF. TALENs enable efficient plant genome engineering. Plant Physiol, 2013, 161(1): 20-27.
[49] Fauser F, Schiml S, Puchta H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in Arabidopsis thaliana. Plant J, 2014, 79(2): 348-359.
[50] Qi YP, Li XH, Zhang Y, Starker CG, Baltes NJ, Zhang F, Sander JD, Reyon D, Joung JK, Voytas DF. Targeted deletion and inversion of tandemly arrayed genes in Arabidopsis thaliana using zinc finger nucleases. G3: Genes|Genom|Genet, 2013, 3(10): 1707-1715.
[51] Christian M, Qi YP, Zhang Y, Voytas DF. Targeted mutagenesis of Arabidopsis thaliana using engineered TAL effector nucleases (TALENs). G3: Genes|Genomes| Genetics, 2013, 3(10): 1697-1705.
[52] Shan QW, Wang YP, Chen KL, Liang Z, Li J, Zhang Y, Zhang K, Liu JX, Voytas DF, Zheng XL, Zhang Y, Gao CX. Rapid and efficient gene modification in rice and Brachypodium using TALENs. Mol Plant, 2013, 6(4): 1365-1368.
[53] Zhou HB, Liu B, Weeks DP, Spalding MH, Yang B. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice. Nucleic Acids Res, 2014, 42(17): 10903-10914.
[54] Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ. A CRISPR/Cas9 toolkit for multiplex genome editing in plants. BMC Plant Biol, 2014, 14: 327.
[55] Xie KB, Minkenberg B, Yang YN. Boosting CRISPR/ Cas9 multiplex editing capability with the endogenous tRNA-processing system. Proc Natl Acad Sci USA, 2015, 112(11): 3570-3575.
[56] Wang T, Wei JJ, Sabatini DM, Lander ES. Genetic screens in human cells using the CRISPR/Cas9 system. Science, 2013, 343(6166): 80-84.
[57] Shalem O, Sanjana NE, Hartenian E, Shi X, Scott DA, Mikkelson T, Heckl D, Ebert BL, Root DE, Doench JG, Zhang F. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science, 2013, 343(6166): 84-87.
[58] Zhou YX, Zhu SY, Cai CZ, Yuan PF, Li CM, Huang YY, Wei WS. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature, 2014, 509(7501): 487-491.
[59] Findlay GM, Boyle EA, Hause RJ, Klein JC, Shendure J. Saturation editing of genomic regions by multiplex homology-directed repair. Nature, 2014, 513(7516): 120-123. [60] Xie F, Ye L, Chang JC, Beyer AI, Wang JM, Muench MO, Kan YW. Seamless gene correction of β-thalassemia mutations in patient-specific iPSCs using CRISPR/Cas9 and piggyBac. Genome Res, 2014, 24(9): 1526-1533.
[61] Baltes NJ, Voytas DF. Enabling plant synthetic biology through genome engineering. Trends Biotechnol, 2015, 33(2): 120-131.
[62] de Lange O, Binder A, Lahaye T. From dead leaf, to new life: TAL effectors as tools for synthetic biology. Plant J, 2014, 78(5): 753-771.
[63] Sanchez JP, Ullman C, Moore M, Choo Y, Chua NH. Regulation of gene expression in Arabidopsis thaliana by artificial zinc finger chimeras. Plant Cell Physiol, 2002, 43(12): 1465-1472.
[64] Morbitzer R, Römer P, Boch J, Lahaye T. Regulation of selected genome loci using de novo-engineered transcription activator-like effector (TALE)-type transcription factors. Proc Natl Acad Sci USA, 2010, 107(50): 21617-21622.
[65] Liu WS, Rudis MR, Peng YH, Mazarei M, Millwood RJ, Yang JP, Xu WZ, Chesnut JD, Stewart CN. Synthetic TAL effectors for targeted enhancement of transgene expression in plants. Plant Biotechnol J, 2013, 12(4): 436-446.
[66] Baazim H. RNA-guided transcriptional regulation in plants via dCas9 chimeric proteins. Bioscience, 2014. http://hdl.handle.net/10754/316715.
[67] Tanenbaum ME, Gilbert LA, Qi LS, Weissman JS, Vale RD. A protein-tagging system for signal amplification in gene expression and fluorescence imaging. Cell, 2014, 159(3): 635-646.
[68] Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F. Genome-scale transcriptional activation by an engineered CRISPRCas9 complex. Nature, 2014, 517(7536): 583-588.
[69] Mahfouz MM, Li LX, Piatek M, Fang XY, Mansour H, Bangarusamy D, Zhu JK. Targeted transcriptional repression using a chimeric TALE-SRDX repressor protein. Plant Mol Biol, 2012, 78(3): 311-321.
[70] Zhang ZH, Wu E, Qian ZJ, Wu WS. A multicolor panel of TALE-KRAB based transcriptional repressor vectors enabling knockdown of multiple gene targets. Sci Rep, 2014, 4: 7338.
[71] Gilbert LA, Larson MH, Morsut L, Liu ZR, Brar GA, Torres SE, Stern-Ginossar N, Brandman O, Whitehead EH, Doudna JA, Lim WA, Weissman JS, Qi LS. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 2013, 154(2): 442-451.
[72] Zalatan JG, Lee ME, Almeida R, Gilbert LA, Whitehead EH, La RM, Tsai JC, Weissman JS, Dueber JE, Qi LS, Lim WA. Engineering complex synthetic transcriptional programs with CRISPR RNA scaffolds. Cell, 2015, 160(1-2): 339-350.
[73] Meister GE, Chandrasegaran S, Ostermeier M. Heterodimeric DNA methyltransferases as a platform for creating designer zinc finger methyltransferases for targeted DNA methylation in cells. Nucleic Acids Res, 2010, 38(5): 1749-1759.
[74] Mendenhall EM, Williamson KE, Reyon D, Zou JY, Ram O, Joung JK, Bernstein BE. Locus-specific editing of histone modifications at endogenous enhancers. Nat Biotechnol, 2013, 31(12): 1133-1136.
[75] Konermann S, Brigham MD, Trevino AE, Hsu PD, Heidenreich M, Cong L, Platt RJ, Scott DA, Church GM, Zhang F. Optical control of mammalian endogenous transcription and epigenetic states. Nature, 2013, 500(7463): 472-476.
[76] Maeder ML, Angstman JF, Richardson ME, Linder SJ, Cascio VM, Tsai SQ, Ho QH, Sander JD, Reyon D, Wilkinson MF, Joung JK. Targeted DNA demethylation and activation of endogenous genes using programmable TALE-TET1 fusion proteins. Nat Biotechnol, 2013, 31(12): 1137-1142.
[77] O' Connell MR, Oakes BL, Sternberg SH, East-Seletsky A, Kaplan M, Doudna JA. Programmable RNA recognition and cleavage by CRISPR/Cas9. Nature, 2014, 516(7530): 263-266.
[78] Zebec Z, Manica A, Zhang J, White MF, Schleper C. CRISPR-mediated targeted mRNA degradation in the archaeon Sulfolobus solfataricus. Nucleic Acids Res, 2014, 42(8): 5280-5288.
[79] Tamulaitis G, Kazlauskiene M, Manakova E, Venclovas Č, Nwokeoji AO, Dickman MJ, Horvath P, Siksnys V. Programmable RNA shredding by the type III-A CRISPR-Cas system of Streptococcus thermophilus. Mol Cell, 2014, 56(4): 506-517.
[80] Staals RHJ, Zhu YF, Taylor DW, Kornfeld JE, Sharma K, Barendregt A, Koehorst JJ, Vlot M, Neupane N, Urlaub H, Heck AJ, Nogales E, Doudna JA, Shinkai A, van der Oost J. RNA targeting by the type III-A CRISPR-Cas Csm complex of Thermus thermophilus. Mol Cell, 2014, 56(4): 518-530.
[81] Yagi Y, Nakamura T, Small I. The potential for manipulating RNA with pentatricopeptide repeat proteins. Plant J, 2014, 78(5): 772-782.
[82] Ramakrishna S, Kim YH, Kim H. Stability of zinc finger nuclease protein is enhanced by the proteasome inhibitor MG132. PLoS One, 2013, 8(1): e54282.
[83] Certo MT, Gwiazda KS, Kuhar R, Sather B, Curinga G, Mandt T, Brault M, Lambert AR, Baxter SK, Jacoby K, Ryu BY, Kiem HP, Gouble A, Paques F, Rawlings DJ, Scharenberg AM. Coupling endonucleases with DNA end-processing enzymes to drive gene disruption. Nat Methods, 2012, 9(10): 973-975.
[84] Miller JC, Tan SY, Qiao GJ, Barlow KA, Wang JB, Xia DF, Meng XD, Paschon DE, Leung E, Hinkley SJ, Dulay GP, Urnov FD, Zhang HS, Holmes MC, Zhang L, Gregory PD, Rebar EJ. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol, 2011, 29(2): 143-148.
[85] Deng D, Yin P, Yan CY, Pan XJ, Gong XQ, Qi SQ, Xie T, Mahfouz M, Zhu JK, Yan N, Shi YG. Recognition of methylated DNA by TAL effectors. Cell Res, 2012, 22(10): 1502-1504.
[86] Valton J, Dupuy A, Daboussi F, Thomas S, Maréchal A, Macmaster R, Melliand K, Juillerat A, Duchateau P. Overcoming Transcription Activator-like Effector (TALE) DNA binding domain sensitivity to cytosine methylation. J Biol Chem, 2012, 287(46): 38427-38432.
[87] Doench JG, Hartenian E, Graham DB, Tothova Z, Hegde M, Smith I, Sullender M, Ebert BL, Xavier RJ, Root DE. Rational design of highly active sgRNAs for CRISPRCas9-mediated gene inactivation. Nat Biotechnol, 2014, 32(12): 1262-1267.
[88] Ren XJ, Yang ZH, Xu J, Sun J, Mao DC, Hu YH, Yang SJ, Qiao HH, Wang X, Hu Q, Deng P, Liu LP, Ji JY, Li JB, Ni JQ. Enhanced specificity and efficiency of the CRISPR/Cas9 system with optimized sgRNA parameters in Drosophila. Cell Rep, 2014, 9(3): 1151-1162.
[89] Qi YP, Zhang Y, Zhang F, Baller JA, Cleland SC, Ryu Y, Starker CG, Voytas DF. Increasing frequencies of site-specific mutagenesis and gene targeting in Arabidopsis by manipulating DNA repair pathways. Genome Res, 2013, 23(3): 547-554.
[90] Mali P, Aach J, Stranges PB, Esvelt KM, Moosburner M, Kosuri S, Yang LH, Church GM. CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering. Nat Biotechnol, 2013, 31(9): 833-838.
[91] Kwon YI, Abe K, Osakabe K, Endo M, Nishizawa-Yokoi A, Saika H, Shimada H, Toki S. Overexpression of OsRecQl4 and/or OsExo1 enhances DSB-induced homologous recombination in rice. Plant Cell Physiol, 2012, 53(12): 2142-2152.
[92] Lin S, Staahl BT, Alla RK, Doudna JA. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. eLife, 2014, 3: e04766.
[93] Baltes NJ, Gil-Humanes J, Cermak T, Atkins PA, Voytas DF. DNA replicons for plant genome engineering. Plant Cell, 2014, 26(1): 151-163.
[94] Byrne SM, Ortiz L, Mali P, Aach J, Church GM. Multi-kilobase homozygous targeted gene replacement in human induced pluripotent stem cells. Nucleic Acids Res, 2015, 43(3): e21.
[95] Shin J, Chen JK, Solnica-Krezel L. Efficient homologous recombination-mediated genome engineering in zebrafish using TALE nucleases. Development, 2014, 141(19): 3807-3818.
[96] Fauser F, Roth N, Pacher M, Ilg G, Sánchez-Fernández R, Biesgen C, Puchta H. In planta gene targeting. Proc Natl Acad Sci USA, 2012, 109(19): 7535-7540.
[97] Yusa K, Rashid ST, Strick-Marchand H, Varela I, Liu PQ, Paschon DE, Miranda E, Ordonez A, Di Santo JP, Lomas DA, Bradley A, Vallier L. Targeted gene correction of α1-antitrypsin deficiency in induced pluripotent stem cells. Nature, 2011, 478(7369): 391-394.
[98] Smith C, Gore A, Yan W, Abalde-Atristain L, Li Z, He CX, Wang Y, Brodsky RA, Zhang K, Cheng LZ, Ye ZH. Whole-genome sequencing analysis reveals high specificity of CRISPR/Cas9 and TALEN-based genome editing in human iPSCs. Cell Stem Cell, 2014, 15(1): 12-13.
[99] Doyon Y, Vo TD, Mendel MC, Greenberg SG, Wang JB, Xia DF, Miller JC, Urnov FD, Gregory PD, Holmes MC. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat Methods, 2011, 8(1): 74-79.
[100] Fu YF, Sander JD, Reyon D, Cascio VM, Joung JK. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol, 2014, 32(3): 279-284.
[101] Guilinger JP, Pattanayak V, Reyon D, Tsai SQ, Sander JD, Joung JK, Liu DR. Broad specificity profiling of TALENs results in engineered nucleases with improved DNA-cleavage specificity. Nat Methods, 2014, 11(4): 429-435.
[102] Gaj T, Guo J, Kato Y, Sirk SJ, Barbas CF. Targeted gene knockout by direct delivery of zinc-finger nuclease proteins. Nat Methods, 2012, 9(8): 805-807.
[103] Cai YJ, Bak RO, Mikkelsen JG, Golub T. Targeted genome editing by lentiviral protein transduction of zinc-finger and TAL-effector nucleases. eLife, 2014, 3: e01911.
[104] Guilinger JP, Thompson DB, Liu DR. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat Biotechnol, 2014, 32(6): 577-582.
[105] Ledford H. US regulation misses some GM crops. Nature, 2013, 500(7463): 389-390.
[106] Jones HD. Regulatory uncertainty over genome editing. Nat Plants, 2015, 1: 14011.
[107] Li L, Piatek M, Atef A, Piatek A, Wibowo A, Fang X, Sabir J, Zhu JK, Mahfouz M. Rapid and highly efficient construction of TALE-based transcriptional regulators and nucleases for genome modification. Plant Mol Biol, 2012, 78(4-5): 407-416.
[108] Johnson RA, Gurevich V, Levy AA. A rapid assay to quantify the cleavage efficiency of custom-designed nucleases in planta. Plant Mol Biol, 2013, 82(3): 207-221.
[109] Beurdeley M, Bietz F, Li J, Thomas S, Stoddard T, Juillerat A, Zhang F, Voytas DF, Duchateau P, Silva GH. Compact designer TALENs for efficient genome engineering. Nat Commun, 2013, 4: 1762.
[110] Wendt T, Holm PB, Starker CG, Christian M, Voytas DF, Brinch-Pedersen H, Holme IB. TAL effector nucleases induce mutations at a pre-selected location in the genome of primary barley transformants. Plant Mol Biol, 2013, 83(3): 279-285.
[111] Curtin SJ, Zhang F, Sander JD, Haun WJ, Starker C, Baltes NJ, Reyon D, Dahlborg EJ, Goodwin MJ, Coffman AP, Dobbs D, Joung JK, Voytas DF, Stupar RM. Targeted mutagenesis of duplicated genes in soybean with zinc-finger nucleases. Plant Physiol, 2011, 156(2): 466-473.
[112] Gurushidze M, Hensel G, Hiekel S, Schedel S, Valkov V, Kumlehn J. True-breeding targeted gene knock-out in barley using designer TALE-nuclease in haploid cells. PLoS One, 2014, 9(3): e92046.
[113] Liang Z, Zhang K, Chen KL, Gao CX. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system. J Genet Genom, 2014, 41(2): 63-68.
[114] Lor VS, Starker CG, Voytas DF, Weiss D, Olszewski NE. Targeted mutagenesis of the tomato PROCERA gene using TALENs. Plant Physiol, 2014, 166(3): 1288-1291.
[115] Jiang WZ, Yang B, Weeks DP. Efficient CRISPR/ Cas9-mediated gene editing in Arabidopsis thaliana and inheritance of modified genes in the T2 and T3 generations. PLoS One, 2014, 9(6): e99225.
[116] Hyun Y, Kim J, Cho SW, Choi Y, Kim JS, Coupland G.Site-directed mutagenesis in Arabidopsis thaliana using dividing tissue-targeted RGEN of the CRISPR/Cas system to generate heritable null alleles. Planta, 2015, 241(1): 271-284.
[117] Schiml S, Fauser F, Puchta H. The CRISPR/Cas system can be used as nuclease for in planta gene targeting and as paired nickases for directed mutagenesis in Arabidopsis resulting in heritable progeny. Plant J, 2014, 80(6): 1139-1150.
[118] Mao YF, Zhang H, Xu NF, Zhang BT, Gao F, Zhu JK. Application of the CRISPR-Cas system for efficient genome engineering in plants. Mol Plant, 2013, 6(6): 2008-2011.
[119] Jiang WZ, Zhou HB, Bi HH, Fromm M, Yang B, Weeks DP. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice. Nucleic Acids Res, 2013, 41(20): e188.
[120] Gao JP, Wang GH, Ma SY, Xie XD, Wu XW, Zhang XT, Wu YQ, Zhao P, Xia QY. CRISPR/Cas9-mediated targeted mutagenesis in Nicotiana tabacum. Plant Mol Biol, 2015, 87(1-2): 99-110.
[121] Upadhyay SK, Kumar J, Alok A, Tuli R. RNA guided genome editing for target gene mutations in wheat. G3: Genes|Genom|Genet, 2013, 3(12): 2233-2238.
[122] Sugano SS, Shirakawa M, Takagi J, Matsuda Y, Shimada T, Hara-Nishimura I, Kohchi T. CRISPR/Cas9 mediated targeted mutagenesis in the liverwort Marchantia polymorpha L. Plant Cell Physiol, 2014, 55(3): 475-481.
[123] Jia HG, Wang N. Targeted genome editing of sweet orange using Cas9/sgRNA. PLoS One, 2014, 9(4): e93806.
[124] Brooks C, Nekrasov V, Lippman ZB, Van Eck J. Efficient gene editing in tomato in the first generation using the clustered regularly interspaced short palindromic repeats/CRISPR-associated9 system. Plant Physiol, 2014, 166(3): 1292-1297.
[125] Weinthal DM, Taylor RA, Tzfira T. Nonhomologous end joining-mediated gene replacement in plant cells. Plant Physiol, 2013, 162(1): 390-400.
[126] de Pater S, Pinas JE, Hooykaas PJJ, van der Zaal BJ. ZFN-mediated gene targeting of the Arabidopsis protoporphyrinogen oxidase gene through Agrobacteriummediated floral dip transformation. Plant Biotechnol J, 2013, 11(4): 510-515.
[127] Cai CQ, Doyon Y, Ainley WM, Miller JC, DeKelver RC, Moehle EA, Rock JM, Rebar EJ, Collingwood TN, Rubin-Wilson B, Gregory PD, Urnov FD, Petolino JF. Targeted transgene integration in plant cells using designed zinc finger nucleases. Plant Mol Biol, 2009, 69(6): 699-709.
[128] Ainley WM, Sastry-Dent L, Welter ME, Murray MG, Zeitler B, Amora R, Corbin DR, Miles RR, Arnold NL, Strange TL, Simpson MA, Cao Z, Carroll C, Pawelczak KS, Blue R, West K, Rowland LM, Perkins D, Samuel P, Dewes CM, Shen L, Sriram S, Evans SL, Rebar EJ, Zhang L, Gregory PD, Urnov FD, Webb SR, Petolino JF. Trait stacking via targeted genome editing. Plant Biotechnol J, 2013, 11(9): 1126-1134.
(责任编委: 张博)
Research progress of genome editing and derivative technologies in plants
Qiwei Shan, Caixia Gao
State Key Laboratory of Plant Cell and Chromosome Engineering, Institute of Genetics and Developmental Biology, Chinese Academy of Sciences, Beijing 100101, China
Genome editing technologies using engineered nucleases have been widely used in many model organisms. Genome editing with sequence-specific nuclease (SSN) creates DNA double-strand breaks (DSBs) in the genomic target sites that are primarily repaired by the non-homologous end joining (NHEJ) or homologous recombi-nation (HR) pathways, which can be employed to achieve targeted genome modifications such as gene mutations, insertions, replacements or chromosome rearrangements. There are three major SSNs—zinc finger nuclease (ZFN), transcription activator-like effector nuclease (TALEN) and clustered regularly interspaced short palindromic repeats/CRISPR-associated 9 (CRISPR/Cas9) system. In contrast to ZFN and TALEN, which require substantial protein engineering to each DNA target, the CRISPR/Cas9 system requires only a change in the guide RNA. For this reason, the CRISPR/Cas9 system is a simple, inexpensive and versatile tool for genome engineering. Furthermore, a modified version of the CRISPR/Cas9 system has been developed to recruit heterologous domains that can regulate endogenous gene expression, such as activation, depression and epigenetic regulation. In this review, we summarize the development and applications of genome editing technologies for basic research and biotechnology, as well as highlight challenges and future directions, with particular emphasis on plants.
genome editing; ZFN; TALEN; CRISPR/Cas9; homologous recombination; off-target mutation
2015-04-13;
2015-06-18
国家自然科学基金项目(编号:31420103912和31271795)和转基因重大专项(编号:2014ZX0801003B)资助
单奇伟,博士,研究方向:遗传学。E-mail: qwshan@icloud.com
高彩霞,博士,研究员,博士生导师,研究方向:遗传学。E-mail: cxgao@genetics.ac.cn
10.16288/j.yczz.15-156
时间:2015-9-1 11:14:07
URL:http://www.cnki.net/kcms/detail/11.1913.R.20150901.1114.006.html
