普陀樟SRAP-PCR反应体系的建立
2014-11-24吕昕谣赵国淼曾燕如宋绪忠
吕昕谣,赵 颖,赵国淼,曾燕如*,宋绪忠,杨 华
(1.浙江农林大学 亚热带森林培育国家重点实验室培育基地,浙江 临安 311300;2.浙江省舟山市林业科学研究院,浙江 舟山 316000;3.浙江省林业科学研究院,浙江 杭州 310023)
普陀樟(Cinnamomum japonicumvar.chenii)系樟科樟属常绿乔木,为舟山海岛特有濒危植物。集中分布在舟山市普陀区的普陀山、朱家尖及其毗邻的悬水小岛上,除此之外的其他区域均未见有该树种的天然分布[1]。普陀樟木材坚实致密,纹理通直,耐腐,耐水湿,是优良的用材树种;且根系发达,抗风性强,且兼具耐盐碱、耐干旱瘠薄等特性,适应性广,非常适宜沿海地区栽种。而目前却缺乏对野生普陀樟致濒原因等各方面的足够了解,难以实施有效的遗传保育。
相关序列扩增多态性(Sequence-related amplifiedpolymorphism, SRAP)是Li和Quiros[2]开发的分子标记技术,该技术通过独特的引物设计对开放阅读框(Open reading frames, ORFs)进行扩增,因个体不同以及物种的内含子、启动子与间隔区长度不等而产生多态性[3]。Ferriol等[4]在观赏南瓜(Cucurbita moschata)中利用SRAP和AFLP两种分子标记进行遗传多样性实验,结果与AFLP标记相比,SRAP标记结果更接近其性状变异。Budak[5]等对野牛草(Buchloe dactyloides)进行ISSR(Inter-simple Sequence Repeat)、SSR(Simple Sequence Repeat)、RAPD(Random Amplified Polymorphic DNA)和SRAP等4种分子标记的遗传多样性研究,结果SRAP表现出更丰富的遗传多样性,是区分能力最强的标记。与其他分子标记相比,SRAP标记技术具有重复性好、多态性高、操作简便、共显性、易测序、引物具有通用性等优越性,已逐渐成为种质资源研究的有效技术,近年来多应用于遗传多样性分析[5]、遗传图谱构建[6]以及重要性状标记[7]等诸多领域。
目前,有关普陀樟分子标记方面的研究尚未见报道。本研究建立了普陀樟的SRAP-PCR反应体系,可为进一步开展普陀樟的遗传多样性及相关遗传研究提供技术支撑,也可为普陀樟基因组分析、分子标记辅助育种等奠定基础。
1 材料与方法
1.1 材料
新鲜健康的普陀樟叶片样品7份采自舟山群岛不同岛屿,带回实验室后清水洗净,放置-40℃冰箱保存待用。

表1 SRAP 引物序列Table1 Sequences of SRAP primers
Taq DNA聚合酶(5U/μL)购自上海申能博彩生物科技有限公司;dNTP、DNA Marker(Fermentas)、SRAP 引物(表1)等均购自生工生物工程(上海)有限公司。
1.2 方法
1.2.1 基因组DNA的提取与检测 采用改良CTAB方法结合TNE的方法[8]提取普陀樟基因组DNA。提取的DNA用1.0%琼脂糖凝胶电泳检测DNA的完整性,用紫外分光光度计(NanoDrop 1000 Spectrophotometer,美国)测定A260、A280,并计算两者的比值,以检验DNA的质量,并根据测定的浓度将DNA稀释到10 ng/μL,-20℃保存备用。
1.2.2 SRAP-PCR反应体系的建立 基于刘浩凯等优化的适用于榧树的SRAP分析方法(含PCR产物电泳)[9]采用L16(45)正交试验设计,对可能影响普陀樟SRAP-PCR反应的Mg2+浓度、dNTPS、Taq DNA聚合酶、引物(F2/R4)和模板DNA用量进行5因素4水平正交试验(表2)。反应总体积为20 μL,实验重复4次,以确定最佳体系组合。

表2 SRAP-PCR正交设计试验L16(45)Table2 L16(45)orthogonal design for SRAP-PCR reaction
反应程序为94℃预变性5 min;共5个循环,每个循环94℃变性45s,35℃退火45s,72℃延伸1 min;随后35个循环,每个循环94℃变性45 s,50℃退火45s,72℃延伸1 min;最后72℃延伸5 min,4℃保存。扩增产物加入3 μL Loading dye混合均匀,以3 000 bp DNA Ladder为参照标准,采用8%非变性聚丙烯酰胺凝胶进行电泳,160V电泳85 min。电泳结束后采用银染法进行染色,显色后将凝胶置于可见光灯箱上观察电泳结果,并拍照保存。
随机选取F6/R7和F6/R8两个引物组合,利用筛选出的普陀樟最优体系,对30份普陀樟DNA材料进行PCR扩增,检测反应体系的稳定性。
利用优化设计确定的SRAP-PCR的最佳反应体系,用表1所示的10个正向引物和10个反向引物组合的100对引物,对4个不同普陀樟样品进行引物组合筛选。
2 结果与分析
2.1 普陀樟基因组DNA的提取
经紫外分光光度计测定,8份普陀樟DNA样品OD260/OD280值均在1.8 ~ 2.0;1%琼脂糖电泳检测结果(图1)显示,DNA条带完整清晰,无拖尾现象,符合分子标记扩增对DNA的要求。

图1 普陀樟DNA的琼脂糖电泳检测图Figure1 Argarose electrophoretogram of genomic DNAs extracted in C.japonicum var.chenii
2.2 SRAP-PCR反应体系的优化
从正交实验结果可以看出,不同的PCR组份组合扩增效果具有明显的差异,其中1 ~ 4、7、8号扩增效果不好,11、12、15、16号位点较清楚,但位点数少,均予以淘汰(图2)。仔细观察发现,9、10号比13、14号扩增的位点数多,而且更加清晰。直观分析10号组合为佳,9号组合次之,二者间无明显差异。虑到9号组合中 Taq 酶的用量为 2 U,因此本着经济有效的原则,最终确定10号组合为最理想的SRAP-PCR反应体系,即20 μL PCR体系包括 2.5 mmol/L Mg2+、0.2 mmol/L dNTPs、1.5 U Taq DNA聚合酶、0.4 μmol/L引物、10 ng模板DNA、2 μL 10×PCR buffer。

图2 正交实验产物电泳图Figure2 Electrophoretogram of PCR products based on the orthogonal design
2.3 SRAP-PCR体系的稳定性检测及引物筛选
SRAP-PCR体系的稳定性检测表明,扩增谱带清晰而稳定,位点数多,且多态性丰富(图3),说明建立的反应体系具有良好的稳定性,适合于普陀樟的SRAP-PCR分析。利用该反应体系,从100对引物组合(表1)中筛选出20对扩增产物清晰、多态性较好的引物(F2R7、F3R7、F4R7、F5R7、F6R7、F8R7、F9R7、F5R1、F5R8、F6R8、F9R6、F2R1、F4R2、F5R1、F5R2、F3R3、F4R3、F6R4、F10R4、F5R5),用于普陀樟后续遗传多样性分析。
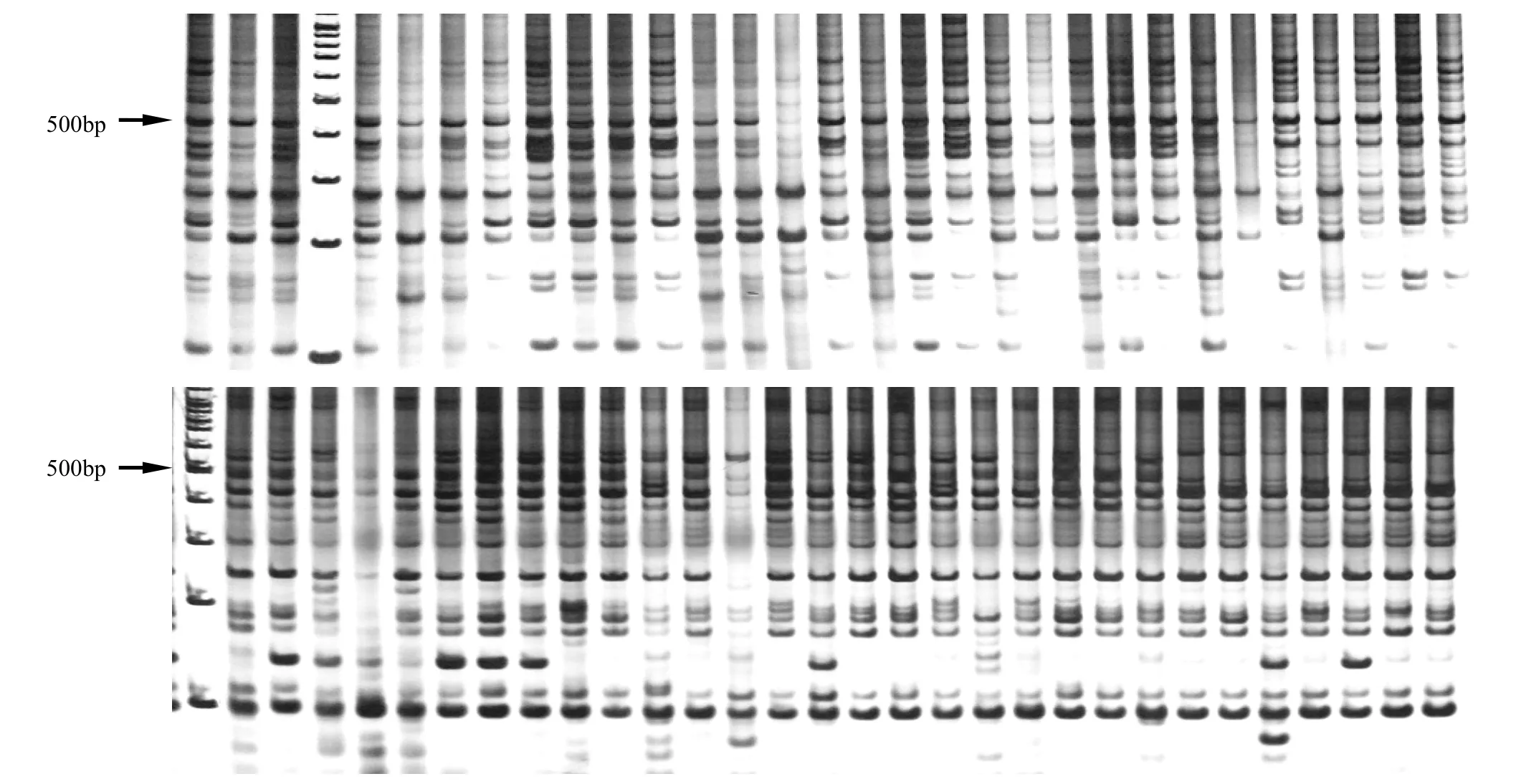
图3 SRAP反应体系稳定性检测(引物对:上:F6R7;下:F6R8)Figure3 Testing of the SRAP reaction (Primer pairs: F6R7 (up) and F6R8(down))
3 结论与讨论
SRAP分子标记是基于PCR反应的一种标记技术,它的结果受到体系中各个因素的影响,且各因素之间还存在相互作用,因此各因素之间的配比只有达到最佳状态时,才能得到稳定、可重复的扩增结果。不同植物适合的SRAP 反应体系不同,所以需要根据物种的不同对反应体系进行优化。
本实验采用了一次性正交实验设计筛选反应体系,并对该体系稳定性进行了检测及初步应用,结果显示扩增位点清晰稳定,多态性好,重复性强,说明本研究建立的普陀樟SRAP-PCR体系稳定可靠、扩增效率高,为日后普陀樟基因组分析、分子标记辅助育种、遗传多样性研究等奠定了基础。
[1]俞慈英,陈叶平,袁燕飞,等.舟山海岛普陀樟等3种特有树种种质资源清查[J].浙江林学院学报,2007,24(4):413-418.
[2]Li G, Quiros C F.Sequence-related amplified polymorphism(SRAP), A new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J].Theor Appl Genet, 2001(103):455-461.
[3]郭大龙,张君玉,李猛,等.葡萄SRAP反应体系优化及引物筛选[J].基因组学与应用生物学,2010,29(2):379-384.
[4]Ferriol M, Pic B, Nuez F.Genetic diversity of a germplasm collection of Cucurbita pepousing SRAP and AFLP markers[J].Theor Appl Genet,2003(107):271-282.
[5]Budak H, Shearman R C, Parmaksiz I,et al.Molecular characterization of Buffalo grass germplasm using sequence-related amplified polymorphism markers[J].Theor Appl Genet, 2004(108):328-334.
[6]王茂芊,白晨.华北地区甜菜品系遗传多样性的SRAP分析[J].内蒙古农业科技,2010(2):29-32, 83.
[7]徐微,张宗文,张恩来,等.大粒裸燕麦(Avena nudaL.)遗传连锁图谱的构建[J].植物遗传资源学报,2013,14(4):673-678.
[8]李华丽,陈美霞,周东新,等.烟草六个重要性状的QTL定位[J].作物学报,2011,37(9):1 577-1 584.
[9]周生财,黄华宏,童再康,等.4种楠木AFLP反应体系优化建立[J].浙江农林大学学报,2013,30(5):789-796.
[10]邱帅,丁信誉,席梦利,等.东方百合SRAP体系优化及引物筛选[J].南京林业大学学报(自然科学版),2013,37(3):6-10.
[11]王燕,龚义勤,赵统敏,等.番茄SRAP-PCR体系优化与品种分子鉴定[J].南京农业大学学报,2007,30(1):23-29.
[12]楚爱香,汤庚国.观赏海棠SRAP-PCR反应体系优化及引物筛选[J].湖北农业科学,2008,47(12):1 394-1 397.
[13]郭庆华,郭美丽,薛芊,等.青葙SRAP体系的建立和优化[J].中草药,2008,39(2):263-266.
[14]王志勇,袁学军,刘建秀,等.狗牙根SRAP-PCR反应体系优化及引物筛选[J].草业学报,2008,17(3):79-85.
[15]路娟,张绍铃,刘庆忠,等.樱桃SRAP-PCR体系优化及其遗传多样性分析[J].果树学报,2009,26(2):163-169.
[16]徐建华,杨虹琦,杨程,等.烟草DNA的提取与SRAP反应体系优化[J].湖南农业大学学报(自然科学版),2009,35(3):257-259.
[17]邹明宏,郭凌飞,曾辉,等.山竹子基因组DNA提取及SRAP反应体系优化[J].经济林研究,2009,27(3):38-41.
[18]赵永国,金梦阳,危文亮.新型植物油莎豆DNA提取与SRAP体系优化[J].中国油料作物学报,2011,33(1):39-43.
[19]彭婵,张新叶,李振芳,等.乌桕SRAP-PCR体系优化与引物筛选[J].湖北林业科技,2011(1):13-17.
[20]杨晨希,王国良.金樱子SRAP-PCR反应体系的建立与优化[J].林业科技开发,2012,26(3):99-103.
[22]刘振,赵洋,杨培迪,等.茶树SRAP反应体系优化及引物筛选[A].湖南省茶叶学会.湖南省茶叶学会2011年学术年会论文集[C].湖南省茶叶学会,2011.
[23]骈瑞琪,倪慧群,陈晓阳.胡枝子SRAP体系优化及在胡枝子亲缘关系中的应用[A].国家林业局,广西壮族自治区人民政府,中国林学会.第二届中国林业学术大会——S2功能基因组时代的林木遗传与改良论文集[C].国家林业局,广西壮族自治区人民政府,中国林学会,2009.
[24]陈莉娉,张小平,李晓红.青檀SRAP-PCR体系优化设计方案[J].生物学杂志,2012,29(5):87-91.
[25]邱文武,韦持章,郭凌飞,等.广西野生桃金娘SRAP体系优化及建立[J].植物研究,2013,33(3):346-350.
