两株高致病性PRRSV湖北分离株ORF5与Nsp2基因序列分析
2014-11-23马慧慧饶清宜漆世华温文生李玉萍谢红玲
杨 雷,马慧慧,饶清宜,漆世华,温文生,李玉萍,谢红玲
(武汉中博生物股份有限公司,武汉430070)
猪繁殖与呼吸综合征(Porcine Reproductive and Respiratory Syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)引起的一种严重危害养猪业的传染病[1]。其主要症状表现为妊娠母猪的流产、死胎、木乃伊胎等繁殖障碍,以及各种年龄猪(特别是仔猪)的呼吸道症状。PRRSV属于动脉炎病毒科,为一种有囊膜的单股正链RNA病毒,基因组全长约 15 kb,含有 9个开放阅读框(ORFs):ORF1a,ORF1b,ORFs2-7[2-3]。 PRRSV 有两种基因型,分别为北美基因型VR-2332和欧洲基因型Lelystad 病毒(LV)[4]。 1996 年郭宝清等[5]首次从国内疑似PRRS感染猪群中分离出PRRSV,从而证实了本病在我国的存在。2006年我国猪群暴发了高致病性猪繁殖与呼吸综合征,高致病性猪繁殖与呼吸综合征是由高度变异的PRRSV引起的猪的烈性传染病,已发病超过200万头猪,大约有400000头的死亡病例[6]。本研究从发病猪场采集病料,分离得到两株高致病性PRRSV毒株,并对其ORF5和Nsp2序列进行分析,为研究流行毒株的变异情况及选择合理的毒株制备疫苗进行免疫,以及预防和控制该病奠定了基础。
1 材料和方法
1.1 病料和细胞 病料:采自湖北某规模化猪场疑似PRRSV感染猪的肺脏和淋巴结。细胞:Marc-145细胞来源于中国兽医药品监察所,由本公司实验室以常规方法培养、保存,外源检测纯净。
1.2 主要试剂 DMEM培养基购自Hyclone公司;胎牛血清购自山东劲牛公司;RNA提取试剂盒、一步法 RT-PCR 试剂盒(Code:DRR064A)、DL8000、DNA胶回收试剂盒均购自TaKaRa公司产品。
1.3 引物设计 根据GenBank中发表的PRRSV美洲型毒株比对结果,分别设计了用于扩增ORF5和Nsp2基因的引物。其中ORF5上下游引物序列为:ORF5-1:5’ -ATGTTGGGGAAGTGCTTGACC-3’,ORF5-2:5’-CTAGAGACGACCCCATAGTTC-3’,预期扩增片段大小为603 bp;Nsp2上下游引物序列为:Nsp2-1:5’-TAGGAAGGTCAGGTCCGATT-3’,Nsp2-2:5’-ACAGGGAGCTGCTTGATGAT-3’,预期扩增片段大小为425 bp。引物由上海桑尼生物科技有限公司合成。
1.4 病毒分离 取1 g疑似PRRSV病死猪组织病料,加10 mL DMEM制成组织匀浆,加青霉素、链霉素各2000 U/mL,反复冻融 3次,4000 r/min 离心20 min,取上清用 0.22 μm 滤膜过滤除菌,按10%的量接种至培养单层的Marc-145细胞,37℃吸附1 h,加10 mL维持液并于37℃恒温箱培养,观察细胞病变情况。待细胞出现典型的细胞病变时,收毒,置-70℃保存。同上方法连续传代,收毒时间稳定后,保存备用。
1.5 一步法RT-PCR 按照RNA提取试剂盒说明书提取病料RNA,按照一步法RT-PCR明书进行PCR扩增,PCR反应体系如下:2×一步法RT-PCR BufferⅢ 12.5 μL、上下游引物各 0.5 μL、Ex-Taq HS 0.5 μL、RT Enzyme Mix Ⅱ 0.5 μL、RAN 2 μL,加去离子水至25 μL。ORF5基因 PCR反应程序:42℃反转录5 min,95℃预变性 10 s,1个循环;95 ℃变性 5 s,64 ℃退火40 s,72 ℃延伸40 s,共35个循环;72℃延伸5 min。Nsp2基因PCR反应程序同ORF5基因。反应结束后分别取PCR产物进行常规电泳检测。
1.6 序列分析 将琼脂糖凝胶电泳含有目的片段的PCR产物进行切胶,按照TaKaRa公司胶回收试剂盒说明书回收目的基因片段。将回收的目的片段送上海生工生物工程有限公司测序,测序结果用DNA MAN软件分析,并和GenBank中其他PRRSV毒株的Nsp2、ORF5基因序列进行同源性比较和分析,国内外参考序列见表1。

表1 不同来源的PRRSV毒株
2 结果
2.1 病毒的分离 从Marc-145细胞上成功分离到2株病毒,分别命名为HBXN和HBHS。培养的正常Marc-145细胞形态较好,轮廓清楚,界限清晰;2株分离毒株在Marc-145细胞上均可致典型CPE,主要表现:刚开始表现为细胞聚集成丛,在随后的2~3 d内表现为固缩、变圆,最后细胞开始脱落(图1)。

图1 正常Marc-145细胞和接毒后Marc-145细胞
2.2 一步法RT-PCR的鉴定 用设计的2对特异性引物分别进行一步法RT-PCR,产物经1%琼脂糖凝胶电泳,可见大小约603 bp和425 bp的特异性扩增片段,与预期结果一致(图2)。目的基因片段经电泳回收,送上海生工生物工程有限公司进行测序,将测序结果与国内外的参考序列进行比对分析。
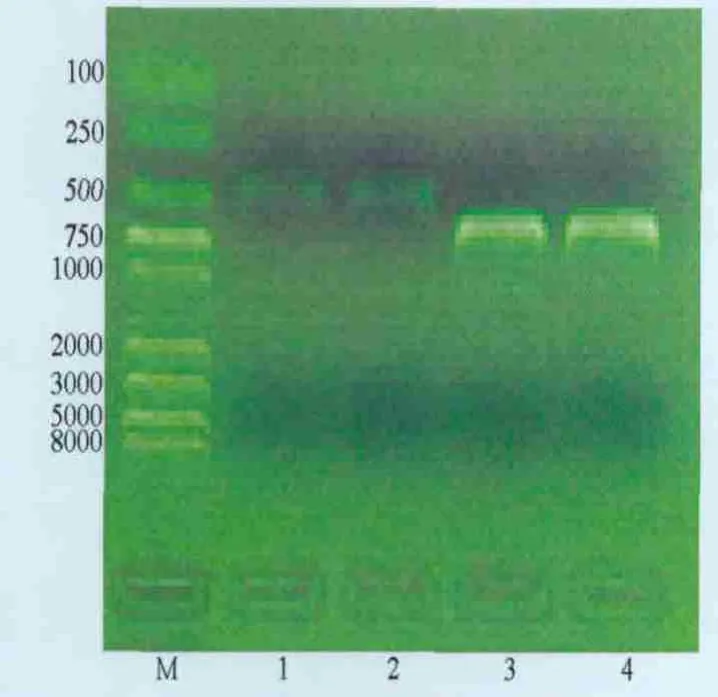
图2 PCR扩增分离毒株目的片段电泳结果
2.3 Nsp2基因序列分析 用DNA MAN软件对PRRSV HBXN株和HBHS株的Nsp2部分基因序列进行分析表明,两株分离株的Nsp2基因均有2个部位出现了缺失,分别为3个核苷酸和连续87个核苷酸的缺失,与国内分离株Nsp2基因序列核苷酸缺失部分的比对结果见图3。核苷酸同源性分析表明,两个分离株之间Nsp2基因核苷酸与氨基酸同源性达到98.8%,与国内分离的高致病性PRRSV毒株JXA1、GD2007、GD2008的氨基酸同源性很高,介于98.5% ~99.5%;而与 NB-04、HUN4、HB-1、WUH1高致病性毒株同源性较低(表2)。遗传进化树分析表明(图4),两个分离株的Nsp2序列与国内分离的高致病性PRRSV毒株JXA1、GD2007、GD2008在同一亚群中,与PRRSV疫苗株pMLV和AMERVAC、NB-04株、HUN4株、HB-1株、WUH1株、LV株存在较远的遗传距离。
2.4 ORF5基因序列分析 用DNA MAN软件对PRRSV HBHS株和HBXN株的ORF5基因序列进行分析表明,两个分离株之间ORF5氨基酸同源性达到98.6%,HBHS株和 2007年国内流行毒株HUN4的氨基酸同源性较高,为99.5%。HBXN株与HK13株的氨基酸同源性较高,为99.0%。HBHS株和HBXN株与国外分离株pMLV株、LV株和VR2332株的氨基酸同源性较低(表3)。系统进化树分析表明(图5),HBHS株和HBXN株的ORF5序列与国内分离的高致病性PRRSV毒株JXA1、GD2007、GD2008、WUH1、HUN4、HK13、HB-1、NB-04的遗传距离很近,同处一个基因群中,而与CH-1a等经典毒株较远。

图3 部分Nsp2基因缺失序列对比分析

表2 Nsp2部分基因序列氨基酸同源性比对分析
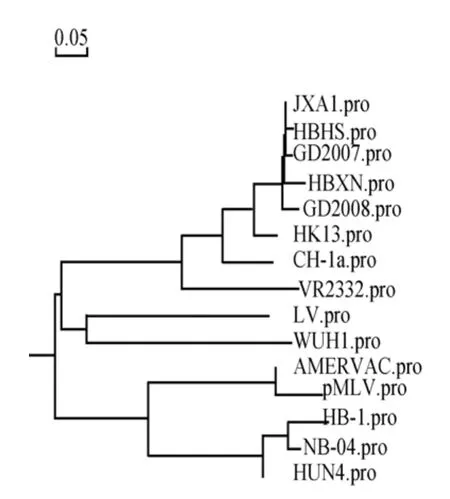
图4 Nsp2部分基因序列系统进化树

图5 ORF5基因序列系统进化树

表3 ORF5基因序列氨基酸同源性比对分析
3 讨论
PRRSV各分离株基因组序列存在不同程度的差异。不同地域分离株基因组序列分析显示,PRRSV有2种基因型,即美洲型(以VR2332为代表株)和欧洲型(以LV为代表株),欧洲型分离株间的差异较小,北美洲型分离株间的差异相对较大。目前,国内外已经完成多株PRRSV的全基因组序列测定[7-8],序列分析的结果表明,PRRSV的变异分布于整个基因组,尤以ORF5基因和Nsp2基因的变异最大[9-10]。因此,对这2个基因变异的分析在一定程度上可以反映病毒基因组序列变异的情况,在研究上常作为比较PRRSV变异的靶基因。
本研究通过对HBHS株和HBXN株的Nsp2基因序列分析表明:此次分离的两株高致病性PRRSV HBHS株和HBXN株都分别缺失了3个碱基和87个碱基,即共缺失30个氨基酸,这与国内分离的高致病性 PRRSV毒株 JXA1、GD2007、GD2008的Nsp2高变区碱基的缺失是一致的,且具有很高的同源性。序列比对发现此次分离到的两个分离株之间也具有很高的同源性,高达98.8%。所分离毒株临床上具有高致病性。
PRRS不同分离株的核苷酸序列存在广泛而且明显的变异,ORF5基因最容易发生变异,由其编码的GP5蛋白是PRRSV所有结构蛋白中具有较强抗原性的蛋白,不仅可诱导产生与病毒的中和反应呈显著相关的中和抗体,还在引起特异性细胞免疫、阻止肺泡巨噬细胞损伤和诱导细胞凋亡等多个方面发挥着重要作用[11]。因此,ORF5基因的变异,引起其编码的GP5蛋白的变化,带来抗原性的改变,最终导致PRRSV相关特性的改变。这可能是免疫猪群不能抵抗变异毒株攻击而发病的原因之一。通过对ORF5序列分析发现,本研究中所分离的HBHS株和HBXN株的ORF5序列与国内分离的高致病性 PRRSV毒株 JXA1、GD2007、GD2008、WUH1、HUN4、HK13、HB-1、NB-04 的遗传距离很近,同处一个基因群中,而与CH-1a等经典毒株较远。说明了分离的毒株ORF5基因在很大程度上发生了变异,该结论为研究流行毒株的变异情况及选择合理的毒株制备疫苗进行免疫,以及预防和控制该病奠定了基础。
[1]Meulenberg J J.PRRSV, the virus[J].Vet Res, 2000, 31(1):1l-21.
[2]Grebennikova T V, Clouser D F, Vorwald A C,et al.Genomic characterization of virulent, attenuated, and revertant passages of a North American porcine reproductive and respiratory syndrome virus strain[J].J Virol, 2004, 321:383-390.
[3]Wootton S, Yoo D, Rogan D.Full-length sequence of a Canadian porcine reproductive and respiratory syndrome virus(PRRSV)isolate[J].Arch Virol, 2000, 145:2297-2323.
[4]Nelsen C J, Murtaugh M P, Faaberg K S.Porcine reproductive and respiratory syndrome virus comparison:divergent evolution on two continents[J].J Virol, 1999, 73:270-280.
[5]郭宝清,陈章水,刘文兴,等.从疑似PRRS流产胎儿分离猪繁殖与呼吸综合征病毒(PRRSV)的研究[J].中国畜禽传染病,1996, 87(2):1-5.
[6]Tian K, Yu X, Zhao T,et al.Emergence of fatal PRRSV variants:unparalleled outbreaks of atypical PRRS in China and molecular dissection of the unique hallmark[J].PLOS One,2007, 2(6):1-10.
[7]Cheon D S, Chae C.Antigenic variation and genotype of isolates of porcine reproductive and respiratory syndrome virus in Korea[J].Vet Rec, 2000, 147(8): 215-218.
[8]Kapur V,Pawlovich T M,Murtaugh M P,et al.Genetic variation in porcine reproductive and respiratory syndrome virus isolates in the midwestern United States[J].J Gen Virol, 1996, 77(6): 1271-1276.
[9]Mateu E, Diaz I.The challenge of PRRS immunology[J].Vet J,2008, 177(3): 345-351.
[10]Mateu E, Diaz I, Darwich L, et al.Evolution of ORF5 of Spanish porcine reproductive and respiratory syndrome virus strains from 1991 to 2005[J].Virus Res, 2006, 115(2): 198-206.
[11]刘 芳,汪铭书,程安春.猪繁殖与呼吸综合征病毒诱导细胞凋亡的研究进展[J].黑龙江畜牧兽医,2005,(10):82-83.
