用于寡核苷酸二级结构预测的热力学数据库研究进展
2014-11-14刘哲言屈武斌张成岗
刘哲言,屈武斌,张成岗
(军事医学科学院放射与辐射医学研究所,蛋白质组学国家重点实验室,全军军事认知与心理卫生研究中心,北京100850)
近年来,以核酸分子杂交为基础的生物技术如聚合酶链反应、DNA印迹、RNA印迹、芯片杂交等在病原微生物检测、临床诊断中应用广泛,其可靠性依赖于寡核苷酸分子与其靶点结合的高稳定性与特异性,而分析这种结合特性的关键在于寡核苷酸与靶分子结合的二级结构的精确预测,否则会导致假阴性或假阳性的检测结果[1-4]。
已有研究显示最近邻模型(Nearest-Neighbor Model,简称NN model)是预测寡核苷酸二级结构最可靠的热力学计算方法[5],该模型指出一个给定碱基对的稳定性依赖于其临近碱基对的稳定性。其基本思想是将核酸分子结合过程中的标准焓变和熵变计算转化为由A、T、G、C所形成的10个完美匹配二聚体以及非完美匹配结构的标准焓变和熵变的累加和,再加上起始和结束单独匹配碱基对GC或AT以及序列对称性等因素的影响[6-7]。然而,由于寡核苷酸单分子自身折叠或者双分子杂交所形成的二级结构具有多样性与复杂性,除完美匹配外,还包含单独错配、连续错配、内环、膨胀环、末端摇摆、CNG重复、GU摆动等多种模式。因此,寡核苷酸二级结构热力学计算的精确性还需要依赖上述多种结构的热力学参数。
鉴于目前的热力学数据分散于不同的文献中[8-11],且不同来源数据可靠性不一,因此本文根据学术界对不同来源数据的认可程度,系统性的综述了近年来广泛用于寡核苷酸二级结构预测的热力学参数及相关计算,并指出当前数据库的局限及未来发展方向,从而为相关人员进行研究提供整合数据库资源,促进寡核苷酸二级结构的精确预测。
1 DNA与DNA相互作用的热力学参数
1.1 完美匹配
针对DNA/DNA相互作用双链中Watson-Crick的 AT、GC配对,SantaLucia于 1998年提出的在1mol/L NaCl环境下的寡核苷酸最近邻热力学参数是与实验数据误差最小的参数表[8],受到广泛应用(见表1)。其中ΔH、ΔS与ΔG分别表示标准焓变、标准熵变和标准自由能,可直接从热力学参数表获取。完美匹配结构的热力学计算如下:
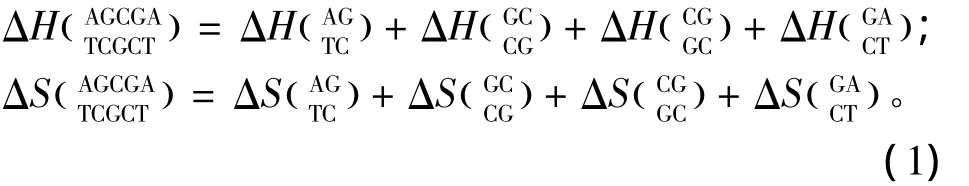
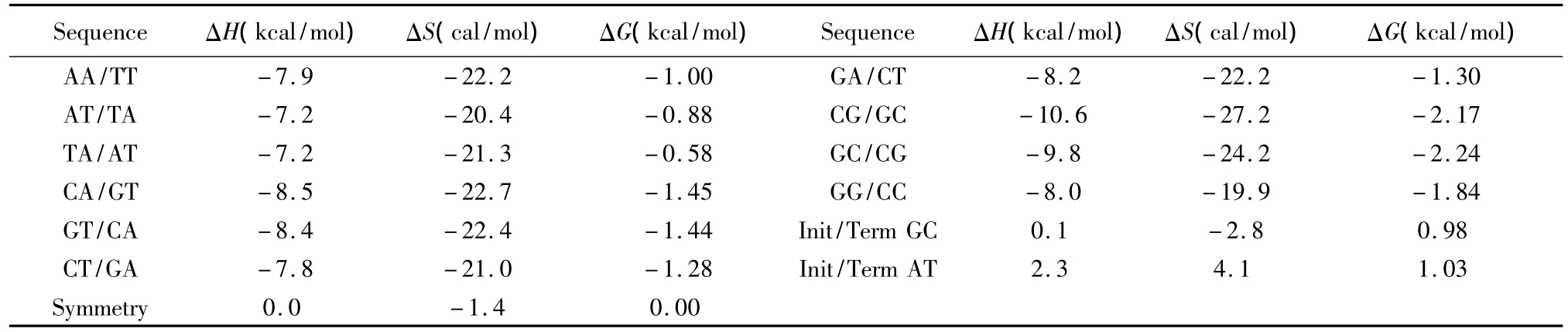
表1 DNA/DNA结合完美匹配最近邻热力学参数(1 mol/L,37℃)Table 1 Nearest-neighbor thermodynamic parameters for DNA/DNA perfect matches(1 mol/L,37℃)
1.2 单独错配
当出现单独错配的模式时,应用 Allawi、SantaLucia以及Peyret等人于1997~1999年提供的热力学数据[9-13](见表2)。单独错配结构的热力学计算如下:
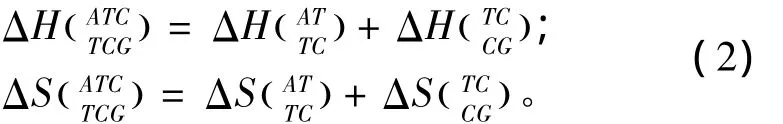
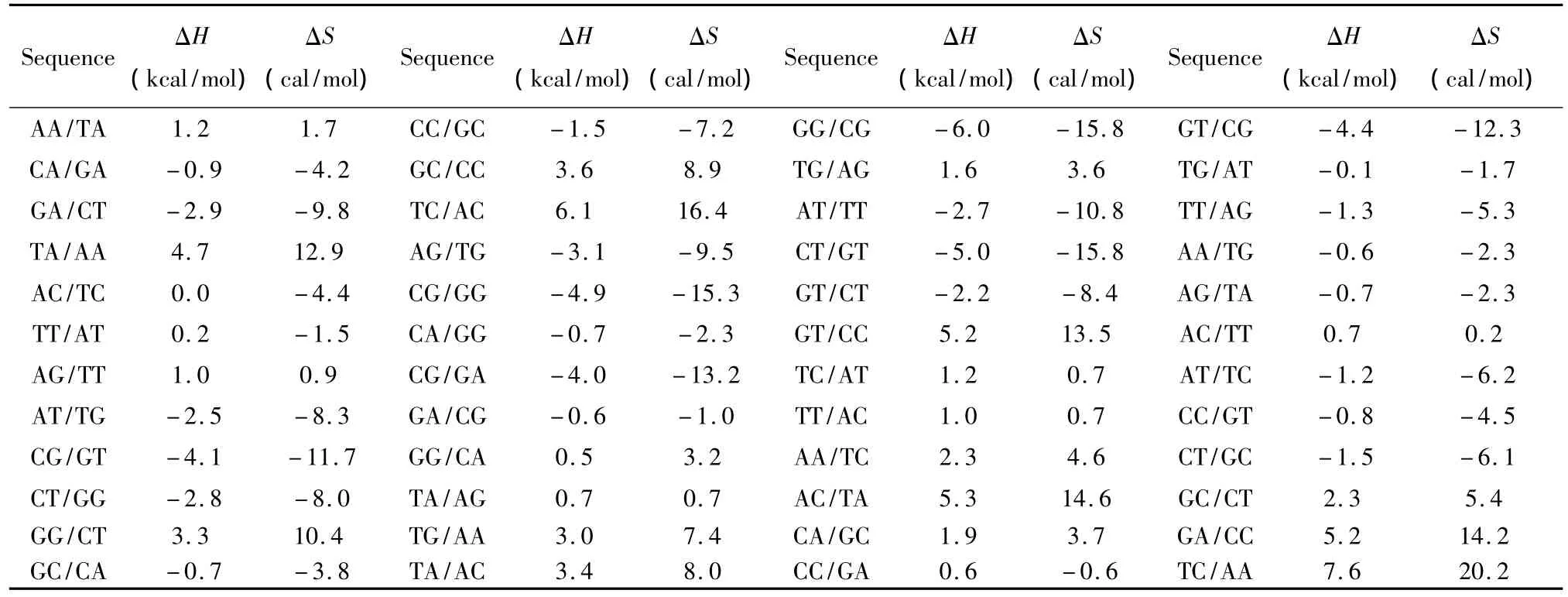
表2 单独错配结构热力学参数(1 mol/L,37℃)Table 2 Thermodynamic parameters for DNA/DNA single mismatch(1 mol/L,37 ℃)
1.3 连续错配
当出现连续错配即两个毗连错配的模式时,应用Allawi、SantaLucia以及Peyret等人于1997~1999年提供的热力学数据[9-13](见表3)。连续错配结构的热力学计算如下:
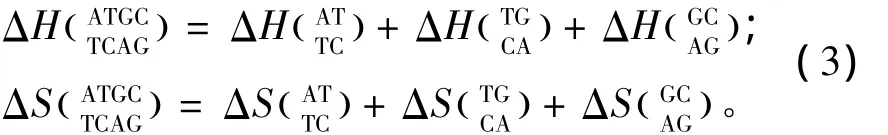
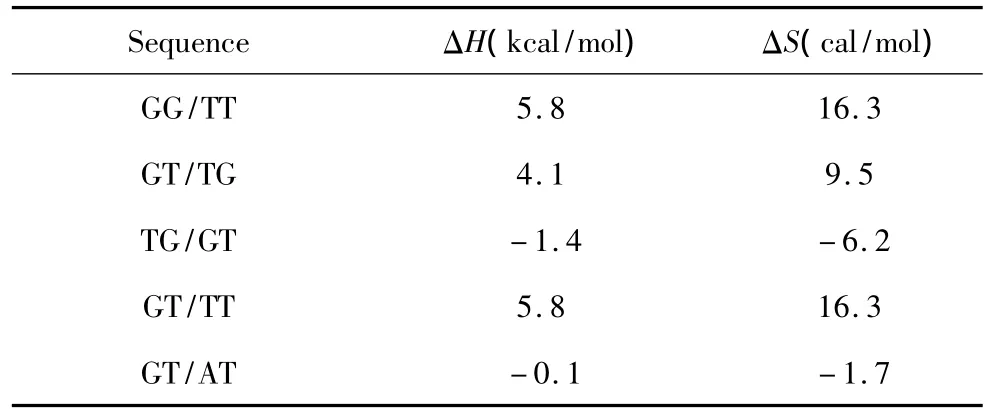
表3 连续错配结构热力学参数(1 mol/L,37℃)Table 3 Thermodynamic parameters for DNA/DNA tandem mismatches(1 mol/L,37 ℃)
1.4 内环
当出现内环即三个及其以上毗连错配的模式时,应用Santalucia和Hicks于2004年提供的热力学数据[14](见表4)。内环结构的热力学计算如下:
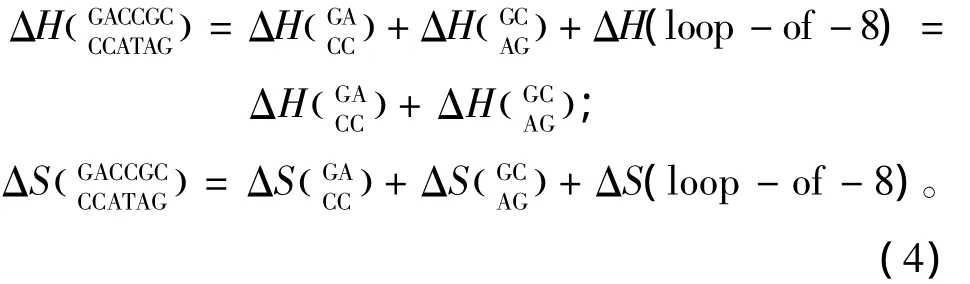
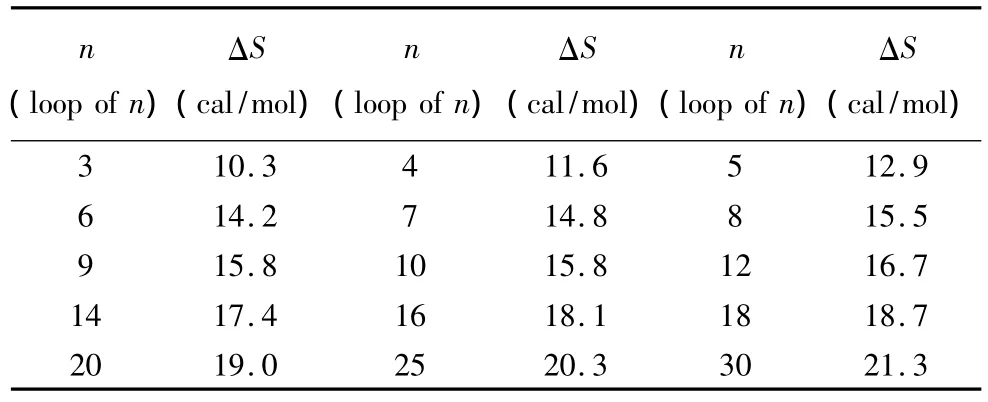
表4 内环结构热力学参数(1 mol/L,37℃)Table 4 Thermodynamic parameters for DNA/DNA internal loop(1 mol/L,37 ℃)
1.5 单独摇摆末端
所谓单独摇摆末端(Single dangling-end),指杂交序列5’或3’末端出现一个未匹配的核酸即空位gap结构(用“-”表示)。当出现这种模式时,应用Bommarito等人于2000年提供的热力学数据[15](见表5)。Single dangling-end结构的热力学计算如下:
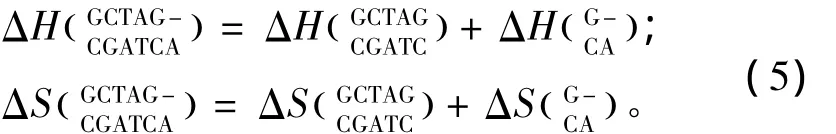
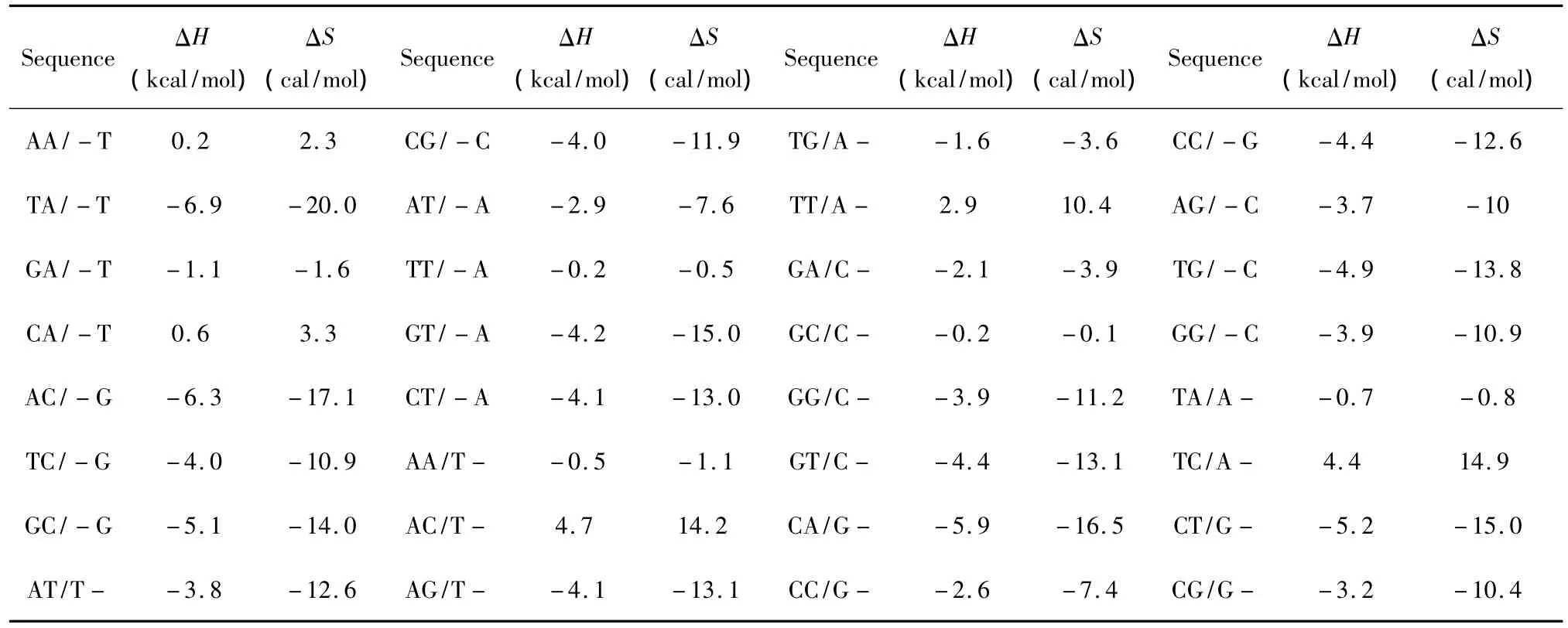
表5 Single dangling-end结构热力学参数(1 mol/L,37℃)Table 5 Thermodynamic parameters for DNA/DNA single dangling-end(1 mol/L,37 ℃)
1.6 长摇摆末端
所谓长摇摆末端(Long dangling-end),指杂交序列5’或3’末端出现连续两个及其以上未匹配的核酸即空位gap结构(用“-”表示)。当出现这种模式时,应用Sugimoto等人于2002年提供的热力学数据[16](见表6)。Long dangling-end结构的热力学计算如下:
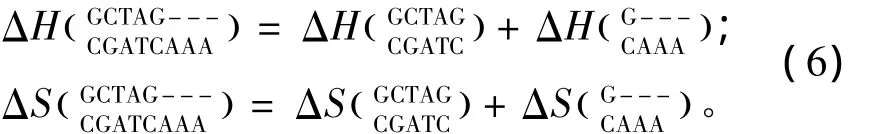
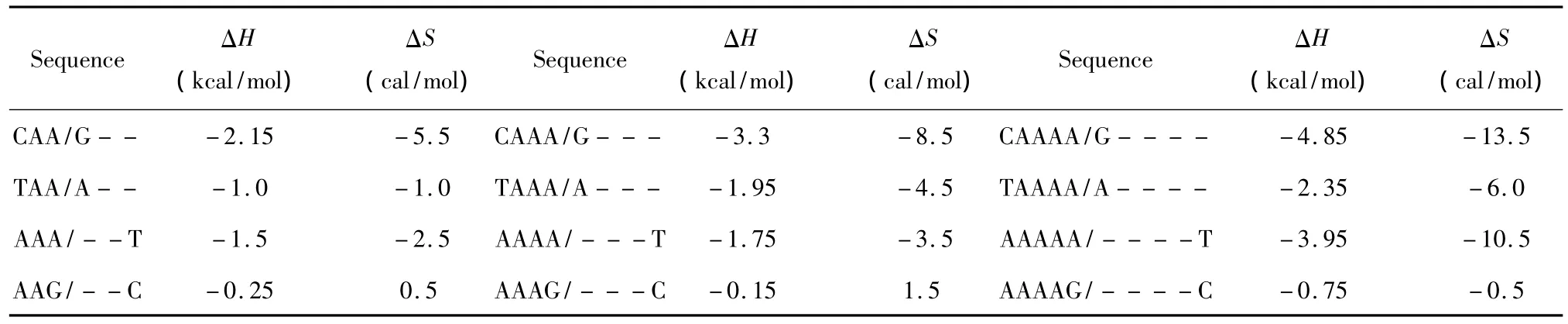
表6 Long dangling-end结构热力学参数(1 mol/L,37℃)Table 6 Thermodynamic parameters for DNA/DNA long dangling-end(1 mol/L,37 ℃)
1.7 单独膨胀环
所谓单独膨胀环(Single bulge loop),指杂交序列内部出现一个未匹配的核酸即空位gap结构(用“-”表示)。当出现这种模式时,应用Tanaka等人于2004年提供的热力学数据[17](见表7)。Single bulge loop结构的热力学计算如下:
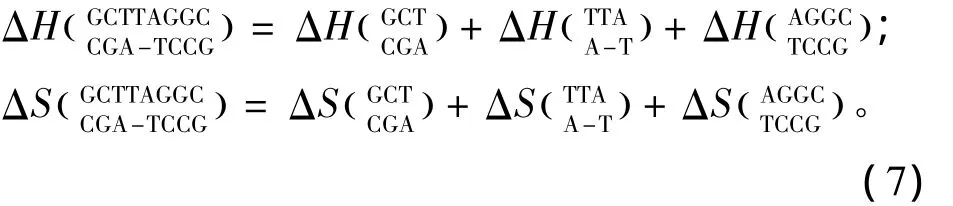
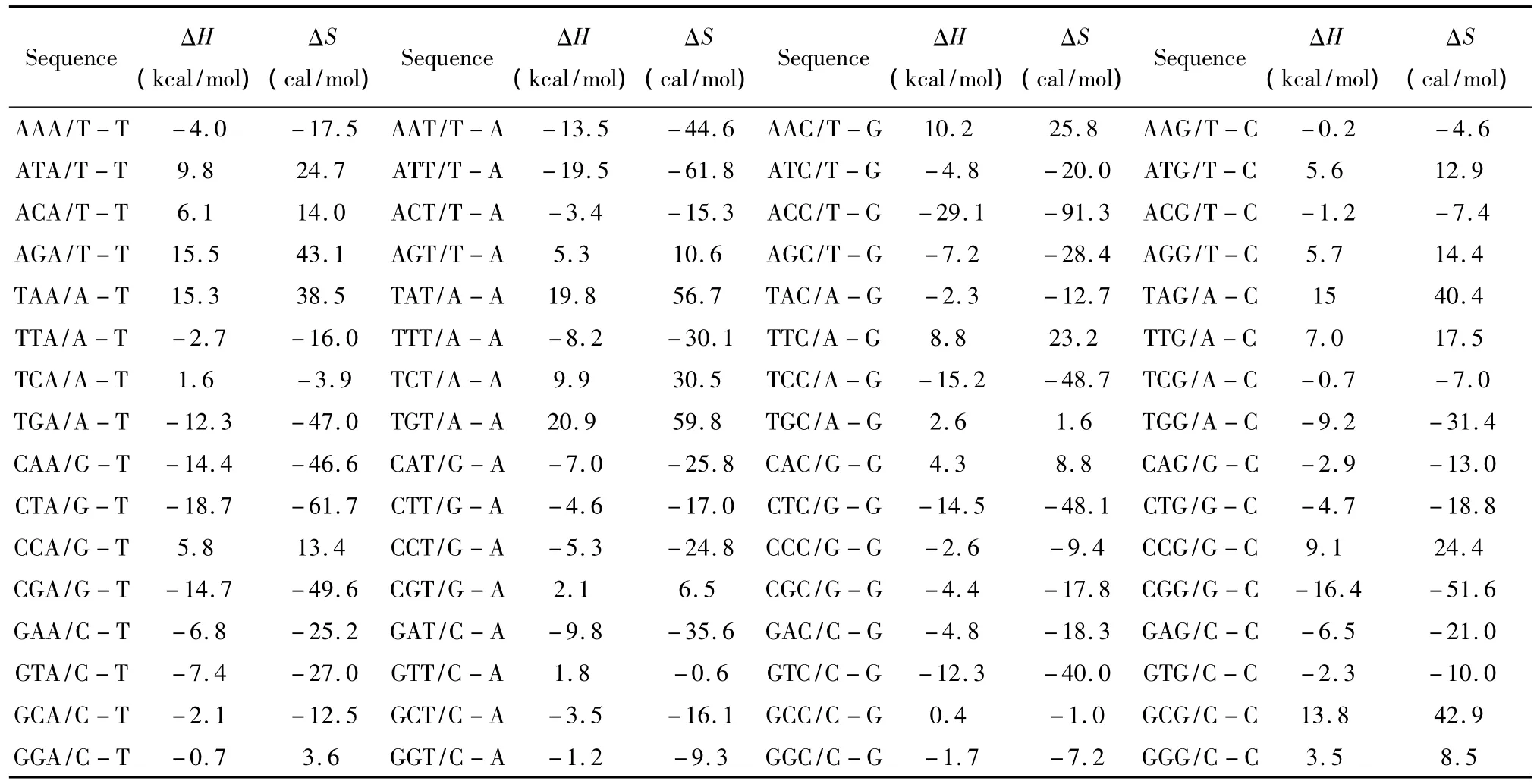
表7 Single bulge loop结构热力学参数(1 mol/L,37℃)Table 7 Thermodynamic parameters for DNA/DNA single bulge loop(1 mol/L,37 ℃)
1.8 长膨胀环
所谓长膨胀环(Long bulge loop),指杂交序列内部出现连续两个及其以上未匹配的核酸即空位gap结构(用“-”表示)。当出现这种模式时,应用Santalucia和Hicks于2004年提供的热力学数据[14](见表8)。Long bulge loop结构的热力学计算如下:

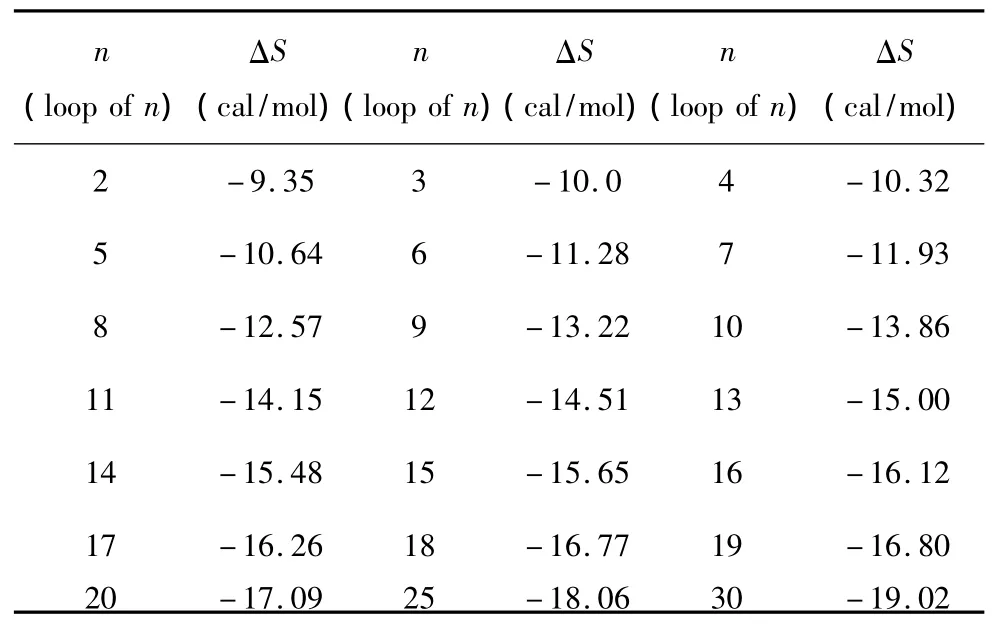
表8 Long bulge loop结构热力学参数(1 mol/L,37℃)Table 8 Thermodynamic parameters for DNA/DNA long bulge loop(1 mol/L,37 ℃)
2 DNA与RNA相互作用的热力学参数
当DNA与RNA相互作用时,完美匹配的最近邻热力学参数表由Sugimoto等人于1995年提出[18](见表9),完美匹配结构的热力学计算同1.1。
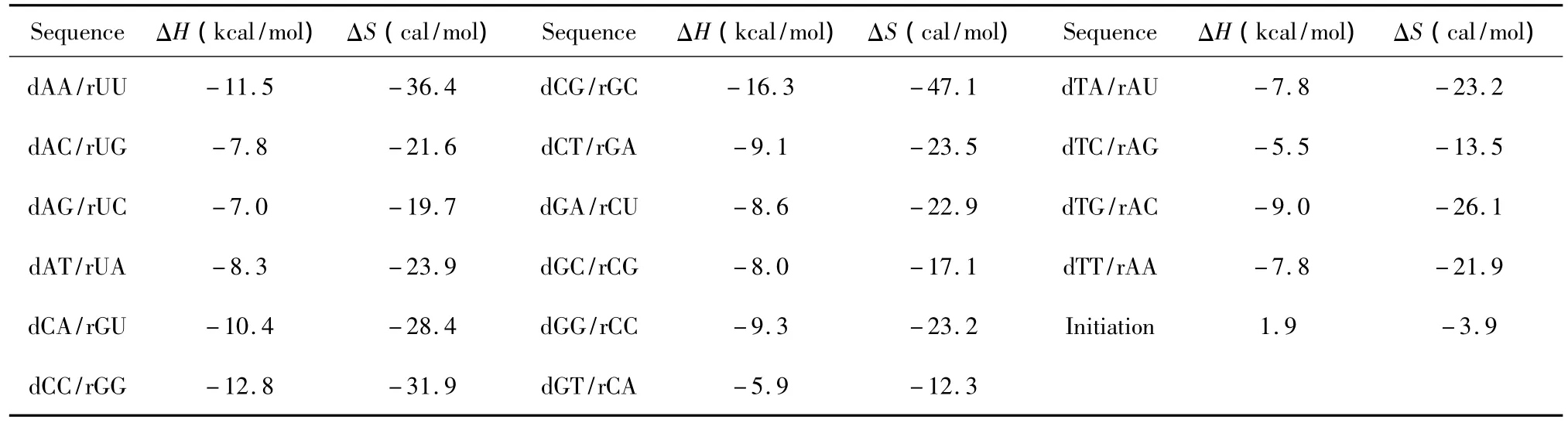
表9 DNA/RNA结合完美匹配最近邻热力学参数(1 mol/L,37℃)Table 9 Thermodynamic parameters for DNA/RNA perfect matches(1 mol/L,37 ℃)
3 RNA与RNA相互作用的热力学参数
3.1 完美匹配
当RNA与RNA相互作用时,完美匹配的最近邻热力学参数表[19]由Xia等人于1998年提出(见表10)。当mRNA与RNA相互作用时,完美匹配的最近邻热力学参数表[20]由Turner等人于2006年提出(见表11)。完美匹配结构的热力学计算同1.1。
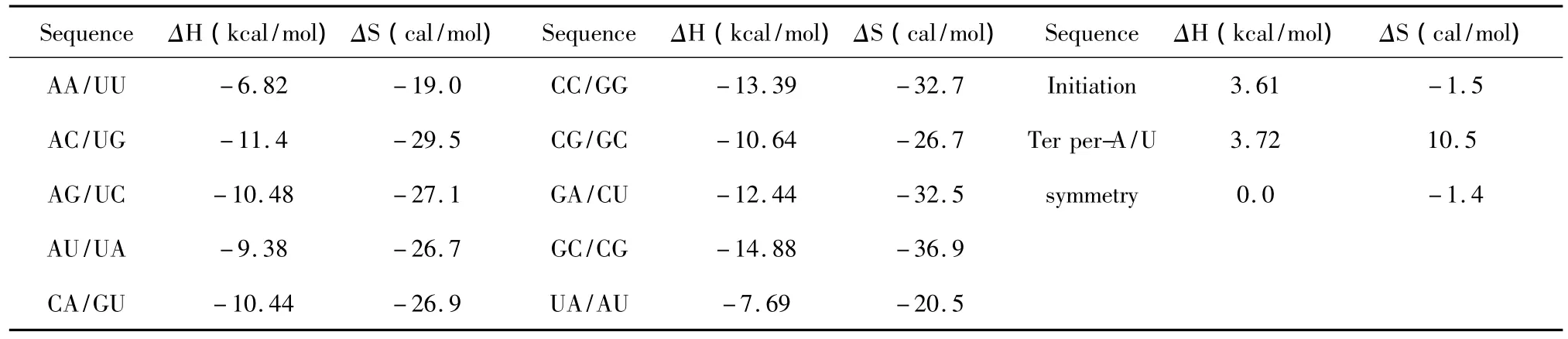
表10 RNA/RNA结合完美匹配最近邻热力学参数表(1 mol/L,37℃)Table 10 Thermodynamic parameters for RNA/RNA perfect matches(1 mol/L,37 ℃)
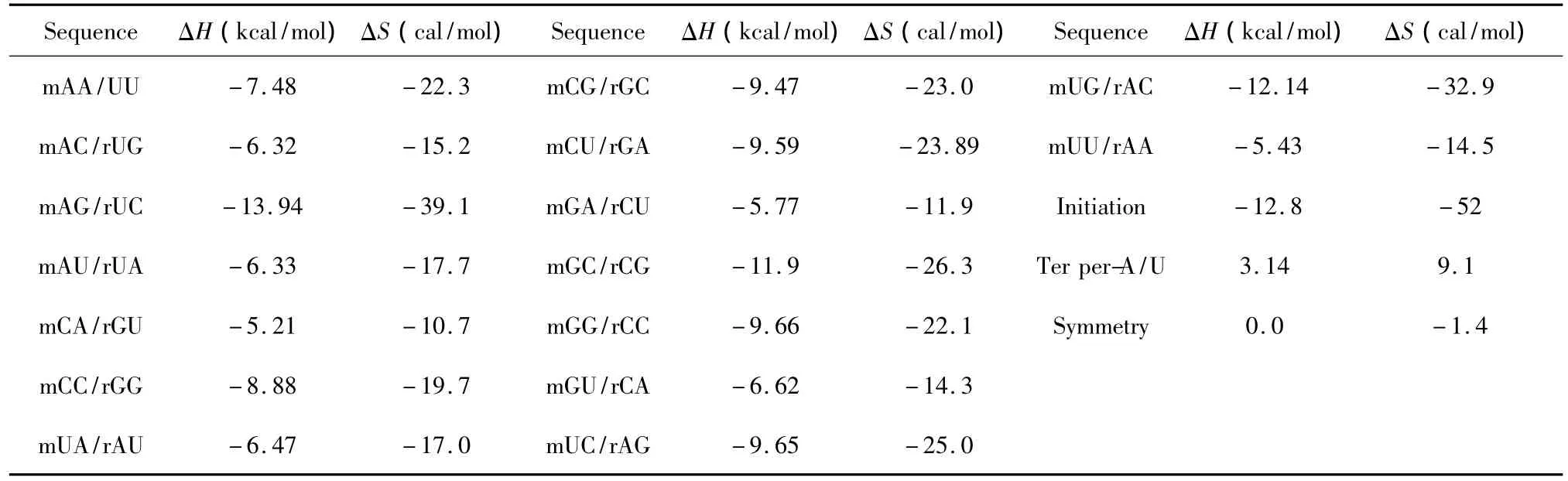
表11 mRNA/RNA结合完美匹配最近邻热力学参数表(1 mol/L,37℃)Table 11 Thermodynamic parameters for mRNA/RNA perfect matches(1 mol/L,37 ℃)
3.2 错配
当出现单独错配的模式时,应用Znosko等人于2008年提供的热力学数据[21](见表12),其中R表示嘌呤碱基,Y表示嘧啶碱基;单独错配结构的热力学计算如下:
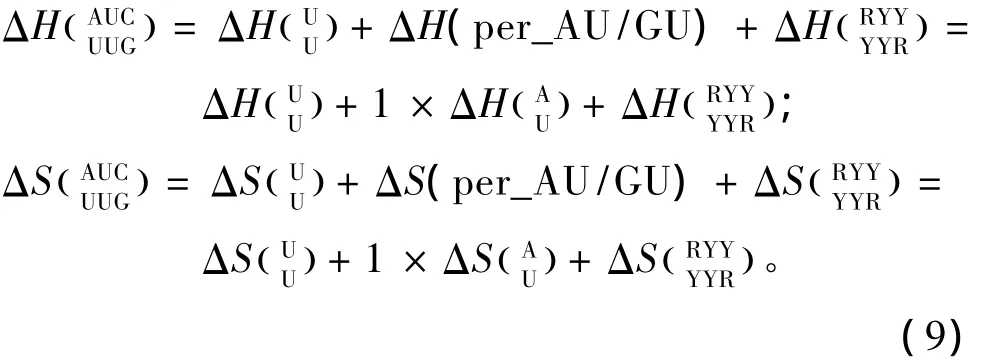
当出现连续错配的模式时,应用Turner等人于1999和2006年提供的热力学数据[22-23](见表13)。连续错配结构的热力学计算如下:
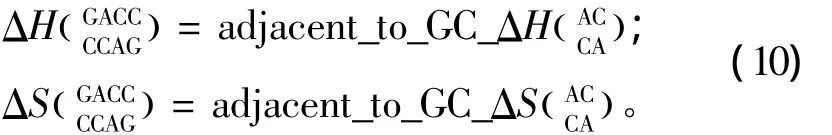
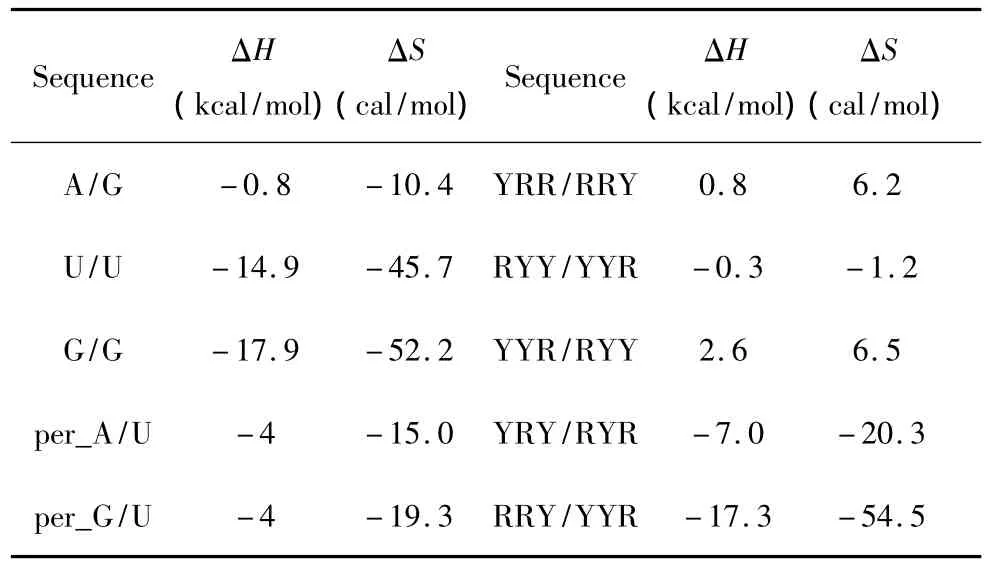
表12 单独错配结构热力学参数(1 mol/L,37℃)Table 12 Thermodynamic parameters for RNA/RNA single mismatch(1 mol/L,37 ℃)
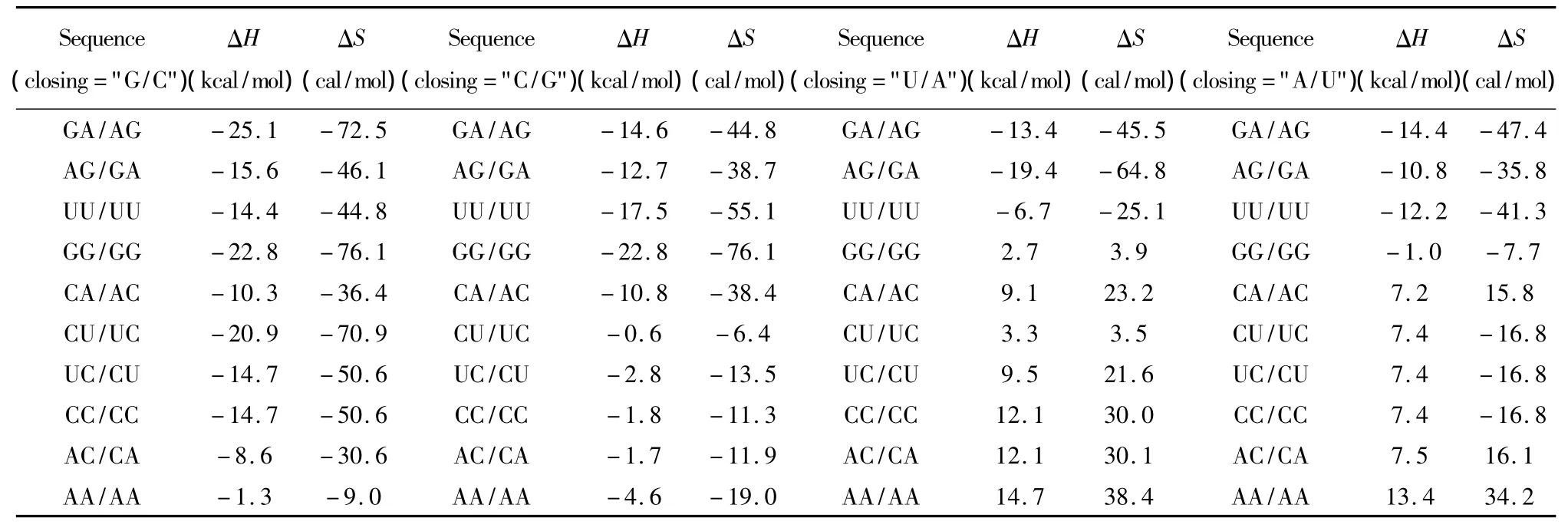
表13 连续错配结构热力学参数表(1 mol/L,37℃)Table 13 Thermodynamic parameters for RNA/RNA tandem mismatches(1 mol/L,37 ℃)
3.3 内环
当出现内环结构,应用Turner等人于1999年和2006年提供的热力学数据[22-23](见表14)。内环结构的热力学计算如下:
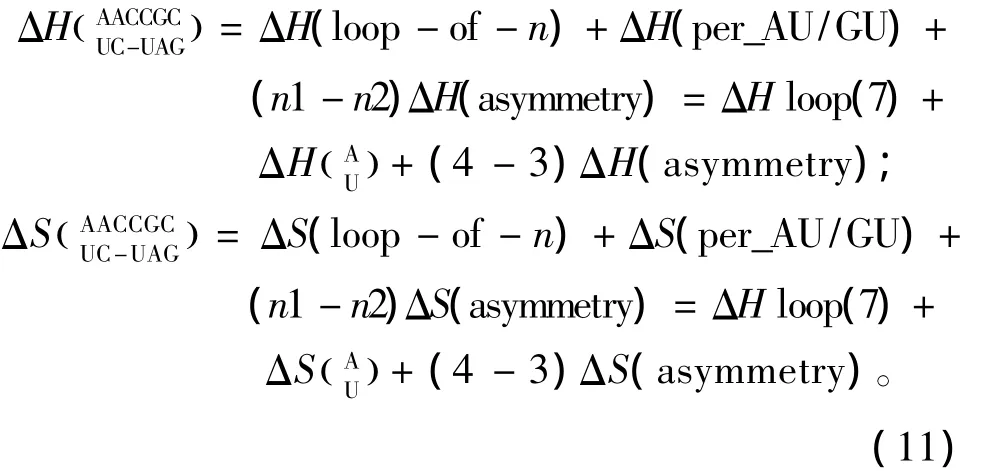
3.4 CNG 重复序列
所谓CNG重复序列,是指一条序列(5’至3’方向)由多个G(CNG)xC的子片段组成的序列,这里x是指CNG重复的数目;N代表一种单独错配类型即N/N。当出现这种模式时,应用Broda等人于2005年提供的热力学数据[24](见表15)。CNG重复结构的热力学计算如下:
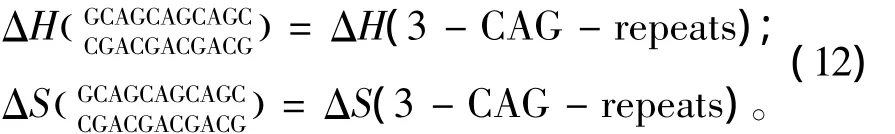
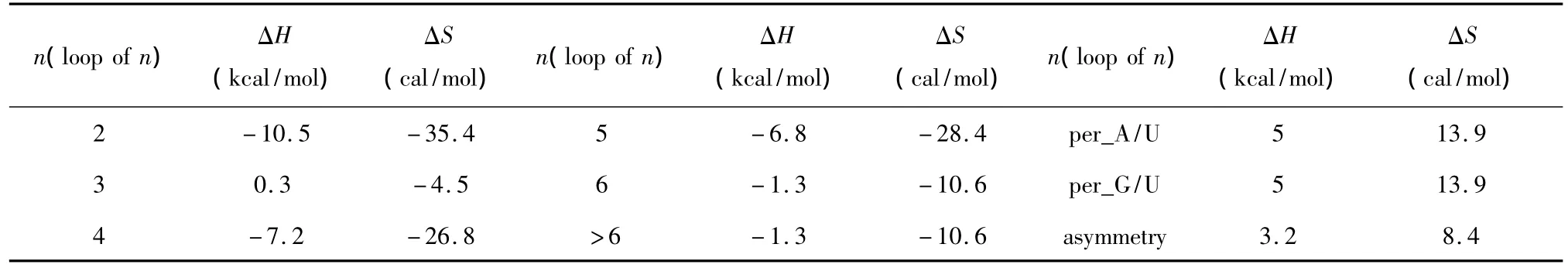
表14 内环结构热力学参数(1 mol/L,37℃)Table 14 Thermodynamic parameters for RNA/RNA internal loop(1 mol/L,37 ℃)
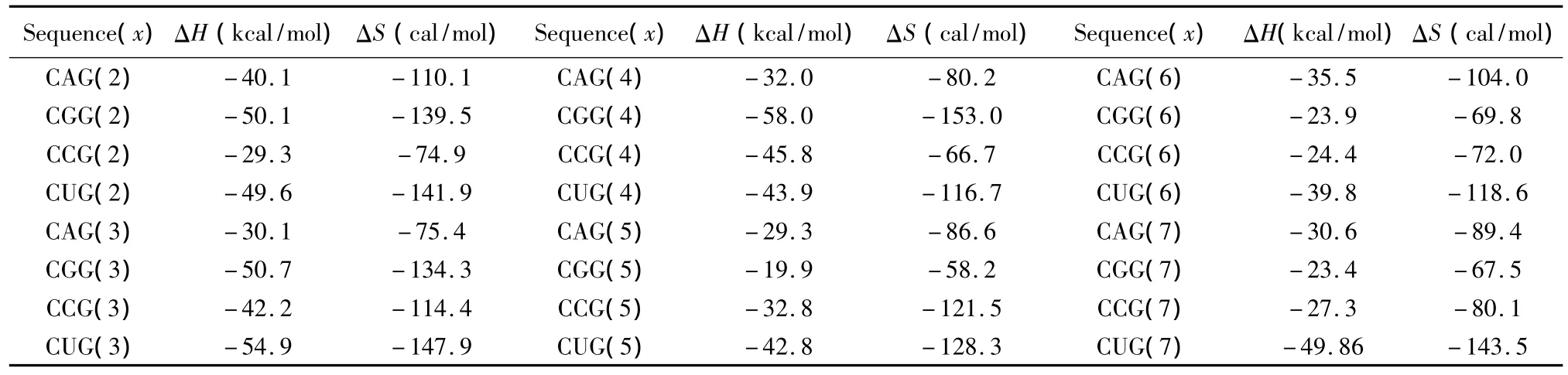
表15 CNG重复结构热力学参数(1 mol/L,37℃)Table 15 Thermodynamic parameters for CNG repeats(1 mol/L,37 ℃)
3.5 GU摆动配对
当RNA/RNA相互作用呈现多个连续的GU配对(非Watson-Crick配对)结构时,应用Turner等人于1999年提供的热力学数据[22](见表16)。GU摆动配对结构的热力学计算如下:
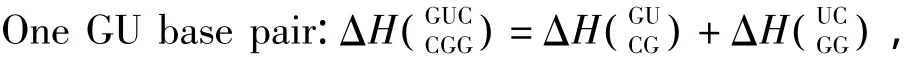
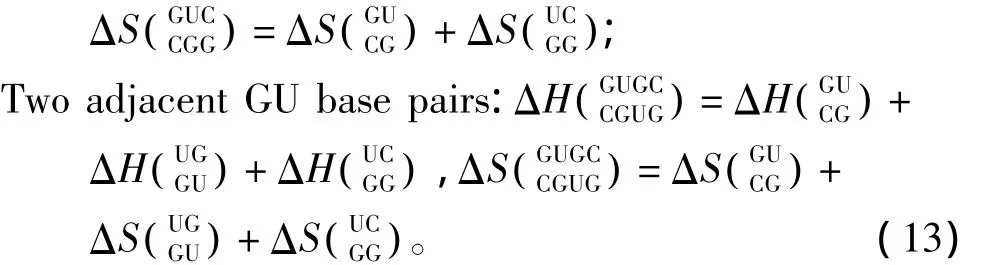
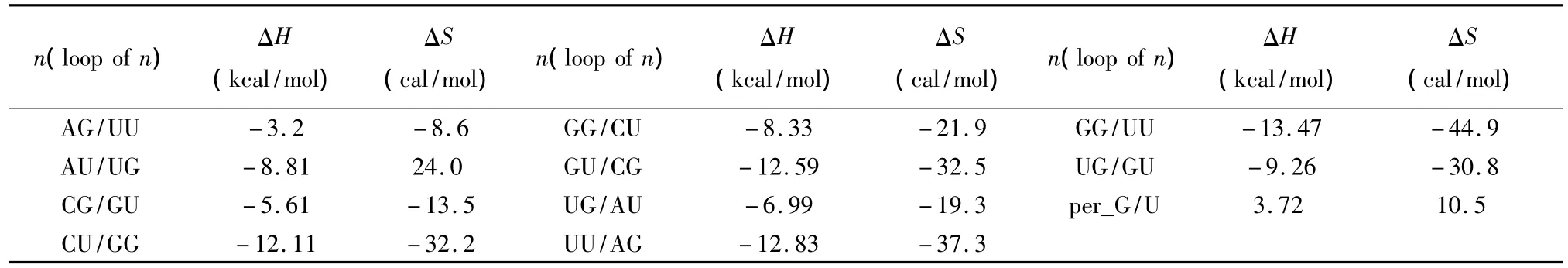
表16 GU摆动配对结构热力学参数(1 mol/L,37℃)Table 16 Thermodynamic parameters for GU wobble base pairs(1 mol/L,37 ℃)
3.6 摇摆
对于Sigle dangling-end模式,应用Serra等人于2006和2008年提供的热力学数据[25-26](见表17);对于Long dangling-end模式,应用Sugimoto与Serra等人于2002和2006年提供的热力学数据[16,25](见表18),其中 R表示嘌呤碱基,Y表示嘧啶碱基。Dangling-end结构的热力学计算同1.5与1.6。
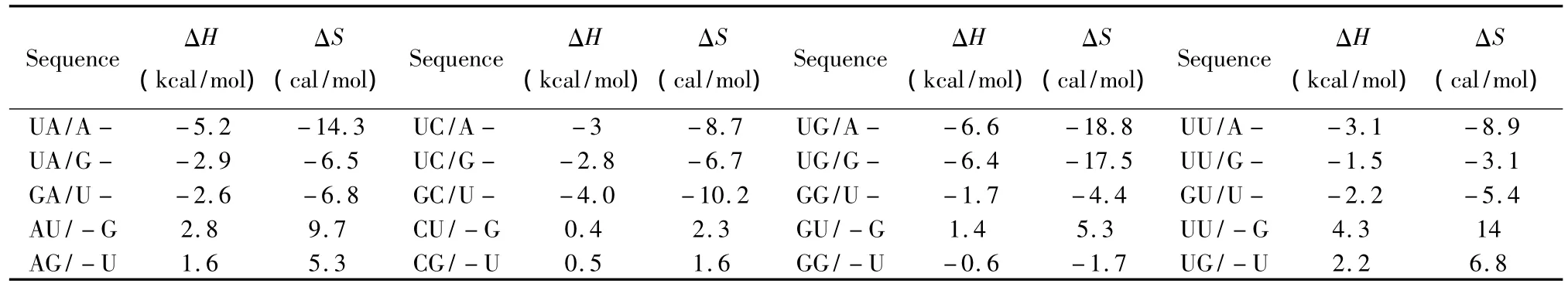
表17 Single dangling-end结构热力学参数(1 mol/L,37℃)Table 17 Thermodynamic parameters forRNA/RNA single dangling-end(1 mol/L,37℃)

表18 Long dangling-end结构热力学参数(1 mol/L,37℃)Table 18 Thermodynamic parameters forRNA/RNA long dangling-end(1 mol/L,37 ℃)
3.7 膨胀环
对于Single bulge loop模式,应用Serra等人于2007年提供的热力学数据[27](见表 19),Single bulge loop结构的热力学计算同1.7;对于Long bulge loop结构应用Turner等人于1999和2006年提供的热力学数据[22-23](见表 20),Long bulge loop 结构的热力学计算如下:
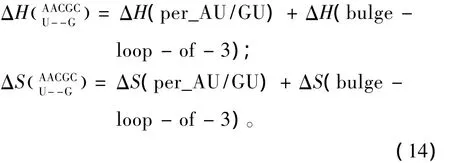
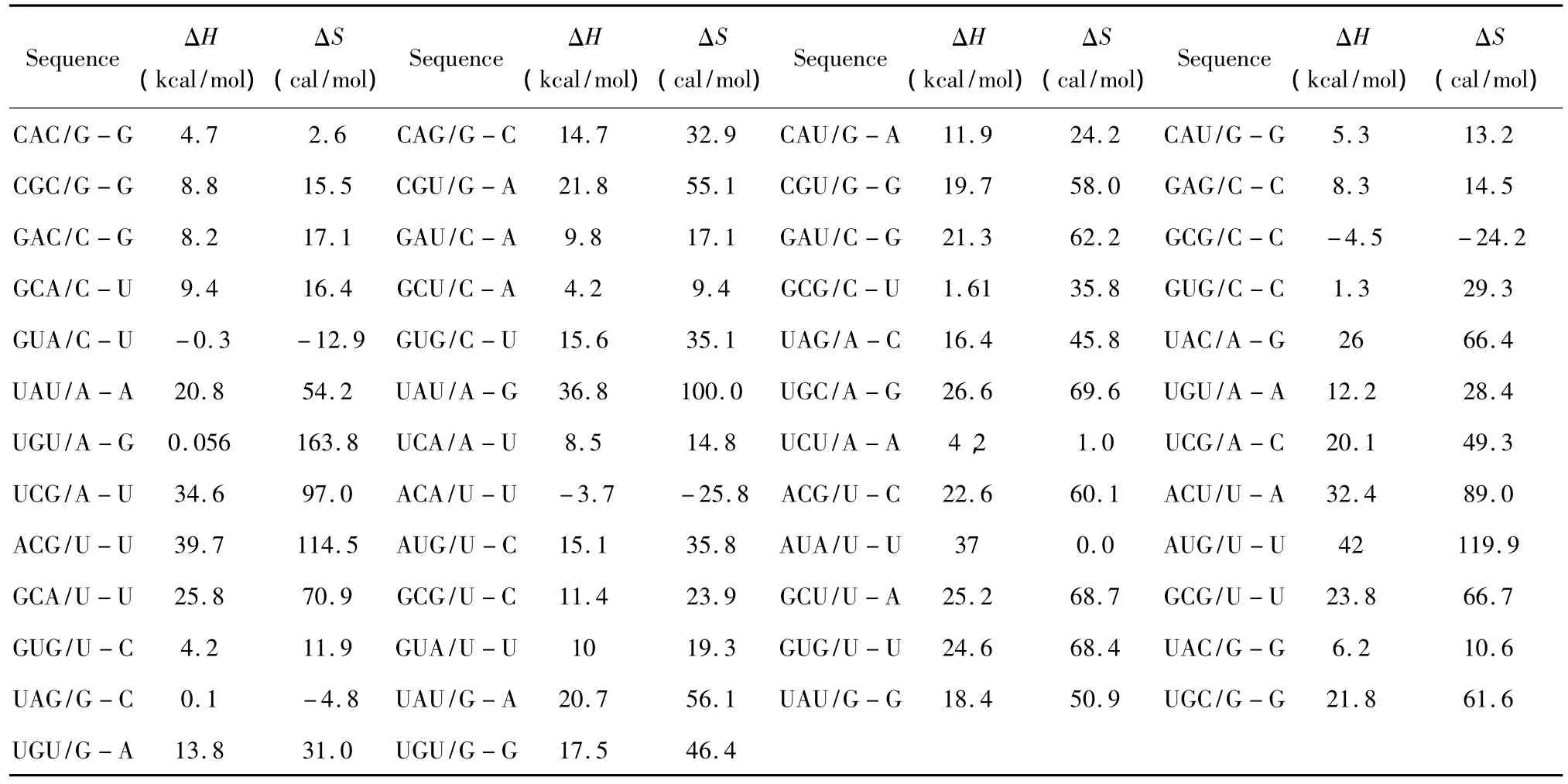
表19 Single bulge-end结构热力学参数(1 mol/L,37℃)Table 19 Thermodynamic parameters for RNA/RNA single bulge-end(1 mol/L,37 ℃)
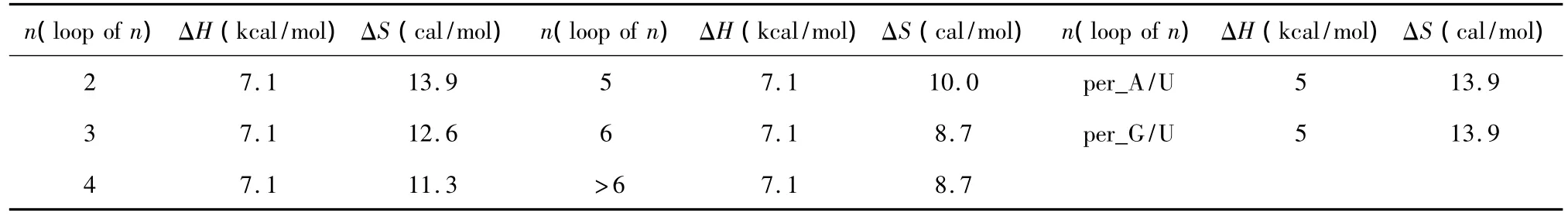
表20 Long bulge-end结构热力学参数(1 mol/L,37℃)Table 20 Thermodynamic parameters forRNA/RNA long bulge-end(1 mol/L,37 ℃)
4 特殊碱基的热力学参数
除上述ATCGU之外,生物体中同样存在一些非正常的碱基如肌苷(Inosine base,I)、羟基腺嘌呤(2_hydroxyadenine pairs,A*)等。目前发现肌苷在RNA/RNA相互作用中以IU匹配的形式出现,相关的热力学数据由Znosko等人于2007年提供[28](见表21);肌苷在DNA/DNA相互作用中的热力学数据由 Santalucia等人于2005年提供(见表22)[29],热力学计算如下:
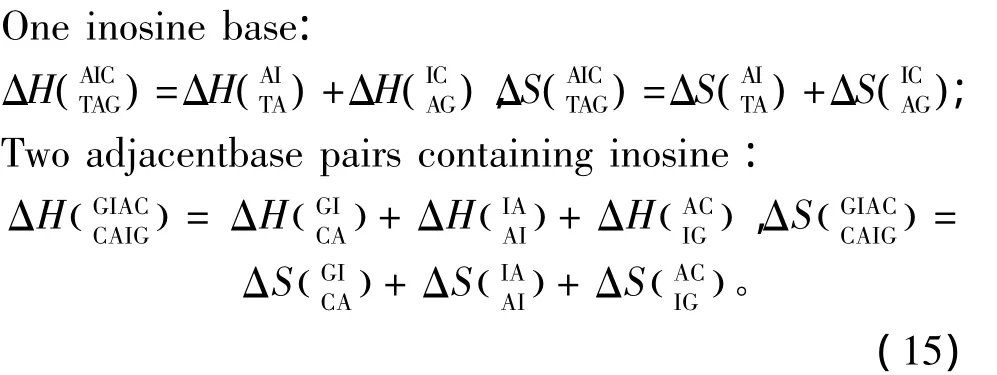
羟基腺嘌呤(A*)的热力学参数由Sugimoto等人于2001年提供[30],通常 A*只在5’-GA*C -3’以及5’-TA*A-3’序列中出现(见表23)。热力学计算如下:
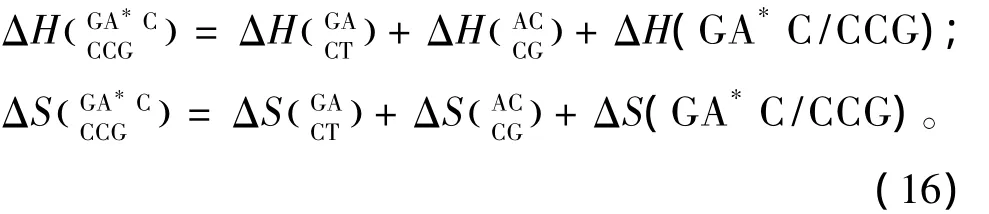
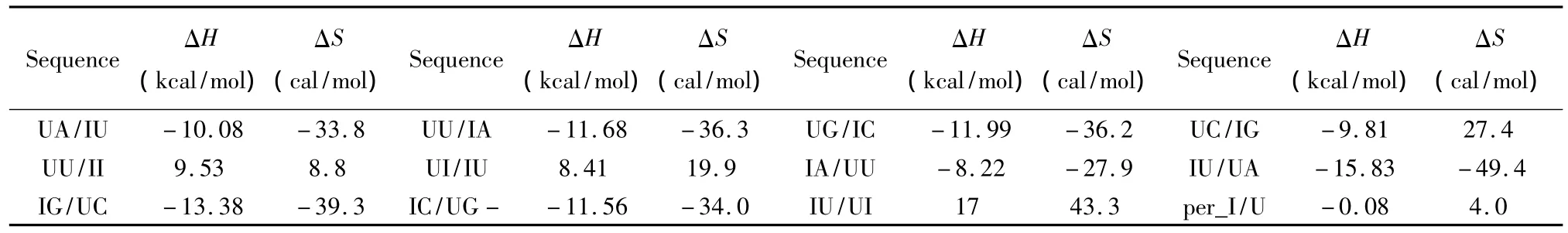
表21 RNA/RNA结合含有I碱基的热力学参数(1 mol/L,37℃)Table 21 Thermodynamic parameters for RNA/RNA inosine base(1 mol/L,37 ℃)
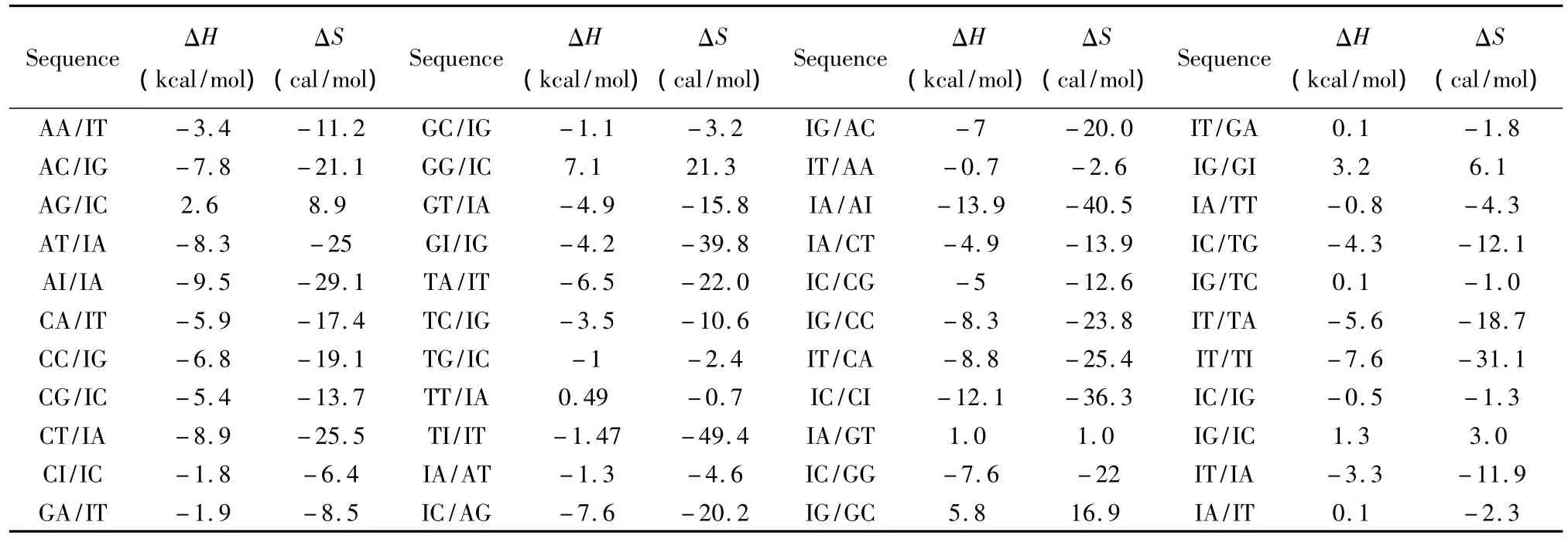
表22 DNA/DNA结合含有I碱基的热力学参数(1 mol/L,37℃)Table 22 Thermodynamic parameters for DNA/DNA inosine base(1 mol/L,37 ℃)
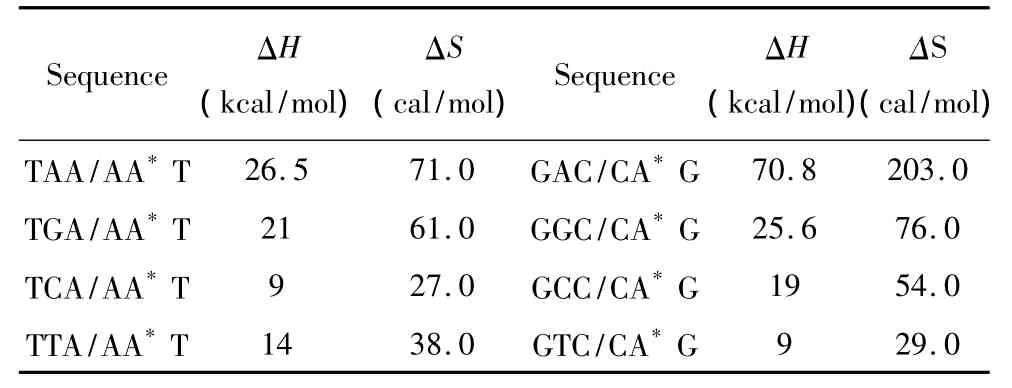
表23 DNA/DNA结合含有A*结构的热力学参数(1 mol/L,37℃)Table 23 Thermodynamic parameters for DNA/DNA A*(1 mol/L,37 ℃)
5 结语与展望
寡核苷酸二级结构的精确预测在分子生物学应用中发挥越来越重要的作用,热力学参数的不断完善使其精确性得到逐步提高。本文针对不同二级结构,综述目前可利用的有效热力学数据库,涵盖完美匹配、错配、内环、膨胀环、末端摇摆、CNG重复、GU摆动配对以及包含肌苷和羟基腺嘌呤等结构。基于本文的数据,用户可以根据不同需求选择合适的数据集,开发自有或者改进现有的寡核苷酸二级结构的预测算法,从而提高以寡核苷酸杂交为基础的分子生物技术的成功率。
然而,生物学过程是复杂的,许多基于核酸杂交的生物技术实验中包含的酶,环境的pH值,Ca2+、Mg2+等离子以及一些变性剂如甲酰胺(formamide)、DMSO(二甲基亚砜)都会显著影响寡核苷酸二级结构的评估[14];并且,除 A、T、C、G、U 正常碱基以及少数报道的特殊碱基外,其他生物分子如经甲基化或者乙酰化修饰后的核苷酸也大量存在于生物体中。因此,未来有必要通过实验补充新的热力学参数,使现有的热力学数据得到充实和完善,以进一步提高热力学计算的精确性,从而促进寡核苷酸二级结构的准确预测。
References)
[1] FURUICHI T,INOUYE S,INOUYE M.Biosynthesis and structure of stable branched RNA covalently linked to the 5'end of multicopy single-stranded DNA of Stigmatella aurantiaca[J].Cell,1987,48(1):55 - 62.
[2] GACY A M,GOELLNER G,JURANIC N,et al.Trinucleotide repeats that expand in human disease form hairpin structures in vitro[J].Cell,1995,81(4):533 - 540.
[3] HIGASHITANI N,HIGASHITANI A,HORIUCHI K.Nucleotide sequence of the primer RNA for DNA replication of filamentous bacteriophages[J].J Virol,1993,67(4):2175-2181.
[4] MOROZOV S,CHERNOV B K,MERITS A,et al.Computer-assisted predictions of the secondary structure in the plant virus single-stranded DNA genome[J].J Biomol Struct Dyn,1994,11(4):837-847.
[5] CROTHERS D M ,ZIMM B H.Theory of the melting transition of synthetic polynucleotides:Evaluation of the stacking free energy[J].J Mol Biol,1964,9:1 -9.
[6] SCHILDKRAUT C.Dependence of the melting temperature of DNA on salt concentration[J].Biopolymers,1965,3(2):195-208.
[7] 张艳春,屈武斌,卢一鸣,等.DNA解链温度(Tm)不同预测方法的比较[J].军事医学,2011(03):231-235.ZHANG Yanchun,QU Wubin,LU Yiming,et al.A Comparison of different melting temperature(Tm)prediction methods for DNA[J].Military Medical Sciences,2011,35(3):231-235.
[8] SANTALUCIA J,JR.A unified view of polymer,dumbbell,and oligonucleotide DNA nearest-neighbor thermodynamics[J].Proc Natl Acad Sci U S A,1998,95(4):1460-1465.
[9] ALLAWI H T,SANTALUCIA J,JR.Thermodynamics and NMR of internal G.T mismatches in DNA[J].Biochemistry,1997,36(34):10581-10594.
[10] ALLAWI H T,SANTALUCIA J,JR.Nearest neighbor thermodynamic parameters for internal G.A mismatches in DNA[J].Biochemistry,1998,37(8):2170 -2179.
[11] ALLAWI H T,SANTALUCIA J,JR.Thermodynamics of internal C.T mismatches in DNA[J].Nucleic Acids Res,1998,26(11):2694-2701.
[12] ALLAWI H T,SANTALUCIA J,JR.Nearest-neighbor thermodynamics of internal A.C mismatches in DNA:sequence dependence and pH effects[J].Biochemistry,1998,37(26):9435-9444.
[13] PEYRET N,SENEVIRATNE P A,ALLAWI H T,et al.Nearest-neighbor thermodynamics and NMR of DNA sequences with internal A.A,C.C,G.G,and T.T mismatches[J].Biochemistry,1999,38(12):3468 -3477.
[14] SANTALUCIA J,JR,HICKS D.The thermodynamics of DNA structural motifs[J].Annu Rev Biophys Biomol Struct,2004,33:415 -440.
[15] BOMMARITO S,PEYRET N,SANTALUCIA J,et al.Thermodynamic parameters for DNA sequences with dangling ends[J].Nucleic Acids Res,2000,28(9):1929 -1934.
[16] OHMICHI T,NAKANO S,MIYOSHI D,et al.Long RNA dangling end has large energetic contribution to duplex stability[J].J Am Chem Soc,2002,124(35):10367-10372.
[17] TANAKA F,KAMEDA A,YAMAMOTO M,et al.Thermodynamic parameters based on a nearest-neighbor model for DNA sequences with a single-bulge loop[J].Biochemistry,2004,43(22):7143-7150.
[18] SUGIMOTO N,NAKANO S,KATOH M,et al.Thermodynamic parameters to predict stability of RNA/DNA hybrid duplexes[J].Biochemistry,1995,34(35):11211 -11216.
[19] XIA T,SANTALUCIA J,JR,et al.Thermodynamic parameters for an expanded nearest-neighbor model for formation of RNA duplexes with Watson-Crick base pairs[J].Biochemistry,1998,37(42):14719 -14735.
[20] KIERZEK E,MATHEWS D H,CIESIELSKA A,et al.Nearest neighbor parameters for Watson-Crick complementary heteroduplexes formed between 2'-O-methyl RNA and RNA oligonucleotides[J].Nucleic Acids Res,2006,34(13):3609-3614.
[21] DAVIS A R,ZNOSKO B M.Thermodynamic characterization of naturally occurring RNA single mismatches with G - U nearest neighbors[J].Biochemistry,2008,47(38):10178-10187.
[22] MATHEWS D H,SABINA J,ZUKER M,et al.Expanded sequence dependence of thermodynamic parameters improves prediction of RNA secondary structure[J].J Mol Biol,1999,288(5):911 -940.
[23]LU Z J,TUMER D H,MATHEWS D H.A set of nearest neighbor parameters for predicting the enthalpy change of RNA secondary structure formation[J].Nucleic Acids Res,2006,34(17):4912 -4924.
[24] BRODA M,KIERZEK E,GDANIEC Z,et al.Thermodynamic stability of RNA structures formed by CNG trinucleotide repeats.Implication for prediction of RNA structure[J].Biochemistry,2005,44(32):10873-10882.
[25] O'TOOLE A S,MILLER S,HAINES N,et al.Comprehensive thermodynamic analysis of 3'double-nucleotide overhangs neighboring Watson-Crick terminal base pairs[J].Nucleic Acids Res,2006,34(11):3338 -3344.
[26] MILLER S,JONES L E,GIOVANNITTI K,et al.Thermodynamic analysis of 5'and 3'single-and 3'double-nucleotide overhangs neighboring wobble terminal base pairs[J].Nucleic Acids Res,2008,36(17):5652 -5659.
[27] BLOSE J M,MANNI M L,KLAPEC K A,et al.Nonnearest-neighbor dependence of the stability for RNA bulge loops based on the complete set of group I single-nucleotide bulge loops[J].Biochemistry,2007,46(51):15123-15135.
[28] WRIGHT D J,RICE J L,YANKER D M,et al.Nearest neighbor parameters for inosine x uridine pairs in RNA duplexes[J].Biochemistry,2007,46(15):4625 -4634.
[29] WATKINS N E,JR,SANTALUCIA J,et al.Nearest-neighbor thermodynamics of deoxyinosine pairs in DNA duplexes[J].Nucleic Acids Res,2005,33(19):6258 -6267.
[30] KAWAKAMI J,KAMIYA H,YASUDA K,et al.Thermodynamic stability of base pairs between 2-hydroxyadenine and incoming nucleotides as a determinant of nucleotide incorporation specificity during replication[J].Nucleic Acids Res,2001,29(16):3289 -3296.
