两种模式生物核小体定位比较研究
2014-11-14丰继华单秋甫陈攀峰单增辉
卢 英,丰继华,单秋甫,陈攀峰,单增辉
(云南民族大学电气信息工程学院,云南昆明650500)
采用模式生物进行研究的理论基础源自于物种间基本生命过程的保守性,即从一个生物得到的知识可能外推到包括人在内的另外一个生物上。例如单细胞生物可以用来研究基本的细胞生化过程,避免了研究多细胞生物体内组织和器官分化所带来的复杂性。但是,采用同样的方式却不能用单细胞生物的代谢来解释控制多细胞生物所需要的每一个过程。每种生物都具有其独特性。因此,对过度外推模式生物得到的结论必须保持谨慎。具体选择哪种模式生物更好,需要根据具体情况而定。
每个模式生物各有利弊。为此,有必要在两个以上不同模式生物中做平行实验。为了全面揭示真核生物核小体的定位特点,我们分别选取一个单细胞生物和一个多细胞生物作为研究对象。原因在于:(1)酵母作为单细胞生物,其基因组较小,核小体的定位特征相对简单,对其染色质结构的研究及认识也较为清楚[1-2],能得到相对准确而清晰的数据结构和图谱。(2)果蝇胚胎期是一个由单细胞到多细胞的分化过渡的阶段,研究这一时期的染色质结构,相对于直接研究分化后的果蝇成体困难要小得多。且与单细胞酵母细胞相比,果蝇胚胎期又具有某些单细胞生物的特点,因而在酵母染色质研究中获得的知识,可用于对果蝇的研究,二者具有可比性。(3)以往研究已经通过实验获得了酵母与果蝇胚胎期的核小体占位率数据和H2AZ核小体定位数据[3],因此研究具有可行性。
通过对现有实验数据和实验条件进行综合考察后,本文最终选取了在遗传研究上最具代表意义的两个模式生物:酿酒酵母(S.cerevisiae)和黑腹果蝇(D.melanogaster)[8]作为研究对象,希望通过比较分析这两种生物的核小体定位数据[9],得出有生物学意义的结论。对今后推广到其他物种的核小体定位模式的研究有着极为重要的现实意义和实际应用价值。事实也证明,通过对酵母和果蝇的相关基因组核小体的图谱分析,我们发现了两者的基因内染色体组织架构具有显著的共同点与差异性。
1 实验方法
1.1 数据准备
本研究数据主要来自两个部分:第一、酵母核小体定位实验数据,包括Lee等人研究中给出的高分辨率核小体占位率实验数据[2],来自NCBI数据库中的酵母16条染色体DNA编码序列、David等人文献中给出的4 792个高置信度酵母基因组实验数据[1],以及Albert等人实验得到的 H2A.Z核小体位置数据[4]。第二部分果蝇的核小体定位的实验数据,包括Mavrich等人在实验中得到的果蝇胚胎期H2AZ核小体定位数据、总体核小体(bulk nucleosome)占位率数据[5],以及 Pavel Tomancak 等人对果蝇胚胎期不同表达模式基因的分类数据[6]。由于以上核小体定位实验数据来自不同实验平台,具有异构性,我们根据研究目的利用信号处理方法对与本研究有关的数据进行了重构。
1.2 数据处理
1.2.1 核小体定位实验数据的处理
(1)高分辨率核小体占位率实验数据的处理
由于Lee等人提供的酵母高分辨率核小体占位率实验数据来自每隔4 bp采样的全基因组微阵列平台 (an Affymetrix tiling microarray)[2],而由 Travis N.Mavric等人提供的果蝇实验数据却是每隔36 bp采样的全基因组微阵列数据[5]。在此,我们通过插值方法得到一个覆盖全基因组每1 bp的核小体占位率数据,以供后续对齐使用。通过三次样条插值(spline),我们得到了精度较高,平滑度较好的包括了酵母16条染色体的实验预备数据。由于果蝇数据的间隔较大(36 bp),因此获得的果蝇6条染色体的实验预备数据精度相对于酵母要低一些。
(2)H2AZ核小体定位数据的处理
由于Albert等人提供的酵母H2A.Z核小体定位原始实验数据和由Travis N.Mavric等人同时提供了果蝇胚胎期基因组的H2A.Z核小体定位原始实验数据,都包括了H2A.Z核小体的起始位点、终止位点以及一些核小体的物理性质数据。正是因为其中核小体的位置以及与其定位强度有关的物理性质恰好符合高斯分布的特性,我们认为具有良好的数理统计规律特性的高斯分布能很好的应用在核小体的定位研究上。我们利用高斯分布图形对核小体定位数据进行重建,这高斯分布公式如下:
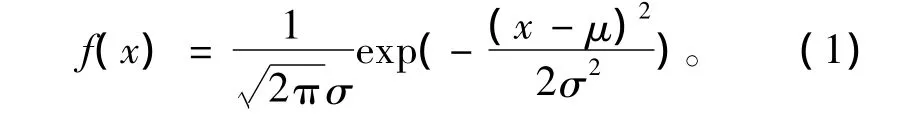
在每一个核小体的位置上构造一个高斯分布图形(见图1),期望值取核小体定位数据的中点,并且基于高斯分布中的“2σ原则”,我们将标准差取为核小体长度的1/4,以最大限度的减少误差。最后,利用实验数据中的峰值数据(peakhigh)作为这个核小体高斯分布的中点值,peakhigh是表示该核小体存在于这个位置的可能性大小的物理性质。
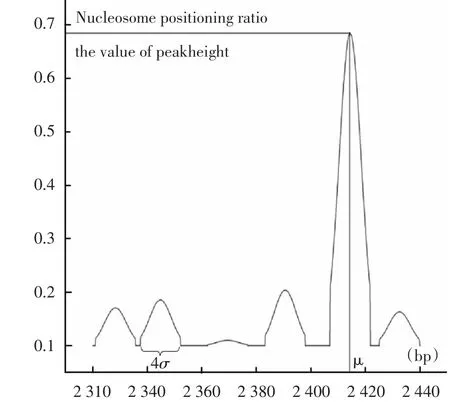
图1 核小体定位的正态分布重构示意图Fig.1 Normal distribution reconstruction graph of the Nucleosome positioning
1.2.2 基因数据的重构
由Pavel Tomancak等人提供的果蝇原始实验数据包括了不同表达模式型的基因的名称数据[6],结合Travis N.Mavric等人提供的基因位置数据[5],分离出不同类表达模式基因的位置,包括广泛表达型(broad)的2 627条基因、限制性表达型(restrict)的2 085条基因、沉默型(silent)的8 941条基因。
1.2.3 基于中心对齐的基因排序
通过前期处理和筛选,得到了酵母和果蝇全体基因组(所有基因)、果蝇广泛表达基因组(胚胎期所有时期均被翻译)、果蝇限制性表达基因组(胚胎期部分时期被翻译,部分时期不被翻译)、果蝇沉默型基因组(胚胎期不被翻译)中的基因位置数据。这些组数据包括基因所在染色体上的位置,基因的起始位点和终止位点,基因正转录(W型)和反转录(C型)情况。
为了得到以上每组基因的一个二维绘图(hot map)。首先分别将每组基因按照基因长度从小到大依次递增的原则进行重排列,并提取每条基因的中心点,这样我们得到各组由短基因到长基因排列的基因位置数据。其次将此类位置数据分别映射到酵母和果蝇的全核小体占位率数据与H2A.Z核小体的定位数据中。在各条染色体上找到每条基因的中心点,并在每个基因的中心点周围,截取上、下游各Nbp的长度范围的核小体占位率数据或在该范围内含H2A.Z核小体的定位数据,取N的原则为各组基因中20%的最长基因的平均值。最后对截取的数据进行中心对齐(其中C型基因对应的数据要进过反向处理)排列,这样就得到了每组基因中心对齐的核小体的定位数据。将对齐后的数据进行二维绘图(hot map),由此得到了各组基因中心对齐的核小体的定位图谱。
1.2.4 图谱
这里用hot map绘图法绘出以下多组二维绘图(hot map),展示了一个明显的、有组织的核小体排列序列。我们从不同层次、从外到内、从顶端到低端、从最短基因到最长基因的去分析,有组织的核小体的剖面图被生动形象的展现出来。
从图2A中很明显的观测到酵母中基因5’端处的边界显示强度明显比基因3’端边界显示强度大,这一点与Cedric Vaillant等人的关于酵母全体基因上发现的规律一致,即在核小体缺失区域(NFR)的基因3’端的存在着5个核小体影响着基因,在核小体缺失区域(NFR)基因5’端则存在着7个核小体的影响着基因[7]。然而,图2B中所显示出来的果蝇全体基因的中心点周围H2A.Z核小体分布图中,基因5’端的边界却与基因3’端的边界显示出轮廓对称,且显示强度也一致,这一点与酵母中观察到的完全不同。在酵母中,一般认为H2A.Z核小体与基因的转录相关,并且倾向于定位在具有转录活性的基因5'端。但是在果蝇胚胎中看到的情况却是;基因5'端核小体缺失区域(NFR)与基因3’端核小体缺失区域(NFR)两侧具有同样数目或至少是数目相差不H2AZ核小体,且边界上的核小体数量也远远超过酵母。
这种从单细胞生物到多细胞生物间的差异性,体现了生物界中的进化特性。从果蝇胚胎的H2A.Z核小体分布看,单细胞生物和多细胞生物有着显著差别。原因在于当果蝇胚胎处于不同发育阶段时,一些基因必须严格按发育程序保持的沉默,否则对幼虫可能是致命的。这就需要果蝇进化出比单细胞生物酵母更为复杂的染色体调控机制。因此,H2A.Z核小体在酵母中的作用可能在果蝇中只得到了部分保留。
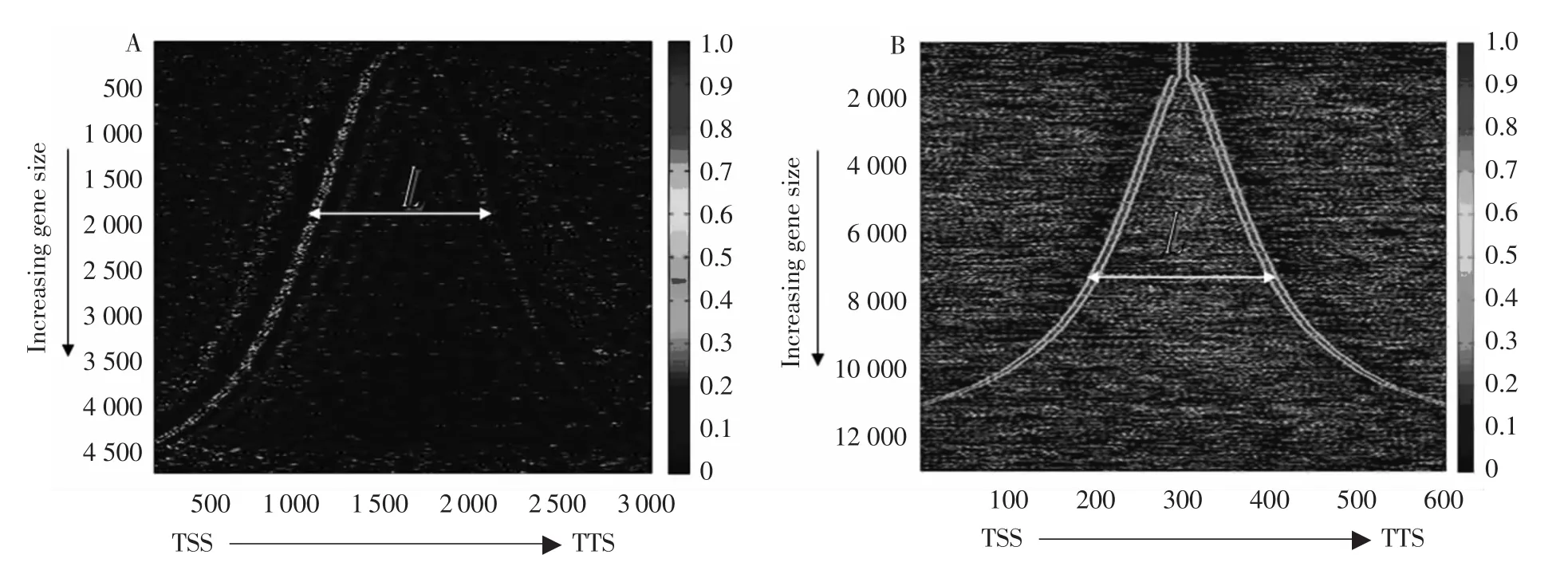
图2 酵母与果蝇全基因中心点周围H2A.Z核小体分布Fig.2 H2A.Z nucleosome distribution around the center point of genes in Yeast and Drosophila
图3为酵母和果蝇果蝇胚胎期沉默型基因中心点周围H2A.Z核小体分布图,这里选取果蝇果蝇胚胎期沉默型基因组图是因为它表现出的特征是最明显的。从图3A中可以发现,当L<1.5 kbp时,在核小体缺失区域的两个边界处呈现出明显的周期性包装,并且出现了一个定义明确有着均匀间隔的核小体数n。随着L的增加,这种晶体状的基因簇在基因区域呈现出与核小体相同的数目n(2<n<10)。对于L>1.5 kbp的基因,周期性核小体的位置是可见的,但因受两边界的诱导,显示周期性规律的中心点处核小体位置所需的强力限制性减弱,从而使周期性出现比较模糊现象。从图3B果蝇中同样可以发现酵母中发现的规律,但是较酵母所表现的出来的更模糊更复杂的图像。

图3 酵母全基因和果蝇胚胎期沉默型基因中心点周围H2A.Z核小体分布Fig.3 H2A.Z nucleosome distribution around the center point of genes in silent gene of Yeast and Drosophila embryo
图4为酵母和果蝇果蝇胚胎期沉默型基因中心点周围核小体占位率分布,从图4A中也发现了前面两组A图中发现的两个核小体排列特征,并且此图更为清晰明确。而图4B中却只能看到一个两个核小体确实区域所组成的轮廓。原因之一可能是酵母高分辨率核小体占位率实验数据来自每隔4 bp采样的全基因组微阵列平台,而果蝇实验数据却是每隔36 bp采样的全基因组微阵列平台,其精度相差太大。原因之二可能就是果蝇较酵母是更高级的生物,其基因组织结构也更复杂,这也解释了前一组图组果蝇的周期性比酵母模糊的疑问。
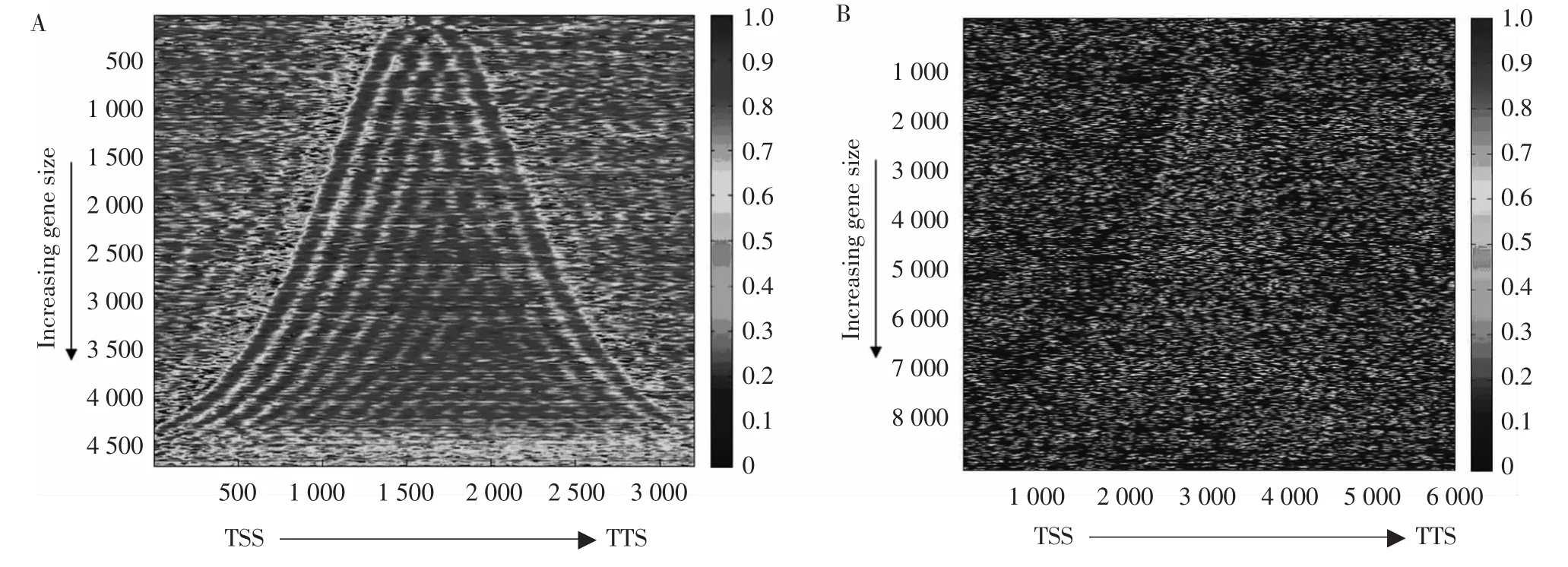
图4 酵母和果蝇胚胎期沉默型全基因中心点周围全部核小体占位率分布Fig.4 The occupancy rate distribution of all nucleosome around the center point of genes in yeast and silent genes in Drosophila
2 结论
本文从高分辨率核小体占位率实验数据和H2AZ核小体定位数据分别去研究酵母和果蝇胚胎期核小体定位及染色质结构,发现不同物种和同一物种中不同表达模式的基因与全体基因上,核小体分布会呈现出不同的特征。酵母全体基因中核小体对NFR的基因3’端影响较NFR基因5’端影响小,果蝇的全体基因中核小体对NFR的基因3’端影响较NFR基因5’端影响却是一样的。酵母与果蝇胚胎期沉默型基因的核小体缺失区域的两个边界中间处都出现了一个明确有着均匀间隔的核小体数n,且随着基因长度L的变长其周期性特性逐渐变模糊,只是果蝇相对来说比酵母的更为复杂和模糊。
上面所述的差异性体现了在‘硬’遗传和‘软’遗传方面,两个物种基因组进化的不同。在‘软’遗传方面,发现了核小体在两个物种启动子和终止子区的核小体缺失区域在范围上的差异性;在‘硬’遗传方面,揭示了两个物种与‘硬’遗传上所呈现的相关的核小体数目与位置上的进化差异。结果表明,从单细胞酵母生物到多细胞果蝇生物间基因组的进化过程中,核小体组织的演化既有变异性,也具有保守性。
References)
[1] LIOR D,WOLFGANG H,GRANOVSKAIA M,et al.A high-resolution map of transcription in the yeast genome[J].Proc Natl Acad Sci U S A,2006,103(14):5320-5325.
[2] WILLIAM L,DESIREE T,NICOLAS B,et al.A high -resolution atlas of nucleosome occupancy in yeast[J].Nat Genet,2007,39(10):1235 -1244.
[3] PECKHAM H E, THURMAN R, FU Y,et al.Nucleosome positioning signals in genomic DNA[J].Genome Res,2007,17(8):1170 -1177.
[4] ALBERT I,MAVRICH T N,LYNN P T,et al.Translational and rotational settings of H2A.Z nucleosomes across the Saccharomyces cerevisiae genome[J].Nature,2007,446(7135):572-576.
[5] MAVRICH T N,IOSHIKHES I P,LI X,et al.Nucleosome organization in the Drosophila genom [J].Nature,2008,7192(453):358 -362.
[6] TOMANCAK P,BERMAN B P,BEATON A,et al.Global analysis of patterns of gene expression during Drosophila embryogenesis[J].Genome Biol,2007,145(8):R14.
[7] VAILLANT C,PALMEIRA L,CHEVEREAU G,et al.A novel strategy of transcription regulation by intra-genic nucleosome ordering[J].Genome Res,2009,59(20):256-287.
[8] 刘素宁,沈杰.果蝇基因组与功能基因研究进展[J].应用昆虫学报,2011,6(48):1559 -1572.LIU Suning,SHEN Jie.Progress in research on fumctional genes in the Drosophila genome[J].Chinese Journal of Applied Entomology,2011,48(6):1559 -1572.
[9] JANSEN A.,VERSTREPEN K J.Nucleosome positioning in saccharomyces cerevisiae[J].Microbiol Mol Biol Rev,2011,75(14):301 -320.
