生物膜干涉法研究STX与适配体间的相互作用
2014-09-14郑欣胡波高顺祥刘德婧孙铭娟王梁华
郑欣,胡波,高顺祥,刘德婧,孙铭娟,王梁华
(中国人民解放军第二军医大学基础部 生物化学与分子生物学教研室,上海 200433)
生物膜干涉法研究STX与适配体间的相互作用
郑欣,胡波,高顺祥,刘德婧,孙铭娟,王梁华Δ
(中国人民解放军第二军医大学基础部 生物化学与分子生物学教研室,上海 200433)
目的研究石房蛤毒素与其适配体间的相互作用。方法采用生物膜干涉法测定石房蛤毒素与适配体间的亲和常数,结合常数及解离常数,以其类似物河豚毒素和随机序列分析该适配体的特异性。结果该适配体可以与石房蛤毒素结合,Kd值为7.44μM,且不与河豚毒素结合。随机序列以较低的亲和力与石房蛤毒素结合。结论该适配体可以进行进一步优化,提高其与石房蛤毒素的亲和力与稳定性,以用于石房蛤毒素的检测,并用于石房蛤毒素的中毒治疗。
适配体;石房蛤毒素;相互作用;膜干涉技术
石房蛤毒素(C10H17N7O4,STX)是目前已知的最强有力的神经毒素之一,由海洋藻类合成后,富集于甲壳类和软体动物中,与多种类似物共同组成麻痹性贝类毒素家族,会导致人类和海洋动物产生麻痹性贝类中毒[1-4]。该毒素主要通过阻断可兴奋细胞表面的钠离子通道产生毒性效应[5]。小鼠腹腔注射、静脉注射以及口服的半数致死量LD50分别是8~10μg/kg,3.4μg/kg和260μg/kg[6]。为了保证人类的健康不受威胁,对石房蛤毒素进行监测显得尤为重要。
然而,检测石房蛤毒素,尤其是快速检测中最关键的部分是识别石房蛤毒素的元件[7]。核酸适配体,一小段寡核苷酸序列(DNA或RNA),能够与多种靶分子如药物,蛋白质,其他无机或有机分子高亲和力高特异性结合,已作为分析识别元件应用于靶分子检测,诊断,治疗等多方面[8-11]。目前,已有大量适配体经由SELEX(指数富集性配体系统进化)的方法从随机寡核苷酸序列库中筛选得到[12]。由于适配体的多种自身优势,包括其稳定性,低免疫原性,易于自动化学合成等,适配体在生物传感检测,诊断,治疗方面很有前景[13]。据报道,已有研究团队筛选得到一条DNA适配体,APTSTX1,可以与STX特异性结合,有潜力作为候选的分析识别元件应用于STX的快速高通量检测中[7]。本文采用生物膜干涉的方法对该序列与STX的相互作用进行动态学研究以及非特异性分析,旨在为后期的适配体优化提供实验基础,推进该适配体在STX检测中的实际应用。
1 材料与方法
1.1 材料和仪器 适配体5'-biotin APTSTX1及随机序列5'-biotin RANDOM均由上海生工生物工程有限公司合成。石房蛤毒素,河豚毒素TTX均购自台湾Algal Science公司。PBS-T(10 mM磷酸盐缓冲液、2.7 mM KCl、140 mM NaCl、0.05%吐温-20、pH 7.4;购自Sigma公司)。实验过程中所有试剂准备使用DEPC水(IDT公司)。生物膜干涉仪器(OctetRED 96系统,ForteBio公司),SSA芯片(加强型链霉亲和素,ForteBio公司)。
1.2 适配体和毒素准备 将冻干5'-biotin APTSTX1及RANDOM溶于PBS-T中,倍比稀释至1μM,95℃热变性5min后立即0℃复性10 min,最后4℃放置,以便后期检测分析使用。按照说明书STX干粉溶于3mM HCl中,配制成238μM的母液,然后倍比稀释至23.80μM,11.90μM和5.95μM(PBS-T)。TTX溶液直接用PBS-T稀释至30μM。
1.3 方法 采用生物膜干涉技术分析适配体与STX间的相互作用[14-15]。整个分析过程可以实时监测。首先采用生物素-链霉亲和素的方法经过平衡(PBS-T,1 min),适配体耦合(5min),解离(PBS-T,5min)和平衡(PBS-T,2min)四个步骤将APTSTX1固定于SSA芯片上。然后,分别经过靶分子STX(23.8μM,11.9μM和5.95μM)和TTX(30μM)的结合以及解离过程(各5min)得到响应值。另外设置3个SSA对照芯片,分别只加入APTSTX1,STX,或者2者都不加的本底对照。同样地,采用上述方法研究RANDOM与STX间的相互作用。对照芯片的响应值借助Octet数据分析软件CFR Part11 Version 6.x从样品芯片的响应值中扣除,同时经过数据拟合得到适配体的结合常数Kon,解离常数Kdis以及亲和常数KD值。
2 结果
2.1 APTSTX1与STX之间的相互作用 本文采用生物膜干涉的方法对APTSTX1与STX之间的相互作用进行实时分析,结果显示STX可以与APTSTX1结合,并且随着STX浓度的升高,响应值升高,利用数据分析软件采用1∶1结合模式对不同浓度STX的响应曲线进行拟合,得到2者的结合解离曲线(见图1),以及亲和常数,结合常数和解离常数(见表1)。
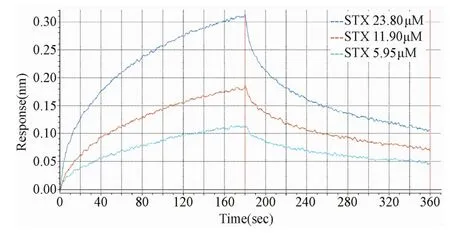
图1 不同浓度的STX(23.80μM,11.90μM和5.95μM)与APTSTX1之间的结合解离曲线Fig.1 The curve of association and dissociation of APTSTX1 with STX at23.80,11.90 and 5.95μM

表1 STX与适配体、随机序列之间的亲和参数Tab.1 Affinity parameters between STX and aptamer,random sequence
2.2 APTSTX1与STX间相互作用的特异性 采用高浓度的河豚毒素TTX以及随机序列RANDOM作为对照进行生物膜干涉分析,以研究适配体APTSTX1与STX间相互作用的特异性。结果显示,高浓度的TTX不能与APTSTX1相结合。而随机序列RANDOM与STX之间有相互作用,经过数据拟合分析得到其亲和常数,结合常数和解离常数(见表1)。如表1所示,随机序列RANDOM与STX之间的亲和力远低于APTSTX1。
3 讨论
NHI研究团队在2013年报道通过磁珠法筛选得到可以与小分子STX相结合的适配体,并通过SPR的方法证实2者可以结合,且随机序列与STX并无非特异性结合,然而该报道并未给出2者的具体KD值。本实验中,首次采用免标记的生物膜干涉技术[14-15]成功进行适配体与小分子靶标之间的相互作用研究,一旦靶分子与传感器表面包被的适配体结合,即引起光学膜层厚度的变化,导致光学膜层2个表面的反射光所形成的干涉光谱的变化。根据响应值,即干涉光谱的相位位移强度(nm),得到了游离状态下的STX与适配体间的亲和参数KD,结合常数kon以及解离常数kdis,实时观察2者之间的作用情况,并对该序列的特异性进行了分析。结果显示,STX与APTSTX1是可以相互结合的,而且是慢结合慢解离的过程。河豚毒素与石房蛤毒素相似,都是带有胍胺基的生物碱类毒素,然而该序列与TTX不能结合,表明APTSTX1可以特异地与STX作用。
与前述报道所得的结果不同的是,本实验结果显示随机序列RANDOM与STX也可以结合,但亲和力低于APTSTX1约5倍,响应值也较低。分析原因,可能是:STX带较强的正电荷,而DNA适配体带强负电荷,2者因为电荷的吸引产生相互作用。而由于靶分子诱导DNA进行再折叠也较常见,随机序列与STX的诱导契合程度不及APTSTX1,故亲和力较低。类似地,通过体外筛选得到的针对带正电靶分子的适配体都可能存在这样的情况。
虽然STX也会与随机序列非特异性结合,但APTSTX1可以特异地结合STX,而不结合TTX,这表明APTSTX1仍可以作为分析识别元件应用于STX的检测或者中毒治疗中。本实验结果提示,该适配体可以进行进一步优化以提高其亲和力和稳定性,从而促进它在各领域的应用。本课题已对该适配体进行了优化,亲和力提高了30倍(数据未发表),有潜力作为分子识别元件用于石房蛤毒素的检测或去除。
[1] Tian L,Cheng J,Chen X,et al.Early developmental toxicity of saxitoxin on medaka(Oryzias melastigma)embryos[J].Toxicon,2014,77:16-25.
[2]Wiese M,D'Agostino PM,Mihali TK,et al.Neurotoxic alkaloids:saxitoxin and its analogs[J].Mar Drugs,2010(8):2185-2211.
[3]Melegari SP,Perreault F,Moukha S,et al.Induction to oxidative stress by saxitoxin investigated through lipid peroxidation in Neuro 2A cells and Chlamydomonas reinhardtii alga[J].Chemosphere,2012,89(1):38-43.
[4]Perreault F,Matias MS,Melegari SP,et al.Investigation of animal and algal bioassays for reliable saxitoxin ecotoxicity and cytotoxicity risk evaluation[J].Ecotoxicol Environ Saf,2011,74(4):1021-1026.
[5]Cusick KD,Sayler GS.An overview on the marine neurotoxin,saxitoxin:genetics,molecular targets,methods of detection and ecological functions[J].Mar Drugs,2013,11:991-1018.
[6]Kawatsu K,Kanki M,Harada T,et al.A highly rapid and simple competitive enzyme-linked immunosorbent assay for monitoring paralytic shellfish poisoning toxins in shellfish[J].Food Chem,2014,162:94-98.
[7]Handy SM,Yakes BJ,DeGrasse Ja,et al.First report of the use of a saxitoxin-protein conjugate to develop a DNA aptamer to a small molecule toxin[J].Toxicon,2013,61:30-37.
[8]Radom F,Jurek PM,Mazurek MP,et al.Aptamers:Molecules of great potential[J].Biotechnol Adv,2013,31:1260-1274.
[9]Reinemann C,Strehlitz B.Aptamer-modified nanoparticles and their use in cancer diagnostics and treatment[J].Swiss Med Wkly,2014,144(January):w13908.
[10]Giannetti a,Tombelli S,Baldini F.Oligonucleotide optical switches for intracellular sensing[J].Anal Bioanal Chem,2013,405(19):6181-6196.
[11]Sharma AK,Heemstra JM.Small-molecule-dependent split aptamer ligation[J].JAm Chem Soc,2011,133(32):12426-12429.
[12]Shigdar S,Macdonald J,Connor MO,et al.Aptamers as Theranostic Agents:Modifications,Serum Stability and Functionalisation[J]. Sensors(Basel),2013,13:13624-13637.
[13]Wang RE,Wu H,Niu Y,et al.Improving the Stability of Aptamers by Chemical Modification[J].Curr Med Chem,2011,18:4126-4138.
[14]Concepcion J,Witte K,Wartchow C,et al.Label-free detection of biomolecular interactions using BioLayer interferometry for kinetic characterization[J].Comb Chem High Throughput Screen,2009,12:791-800.
[15]Zhang M,Jiang XQ,Le HN,et al.Dip-and-read method for label-free renewable sensing enhanced using complex DNA structures[J].ACS,Appl Mater Interfaces,2013,5(3):473-478.
(编校:吴茜)
Bio-Layer Interferometry in study of interaction between STX and aptamer
ZHENG Xin,HU Bo,GAO Shun-xiang,LIU De-jing,SUN Ming-juan,WANG Liang-huaΔ
(Department of Biochemistry and Molecular Biology,College of Basic Medical Sciences,Second Military Medical University,Shanghai200433,China)
ObjectiveTo study the interaction between STX and its aptamer.MethodsBio-Layer Interferometry was used to obtain the affinity constant,binding constant and dissociation constant of the aptamer against saxitoxin.An analogue of saxitoxin,tetrodotoxin,and a random sequence were analyzed as controls to study the specificity of the aptamer.ResultsThe aptamer could bind with saxitoxin with a Kd value of7.44μM withoutany interaction with tetrodotoxin.The random sequence could bind with saxitoxin with low affinity.ConclusionThe aptamer can be further optimized to improve its affinity and stability for the application in monitoring or poisoning treatment of saxitoxin.
Aptamer;saxitoxin;interaction;Bio-Layer Interferometry
R319
A
1005-1678(2014)09-0017-03
国家自然科学基金(2013AA092904)
郑欣,女,硕士,研究方向:海洋生物毒素,E-mail:dearjanna@126.com;通信作者,王梁华,男,博士、副教授,研究方向:海洋生物毒素,E-mail:wsh928@hotmail.com。
