新型甲型H7N9流感病毒PB1- F2蛋白基因进化和变异分析
2014-09-12资海荣李伟周丹李婕宣杨郭艳卫平民
资海荣,李伟,周丹,李婕,宣杨,郭艳,卫平民
(东南大学 公共卫生学院,江苏 南京 210009)
禽流感(Bird Flu或Avian Influenza)是禽流行性感冒的简称,它是由甲型流感病毒的一种亚型(也称禽流感病毒)引起的一种急性传染病,也能感染人类,被国际兽疫局定为甲类传染病,又称真性鸡瘟或欧洲鸡瘟。到目前为止,已报道[1- 6]的可引起人类感染的禽流感病毒有H7N7、H9N2 和H5N1。自从1997年在香港暴发H5N1以来,此病症引起世界各国卫生组织的高度关注。以往仅在禽类发现甲型H7亚型病毒感染,2013年2月底开始,我国东部省市地区陆续首次发现了由甲型H7N9禽流感病毒引起人感染的病例[7]。截至2013年8月30日,新型甲型H7N9禽流感散发涉及12个城市(包括上海、安徽、浙江、江苏、广州、山东、湖南、河南、江西、福建、河北、北京),确诊感染病例达135例,其中死亡病例45例。进一步研究[8]表明,H7N9流感病毒能够发生有限的人传人。
甲型流感病毒系单股负链RNA病毒,其基因组包括8个独立片段:PB2、PB1、PA、HA、NP、NA、M、NS,它们依次编码多聚酶成分(PB2、PB1、PA)、血凝素(HA)、核衣壳蛋白(NP)、神经氨酸酶(NA)、基质蛋白(M1)、膜蛋白(M2)、非结构蛋白(NS1、NEP)[9- 10]。2001年,Chen等[11]在研究MHCⅠ类分子递呈抗原肽时发现了一种不属于流感病毒任何已知开放阅读框(open reading frame,ORF)编码的多肽——PB1- F2蛋白,该蛋白是导致甲型流感病毒致病性的关键蛋白之一。 研究[12]显示,PB1- F2蛋白可以诱导宿主细胞线粒体途径的细胞凋亡,部分流感病毒毒株的PB1- F2蛋白可以提高流感病毒聚合酶活性,从而促进病毒在宿主细胞中复制。同时,完整的PB1- F2蛋白上存在特异的抗原表位,能诱导宿主体内免疫T细胞的表达,引起其免疫水平的改变,这与二次细菌感染引发的肺损伤具有显著关联;还发现随着基因的进化,有部分甲型流感病毒的亚型在PB1- F2序列上提前出现了终止密码子,导致PB1- F2蛋白的断裂,形成PB1- F2长度的多态性,而且从时间上呈现了一定的进化特征。截短型PB1- F2蛋白仍保留部分功能,如A/Beijing/11/1956(H1N1);已发现A/Hong Kong/1/1968(H3N2)的完整PB1- F2蛋白具有促炎症反应功能,H7N9作为新型甲型流感病毒的亚型之一,其PB1- F2蛋白的进化现状值得监测并探讨。本研究收集IRD数据库上的自2013年2月19日到2013年10月31日的H7N9流感毒株63株,利用MEGA 5.2.1软件进行分析,旨在对新型甲型H7N9流感病毒的PB1基因进化趋势进行分析,并对H7N9高致病性机制进行探讨。
1 资料与方法
1.1 资料来源
流感病毒基因序列通过 The NIAID Influenza Research Database(IRD) online(http://www.fludb.org)获得,下载自2013年2月19日到2013年10月31日的新型甲型H7N9流感病毒PB1基因序列63条,其中上海22株、江苏13株、安徽1株、山东7株、广东2株、浙江8株、湖南2株、江西2株、河南2株、福建2株、台湾2株;环境样本毒株13株,禽流感毒株33株(其中鸡源26株,鸭源2株,鸽子源5株),人流感17株。
1.2 构建进化树
利用Molecular Evolutionary Genetics Analysis(MEGA)Version 5.2.1 软件进行核苷酸序列比对,采用邻接(neighbor- joining,NJ)法(bootstraps=1 000)绘制H7N9流感病毒PB1基因的系统进化树,分析2013年新型甲型H7N9流感病毒PB1基因的进化状况。
1.3 PB1- F2氨基酸位点改变
利用MEGA 5.2.1 软件进行氨基酸序列比对,对PB1- F2氨基酸位点改变进行分析。
1.4 各型氨基酸构成比例
利用MEGA 5.2.1软件进行氨基酸序列比对,对PB1- F2蛋白中各类氨基酸构成比进行运算、输出。Excell表格对酸性氨基酸、碱性氨基酸、疏水性氨基酸、亲水性氨基酸的构成比进行计算,利用PASW 18.0 进行统计学分析。
2 结果与分析
2.1 H7N9流感病毒PB1系统进化树
见图1。
从图1 PB1系统进化树可以发现,63株新型甲型H7N9流感毒株主要进化为两大系别,18株病毒株属于系别Ⅰ,45株病毒株属于系别Ⅱ。系别Ⅰ与系别Ⅱ之间的核苷酸同源性以及氨基酸同源性分别为 96.3%~100%和98.6%~100%,系别Ⅰ核苷酸同源性以及氨基酸同源性分别为96.8%~100%和98.6%~100%,系别Ⅱ核苷酸同源性以及氨基酸同源性分别为98.5%~100%和99.2%~100%。
2.2 H7N9流感病毒中发现PB1- F2断裂株存在
将下载的PB1序列通过MEGA 5.2.1软件进行比对,在PB1的氨基酸第95~367从起始密码子ATG的位点到TAA或者TGA的终止密码子中截选出来,利用MEGA 5.2.1软件进行氨基酸序列比对,在63株毒株中发现有17株序列同时在氨基酸第26位提前出现了终止密码子,导致PB1- F2蛋白在第25位氨基酸后出现断裂。出现截短型PB1- F2蛋白毒株中分别有环境样本6株,其中3株来自山东,2株来自河南,1株来自上海;禽流感8株,其中7株为鸡源性,1株为鸽子源性;人流感3株,分别为A/Nanchang/1/2013、A/Changsha/1/2013、A/Changsha/2/2013。具体见图2。
2.3 H7N9流感病毒的氨基酸位点改变
通过对H7N9流感病毒PB1- F2病毒的氨基酸序列进行分析,发现本研究中的63株毒株中共有23处位点发生了改变。由图2可以看出,PB1- F2蛋白截短型毒株基因发生突变的位点明显多于PB1- F2蛋白完整型毒株,且PB1- F2蛋白截短型毒株基因的突变位置相对较固定。然而PB1- F2蛋白截短型毒株中A/chicken/Rizhao/875/2013的发生突变的位点相对于其他毒株要多。完整型PB1- F2蛋白毒株相对较少,却也同样存在发生突变位点较高的毒株,比如A/chicken/Shanghai/S1080/2013发生突变的位点却相对较多,此外还有A/chicken/Zhejiang/SD019/2013、A/Guangdong/1/2013、A/duck/Zhejiang/SC410/2013、A/duck/Zhejiang/SC410/2013。
2.4 H7N9流感病毒PB1- F2各类氨基酸构成
见表1。
氨基酸根据其结构与特性可分为疏水性氨基酸、亲水性氨基酸、酸性氨基酸、碱性氨基酸[13],63株流感毒株中构成PB1- F2蛋白的各类氨基酸比例见表1。经方差分析,完整型PB1- F2蛋白毒株与截短型PB1- F2蛋白毒株的各类氨基酸构成比差异有统计学意义(F1=44.25,P1<0.01;F2=17.83,P2<0.01;F3=719.02,P3<0.01;F4=1 016.19,P4<0.01)(1=疏水性氨基酸,2=亲水性氨基酸,3=酸性氨基酸,4=碱性氨基酸)。数据显示,由于PB1- F2蛋白的断裂导致构成各类氨基酸的比例发生改变。
表1 2013年H7N9流感毒株PB1-F2蛋白各类氨基酸比例
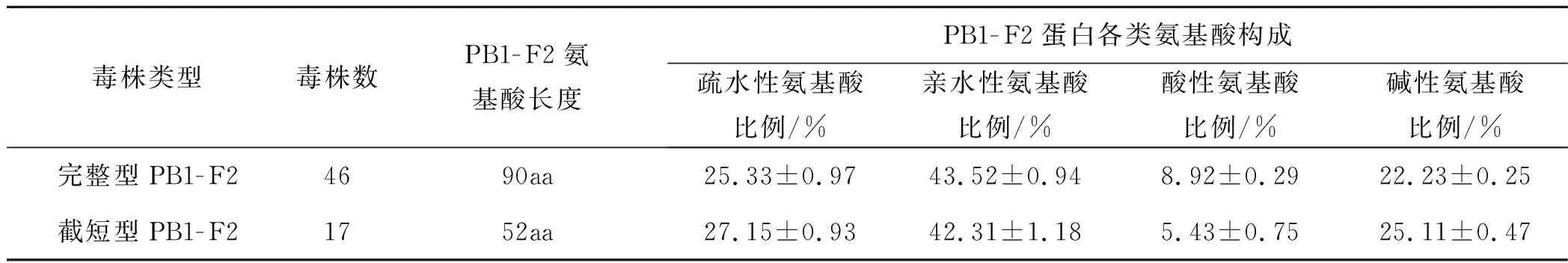
毒株类型毒株数PB1-F2氨基酸长度PB1-F2蛋白各类氨基酸构成疏水性氨基酸比例/%亲水性氨基酸比例/%酸性氨基酸比例/%碱性氨基酸比例/%完整型PB1-F24690aa25.33±0.9743.52±0.948.92±0.2922.23±0.25截短型PB1-F21752aa27.15±0.9342.31±1.185.43±0.7525.11±0.47
3 讨 论
通过对甲型H7N9流感病毒PB1系统进化树分析可以发现,系别 Ⅰ、Ⅱ的划分主要是依据PB1- F2蛋白断裂与否。系别 Ⅰ主要是完整型PB1- F2蛋白毒株,除A/chicken/Rizhao/875/2013外。系别Ⅱ主要是截短型PB1- F2蛋白毒株,A/chicken/Shanghai/S1358/2013、A/chicken/Jiangsu/SC099/2013除外。本研究结果表明,截短型PB1- F2蛋白已经成为新型甲型H7N9进化的重要特征。

● 代表人H7N9毒株,◇代表环境样本毒株;分支处数据是Bootstrap检验的可信度;0.005标尺代表单位长度内核苷酸差异水平
图1 2013年期间63株甲型H7N9流感毒株PB1系统进化树

方框内*为终止密码子部位,即发生断裂部位
图2 2013年甲型H7N9流感毒株PB1-F2氨基酸序列特征
PB1- F2基因有40个左右的位置可以通过单个核甘酸的突变提前引入终止密码子,从而编码不同长度的C端截短型PB1- F2蛋白[14]。McAuley等[15- 16]采用一系列方法,对来源于不同毒株、完整长度的PB1- F2蛋白的相关功能分别进行研究,通过反向遗传方法,将PR8病毒的7个基因片段(PB2、PA、HA、NP、NA、M、NS)和不同来源的PB1片段[A/Brevig Mission/1/1918(H1N1)、A/Vietnam/1203/2004(H5N1)]分别克隆至pHW2000质粒,将每一种PB1片段和PR8病毒的7个基因片段进行重组转化,合成多种来源PB1片段的重组病毒;再对PR8和新病毒进行细胞和小鼠致病性实验,比较不同毒株PB1- F2蛋白的促细胞凋亡作用、致小鼠肺部炎症及小鼠二次肺炎链球菌感染的严重程度,并监测病毒感染细胞时聚合酶活性的变化;又合成既定长度PB1- F2的C端肽链(第67至90位aa)。研究结果发现,来源于不同毒株的完整PB1- F2蛋白的功能存在一定差异;截短型PB1- F2蛋白仍保留部分功能,如A/Beijing/11/1956(H1N1),PB1- F2蛋白长度为57aa,截短型PB1- F2蛋白能增加病毒在293T细胞中的聚合酶活性。以上差异同样被其他相关研究所证实[17- 19 ]。
在本研究中的63株新型甲型H7N9流感毒株中发现17株出现断裂,且均生在PB1- F2的第25位氨基酸后,提示H7N9流感病毒在25位氨基酸后发生断裂是比较固定的断裂位点,而H7N9流感PB1- F2截短型毒株的出现对于H7N9致病性的影响尚需要进一步研究。
已有研究[20]发现,Brevig Mission(1918年,H1N1)与香港(1997年,H5N1)均有发现流感毒株中PB1- F2蛋白的第66位碱基发生点突变,产生氨基酸N66S,并证实其具可提升流感病毒的致病性。本研究中63株新型甲型H7N9流感毒株中第66位均为N,未发生碱基突变。有研究[21- 22]发现,H7N9人流感病毒是一个来自于H7N3(A/duck/Zhejiang/12/2011,ZJ12亚型)、H7N9(A/wild bird/Korea/A14/2011,KO14亚型)、H9N2(A/brambling/Beijing/16/2012- like viruses)的禽流感病毒重配株。其中6个内部基因(M、NS、NP、PB1、PB2、PA)则来自H9N2。现有关于H9N2基因进化的研究[14]发现,H9N2进化中存在66N与N66S毒株,本研究中未发现N66S毒株,提示在进行新型甲型H7N9的重配过程中,参与重配的H9N2属于66N毒株。
本研究通过对自2013年2月19日到2013年10月31日所有的甲型H7N9流感病毒毒株PB1- F2蛋白核酸以及氨基酸序列的分析,发现在H7N9流感毒株中存在着截短型PB1- F2蛋白毒株,并且H7N9流感病毒毒株PB1- F2蛋白断裂位点均在25位氨基酸后,这将成为H7N9流感毒株进化过程中重要的特征。本研究结果发现,甲型H7N9流感病毒截短型PB1- F2蛋白表现为更加明显的突变。基于研究结果猜想,截短型流感毒株易于导致基因结构的不稳定,从而使流感毒株易于发生基因突变,进而利于流感病毒的复制与传播。这一猜想有待进一步研究和验证。此外,完整型PB1- F2蛋白与截短型PB1- F2蛋白氨基酸构成存在明显的差异,这种差异对于H7N9流感病毒的致病性机制的影响也有待进一步的研究。
[1] LUPIANI B,REDDY S M.The history of avian influenza[J].Comp Immunol Microbiol Infect Dis,2009,32(4):311- 323.
[2] FOUCHIER R A,SCHNEEBERGER P M,Rozendaal F W,et al.Avian influenza A virus(H7N7)associated with human conjunctivitis and a fatal case of acute respiratory distress syndrome[J].Proc Natl Acad Sci USA,2004,101(5):1356- 1361.
[3] KOOPMANS M,WILBRINK B,CONYN M,et al.Transmission of H7N7 avian influenza A virusto human beings during a large outbreak in commercial poultry farms in the Netherlands[J].Lancet,2004,363(9409):587- 593.
[4] LIN Y P,SHAW M,Gregory V,et al.Avian- to- human transmission of H9N2 subtype influenza A viruses:relationship between H9N2 and H5N1 human isolates[J].Proc Natl Acad Sci USA,2000,97(17):9654- 9658.
[5] PEIRIS J S,de JONG M D,GUAN Y.Avian influenza virus(H5N1):a threat to human health[J].Clin Microbiol Rev,2007,20(2):243- 267.
[6] GAMBOTTO A,BARRATT- BOYES S M,de JONG M D,et al.Human infection with highly pathogenic H5N1 influenza virus[J].Lancet,2008,371(9622):1464- 1475.
[7] 刘春艳,艾军红.甲型H7N9禽流感病毒的病毒学特征[J].中国当代儿科杂志,2013,15(6),405- 408.
[8] Avian influenza A H7N9 in zhejiang,China[DB/OL]published online. April.25,2013,http://dx.doi.org/10.1016/s0140- 6736(13)60936- 8.
[9] WEBSTER R G,BEAN W J,GORMAN OT,et al.Evolution and ecology of influenza A viruses[J].Micro Rev,1992,56(1):152- 179.
[10] McHARDY A C,ADAMS B.The role of genomics in tracking the evolution of influenza A virus[J].PLoS Pathog,2009,5(10):e1000566.
[11] CHEN W,CALVO P A,MALIDE D,et al.A novel influenza A virus mitochondrial protein that induces cell death[J].Nature Medicine,2001,7(12):1306- 1312.
[12] 资海荣,李伟,李婕,等.甲型流感病毒PB1- F2蛋白功能的研究进展[J].东南大学学报:医学版,2013,32(5):631- 635.
[13] 庄荣辉.生物化学基础[M/OL].台湾:网页系统之生物化学基础,2001,1.
[14] 黄艳艳,胡北侠,文心田.A型流感病毒中国分离株PB1- F2基因进化分析[J].中国农业科学,2009,42(6):2156- 2163.
[15] McAULEY J L,CHIPUK J E,BOYD K L,et al.PB1- F2 proteins from H5N1 and 20 century pandemic influenza viruses cause immunopathology[J].PLoS Pathog,2010,6:e1001014.
[16] McAULEY J L,ZHANG K,MCCULLERS J A.The effects of influenza A virus PB1- F2 protein on polymerase activity are strain specific and do not impact pathogenesis[J].J Virol,2010,84(1):558- 564.
[17] CHEN C J,CHEN G W,WANG C H,et al.Differential localization and function of PB1- F2 derived from different strains of influenza a virus[J].J Virol,2010,84(19):10051- 10062.
[18] HAI R,SCHMOLKE M,VARGA Z T,et al.PB1- F2 expression by the 2009 pandemic H1N1 influenza virus has minimal impact on virulence in animal models[J].J Virol,2010,86(9):4442- 4450.
[19] KOSIK I,KREJNUSOVA I,BYSRRICKA M,et al.N- terminal region of the PB1- F2 protein is responsible for increased expression of influenza A viral protein PB1[J].Acta Virologica,2011,55:45- 53.
[20] CONENLL G M,TISONCIK J R,ROSENZWEIG E,et al.A single N66S mutation in the PB1- F2 protein of influenza avirus increases virulence by inhibiting the early interferon responseinvivo[J].J Virol,2011,85(2):652- 662.
[21] GAO R,CAO B,HU Y,et al.Human infection with a novel avian- origin influenza A(H7N9) virus[J].N Engl J Med,2013,368:1888- 1897.
[22] CHENY,LIANG W,YANG S,et al.Human infections with the emerging avian influenza A H7N9 virus from wet market poultry:clinical analysis and characterization of viral genome[J].Lancet,2013,6736(13):60903- 60904.
