2010年广州市甲型H1N1流感病毒分离株NA基因变异分析
2014-09-11许沙沙常彦敏冯发深张定梅曹开源
许沙沙,常彦敏,徐 霖,冯发深,何 霞,王 铸,张定梅,曹开源
(1.衡水市哈励逊国际和平医院检验科,河北 衡水 053000;2.郑州大学附属肿瘤医院检验科,河南 郑州 450000;3.中山大学临床检验标准化研究中心,广东 广州 510080)
流感病毒是有包膜的单股负链RNA病毒,属于正黏病毒科(orthomyxoviridae),根据核衣壳蛋白和基质蛋白不同,分为甲(A)、乙(B)、丙(C)3型[1]。甲型H1N1流感于2009年3月在墨西哥爆发,最初被称为“人感染猪流感”,世界卫生组织将流感大流行警告级别提高为6级[2],2009年4月中国卫生部发布公告,明确将猪流感更名为“甲型H1N1流感”。据世界卫生组织统计,截至2010年3月19日,这种新病毒已经波及213个国家,共造成16813例死亡[3]。许多学者认为华南地区是世界流感流行的一个主要起源地,因此加强对甲型H1N1流感的监测对我国传染病防控具有重要意义[4]。神经氨酸酶(neuraminidase,NA)是流感病毒表面重要的糖蛋白,是甲型流感病毒主要抗原之一,对于流感病毒从感染细胞中的释放非常重要。为了监测甲型H1N1流感NA基因的变异情况,及时发现具有流行病学意义的耐药株,我们对2010年从广州采集的1194份呼吸道标本中分离到的6株甲型H1N1病毒株NA基因进行测序,与2009年的代表株进行比对,对其耐药位点和同源性进行分析,了解其变异情况,为今后流感的监控和防治提供参考资料。
材料和方法
一、标本来源
2010年2到12月广州市中山大学附属二院和三院发热门诊病例咽拭子标本1194份,病例纳入标准为:发热3 d以内,体温≥38℃;伴有咳嗽或咽喉疼痛等急性上呼吸道感染症状。
二、毒株
2010年本实验室分离的甲型H1N1流感病毒和2009年3月本实验室从广州市第1例甲型H1N1流感病毒感染患者体内分离的毒株,名称为A/Guangdong/03/2009(H1N1)(GenBank登录号GQ250161);从GenBank中选取北京、上海、广东三地在2009年甲型H1N1流感流行时的NA序列共9株,用以基因分型和比较,见表1。

表1 NA基因用以进化分析的毒株
三、RNA的提取和cDNA的合成
对采集的咽拭子标本进行处理,各取200 μL提取RNA(QIAmp MiniElute Virus Spin试剂盒),逆转录合成 cDNA(Invitrogen,SuperscriptⅢ first strand逆转录试剂盒),操作步骤参照说明书进行,反应条件为:25℃ 10 min,50℃ 50 min,85℃5 min,37 ℃ 20 min。
四、甲型流感检测
对1194份标本的cDNA全部进行甲型流感病毒检测。
1.引物 参照国家流感中心下发的引物,目标片段为M基因,上游引物5'-TTCTAACCGAGGTCGAAACG-3',下游引物 5'-ACAAAGCGTCTACGCTGCAG-3',特异产物长度235 bp。
2.聚合酶链反应(polymerase chain reaction,PCR)反应体系 TaKaRa ExTaq mix 12.5 μL,焦碳酸二乙酯水 7.5 μL,上、下游引物各 2 μL;cDNA模板 1 μL。反应条件:95 ℃ 5 min,94 ℃30 s,52 ℃ 30 s,72 ℃ 1 min,35 个循环,72 ℃10 min。
五、甲型H1N1流感病毒检测
对甲型流感病毒检测阳性的标本进行甲型H1N1流感病毒特异PCR检测。
1.引物 参照国家流感中心下发的引物,目标片段为HA基因,上游引物5'-AATAACATTAGAAGCAACTGG-3',下游引物5'-AGGCTGGTGTTTATRGCACC-3',特异产物长度153 bp。
2.PCR 同上。
六、NA克隆和序列测定
参考GenBank中甲型H1N1流感病毒株NA序列(GenBank登陆号GQ250162)分别设计分段引物 NA-1和 NA-2,NA-1引物扩增1~643 bp,NA-2引物扩增 505~1410 bp,NA基因全长1410 bp,引物序列见表2。

表2 NA基因扩增引物
PCR 反应体系:TaKaRa ExTaq mix 25 μL,焦碳酸二乙酯水 15 μL,上下游引物各 4 μL,cDNA模板2 μL。反应条件:95 ℃ 5 min,94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,35 个循环,72 ℃ 10 min。
胶回收试剂盒(TAKATA)回收PCR产物,连接到pMD 18-T Vector(TAKATA),转化 DH-5α感受态细胞,用Amp+LB筛选阳性菌落提取重组质粒,PCR初步鉴定重组质粒,送上海英骏公司测序。
七、变异分析
利用 Clustal 1.83软件[5]对测序毒株 HA基因序列和氨基酸序列进行比对和分析,利用Glyc 1.0 Server(http://www.cbs.dtu.dk/services/Net-NGlyc/)在线软件预测其糖基化位点,并对其耐药位点进行分析。
八、系统进化分析
将分离的6株广州甲型H1N1流感病毒分离株和从GenBank中筛选的9株甲型H1N1流感病毒的 HA基因用 MEGA4.0软件[6]近邻连接法(neighbor-joining method)构建系统进化树。
结 果
一、标本流行病学资料
1194份咽试子标本中检测甲型流感病毒327份,其中检测到H1N1流感病毒6株。
二、甲型流感病毒PCR检测结果
PCR扩增结果表明成功扩增大小约235 bp的甲型流感病毒特异性片段,见图1。
三、H1N1流感病毒PCR检测结果
PCR扩增结果表明成功扩增大小约153 bp的甲型H1N1流感病毒特异性片段,见图2。
四、甲型H1N1流感病毒的NA基因PCR扩增及测序
6株甲型H1N1流感病毒分离株NA-1段基因的逆转录PCR产物全长643 bp,见图3。6株甲型H1N1流感病毒的NA-2段基因的逆转录PCR产物全长905 bp,与理论值相符,见图4。回收PCR扩增产物,与T载体连接,由上海英骏公司进行测序(结果略)。

图1 甲型流感病毒电泳结果(部分结果)
图2 甲型H1N1流感病毒电泳结果

图3 NA-1片段逆转录PCR电泳结果
五、变异分析
1.NA基因同源性分析 2009年流感病毒代表株和2010年流感病毒分离株共15株,将其NA基因共1410个碱基,用Clustalx 1.83进行序列比对,用BioEdit软件得到15株NA基因的氨基酸相似性矩阵,见表3。2010年分离株和2009年代表株氨基酸相似性较高,介于0.985~0.999。

图4 NA-2片段逆转录PCR电泳结果

表3 NA核酸相似性矩阵
2.NA系统进化树分析 根据NA基因全长序列(1410 bp),对2009年代表株和2010年分离株共15株甲型H1N1流感病毒株进行了系统进化分析,包括本研究分离到的6株,还有选取的2009年代表株。在系统进化树上,15株毒株分成了3个分支,见图5。
3.氨基酸变异分析 2010年分离的6株甲型H1N1流感病毒的NA基因与2009年中国大陆的代表株比对,6株分离株共有30个碱基位点发生突变,其中有义突变为16个,3个位点和NA活性相关,其中222位氨基酸的变异位于NA活性位点上,未发现H275Y耐药位点的变异,见表4。

图5 甲型H1N1流感病毒NA基因系统进化树

表4 2010年分离株和2009年代表株NA片段氨基酸比对结果
4.糖基化位点变异分析 分别将2010年广州市分离到的6株甲型H1N1病毒株和2009年代表株NA基因进行糖基化位点的预测。结果发现4株甲型H1N1病毒株的糖基化位点和2009年代表株相比并没有明显的变化。
5.NA蛋白结构建模 通过蛋白分析专家系统EXPASY(http://ca.expasy.org/)提供的蛋白组学和序列分析工具SWISS-MODEL,模拟构建各NA蛋白的空间构象。3株病毒株在NA活性位点222位、228位和425位等氨基酸位点处发生了变异,见图6。

图6 NA蛋白三维结构
讨 论
NA蛋白是流感病毒包膜上重要的糖蛋白,能切断细胞表面的唾液酸,促使病毒从感染细胞膜上释放,防止子代病毒自身凝集,促进病毒扩散并增强其感染能力。由于NA在流感病毒复制和传播中起重要作用,且其活性中心的氨基酸组成高度保守,因此是研制抗流感药物的重要靶点。
目前抗流感药物有2种:一种为M2离子通道阻滞剂,如金刚烷胺,但是其副作用很大,而且很容易产生耐药株,有学者报道现在流感病毒对烷胺类的耐药现象十分严重[7-8]。而2009年甲型H1N1流感病毒对烷胺类耐药情况同样严重,主要是在NA第31位氨基酸的变异(S31N)[9]。在这种情况下NA抑制剂成为目前抗流感病毒研究的热点[10],而且奥司他韦对甲型流感和乙型流感都有效,不易引起耐药性且耐受性好。但是自从奥司他韦应用于临床以来,不断有耐药株出现,耐药率逐年上升,耐药率在成人为0.4% ~1%[11],在儿童为4% ~8%,其中日本儿童的耐药率高达18%。
流感病毒NA的活性位点结构示意图见图7[12-13],NA催化位点以及周围相关位点高度保守[14-15]。直接参与催化作用的活性区域有5个,分别为 S1~S5,S1由3个氨基酸残基(R118、R292、R371)组成,S2由2个氨基酸残基(E227、E119)组成,S3由2个氨基酸残基(W178、I222)组成,S4 由3 个氨基酸残基(I222、R224、A246)组成,S5由2个氨基酸残基(A246、E276)组成。间接参与催化作用的位点有 R156、Y406、S179、D198、E228、N294、E425 等氨基酸位点。
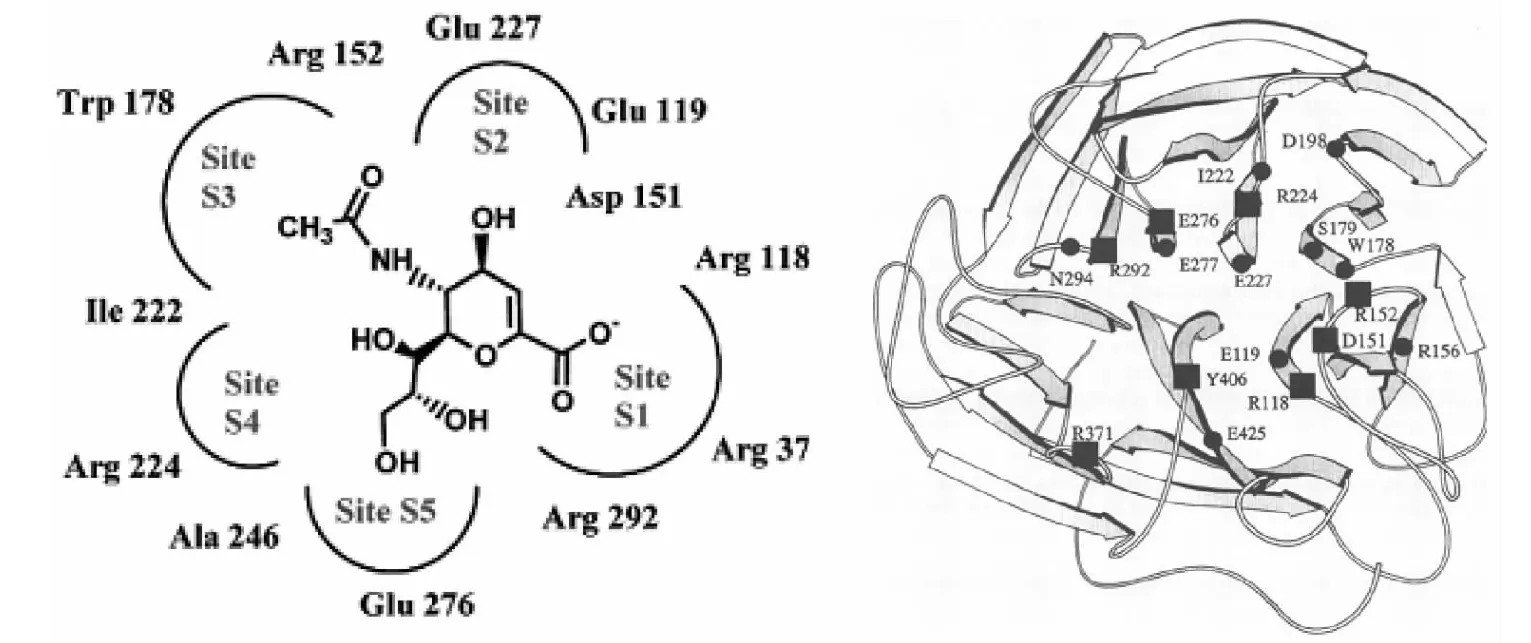
图7 流感病毒NA的活性位点结构示意图
这些位点都直接或间接地参与NA的催化,所以此处氨基酸如果发生突变,就会对NA的活性产生影响,减低流感病毒对NA抑制剂的敏感性,甚至会导致对NA抑制剂的耐药。通过对本研究分离到的6株甲型H1N1流感病毒的NA基因的测序,氨基酸序列分析发现部分毒株出现N42S、N59D、Q313K等的氨基酸位点变异,这些均未涉及NA催化位点。但是在A/Guangdong/ZS03/2010(H1N1)毒株中出现E228G氨基酸突变,A/Guangdong/ZS04/2010(H1N1)毒株出现E425G氨基酸突变,这些位点都与NA的活性有关,但这些毒株其耐药性是否有变化,有待进一步研究。
同时在 A/Guangdong/ZS01/2010(H1N1)毒株中出现N222S氨基酸的突变(虽然与文献报道I222位突变的碱基不一样,但是222位氨基酸还是存在突变的)。许多研究表明222位氨基酸的改变会导致流感病毒对 NA抑制剂的耐药[16]。因此需继续对其耐药性密切监测。
从NA三维结构图中可以看到NA活性位点组成一个疏水性的口袋状结构,可以结合NA抑制剂奥司他韦等药物,起到抗病毒的作用。如果疏水性口袋状结构的氨基酸位点发生变异,会导致无法结合奥司他韦等药物,则会产生流感病毒耐药株。尽管结构图中只有少部分氨基酸的突变,但突变位点在NA活性位点上可能对其耐药性产生影响,需后续试验继续研究。
许多研究表明,275位氨基酸的变异会对奥司他韦产生耐药[17],通过比较分析2010年分离到的甲型H1N1流感病毒,均未发现275位的变异,提示在2010年广州散发的甲型H1N1流感病毒对NA抑制剂仍然敏感。2010年分离株NA糖基化位点与2009年代表株比较并没有变化,与Saxena等[18]的报道相同。同时发现2010年分离株与 2009年代表株同源性较高(0.985~0.999),提示NA基因仍是同一来源,未发生较大变异。对NA系统进化树分析发现,2009年代表株为一支,而2010年分离株为一支,说明2010年与2009年相比,NA基因有部分碱基发生变异。
通过以上分析,NA蛋白275位氨基酸并未出现变异,提示还没有出现对奥司他韦的耐药株,但是在NA活性中心周围已经存在有变异的氨基酸,可能会对其耐药性有影响。同时随着奥司他韦在临床上的广泛应用,甲型H1N1流感病毒耐药株在世界上其他国家不断有报道,更应该继续加强对甲型H1N1流感病毒的耐药性监测以及序列分析工作,在分子遗传进化分析的基础上,及时发现有流行病学意义的耐药株,为国家制定甲型H1N1流感防控策略提供参考。
[1]Fouchier RA,Munster V,Wallensten A et al.Characterization of a novel influenza A virus hemagglutinin subtype(H16)obtained from black-headed gulls[J].J Virol,2005,79(5):2814-2822.
[2]Smith GJ,Vijaykrishna D,Bahl J,et al.Origins and evolutionary genomics of the 2009 swine-origin H1N1 influenza A epidemic[J]. Nature,2009,459(7250):1122-1125.
[3]WHO.Pandemic(H1N1)2009[EB/OL].(2010-03-09)[2010-05-06].http://www.who.int/csr/disease/swinflu/laboratory 19_03_2010/en/.
[4]Shortridge KF.Severe acute respiratory syndrome and influenza:virus incursions from southern China[J].Am J Respir Crit Care Med,2003,168(12):1416-1420.
[5]Thompson JD,Higgins DG,Gibson TJ.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[6]Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599.
[7]Bright RA,Shay DK,Shu B,et al.Adamantane resistance among influenza A viruses isolated early during the 2005-2006 influenza season in the United States[J].JAMA,2006,295(8):891-894.
[8]蓝 雨,李 梓,董丽波,等.中国H3N2亚型流感病毒烷胺类药物耐药性研究[J].中华实验和临床病毒学杂志,2006,20(2):21-23.
[9]Centers for Disease Control and Prevention(CDC).Update:drug susceptibility of swine-origin influenza A(H1N1)viruses,April 2009[J].MMWR Morb Mortal Wkly Rep,2009,58(16):433-435.
[10]Smith BJ,McKimm-Breshkin JL,McDonald M,et al.Structural studies of the resistance of influenza virus neuramindase to inhibitors[J].J Med Chem,2002,45(11):2207-2212.
[11]Monto AS,McKimm-Breschkin JL,Macken C,et al.Detection of influenza viruses resistant to neuraminidase inhibitors in global surveillance during the first 3 years of their use[J].Antimicrob Agents Chemother,2006,50(7):2395-2402.
[12]Colman PM,Hoyne PA,Lawrence MC.Sequence and structure alignment of paramyxovirus hemagglutinin-neuraminidase with influenza virus neuraminidase[J].J Virol,1993,67(6):2972-2980.
[13]Langedijk JP,Daus FJ,van Oirschot JT.Sequence and structure alignment of paramyxoviridae attachment proteins and discovery of enzymatic activity for a morbillivirus hemagglutinin[J].J Virol,1997,71(8):6155-6167.
[14]Hurt AC,Ernest J,Deng YM,et al.Emergence and spread of oseltamivir-resistant A(H1N1)influenza viruses in Oceania,South East Asia and South Africa[J].Antiviral Res,2009,83(1):90-93.
[15]Ferraris O,Lina B.Mutations of neuraminidase implicated in neuraminidase inhibitors resistance[J].J Clin Virol,2008,41(1):13-19.
[16]Deyde VM,Nguyen T,Bright RA,et al.Detection of molecular markers of antiviral resistance in influenza A(H5N1)viruses using a pyrosequencing method[J].Antimicrob Agents Chemother,2009,53(3):1039-1047.
[17]Kiso M,Shinya K,Shimojima M,et al.Characterization of oseltamivir-resistant 2009 H1N1 pandemic influenza A viruses[J].PLoS Pathog,2010,6(8):e1001079.
[18]Saxena SK,Mishra N,Saxena R,et al.Structural and antigenic variance between novel influenza A/H1N1/2009 and influenza A/H1N1/2008 viruses[J].J Infect Dev Ctries,2010,4(1):1-6.
