小鼠α-synuclein基因内含子1甲基化定量检测方法的建立及不同脑区甲基化水平的比较研究
2014-08-14王丹慧蔡彦宁
陆 璐,张 兰,王丹慧,蔡彦宁
(首都医科大学宣武医院药物研究室,神经变性病教育部重点实验室,北京 100053)
众多研究表明,α-突触核蛋白(alpha-synuclein,α-syn)与神经退行性疾病密切相关。例如,异常聚集和纤维化的α-syn是构成路易体的主要成分,而后者是帕金森病(PD)和路易体痴呆的主要标志[1]。又如,α-syn基因突变可以导致家族性PD[2-4]。此外,α-syn的过度表达能够引起细胞内氧化应激水平的增加,进而促进α-syn的聚集和一系列的病理变化[5]。
α-syn的转录调控极为复杂并且与疾病相关。研究表明,α-syn启动子区多态性能够通过影响α-syn的转录,增加发生PD的风险[6]。而α-syn的二倍体、三倍体均能增强其转录,并使罹患PD风险增加[7, 8]。最近,α-Syn的另一种转录调控方式逐渐被揭示并受到重视。多项研究显示,人α-Syn内含子1存在一个CpG岛。PD患者脑组织以及PD患者外周血淋巴细胞中,该CpG岛的甲基化水平显著下降[9, 10]。该CpG岛的低甲基化能够导致α-Syn的高表达[9, 11, 12]。这些结果提示,CpG岛的甲基化程度参与人调控α-Syn的表达,并与PD的发生相关联。研究还显示,正常脑组织中,该CpG岛在皮层、纹状体、黑质中均高度甲基化;而PD患者脑组织中,该CpG岛在皮层、纹状体高度甲基化;在黑质中几乎不存在甲基化[9]。这一结果提示该CpG岛的甲基化可能与PD中多巴胺能神经元特异性损伤相关。啮齿类动物中,α-Syn内含子1区段同样发挥着重要的转录调控作用,并且该区段同样富含CpG位点[13]。目前仍不明确,小鼠α-syn的转录是否也受内含子1区段甲基化水平调控。阐明这一问题将有助于解析PD小鼠模型与PD患者间的异同,有助于辅助正确解读基于PD小鼠模型的研究成果。本研究分析了小鼠α-Syn内含子1的CpG岛,建立了定量分析其甲基化水平的检测方法,检测了不同脑区小鼠α-Syn基因CpG岛甲基化水平。
1 材料和方法
1.1 实验动物
清洁级C57BL/6雄性小鼠,2月龄10只,由斯贝福(北京)实验动物科技有限公司提供,动物合格证号:SCXK(京)2011-0004。饲养条件:每日光照12 h,黑暗12 h,室温23℃,任意摄取水食。相同时间全部处死后取小鼠新鲜脑组织并按解剖结构分离皮质、海马、纹状体及中脑并冻存于-80℃待用。实验全过程对动物的饲养与取材均遵守实验动物管理与保护的有关政策和规定。
1.2 主要试剂和仪器
QIAamp DNA mini试剂盒(德国Qiagen公司),Wizard DNA纯化试剂盒(美国Promega公司),HS Taq (日本Takara公司),PyroMark gold Q96 reagents(德国Qiagen公司),Streptavidin SepharoseTMBeads(美国GE公司),Nanodrop 2000分光光度计(美国Thermo公司),PTC-l00基因扩增仪(美国MJ research公司),PyroMark Q96 ID焦磷酸测序仪(德国Qiagen公司)。
1.3 基因组DNA的提取和偏重亚硫酸氢盐处理
按照QIAamp DNA mini试剂盒说明书提取冻存的不同脑区小鼠组织基因组DNA。使用偏重亚硫酸氢钠和对苯二酚对基因组DNA进行处理,通过该处理,未甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶保持不变。处理过程如下,1 μg基因组DNA使用0.3 mol/L的NaOH进行变性。而后使用新鲜配置的偏重亚硫酸氢钠(终浓度3.0 mmol/L, pH 5.0)与对苯二酚的混合溶液(终浓度0.5 mmol/L, pH 5.0)与基因组DNA混合,石蜡油覆盖,避光50 ℃孵育16 h。继而使用Wizard DNA 纯化试剂盒纯化DNA。最后加入0.3 mol/L的NaOH在37 ℃孵育15 min结束反应,并乙醇沉淀。
1.4 小鼠α-syn内含子1区段CpG岛分析
使用在线软件MSPPrimer (http://www.mspprimer.org/cgi-bin/design.cgi)[14]分析小鼠α-syn基因内含子1及其侧翼序列。
1.5 焦磷酸测序引物设计
使用PyroMark assay design 2.0(德国Qiagen公司)设计扩增引物和测序引物。正向扩增引物为:5’-GGGGAGAGGAGTTGATAAATTAGTTG-3’;反向扩增引物使用Biotin标记:5’- TCCTAATTTCCCC AATCTAATTTCA-3’;测序引物为:5’-GTGAAGG AGTTAGGGA-3’。引物由上海生工生物技术有限公司合成。扩增产物长度160 bp,可以定量分析3个CpG位点的甲基化水平。待分析序列为:GTTAGAGYGTTYGGTAGTAGGTAGTAGAYGGTA,其中Y代表CpG位点。
1.6 聚合酶链反应
经亚硫酸氢盐处理的基因组DNA作为模板,使用HS Taq进行扩增。扩增条件为:94℃预变性2 min; 94℃变性20 s,58℃退火20 s, 72℃延伸20 s,共50循环;最后72℃延伸5 min。
1.7 焦磷酸测序(Pyrosequencing)
将生物素标记的PCR产物与亲和素标记的微珠相混合,利用真空预处理装置使分离纯化生物素标记的单链DNA,转移入PyroMark Q96 ID测序仪中。生物素标记的单链DNA作为模版,使用0.6 μmo/L测序引物,按照PyroMarkQ96使用说明书进行测序操作。
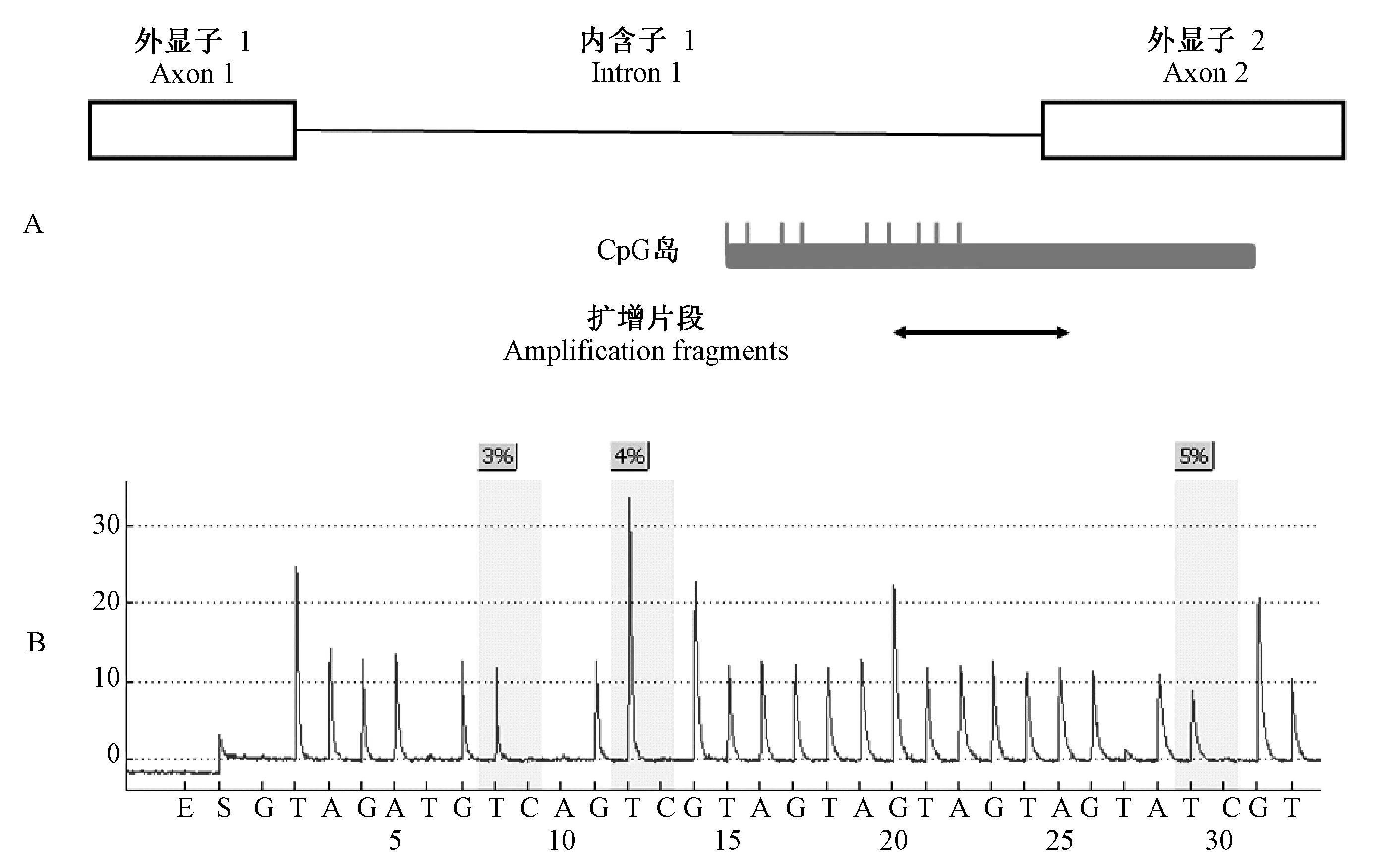
注:(A)小鼠内含子1及其侧翼区域。其中上层方框代表外显子,上层直线代表内含子1;中层灰框代表CpG岛区域,灰框上方灰色竖条代表内含子1中的CpG位点;下层双箭头直线为焦磷酸测序扩增的区域。(B)焦磷酸测序示意图,其中灰色区域为CpG位点,蓝底数字显示每个位点的甲基化百分比。
2 结果
小鼠α-syn基因内含子1长度为674 bp,连同其上下游的外显子1和2共计1 131 bp。经在线软件MSPPrimer分析,与人α-syn基因相似,该区段存在一个CpG岛,该CpG岛覆盖了内含子1的后半段和外显子2的大部分(图1A)。设计焦磷酸测序检测,扩增片断长度为160 bp,电泳显示扩增产物与预期片段大小一致。使用SssI 甲基转移酶处理并经亚硫酸氢盐处理的基因组DNA作为模板,同样扩增获得160 bp 扩增子。而使用未经亚硫酸氢盐处理的基因组DNA作为模板,无扩增产物。表明扩增条件特异(图2)。通过焦磷酸测序能够定量检测3个CpG位点的甲基化水平。焦磷酸测序结果如图1B所示,灰色区域为CpG位点。根据焦磷酸测序中胞嘧啶的数目与胞嘧啶和胸腺嘧啶之和的数目相比即可获得该位点甲基化百分比。每个CpG位点的甲基化百分比由仪器自动计算并显示于CpG位点顶部的蓝色区域。使用焦磷酸测序法对不同脑区的甲基化水平进行检测,如图3所示,成年小鼠皮质、中脑、纹状体、海马等四个脑区该CpG岛平均甲基化水平均低于2%,不同脑区间无明显差别。

注:不同小鼠脑区基因组DNA经偏重亚硫酸氢盐处理,而后作为模板进行扩增。扩增产物160 bp。SssI甲基转移酶处理的基因组DNA作为阳性对照,未经偏重亚硫酸氢盐处理的基因组DNA为阴性对照。
3 讨论
DNA甲基化通过调控基因表达,影响生物生理与行为,是表观遗传学研究的一个重要方面[15]。近年来发展和建立了多种DNA甲基化的检测手段,最常见的是甲基化特异性PCR和克隆测序。焦磷酸测序是一种基于即时测序的技术,可以对甲基化程度进行精确定量。相比其他DNA甲基化检测手段,焦磷酸测序操作简便、结果重复性高也更加准确[16]。因此本研究选择建立基于焦磷酸测序的DNA甲基化检测方法。
多项研究均表明人α-syn内含子1区存在CpG岛,人α-syn的表达受到该CpG岛甲基化水平的调控,并且正常脑组织中多个脑区均呈高甲基化状态[9-12]。我们的研究表明,尽管小鼠α-syn内含子1区也存在CpG岛,但该CpG岛在所检测的四个脑区中均呈低甲基化状态,甲基化水平均不超过2%。这一结果与人脑组织中40-100%的甲基化水平差异明显[9]。提示小鼠脑组织中α-syn的转录调控机制与人类存在明显差异。与此相吻合,人及灵长类动物脑组织中α-syn表达水平随增龄而增加[17],而在啮齿类中α-syn表达量随增龄而减少[18]。这种差异很可能与α-syn内含子1CpG岛甲基化调控存在关联。本研究对于指导选择合适的动物和细胞模型也具有重要意义。例如,有研究显示DNA甲基化酶抑制剂能够上调α-syn的表达,从而加强MPP+以及6-OHDA等毒素的毒性[12]。研究者认为DNA甲基化酶抑制剂可能是治疗PD的靶点。而我们的结果提示小鼠以及小鼠来源的细胞,由于其中α-syn的表达不受DNA甲基化的调控,可能不适合该项目的后续研究。

图3 不同小鼠脑区α-syn内含子1CpG岛甲基化百分比
总之,本研究建立了定量分析小鼠α-syn内含子1CpG岛甲基化水平的焦磷酸测序方法。本研究还提示小鼠与人α-syn的表观遗传调控存在明显差异。
参考文献:
[1] Spillantini MG, Crowther RA, Jakes R, et al. Alpha-synuclein in filamentous inclusions of Lewy bodies from Parkinson’s disease and dementia with Lewy bodies [J]. Proc Natl Acad Sci U S A, 1998, 95(11): 6469-6473.
[2] Polymeropoulos MH, Lavedan C, Leroy E, et al. Mutation in the alpha-synuclein gene identified in families with Parkinson’s disease [J]. Science, 1997, 276(5321):2045-2047.
[3] 张丽, 陈炜, 张旭, 等. 携带A53T突变人α突触核蛋白转基因帕金森病大鼠模型的建立 [J]. 中国比较医学杂志, 2013, 23(10): 1-6.
[4] Zarranz JJ, Alegre J, Gómez-Esteban JC, et al. The new mutation, E46K, of alpha-synuclein causes Parkinson and Lewy body dementia [J]. Ann Neurol, 2004, 55(2):164-173.
[5] 张丽, 张连峰. α-synuclein致病机制和转基因帕金森疾病模型研究进展 [J]. 中国比较医学杂志, 2012, 22(11): 63-67.
[6] Maraganore DM, de Andrade M, Elbaz A, et al. Collaborative analysis of alpha-synuclein gene promoter variability and Parkinson disease [J]. JAMA, 2006, 296(6):661-670.
[7] Ross OA, Braithwaite AT, Skipper LM, et al. Genomic investigation of alpha-synuclein multiplication and parkinsonism [J]. Ann Neurol, 2008, 63(6):743-750.
[8] Miller DW, Hague SM, Clarimon J, et al. Alpha-synuclein in blood and brain from familial Parkinson disease with SNCA locus triplication [J]. Neurology, 2004, 62(10):1835-1838.
[9] Matsumoto L, Takuma H, Tamaoka A, et al. CpG demethylation enhances alpha-synuclein expression and affects the pathogenesis of Parkinson’s disease [J]. PLoS One, 2010, 5(11):e15522.
[10] Tan YY, Wu L, Zhao ZB, et al. Methylation of α-synuclein and leucine-rich repeat kinase 2 in leukocyte DNA of Parkinson’s disease patients [J]. Parkinsonism Relat Disord, 2013, doi: 10.1016/j.parkreldis.2013.12.002. [Epub ahead of print].
[11] Jowaed A, Schmitt I, Kaut O, et al. Methylation regulates alpha-synuclein expression and is decreased in Parkinson‘s disease patients’ brains [J]. Neurosci, 2010, 30(18): 6355-6359.
[12] Wang Y, Wang X, Li R, et al. A DNA methyltransferase inhibitor, 5-aza-2′-deoxycytidine, exacerbates neurotoxicity and upregulates Parkinson’s disease-related genes in dopaminergic neurons[J]. CNS Neurosci Ther, 2013, 19(3):183-190.
[13] Clough RL, Dermentzaki G, Stefanis L. Functional dissection of the alpha-synuclein promoter: transcriptional regulation by ZSCAN21 and ZNF219 [J]. J Neurochem, 2009, 110(5):1479-1490.
[14] Brandes JC, Carraway H, Herman JG. Optimal primer design using the novel primer design program: MSPprimer provides accurate methylation analysis of the ATM promoter [J]. Oncogene, 2007, 26(42):6229-6237.
[15] Johnson AA, Akman K, Calimport SR, et al. The role of DNA methylation in aging, rejuvenation, and age-related disease [J]. Rejuvenation Res, 2012, 15(5):483-494.
[16] Tost J, Gut IG. DNA methylation analysis by pyrosequencing [J]. Nat Protoc, 2007, 2(9):2265-2275.
[17] Xuan Q, Xu SL, Lu DH, et al. Increased expression of α-synuclein in aged human brain associated with neuromelanin accumulation [J]. Neural Transm, 2011, 118(11):1575-1583.
[18] Mak SK, McCormack AL, Langston JW, et al. Decreased alpha-synuclein expression in the aging mouse substantia nigra [J]. Exp Neurol, 2009, 220(2):359-365.
