烟气脱硝尾液厌氧氨氧化处理中微生物群落结构分析
2014-07-19李津于德爽赵丹汪晓晨
李津,于德爽,赵丹,汪晓晨
青岛大学环境科学与工程学院,山东 青岛 266071
烟气脱硝尾液厌氧氨氧化处理中微生物群落结构分析
李津,于德爽,赵丹,汪晓晨
青岛大学环境科学与工程学院,山东 青岛 266071
李津, 于德爽, 赵丹, 等. 烟气脱硝尾液厌氧氨氧化处理中微生物群落结构分析. 生物工程学报, 2014, 30(12): 1865–1875.
Li J, Yu DS, Zhao D, et al. Microbial community in the Anammox process of thermal denitration tail liquid. Chin J Biotech, 2014, 30(12): 1865–1875.
采用厌氧序批式反应器处理火电厂烟气脱硝尾液并研究厌氧氨氧化微生物群落结构。通过扫描电镜观察发现,优势菌群为椭圆形的球菌且具有下凹的火山口状,直径约为0.7 μm。通过污泥总DNA提取、PCR扩增、阳性克隆验证和测序等分子生物学手段得到反应器内菌群16S rRNA基因序列,建立系统发育树和克隆文库。结果表明,在细菌通用引物27F-1 492R PCR扩增的体系中,得到85个克隆子,分为21个OTU,各菌群所占的比例为:变形菌门Proteobacteria:61.18%;酸杆菌门Acidobacteria:17.65%;绿菌门Chlorobi:8.24%;绿弯菌门Chlorofexi:5.88%;芽单胞菌门Gemmatimonadetes:3.53%;硝化螺旋菌门Nitrospirae:2.35%;浮酶状菌门Planctomycetes:1.18%。而在厌氧氨氧化菌特异引物pla46rc-630r; AMX368-AMX820 PCR扩增的体系中有45个克隆子,分为3个OTU,其中Candidatus brocadia sp.占有95.6%,未知菌种4.4%。
烟气脱硝尾液,厌氧氨氧化,16S rDNA克隆文库,群落结构,Candidatus brocadia sinica
厌氧氨氧化 (Anammox) 是一个自养过程,因不需要投加碳源可以节省运行成本[1-2]。厌氧氨氧化菌属于浮霉状菌目Planctomycetales的厌氧氨氧化菌科,该科有5个属,10个种,皆为化能自养型厌氧菌并且具有厌氧氨氧化功能[3]。在这5个厌氧氨氧化菌属中当前有4个Anammox菌属是从活性污泥中得到的,分别是“Kuenenia sp.”[4-5],“Brocadia sp.”[6],“Anammoxogiobus sp.”[7]和“Jettenia sp.”[8]。第5个厌氧氨氧化菌属“Candidatus scalindua”[9-10]通常可以在自然生境中发现,尤其是在海底沉积物和海洋最低含氧带。
Anammox菌形态多样,呈球形、卵形等,直径0.8−1.1 μm[11]。该菌是革兰氏阴性菌;细胞外无荚膜;细胞壁表面有火山口形状的结构;少数有菌毛。细胞内分隔成3部分:厌氧氨氧化体 (Anammoxsome)、核糖细胞质 (Riboplasm)及外室细胞质 (Paryphoplasm)[12]。
Anammox菌是一类难培养微生物,其生长速率较慢,倍增时间约为11 d,采用稀释分离、平板划线分离、显微单细胞分离等传统微生物分离方法均未分离成功[13]。这为ANAMMOX菌的酶学研究以及功能基因研究等带来了不便。然而由于分子生物学技术可以直接从环境样品中提取微生物的总DNA,不需要对微生物进行分离培养,从而成为研究厌氧氨氧化菌的最佳手段,如多聚酶链式反应 (PCR) 、荧光原位杂交 (FISH) 、DNA克隆测序、及梯度凝胶电泳 (DGGE) 等。其中16S rRNA基因序列分析技术是分子生态学领域的一项重要技术,已被广泛用于研究微生物的分类、鉴定以及微生物群落分析,且在很多方面都取得了显著成果[14]。
火电厂烟气脱硝尾液具有较高的氨氮浓度(100−125 mg/L)、较低的B/C比 (低于0.1),较高的进水温度 (30−40 ℃),适合于采用Anammox工艺脱氮。试验采用ASBR反应器研究了火电厂烟气脱硝尾液Anammox过程,经过228 d培养及驯化实现了稳定的厌氧氨氧化运行。取少量厌氧氨氧化活性污泥,通过DNA提取、PCR扩增等一系列分子生物学手段获得反应器内菌群的16S rRNA基因序列,通过构建克隆文库法对反应器内微生物种群进行了多样性分析,为火电厂烟气脱硝尾液的厌氧氨氧化处理提供指导。
1 材料与方法
1.1 污泥来源
实验污泥取自水污染控制实验室稳定运行的ASBR厌氧氨氧化反应器,反应器装置如图1所示。反应器采用有机玻璃制成,有效容积7 L,反应器外壁采用黑布包裹,通过水浴加热,使反应器内温度控制在35 ℃。反应器进水氨氮和亚硝酸盐氮分别为115 mg/L和140 mg/L左右,进水pH值为7.5,水力停留时间为8 h,混合液污泥浓度保持在3 500 mg/L。在此条件下,反应器对氨氮和亚硝酸盐氮的去除率分别为91.5%和97.0%,脱氮性能良好。其中进出水各项指标如表1所示。

图1 ASBR厌氧氨氧化反应器流程图Fig. 1 Flowchart of ASBR Anammox reactor.

表1 进出水各项指标Table 1 Indicators of influent and effluent
1.2 主要试剂及仪器
WisespinCF-1离心机;BIO-RAD PCR扩增仪;DYY-7型电泳仪;水浴锅;台式紫外透射仪;移液枪;WH-3型漩涡振荡仪;微型漩涡混合仪;低速离心机;迷你离心机。
MO BIO土壤DNA提取试剂盒,2×Taq PCR MasterMix,普通琼脂糖凝胶DNA回收试剂盒(离心柱型),PMD18-T载体,大肠杆菌DH5α感受态细胞。核酸染料,MSP,ALUⅠ限制性内切酶。
引物由上海博尚生物科技有限公司合成,上游引物27F,Pla46rc, AMX368;下游引物1492R,630r,AMX820。引物的序列见表2。
1.3 方法
1.3.1 污泥总细菌DNA基因组的提取与PCR扩增
污泥总DNA基因组的提取按照土壤基因DNA提取试剂盒说明书操作,完成后取5 μL收集好的DNA溶液用1%的琼脂凝胶电泳检测,其余分装至微量离心管中−20℃保存。
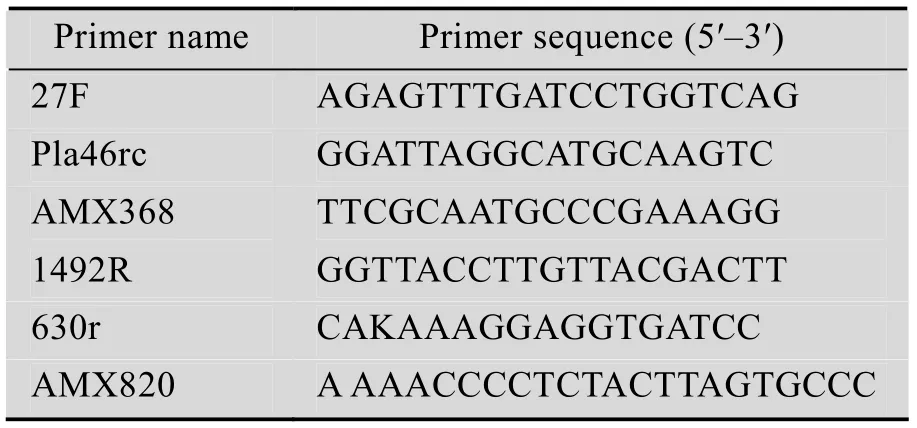
表2 引物序列Table 2 Sequences of primers
分别采用细菌通用引物 (27F-1492R) 和厌氧氨氧化特异引物 (pla46rc-630r; AMX368-AMX820) 对污泥样品总DNA进行16S rRNA基因片段的PCR扩增。其中通用引物扩增体系及反应程序如下:细菌16S rRNA基因片段的PCR反应体系 (50 μL):1 μL引物27F (10 μmol/L),1 μL引物1492R (10 μmol/L),25 μL 2×Taq PCR MasterMix,2 μL细菌DNA模板,适量ddH2O补至50 μL。PCR反应程序:95 ℃预变性5 min,94 ℃变性40 s,52 ℃复性40 s,72 ℃延伸90 s,30个循环,72 ℃延伸7 min。PCR产物用1%的琼脂糖凝胶电泳检测,PCR产物以普通琼脂糖凝胶DNA回收试剂盒进行纯化。
采用浮霉状菌通用引物和细菌通用引物对pla46rc-630r及厌氧氨氧化菌特异引物对AMX368-AMX820对污泥样品总DNA进行巢式PCR,首先采用pla46rc-630r引物对进行第一轮PCR扩增,将第一轮得到的目的片段条带纯化后再作为模板以引物对AMX368-AMX820进行第二轮扩增。PCR反应体系 (50 μL):1 μL上游引物 (10 μmol/L),1 μL下游引物(10 μmol/L),25 μL 2×Taq PCR MasterMix,2 μL细菌DNA模板,适量ddH2O补至50 μL。反应条件[15-16]:95 ℃预变性3 min;95 ℃变性1 min,56 ℃ (第一轮PCR 扩增) /52 ℃ (第二轮PCR扩增) 退火1 min,72 ℃延伸1 min,30个循环;72 ℃延伸10 min。PCR 产物用1%的琼脂糖凝胶电泳进行检测。
1.3.2 PCR产物的连接与克隆
微量离心管中依次加入:1 μL PMD18-T,2 μL PCR纯化产物,2 μL ddH2O,5 μL SolutionⅠ(EDTA 5g/L,FeSO4·7H2O 5g/L),低速离心后16 ℃反应16 h。然后全量 (10 μL) 加入至50 μL DH5α感受态细胞中冰浴30 min,42 ℃加热60 s,再在冰中放置1 min,加入890 μL LB培养基,37 ℃振荡培养60 min。最后取50 μL上述连接产物涂布至含有X-gal、IPTG、Amp的LB固体培养基上37 ℃培养14 h后进行蓝白筛选,随即挑选150个白色菌株 (细菌通用引物体系挑选100个,厌氧氨氧化特异引物体系挑选50个) 接种至5 mL含有Amp的LB液体培养基中,37 ℃振荡培养12 h。

图2 厌氧氨氧化菌颗粒污泥的扫描电镜照片Fig. 2 SEM photo of Anammox bacteria granular sludge.
1.3.3 PCR法验证阳性克隆及酶切
取200 μL上述菌液进行沸水浴10 min,冰浴5 min制得PCR反应模板。PCR反应体系(50 μL):1 μL引物M-13-47 (10 μmol/L),1 μL引物RV-M (10 μmol/L),25 μL 2×Taq PCR MasterMix,2 μL细菌DNA模板,适量ddH2O补至50 μL。反应条件:94 ℃ 3 min ,94 ℃ 30 s,55 ℃ 45 s,72 ℃ 1 min 30 s ,32个循环,72 ℃5 min。PCR产物用1%的琼脂糖凝胶电泳检测。
采用MSP,ALUⅠ两种限制性内切酶对挑选出的阳性克隆进行酶切反应 (37 ℃,2 h),将两种酶切条带都分别相同的克隆子认定为同一个菌种。酶切结果用1%的琼脂糖凝胶电泳检测。
2 结果
2.1 形态学观察
取样时反应器内污泥总体呈现红褐色,在取样瓶壁上附着很多红色颗粒污泥,挑选红色颗粒进行扫描电镜结果如图2所示,颗粒内的菌群为椭圆形球菌聚集体。球形菌具有下凹的火山口状,直径约为0.7 μm,该结果与多数文献报道的厌氧氨氧化菌呈球形、卵形,直径在0.8−1.1 μm的十分相似[17-19]。
2.2 污泥细菌总DNA提取
污泥样品提取的总DNA经凝胶电泳检测,测得DNA浓度为:50.5 ng/μL。A260/A280=1.619,该比值用于评估所提取的DNA纯度,一般认为A260/A280在1.8±0.2范围内说明提取的DNA较为纯净,比值过低表示存在蛋白质或者酚类物质的影响。本次总DNA该比值为1.619,说明所提取的DNA纯度较高,不需要纯化即可进行后续实验。
2.3 细菌16S rRNA基因片段的PCR扩增
采用细菌通用引物27F-1492R,扩增结果产生一条1 500 bp左右的条带,与预期结果一致。采用厌氧氨氧化菌特异引物 (pla46rc-630r) 以及 (AMX368-AMX820),得到目的条带在500 bp左右。张亚平等[20]采用相同引物得到目的片段大小为470 bp左右,与本实验结果相似。本研究采用巢式PCR对厌氧氨氧化菌进行分子生物学检测,第二轮扩增时上游和下游引物均采用了Anammox菌的特异引物,该特异引物可以扩增出不同种类的Anammox菌序列[1,20],从而对反应器内厌氧氨氧化菌的多样性分析更全面。
2.4 克隆文库分析
经过阳性克隆筛选,采用通用引物对27F-1492R的PCR体系共得到85个阳性克隆子,经MSP,ALUⅠ酶切后共得到30个酶切类型,每个酶切类型挑选1−2个阳性克隆子送去测序,得到45个有效序列。去除两端的载体序列,所得序列长度均在1 400−15 00 bp之间。将测序得到的序列输入GenBank数据库中进行Blast同源性比对,寻找相似度最高及具有明确分类地位的已知16S rRNA基因序列,将所测序列与已知序列用Mega软件进行聚类分析,采用Neighbor-Joinin法构建系统发育树,结果如图3所示。将序列比对结果与同一个相关序列相似度相同的克隆子定义为一个操作单元 (OTU),共得到21个OTU,建立克隆文库表格如表3所示。
通过Blast检索发现,目前鉴定的厌氧氨氧化菌16S rRNA基因全序列中没有与引物27F-1492R相匹配的序列,所以认为引物27F-1492R对厌氧氨氧化菌并不具有通用性。文库中主要菌群种类分布在变形菌门Proteobacteria,酸杆菌门Acidobacteria,绿菌门Chlorobi,绿弯菌门Chlorofexi,芽单胞菌门Gemmatimonadetes浮霉状菌门Planctomycetes及硝化螺旋菌门Nitrospirae。

图3 反应器内细菌克隆的系统发育树Fig. 3 Phylogenetic tree of bacteria in the reactor.
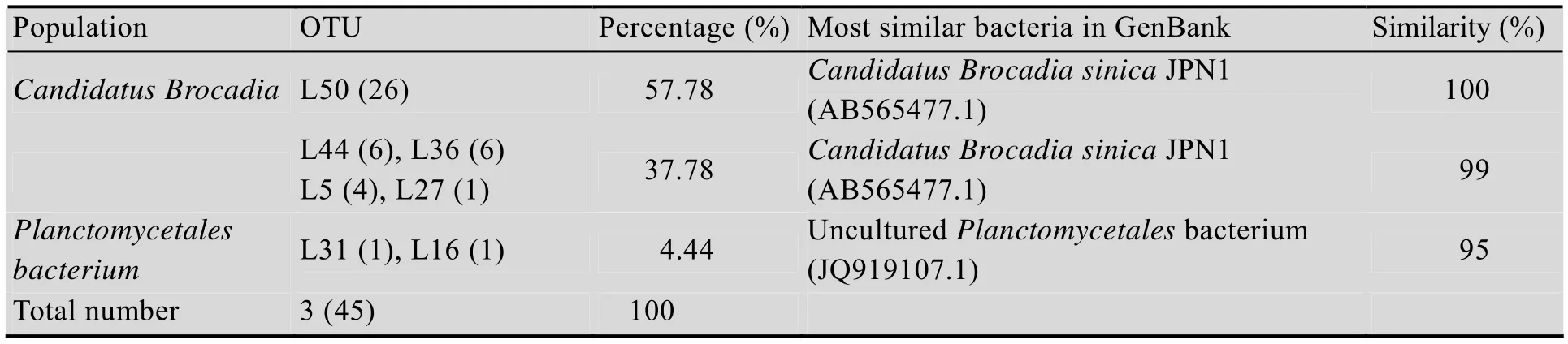
表3 27F-1492R体系16S rDNA克隆文库结果Table 3 16S rDNA clone library results of 27F-1492R system
其中变形菌门Proteobacteria所占的比例最高,共52个克隆子,丰度为61.18%,为样品污泥除厌氧氨氧化菌之外的优势菌群。变形菌门中检测到两个亚纲的菌群,β-Proteobacteria和γ-Proteobacteria,其中β-Proteobacteria内菌种占变形菌门的84.62%,是变形菌门中的主要亚纲,γ-Proteobacteria有8个克隆子,占变形菌门的15.38%。在β-Proteobacteria中归属红环菌科Rhodocyclaceae的克隆子有35个,伯克氏菌科Burkholderiaceae有9个克隆子。归属γ-Proteobacteria的主要菌属有肠杆菌属Enterobacter sp. 和不动杆菌属Acinetobacter sp.以及Raoultella sp.。
酸杆菌门Acidobacteria是反应器内除了变形菌门的另一大菌群,共有15个克隆子占17.65%的比例。其余菌门按比例依次是绿菌门Chlorobi为8.24%,绿弯菌门Chlorofexi为5.88%,芽单胞菌门Gemmatimonadetes为3.53%,硝化螺旋菌门Nitrospirae为2.35%。浮霉状菌门Planctomycetes为1.18%。
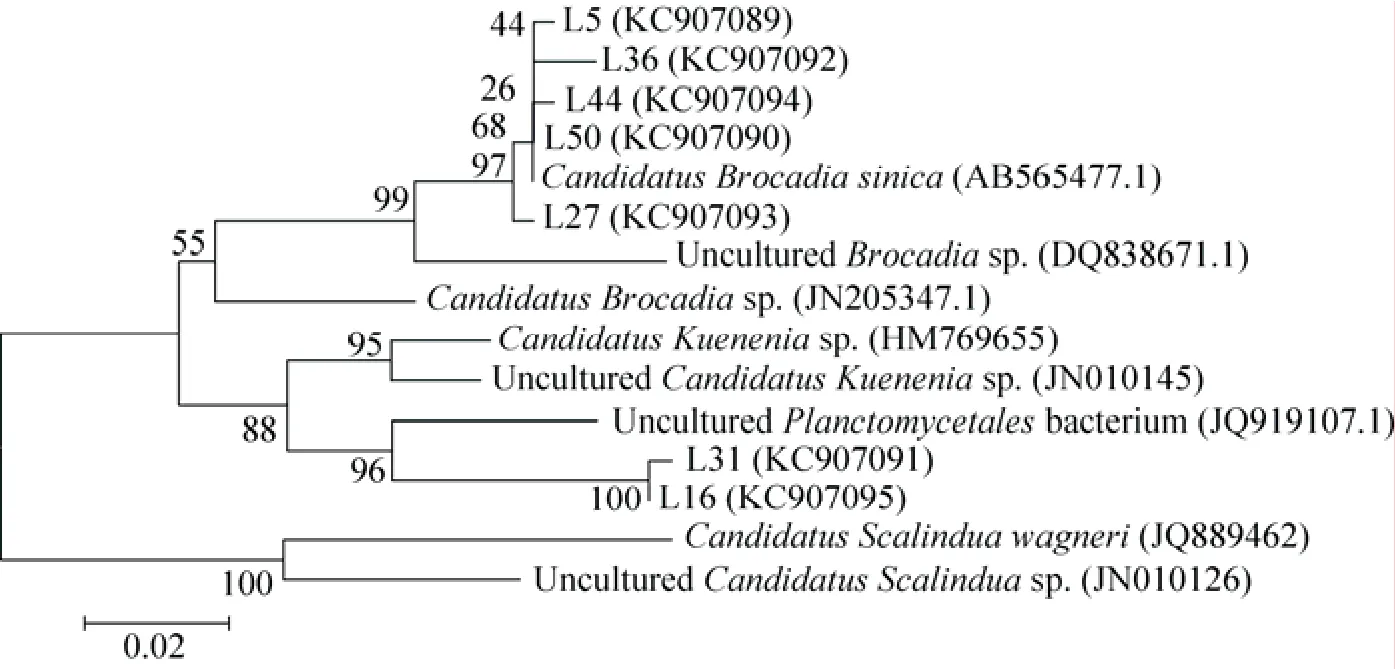
图4 反应器内厌氧氨氧化菌克隆的系统发育树Fig. 4 Phylogenetic tree of anaerobic ammonia oxidizing bacteria in the reactor.
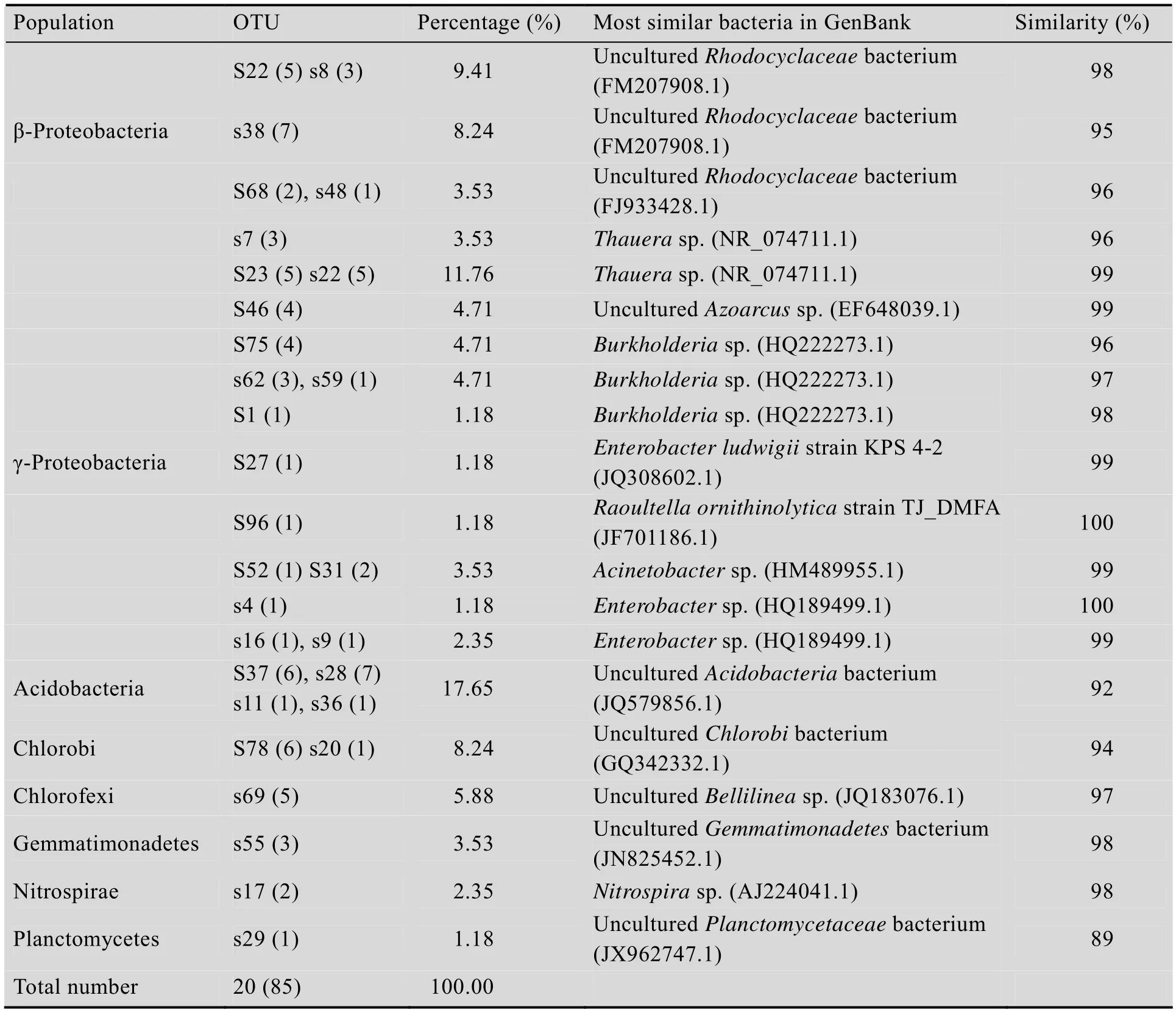
表4 pla46rc-630r,AMX368-AMX820体系16S rDNA克隆文库结果Table 4 16S rDNA clone library results of pla46rc-630r, AMX368-AMX820 system
采用厌氧氨氧化菌特异引物(pla46rc-630r,AMX368-AMX820) 的体系中,共挑选出45个阳性克隆,经酶切后共有7个酶切类型,每个酶切类型挑选1−2个阳性克隆子测序,共得到9个有效序列,根据这些序列在GenBank中检索结果构建的系统发育树和克隆文库如图4和表4所示。厌氧氨氧化反应器内主要的厌氧氨氧化菌群分为3个OTU,其中分布在Candidatus Brocadia sp. 的占2个OTU,有43个克隆子,占总克隆子数的95.6%,与相关序列的相似度分别为100%和99%,说明这43个克隆子所代表的菌株基本属于Candidatus Brocadia sp.,其中有26个克隆子与Candidatus Brocadia sinica JPN1同源相似度达100%,可以认为反应器内存在的大部分厌氧氨氧化菌属于Candidatus Brocadia sinica。而第3个OTU克隆子数为2个,占4.4%;与Uncultured Planctomycetales bacterium的相似度为95%,说明这两个菌种可能是尚未被鉴定的菌种。
3 讨论
采用厌氧氨氧化工艺处理烟气脱硝尾液,反应器内除了厌氧氨氧化菌外还有其他菌群。其中,变形菌门中的β-Proteobacteria菌群占的比例最大。已有研究表明,β-Proteobacteria 是脱氮活性污泥系统中数量最丰富的优势菌群[21],其大多数趋向于在厌氧环境下生存[22]。本研究发现的β-Proteobacteria的菌群主要分布在红环菌目Rhodocyclates和伯克氏菌目Brukholderiales,这与方芳等[23]的研究结果类似。本研究的红环菌目Rhodocyclates中存在有Thauera sp.,而该菌属属于反硝化细菌[24-25],说明反硝化作用也在烟气脱硝尾液的脱氮过程中发挥作用。
除了变形菌门,反应器内还存在着酸杆菌门、绿菌门、绿弯菌门、芽单胞菌门、浮霉状菌门和硝化螺旋菌门。研究表明,绿弯菌门是厌氧污泥消化器中的主要微生物[26],并且厌氧氨氧化系统中存在绿弯菌门细菌对反应器内污泥的颗粒化起到一定的作用[5,27]。然而本研究中污泥始终呈松散状态,没有颗粒污泥的形成,这或许与本研究所采用的烟气脱硝尾液特殊的废水水质有关。
采用厌氧氨氧化工艺处理烟气脱硝尾液,厌氧氨氧化反应器内95.6%的厌氧氨氧化菌属于Candidatus Brocadia sp.,其中57.8%与Candidatus Brocadia sinica的同源相似性达100%。有研究表明,在某一特定的生长环境中,厌氧氨氧化菌的生物多样性较低,通常只有一个种属的厌氧氨氧化细菌占据优势[7,28],每种厌氧氨氧化菌都有特定的生存环境。Candidatus Brocadia sinica更适合处理烟气脱硝尾液。
4 结论
采用ASBR反应器处理火电厂烟气脱硝尾液并研究了Anammox微生物群落结构。通过扫描电镜观察发现,优势菌群为椭圆形的球菌且具有下凹的火山口状,直径约为0.7 μm。通过构建克隆文库发现厌氧氨氧化反应器内95.6%的厌氧氨氧化菌属于Candidatus Brocadia sp.,Candidatus Brocadia sinica更适合处理烟气脱硝尾液。反应器内除了厌氧氨氧化菌外还有其他菌群,其中变形菌门中的β-Proteobacteria菌群占的比例最大,反硝化作用也在烟气脱硝尾液的脱氮过程中发挥作用。
REFERENCES
[1] van de Graff AA, Mulder A, de Bruijn P, et al. Anaerobic oxidation of ammonium is a biologically mediated process. Appl Environ Microb, 1995, 61(4): 1246−1251.
[2] Abma WR, Schultz CE, Mulder JW, et al. Full-scale granular sludge anammox process. Water Sci Technol, 2007, 55: 27−33.
[3] Starr MP, Schmidt JM. Bergey's Manual of Systematic Bacteriology. New York: Springer, 1995: 56−57.
[4] Schmid M, Twachtmann U, Klein M, et al. Molecular evidence for genus level diversity of bacteria capable of catalyzing anaerobic ammonium oxidation. Syst Appl Microbiol, 2000, 23(1): 93−106.
[5] Strous M, Pelletier E, Mangenot S, et al. Deciphering the evolution and metabolism of an ANAMMOX bacterium from a community genome. Nature, 2006, 440(6): 790−794.
[6] Kuenen JG, Jetten MSM. Extraordinary anaerobic ammonium-oxidizing bacteria. Appl Environ Microb, 2001, 67: 456−463.
[7] Kartal B, Rattray J, van Niftrik LA, et al.Candidatus “Anammoxoglobus propionicus” a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria. Syst Appl Microbiol, 2007, 30(1): 39−49.
[8] Quan ZX, Rhee SK, Zuo JE, et al. Diversity of ammonium-oxidizing bacteria in a granular sludge anaerobic ammonium-oxidizing (anammox) reactor. Environ Microb, 2008, 10(11): 3130−3139.
[9] Kuypers MMM, Slikers AO, Lavik G, et al. Anaerobic ammonium oxidation by anammox bacteria in the Black Sea. Nature, 2003, 422: 608−611.
[10] Schmidt I, Sliekers O, Schmid M, et al. New concepts of microbial treatment processes for the nitrogen removal in wastewater. FEMS Microbiol Rev, 2003, 27(4): 481−492.
[11] van Niftrik L, Geerts WJC, van Donselaar EG, et al. Linking ultrastructure and function in four genera of anaerobic ammonium-oxidizing bacteria: cell plan, glycogen storage, and localization of cytochrome cproteins. J Bacteriol, 2008, 190(2): 708−717.
[12] van Niflrik LA, Fuerst JA, Damste JS, et al. The anammoxosome: an intracytoplasmic compartment in anammox bacteria. FEMS Microbiol Lett, 2004, 233(1): 7−13.
[13] Strous M, Heijnen JJ, Kuenen JG, et al. The sequencing batch reactor as a powerful tool for the study of slowly growing anaerobic ammoniumoxidizing microorganism. Appl Microbiol Biotechnol, 1998, 50: 589−596.
[14] Tan FX, Ye C. Molecular identification of 2 strains of Amromonas based on 16S rDNA sequence. J Yangtze Univ, 2011, 8(7): 257−262 (in Chinese).谭凤霞, 叶聪. 基于16S rDNA序列的2株单胞菌属细菌的分子鉴定. 长江大学学报, 2007, 8(7): 257−262.
[15] Shen LD, Hu BL, Zheng P, et al. Molecular detection of anammox bacteria in the sediment of West Lake, Hangzhou. Acta Sci Circum, 2011, 31(8): 1609−1615 (in Chinese).沈李东, 胡宝兰, 郑平, 等. 西湖底泥中厌氧氨氧化菌的分子生物学检测. 环境科学学报, 2011, 31(8): 1609−1615.
[16] Fan GN, Zhu GB, Wang Y, et al. New functional microorganisms in nitrogen cycle restoration of river riparian ecosystems. Acta Sci Circum, 2010, 30 (8) : 1558–1563 (in Chinese).范改娜, 祝贵兵, 王雨, 等. 河流湿地氮循环修复过程中的新型功能微生物. 环境科学学报, 2010, 30(8): 1558−1563.
[17] Dalsgaard T, Canfield DE, Petersen J, et al. N2production by the anammox reaction in the anoxic water column of Golfo Dulce, Costa Rica. Nature, 2003, 422(6932): 606−608.
[18] Jetten M, Strous M, van de Pas-Schoonen KT, et al. The anaerobic oxidation of ammonium. FEMS Microbiol Rev, 1999, 22(6): 421−437.
[19] Egli K, Bosshard F, Werlen C, et al. Microbial composition and structure of a rotating biological contactor biofilm treating ammonium-rich wastewater without organic Carbon. Microb Ecol, 2003, 45(4): 419−432.
[20] Zhang YP, Luan XH. Molecular evidence for ammonia oxidation bacteria in the sediments of shallow lake: A case study in Yangcheng Lake.Acta Sci Circum,2012, 32(1): 182−189 (in Chinese).张亚平, 阮晓红. 浅水湖泊(阳澄湖)沉积物氨氧化菌的分子证据. 环境科学学报, 2012, 32(1): 182−189.
[21] Lee HW, Lee SY, Lee JW, et al. Molecular characterization of microbial community in nitrate-removing activated sludge. FEMS MicrobEcol, 2002, 41(2): 85−94.
[22] Tao TS, Yang RF, Dong XZ, et al. Systematics of prokaryotes. Beijing: Chemistry Industry Press, 2007: 97−99 (in Chinese).陶天申, 杨瑞馥, 东秀珠, 等. 原核生物系统学.北京: 化学工业出版社, 2007: 97−99.
[23] Fang F, Wang SM, Feng CJ, et al. Community structure of denitrifying bacteria in a hybrid AS-biofilm process under aerobic condition. Chin J Ecol, 2011, 30(3): 430−437 (in Chinese).方芳, 王淑梅, 冯翠杰, 等. 好氧条件下复合生物膜-活性污泥反应器中的反硝化菌群结构. 生态学杂志, 2011, 30(3): 430−437.
[24] Wang YH, Ma XY, Liu CF, et al. The bacterial diversity in boifilms associated with media of anammox bioreactors. J Dalian Ocean Univ, 2011, 26(6): 500−506.王银华, 马欣悦, 刘长发, 等. 厌氧氨氧化反应器载体生物膜细菌多样性的研究. 大连海洋大学学报, 2011, 26(6): 500−506.
[25] Liu B, Zhang F, Feng X, et al. Thauera and Azoarcusas functionally important genera in a denitrifying quinoline-removal bioreactor as revealed by microbial community structure comparison. FEMS Microb Ecol, 2006, 55(2): 274−286.
[26] Rivière D, Desvignes V, Pelletier E, et al. Towards the definition of a core of microorganisms involved in anaerobic digestion of sludge. ISME J, 2009, 3(6): 700−714.
[27] Li XR, Du B, Fu HX, et al. The bacterial diversity in an anaerobic ammonium-oxidizing (ANAMMOX) reactor community. Syst Appl Microbiol, 2009, 32(4): 278−289.
[28] van de Vossenberg J, Rattray JE, Geerts W, et al. Enrichment and characterization of marine anammox bacteria associated with global nitrogen gas production. Environ Microb, 2008, 10(11): 3120−3129.
(本文责编 郝丽芳)
Microbial community in the Anammox process of thermal denitration tail liquid
Jin Li, Deshuang Yu, Dan Zhao, and Xiaochen Wang
School of Environmental Science and Engineering, Qingdao University, Qingdao 266071, Shandong, China
An anaerobic sequencing batch reactor (ASBR) was used to treat thermal denitration tail liquid and microbial community was studied. Activated sludge was taken from the reactor for scanning electron microscope analysis. The images showed that the dominant cells in the flora were oval cocci. Its diameter was about 0.7 μm. Through a series of molecular biology methods such as extracting total DNA from the sludge, PCR amplification, positive clone authentication and sequencing, we obtained the 16S rDNA sequences of the flora. Phylogenetic tree and clone library were established. The universal bacteria primers of 27F-1492R PCR amplification system obtained 85 clones and could be divided into 21OTUS. The proportions were as follows: Proteobacteria 61.18%; Acidobacteria 17.65%; Chlorobi 8.24%; Chlorofexi 5.88%; Gemmatimonadetes 3.53%; Nitrospirae 2.35% and Planctomycetes 1.18%. The specific anammox bacterial primers of pla46rc-630r and AMX368-AMX820 PCR amplification system obtained 45 clones. They were divided into 3 OTUS. Candidatus brocadia sp. occupied 95.6% and unknown strains occupied 4.4%.
thermal denitration tail liquid, Anammox, 16S rDNA clone library, molecular community, Candidatus brocadia sinica
April 3, 2014; Accepted: August 13, 2014
Jin Li. Tel: +86-532-85955529; E-mail: ljin0532@126.com
Supported by: National Natural Science Foundation of China (Nos. 51278258, 51478229), Qingdao Municipal Applied Fundamental Research Program (No. 13-1-4-203-jch).
国家自然科学基金 (Nos. 51278258, 51478229),青岛市应用基础研究项目 (No. 13-1-4-203-jch) 资助。
Received: April 3, 2014; Accepted: August 13, 2014
Supported by: National Natural Science Foundation of China (Nos. 51278258, 51478229), Qingdao Municipal Applied Fundamental Research Program (No. 13-1-4-203-jch).
Corresponding author: Jin Li. Tel: +86-532-85955529; E-mail: ljin0532@126.com
国家自然科学基金 (Nos. 51278258, 51478229),青岛市应用基础研究项目 (No. 13-1-4-203-jch) 资助。
