兔豆状囊尾蚴TpRS1基因的克隆及其序列分析
2014-07-12韩乐乐李作磊苟惠天孙晓林
韩乐乐 李作磊 苟惠天 孙晓林
(甘肃农业大学动物医学院,兰州 730070)
豆状囊尾蚴是带科(Taeniidae)带属(Taenia)的豆状带绦虫(Taenia pisiformis)的中绦期蚴虫,常寄生于家兔等啮齿动物的肝脏、大网膜、肠系膜和腹腔内引起其豆状囊尾蚴病[1]。当终末宿主(如犬、狼、狐等)吞食了感染豆状囊尾蚴的中间宿主组织后,豆状囊尾蚴在其小肠中发育为豆状带绦虫[2]。该病呈世界性分布[3-5],我国各地均有发生[6]。该病的流行,不仅对公共卫生及兔肉食品安全构成严重威胁,而且给养殖业的发展造成严重影响。
目前,带科绦虫免疫诊断抗原的研究主要集中在一些低分子量抗原方面[7-10]。研究证明,在带科绦虫囊尾蚴病诊断中具有较好应用价值,如10、14和18 kD等抗原都属于8 kD基因家族成员,只是糖基化修饰程度的差异而形成大小不同的蛋白分子。Ferrer等[7,11]对 8 kD抗原家族基因进行研究发 现,Ts8ku糖蛋白显示出良好的免疫反应性,在猪囊尾蚴病诊断方面具有较高的应用价值。Sako等[12]使用多房棘球蚴Em18重组抗原建立的免疫层析检测方法敏感性和特异性均达到94%以上。Yong等[13]对猪带绦虫RS1基因进行研究,发现其能够与宿主的脂肪酸结合从而获得能源,有助于绦虫在宿主体内寄生,然而这种结合作用同时会受到RS1抗原的特异性抗体的抑制作用。Lee等[14]通过研究猪带绦虫的150 kD疏水性结合蛋白(HLBP)调节脂肪酸吸收的机制,结果表明HLBP调节宿主脂肪酸的吸收可能是寄生虫生存的关键因素。迄今为止,国内外对有关豆状囊尾蚴(Cysticercus pisiformis)免疫学诊断方面的研究工作尚少,仅有1992年倪泽成等对其粗提抗原进行研究的初步报道,但粗提抗原在纯化方法上的缺陷限制了其推广应用[7]。
本研究初次以低分子量抗原RS1为靶基因,通过对GenBank中登录的猪带科绦虫RS1基因序列进行比对,选择保守区设计引物,进行RT-PCR,克隆到具有完整开放阅读框(ORF)的cDNA分子RS1,旨在通过对目的基因及其编码的蛋白结构和特征进行全面的生物信息学分析,为该基因的克隆表达及免疫学特性的初步研究提供理论依据,从而为其生物学功能研究及在免疫诊断与疫苗方面的应用奠定基础。
1 材料与方法
1.1 材料
1.1.1 虫株与菌株 豆状囊尾蚴采自人工感染兔的大网膜等处;无菌生理盐水冲洗3-4次,-70℃保存备用。E. coliJM109由本实验室保存。pMD18-T克隆载体购自大连宝生物工程有限公司。
1.1.2 主要试剂 Trizol试剂购自Invitrogen公司;RNA simple Total RNA Kit、TIANgel Midi Purification Kit和质粒小量提取试剂盒购自天根生化科技(北京)有限公司;TaqDNA聚合酶、DL2000 Marker购自北京康润诚业生物科技有限公司;其他常规试剂为进口或国产分析纯产品。
1.2 方法
1.2.1 兔豆状囊尾蚴TpRS1基因的克隆 根据GenBank中登录的猪带绦虫TSOL RS1基因序列(登录号DQ8650721),用Oligo软件设计一对引物,上游引物:5'-ATGCGTGCCTACATTGTGCTT-3';下游引物:5'-GCATGAAAGTTGACAAGTTAAGCAG-3',引物由上海生工生物工程有限公司合成。按照Trizol试剂说明抽提兔豆状囊尾蚴总RNA。将提取的总RNA按照RNA simple Total RNA Kit说明合成cDNA,用上下游引物进行PCR扩增。反应程序为:94℃ 4 min;94℃ 30 s,50℃ 30 s,72℃ 45 s,35个循环;72℃10 min。取5 μL PCR产物,于1.5%琼脂糖凝胶中电泳,检测扩增片段的长度。电泳检测后目的基因按TIANgel Midi Purification Kit试剂盒操作说明回收纯化。将纯化后的PCR产物与pMD18-T克隆载体于4℃连接过夜,将连接产物转化至E. coliJM109感受态细胞中,并涂布于Amp+LB固体平板,37℃培养14-16 h,鉴定为阳性克隆送测序公司测序。
1.2.2 兔豆状囊尾蚴TpRS1基因的ORF序列分析 在 http://www.ncbi.nlm.nih.gov/g orf/gorf.html上利用ORF finder软件确定兔豆状囊尾蚴基因TpRS1完整的ORF序列,并推导编码蛋白的氨基酸序列。
1.2.3 兔豆状囊尾蚴TpRS1基因的生物信息学分析
1.2.3.1 基本理化性质分析 利用Protparam(http://expasy.org/tools/protparam.html)计算蛋白质的理论分子质量、理论等电点、氨基酸组成等。利用ProtScale(http://expasy.org/tools/protscale.html)分析蛋白质亲、疏水性。
1.2.3.2 信号肽和跨膜区分析 利用SignalP3.0(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽位点。应用TMHMM Serverv.2.0(http://www.cbs.dtu.dk/services/TMHMM/)在线分析程序预测跨膜区域。
1.2.3.3 二级结构和功能预测 应用Predictprotein(http://www.predictprotein.org)对蛋白二级结构及抗原表位作进一步分析。应用MotifScan(http://hits.isb-sib.ch/cgi-bin/PFSCAN)预测蛋白糖基化位点。
1.2.3.4 同源性比较及系统进化分析 用BlastP软件对兔豆状囊尾蚴TpRS1氨基酸序列与GenBank中其他带科绦虫的同源性进行比对。运用MEGA5中的Maximum parsimony(MP)法软件构建系统进化树并进行分析。
2 结果
2.1 兔豆状囊尾蚴TpRS1基因的克隆
以提取的兔豆状囊尾蚴总RNA为模板进行RTPCR扩增,经1.5%琼脂糖凝胶电泳观察,在约250 bp出现特异性条带(图1),与理论预测值大小相符。

图1 PCR产物鉴定
2.2 兔豆状囊尾蚴TpRS1基因的ORF序列分析
使用NCBI的ORF Finder程序对克隆的兔TpRS1基因序列进行开放性阅读框分析并推测其氨基酸序列,结果(图2)显示,TpRS1含有长度为258 bp的开放阅读框,含有起始密码子ATG和终止密码子TAA,编码85个氨基酸。
2.3 兔豆状囊尾蚴TpRS1蛋白的生物信息学分析
2.3.1 理化性质 TpRS1所编码的85个氨基酸残基组成的多肽的理论分子质量为9.6 kD,理论等电点为9.07,原子组成是C431H710N110O122S6。该蛋白质由19种氨基酸组成,其中Lys含量最丰富,不含Gln,负电荷残基(Asp+Glu)12个,正电荷残基(Arg+Lys)15个。脂肪系数为95.29,在溶液中的不稳定指数(Instability index)为37.58,低于阈值40,表明该蛋白状态稳定。
2.3.2 TpRS1基因编码蛋白的疏水性/亲水性预测和分析 运用ProtScale工具对兔豆状囊尾蚴TpRS1基因编码蛋白进行亲水性和疏水性分析。依据氨基酸分值越低亲水性越强和分值越高疏水性越强的规律可知:兔豆状囊尾蚴TpRS1基因编码蛋白多肽链第22位Pro具有最低的分值-1.600和最强的亲水性;第87位Cys具有最高的分值3.133和最强的疏水性。平均亲水性为-0.021,表明该蛋白可能是一个疏水性蛋白(图3)。该结果与DNASTAR软件分析结果基本一致。
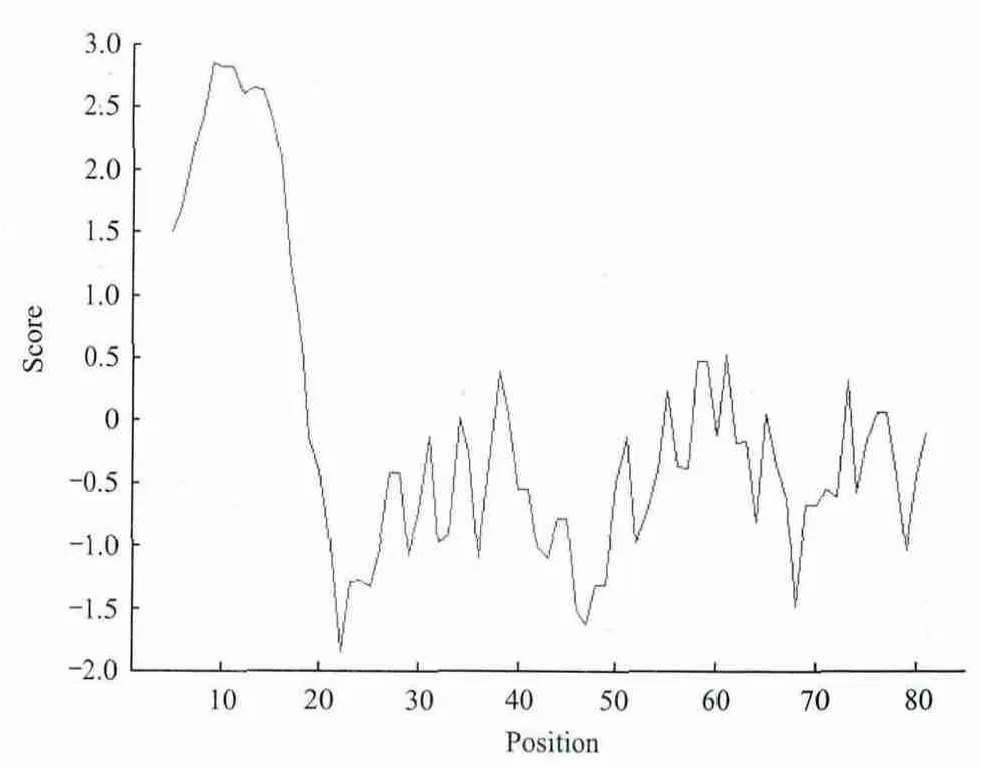
图3 TpRS1基因编码蛋白疏水性分析
2.3.3 信号肽和跨膜区分析 用SignalP 3.0对TpRS1基因编码蛋白进行信号肽分析,结果表明该蛋白不存在信号肽,不属于分泌蛋白。用TMHMM2.0分析跨膜结构发现此编码蛋白所有氨基酸部分位于膜表面,部分位于膜内(图4),证实TpRS1基因编码蛋白是一种跨膜蛋白。
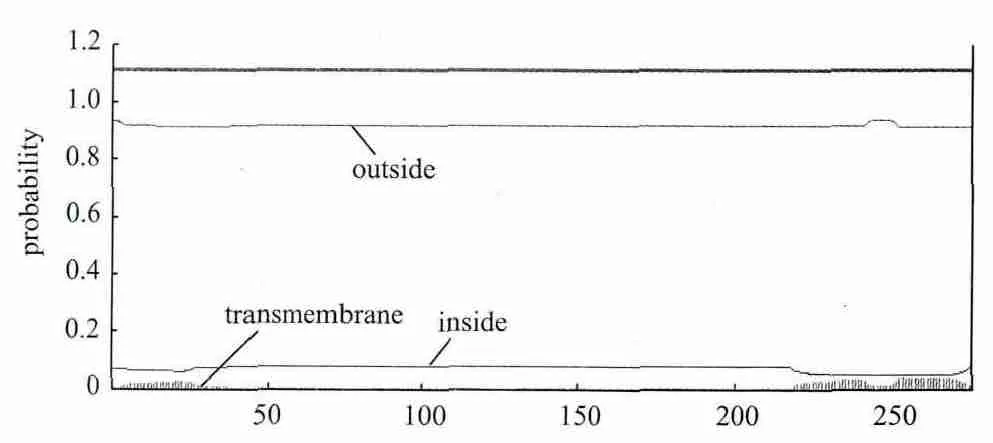
图4 TpRS1基因编码蛋白的信号肽分析
2.3.4 二级结构及其蛋白功能的预测 预测结果(图5)显示,该蛋白的二级结构中α螺旋占70.59%,β折叠占5.88%,其余23.53%为无规卷曲,属于混合型二级结构。该蛋白基序无N-糖基化位点。抗原位点预测显示,其氨基酸序列的2、44、45、66、72和76位为抗原表位(图6)。

图5 TpRS1二级结构的模拟图
2.3.5 TpRS1蛋白的同源性比对及系统进化分析 在GenBank进行Blast查询发现,TpRS1为尚未报道的新序列。通过与GenBank中登录的其他带科绦虫的氨基酸序列进行同源性分析显示,TpRS1编码的氨基酸序列与泡状带绦虫(ACI42356)的氨基酸序列相似性最高达到77%;与猪带绦虫(AEP03198)、多 头带绦虫(ACI42323)及细粒棘球蚴(AC132106)的序列相似性都为72%(图7)。
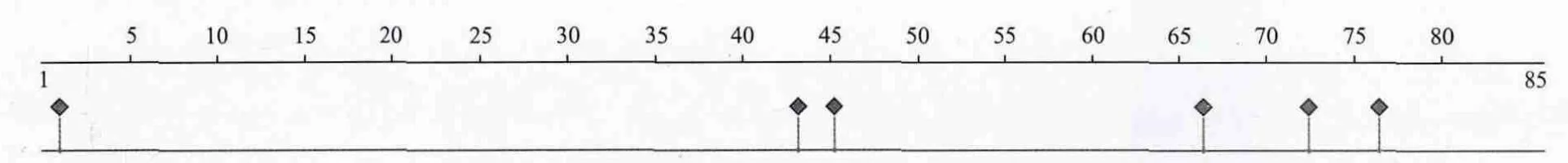
图6 TpRS1抗原位点的预测

图7 TpRS1推测的氨基酸序列与其他带科绦虫的同源性分析
根据BLAST比对结果,以细粒棘球蚴作为外群,选取部分绦虫的氨基酸序列构建系统进化树,TpRS1与泡状带绦虫氨基酸序列同源性最高(图8)。
3 讨论
豆状囊尾蚴病作为家兔的一种重要的寄生虫病,国内广泛流行,但相关研究尚少。目前,主要根据流行病学、临床症状及实验室检查等方面对豆状囊尾蚴病进行综合诊断[15]。免疫学诊断方面的研究并不多。免疫诊断依赖于高特异性的抗原。本试验选取低分子量抗原基因RS1进行克隆,首次克隆兔豆状囊尾蚴TpRS1基因,并最终成功构建了该基因的重组质粒,为获得兔豆状囊尾蚴TpRS1抗原蛋白奠定了基础。在GenBank进行Blast查询,发现该基因核苷酸序列的ORF与猪带绦虫RS1的相同,均为258 bp,含有起始密码子和终止密码子均为ATG和TAA,提示RS1基因可能由同一个祖先基因进化而来。
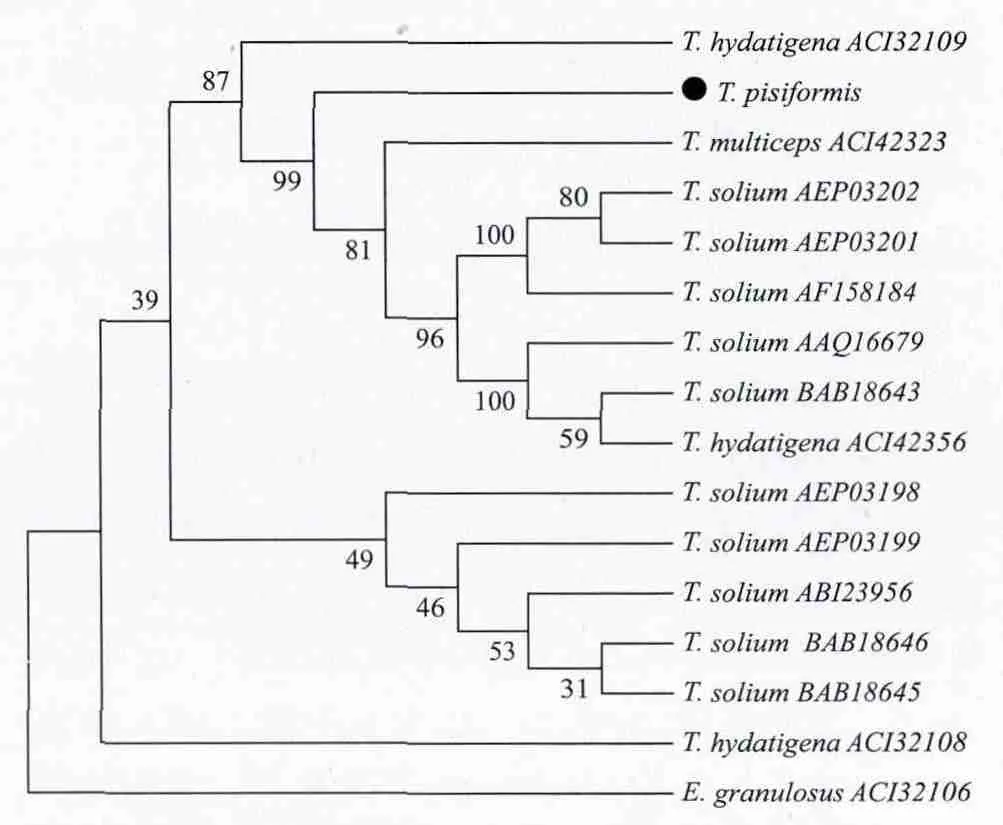
图8 MP法构建的TpRS1基因系统进化树
生物信息学是目前生物学领域发展最为迅速的学科,利用已建立的技术平台和大量的数据库体系,可以迅速准确地预测目的基因或蛋白质的特性、定位以及功能等关键信息。本试验从跨膜区分析、亲水疏水性分析、信号肽分析、抗原位点及二级结构分析对该蛋白的性质功能作出系统预测,TpRS1编码的氨基酸序列与泡状带绦虫的氨基酸序列最相似,同源性达77%;与猪带绦虫、多头带绦虫及细粒棘球蚴也有很高的同源性,都为72%。且与物种进化的结果相一致,反映了RS1基因编码产物在不同物种结构上的稳定性对生物体的功能重要性。预测发现其没有信号肽序列,说明该蛋白可能为包涵体表达蛋白,从而为蛋白质表达后蛋白的获得部分提供了依据。该蛋白为跨膜蛋白,有一定的跨膜区,这些区段一般会形成α螺旋,穿越细胞膜脂质双层结构而将该膜蛋白定位于细胞膜上,这与该蛋白二级结构中主要为α螺旋的预测结果一致。在机体内疏水性残基一般埋在蛋白内部,而亲水性残基位于表面,因此蛋白的亲水部位与蛋白的抗原位点有密切的联系,最高亲水性区域常位于抗原决定簇内部或其附近[16]。经亲水性与抗原表位的共同分析发现该蛋白可能是一个疏水性蛋白,但有6处抗原位点,由此揭秘出该蛋白具有强大的生物学功能,为进一步对该基因的表达及生物学功能的研究奠定基础,也为该蛋白的免疫诊断、疫苗方面的研究提供理论依据。
4 结论
本研究成功克隆了兔豆状囊尾蚴TpRS1基因,该基因序列含有一个由258个核苷酸组成的开放阅读框,编码85个氨基酸。利用生物信息学软件,成功预测并分析了TpRS1基因编码蛋白的基本理化性质、疏水性、信号肽、二级结构、抗原位点及与其他带科绦虫的同源性等。
[1] 汪明.家畜寄生虫学[M] . 第3版.北京:中国农业出版社,2003:117-118.
[2] Toral-Bastida E, Garza-Rodriguez A, Jimenez-Gonzalez DE, et al.Development ofTaenia pisiformisin golden hamster(Mesocricetus auratus)[J] . Parasit Vectors, 2011, 4:147.
[3] Kyngdon CT, Gauci CG, Rolfe RA, et al.In vitrooncosphere-killing assays to determine immunity to the larvae ofTaenia pisiformis,Taenia ovis,Taenia saginata, andTaenia solium[J] . J Parasitol,2006, 92(2):273-281.
[4] 杨世广, 汪志楷, 施宝坤.豆状带绦虫的人工感染及其发育史[J] .中国兽医科技, 1986, 16(6):16-19.
[5] 周永学, 杜爱芳, 张雪娟, 等.我国兔豆状囊尾蚴病的研究进展[J] .中国养兔杂志, 2006(1):25-28.
[6] 张守发, 鞠玉琳, 李学一, 等.不同抗原在兔豆状囊尾蚴病血清学诊断中的研究[J] .延边大学农学学报, 1994, 16(2):98-102, 108.
[7] Ferrer E, Bonay P, Cuevas MF, et al. Molecular cloning and characterisation of Ts8B1, Ts8B2 and Ts8B3, three new members of theTaenia soliummetacestode 8 ku diagnostic antigen family[J] .Mol Biochem Parasitol, 2007, 152(1):90-100.
[8] Assana E, Kanobana K, Tume CB, et al. Isolation of a 14 ku antigen fromTaenia soliumcyst fluid by HPLC and its evaluation in enzyme linked immunosorbent assay for diagnosis of porcine cysticercosis[J] . Res Vet Sci, 2007, 82(3):370-376.
[9] Hancock K, Khan A, Williams FB, et al.Characterization of the 8-kilodalton antigens ofTaenia soliummetacestodes and evaluation of their use in an enzyme-linked immunosorbent assay for serodiagnosis[J] . J Clin Microbiol, 2003, 41(6):2577-2586.
[10] Dang Z, Watanabe J, Kajino K, et al. Molecular cloning and characterization of a T24-like protein inEchinococcus multilocularis[J] .Mol Biochem Parasitol, 2009, 168(1):117-119.
[11] Ferrer E, Mart nez-Escribano JA, Barderas ME, et al. Peptide epitopes of theTaenia soliumantigen Ts8B2 are immunodominant in human and porcine cysticercosis[J] . Mol Biochem Parasitol,2009, 168(2):168-171.
[12] Sako Y, Fukuda K, Kobayashi Y, et al. Development of an immunochromatographic test to detect antibodies against recombinant Em18 for diagnosis of alveolarEchinococcosis[J] . J Clin Microbiol,2009, 47(1):252-254.
[13] Kim SH, Bae YA, Yang Y, et al. Paralogous proteins comprising the 150 kDa hydrophobic-ligand-binding-protein complex of theTaenia soliummetacestode have evolved non-overlapped binding affinities toward fatty acid analogs[J] . International Journal for Parasitology, 2011, 41(11):1207-1215.
[14] Lee EG, Kim SH, Bae YA, et al. A hydrophobic ligand-binding protein of theTaenia soliummetacestode mediates uptake of the host lipid:Implication for the maintenance of parasitic cellular homeostasis[J] . Proteomics, 2007, 7(21):4016-4030.
[15] 许伟.家兔囊虫病的诊断与防治[J] .畜牧兽医杂志, 2009,28(2):134.
[16] Hopp TP, Woods KR. Prediction of protein antigenic determinants from amino acid sequences[J] . Proc Natl Acad Sci USA, 1981,78(6):3824-3828.
