粉纹夜蛾中肠微生物中产AHLs群体感应信号分子菌株的分离鉴定
2014-07-12黄媛媛马宏贾振华黄亚丽宋水山张霞
黄媛媛 马宏 贾振华 黄亚丽 宋水山 张霞
(1.河北省科学院生物研究所,石家庄 050081;2.河北省主要农作物病害微生物控制工程技术研究中心,石家庄 050081)
农业生产中由害虫造成的损失占农作物总损失的15%以上,因此对害虫的防治是农业生产中的一项重要任务。传统的害虫防治主要为化学防治,但长期使用化学农药会影响人类健康并对环境造成污染。而生物农药以其简便有效,对环境和生物安全的优点,引起了世人的高度重视[1]。目前苏云金芽孢杆菌(Bacillus thuringiensis,Bt)是世界上研究最为深入、应用面最广的杀虫微生物。但在实际应用中由于长期使用Bt,使得多种害虫对Bt产生了抗性,其中夜蛾科害虫粉纹夜蛾(Trichoplusia ni)对Bt制剂产生抗性的防治尤为棘手[2]。群体感应(Quorum sensing,QS)系统是指细菌细胞根据密度变化进行基因表达调控的生理行为[3],群体感应参与多种生物学功能调节,如调控细菌毒性因子产生、抗生素合成、生物膜形成等[4],调节植物生长、提高植物抗病性、对耐非生物胁迫中的作用等[5]。近年来有研究发现昆虫中肠细菌也具有群体感应现象,2007年,Guan 等[6]鉴定了来自舞毒蛾幼虫中肠免培养细菌的信号分子合成基因,该研究属首次从免培养细菌中鉴定新的群体感应诱导子类别。之后,Borlee等[7]研究了菜粉蝶幼虫中肠土著微生物和致病微生物产群体感应信号分子的情况,该研究证实在菜粉蝶幼虫的碱性肠道环境中细菌之间的信号分子AHL是可以进行交流的,并且有利于PAO1的致病性。粉纹夜蛾作为昆虫中肠防御体系研究最为深入的昆虫,主要集中在中肠围食膜蛋白结构和Bt杀虫蛋白受体方面,而对粉纹夜蛾中肠微生物群体感应的研究尚未见系统报道。
从细菌群体感应现象出发,探索粉纹夜蛾中肠中产信号分子的微生物与Bt杀虫剂的相互作用,是强化Bt的杀虫作用、增强害虫生物防治效果的一个新途径,具有重要的理论和实际意义。本研究从粉纹夜蛾中肠中进行产群体感应信号分子菌株的筛选,通过形态观察,生理生化鉴定和分子生物学鉴定来确定该菌的种属,并分析其产生信号分子的种类。该研究的进行对明确和阐明粉纹夜蛾中肠细菌群体感应系统对Bt杀虫活性的可能作用与分子机理,有望为提高生物杀虫剂防效和预防害虫对生物杀虫剂的抗性提供新的途径与新的思路。
1 材料与方法
1.1 材料
大肠杆菌(Escherichia coli)DH5α,紫色杆菌(Chromobacterium violaceum)CV026由本实验室保存,pMD18-T购自宝生物工程(大连)有限公司,粉纹夜蛾幼虫由河北农业大学郭巍教授实验室提供,信号分子标准品(C6-HSL、3O C6-HSL)购自Sigma公司。
1.2 方法
1.2.1 粉纹夜蛾中肠微生物菌株分离 选取健康粉纹夜蛾幼虫3条,杀死后75 %的酒精中消毒10 s、0.1% 升汞液中消毒2 min,灭菌水冲洗3次,之后在无菌条件下解剖出中肠组织研磨,采用梯度稀释法将研磨液稀释至1×10-6、1×10-7、1×10-8,并吸3个浓度的稀释液各100 μL分别涂布于LB平板中,各稀释度重复3 次,置于30℃恒温培养箱培养72 h,观察记录菌株生长情况,然后挑取菌落形态差异明显的菌落,依次编号并进行多次纯化,得到单菌落后LB试管保存备用[8]。
1.2.2 产AHLs群体感应信号分子菌株的筛选 将过夜培养的报告菌CV026与50 mL 含有1.5%琼脂的LB 培养基混合,倒平板。在平板底部画出十字方格,将保存的从粉纹夜蛾消化道中分离出来的不同菌株用牙签点接在每个方格中,30℃恒温培养,定时观察各个菌株的生长及产生色素情况,如果菌株周围产生紫色则初步确定该菌株为产生信号分子的菌株。
1.2.3 菌落及菌体形态观察 将筛选出来的产信号分子的菌株划线接种到LB固体培养基中,30℃恒温培养48 h,观察并记录LB培养基上出现的单菌落形态。挑取单菌落的菌体进行Gram染色,在光学显微镜下观察菌体形状、染色、产芽孢等情况[9]。
1.2.4 微生物菌株的16S rDNA扩增及扩增片段的测序 将产AHLs信号分子的菌株接种到LB培养基中,30℃过夜培养,离心获得菌体后采用天根基因组DNA小提试剂盒提取基因组DNA。以提取的细菌DNA 作为模板,以16S rDNA通用引物:5'-AGAGTTTGATCATGGCTCAG-3'和II:5'-GGTTACCTTGTTACGACTT-3'为引物对进行PCR扩增,PCR反应体系为(20 μL):10× Buffer 2 μL,10 mmol/L dNTP 2 μL,rTaq 0.2 μL,模板 1 μL,上下游引物各1 μL,灭菌双蒸水12.8 μL。PCR扩增程序:94℃预变性5 min;94℃变性1 min,56℃退火1 min,72℃延伸1 min,30个循环;72℃延伸10 min。反应结束后1%琼脂糖凝胶电泳检测PCR产物,将扩增正确的PCR产物送上海生工生物技术有限公司测序。
1.2.5 菌株16S rDNA序列的系统发育分析 将菌株的16S rDNA测序结果输入到GenBank中,用BLAST程序与数据库中的所有序列进行比较,利用Clustal 软件进行多序列比对并计算进化距离,采用Mega 5.2 软件进行系统发育树的构建。
1.2.6 菌株生理生化反应测定 基于细菌的16S rDNA分子生物学方法鉴定的初步结果,参考伯杰氏细菌鉴定手册,进行蒙氏肠球菌特异性生理生化反应,包括运行性测定液体基础培养基、耐盐性试验、是否产H2S、不同糖类利用情况、乙酰甲基甲醇试验(V-P试验)、甲基红试验(M.R试验)、吲哚试验、硝酸盐还原试验测定等,进行细菌生理生化鉴定[10]。
1.2.7 信号分子的提取及鉴定 将产AHLs菌株接种到LB培养基中过夜培养,菌液5 000 r/min离心3 min,吸取上清液后加入等体积二氯甲烷进行萃取,将有机相旋转蒸发后重溶于1 mL乙腈中,即得到信号分子样品。用微量移液器将4 μL样品点在C18反相薄层板上,用甲醇水(6040,V/V)的展开剂展开,充分展开后蒸发溶剂,用混有C. violaceum026检测菌株的软琼脂覆盖在薄层板上,30℃过夜培养后观察试验结果的显色情况[11]。
2 结果
2.1 产群体感应信号分子菌株的分离
通过稀释涂平板的方法从粉纹夜蛾中肠中共分离出细菌120株,将这些菌株为备用菌株进行产信号分子菌株的筛选。
产AHLs信号分子菌株的分离采用平板筛选法,用的报告菌为Chromobacterium violaceumCV026,通过该方法,从120株菌株分离出2株产信号分子的菌株(图1),菌株编号分别为Tni-9和Se-9。
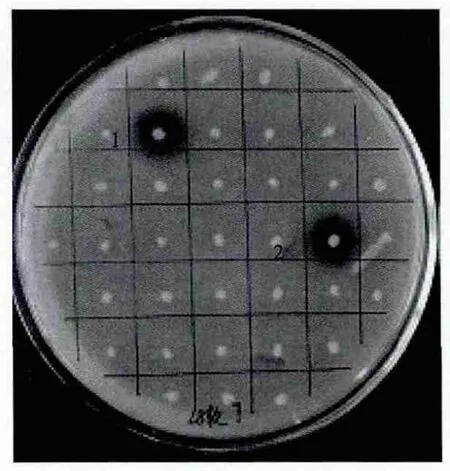
图1 产群体感应信号分子的菌株筛选结果
2.2 产群体感应信号分子菌株的鉴定
2.2.1 菌株的培养特征和形态特征 将菌株Tni-9和Se-9接种在LB平板上,30℃培养48 h观察其菌落形态,Tni-9和Se-9菌落近圆形,边缘啮蚀状,颜色为乳白色,表面光滑无光泽不透明。将菌株Tni-9和Se-9进行革兰氏染色后在显微镜下观察其形态为圆形或卵圆形,单个或成对排列且革兰氏染色为阳性(图2,图3)。
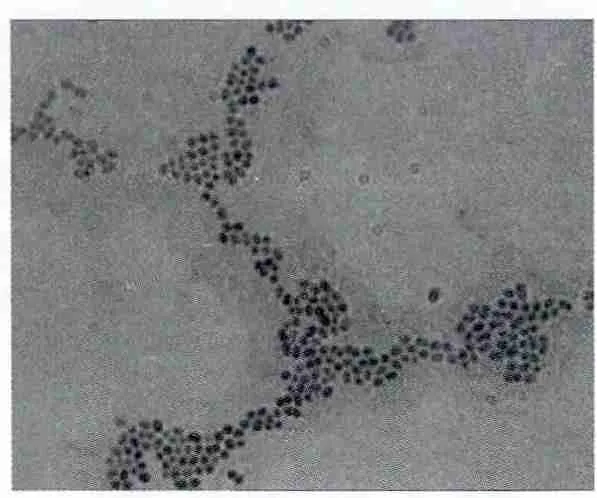
图2 Tni-9显微镜下镜检照片(100ⅹ)

图3 Se-9显微镜下镜检照片(100ⅹ)
2.2.2 菌株Tni-9、Se-9的16S rDNA基因鉴定 扩增菌株Tni-9、Se-9的16S rDNA基因得到一条明亮的特异条带,长约1 500 bp(图4)。获得了Tni-9、Se-9菌株16S rDNA 核酸序列(GenBank登录号分别为:KF551916,KF551917)。根据16S rDNA序列对Tni-9、Se-9相近物种进行聚类分析及系统发育树的构建,结果(图5)表明Tni-9、Se-9的16S rDNA序列与蒙氏肠球菌的序列同源性最近,初步鉴定这两个菌株属于蒙氏肠球菌。

图4 菌株Tni-9、Se-9 16s rDNA-PCR 电泳图
2.2.3 菌株的生理生化特征鉴定 根据上述的分子鉴定结果选取菌株Se-9进行菌株的生理生化检测,结果如表1所示,菌株Se-9可发酵的碳水化合物种类广泛,接触酶阴性,在6.5% NaCl中可以生长,V-P反应阳性。参照《常见细菌系统鉴定手册》鉴定菌株Se-9为蒙氏肠球菌。
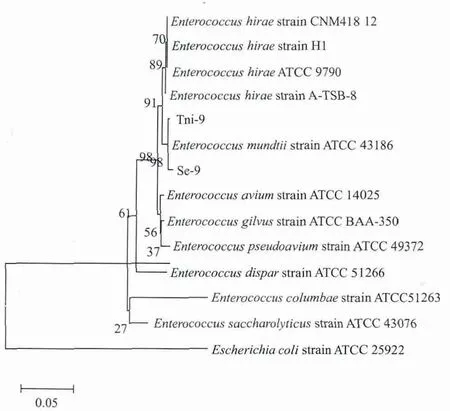
图5 Tni-9和Se-9的系统发育进化树

表1 Se-9主要生理生化特性
2.3 菌株Se-9产信号分子种类的确定
以3OC6-HSL和C6-HSL标准样品为对照进行薄层层析,分析Se-9菌株产生信号分子的种类,由图6可以看出两个菌株在3OC6-HSL和C6-HSL标准样品所在的相应位置都出现了一个紫色斑点,说明该菌株产生3OC6-HSL和C6-HSL两种信号分子。
3 讨论
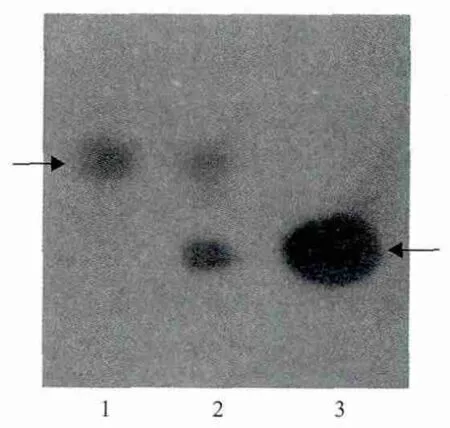
图6 菌株产信号分子种类的薄层层析分析
昆虫肠道栖息着大量的微生物。随着近年来研究肠道微生物的方法不断进步,尤其是基于16S rDNA的分子生物学方法的应用,人们对肠道微生物的了解逐渐加深。肠道微生物对昆虫寄主的作用包括提供营养、利用拓殖抗性抵抗外来微生物侵袭、参与多重营养关系、引起昆虫免疫反应等。粉纹夜蛾是昆虫中肠防御体系研究最为深入的昆虫,但主要集中在中肠围食膜蛋白结构和Bt杀虫蛋白受体方面,对农业上重要害虫粉纹夜蛾中肠微生物群体感应的研究尚未见系统报道。本研究从粉纹夜蛾中肠中分离出120个菌株,再利用luxI/luxRQS 系统的微生物传感菌紫色杆菌(Chromobacterium violaceum)CV026分离出两株可以产信号分子的菌株Tni-9和Se-9。对这两个菌株进行了生理生化分析,通过16S rDNA扩增得到一条大小为1 500 bp左右的条带。在NCBI基因库中进行比对,发现这两个菌株与蒙氏肠球菌(Enterococcus mundtiiATCC 43186)的16S rDNA核酸序列同源性最高达到99%。将测序结果用BLAST软件与GenBank 中的相关属种的16S rDNA序列进行比对,以大肠杆菌为外群,用Mega5.2 软件构件系统发育树。100次比对中98次都与蒙氏肠球菌聚合到同一个分支,鉴定这两个菌株属于蒙氏肠球菌。提取菌株Se-9所产生的信号分子,通过薄层层析与微生物传感菌相结合的方法对菌株产生的信号分子进行定性分析,结果显示Se-9可以产生两种信号分子C6-HSL和3OC6-HSL。该菌株的获得,为下一步利用转座子标签技术构建群体感应缺失突变体,并在缺少及富含该产信号分子菌株条件下测定昆虫的杀虫活性奠定基础(本试验正在进行中)。整个研究的目的是以粉纹夜蛾为研究对象,将细菌群体感应系统和昆虫中肠防御体系联系起来,探索群体感应系统对微生物杀虫活性影响,为农业生产中昆虫生物防治提供理论依据和新的思路。
4 结论
从粉纹夜蛾中肠中分离出120个菌株,采用群体感应报告菌株CV026进行复筛,分离出两株可以产信号分子的菌株Tni-9和Se-9。对这两株菌进行形态学分析、16S rDNA 鉴定及生理生化分析,鉴定这两株菌为蒙氏肠球菌。提取Se-9的信号分子,通过薄层层析分析显示Se-9可以产生两种信号分子C6-HSL和3OC6-HSL。
[1] 刘廷辉, 郭喂, 申建茹.苏云金芽孢杆菌菌株GS8菌株的生物学特性及其cry基因型鉴定[J] 农药学学报, 2009, 11(1):92-97.
[2] Janmaat AF, Myers J. Rapid evolution and the cost of resistance to Bacillus thruingiensis in greenhouse populations of cabbage loopers,Trichoplusia ni[J] .Proceedings of the Royal Society, 2003, 270(1530):2263-2270.
[3] Thomanek H, Schenk ST, Stein E, et al. ModifiedN-acyl-homoserine lactones as chemical probes for the elucidation of plant-microbe interactions[J] . Org Biomol Chem, 2013, 11:6994-7003.
[4] Parsek MR, Val DL, Hanzelka BL, et al. Acyl homoserine-lactone quorum-sensing signal generation[J] . Proceeding of the National Academy of the Sciences, 1999, 96:4360-4365.
[5] 边子睿, 刘方, 张霞, 等. GCR1和GCR2在N-酰基高丝氨酸内酯调节拟南芥根生长中的作用[J] . 植物生理学报, 2011, 47(2):175-180.
[6] Guan C, Ju J, Borlee BR, et al. Signal mimics derived from a metagenomic analysis of the gypsy moth gut microbiota[J] .Applied and Environmental Microbiology, 2007, 73:3669-3676.
[7] Borlee BR, Geske GD, Robinson CJ, et al. Quorum-sensing signals in the microbial community of the cabbage white butterfly larval midgut[J] . International Society for Microbial Ecology, 2008, 2(11):1101-1111.
[8] 刘玉升, 叶保华, 吕飞, 等.豆天蛾幼虫肠道细菌分离及初步鉴定研究[J] .山东农业大学学报:自然科学版, 2006, 37(3):345-348.
[9] 周德庆.微生物学实验手册[M] .上海:上海科学技术出版社,1986:65-70, 178-180.
[10] 万国福.鸡肠道菌的分离、鉴定和微生态制剂的研究[J] .工艺与设备, 2008, 7:24-27.
[11] 黄媛媛、边子睿、宋水山. 胡萝卜软腐欧文氏菌中信号分子合成酶expI基因的克隆、表达及其分析[J] .生物学杂志,2012, 29(4):60-64.
