N-糖苷酶基因在脑膜炎败血伊丽莎白金菌临床分离株中的分布
2014-06-27李蒙孙桂芹陈菁李天胜王蕾策力木格陈力
李蒙,孙桂芹,陈菁,李天胜,王蕾,策力木格,陈力
复旦大学基础医学院教育部/卫生部医学分子病毒学重点实验室,上海 200032
脑膜炎败血伊丽莎白金菌(Elizabethkingiameningoseptica,EM)是非发酵革兰阴性杆菌,可广泛分布于土壤和水环境中[1]。该菌又名脑膜脓毒金黄杆菌(Flavobacteriummeningosepticum, FM;Chryseobacteriummeningosepticum, CM),2005年后基于16S rRNA序列分析后被归于伊丽莎白金菌属[2]。EM为条件致病菌,可引起新生儿脑炎和败血症[3,4]。植入性医疗器械或其他污染因素[5]使其院内获得性感染病例增多,尤其是在重症监护室(intensive care unit, ICU)中。据报道,中国台湾地区EM感染的发病率从1999年的7.5/10万入院病例上升至2006年的35.6/10万入院病例[6];2006~2007年中国非发酵革兰阴性杆菌耐药监测网结果显示,EM临床分离株占非发酵革兰阴性杆菌临床分离株的1.02%[7]。EM对医院常用的头孢类、碳青霉烯类、氨基糖苷类抗生素呈现天然耐多药[8],感染后2周内病死率高达25%以上[6,9]。迄今对EM的研究多集中在临床病例报道和耐药性研究,关于其快速检测方法和特征性分子机制的研究较少。
N-糖苷酶(peptide:N-glycanase, PNGase)是去糖基化酶的一种,专一识别N-糖蛋白或N-糖肽中的Asn-X-Ser/Thr序列,将Asn上连接的糖链从底部完整切除[10]。PNGase最早于1977年从杏仁中发现,并命名为PNGase A,随后发现其在酵母、线虫、拟南芥、鱼、小鼠、人等真核生物中广泛存在,参与生长、发育及降解错误折叠的糖蛋白等过程[11-14]。但在原核生物中PNGase极为罕见,仅在EM中发现了此类酶,并命名为PNGase F[15]。PNGase F是糖生物学研究中最常用的工具酶,对研究细胞表面糖蛋白结构与功能的关系、糖链对糖蛋白功能的影响和糖链结构的分析具有重要意义[16-19],但PNGase F在EM中的生物学功能仍未知。
本实验室前期对临床分离的一株EM菌株FMS-007进行全基因组测序(GenBank序列号:CP006576)分析并验证功能,发现该菌株中可能同时存在2种PNGase,即PNGase F和PNGase F-Ⅱ。FMS-007菌株中的PNGase F-Ⅱ与PNGase F在核苷酸水平和氨基酸水平的同源性不高,分别为46.9%和21.8%,但蛋白结构与PNGase F相似,并具有与PNGase F相同的切除糖肽和糖蛋白上N-连接寡糖链的功能(已投稿)。PNGase F和PNGase F-Ⅱ这2个同工酶同时存在预示PNGase可能在EM的生活史中具有重要生理功能。本研究利用核酸检测技术,分析了PNGase F和PNGase F-Ⅱ在浙江省3家三级甲等医院65株EM临床分离株中的分布情况,为建立特异聚合酶链反应(polymerase chain reaction,PCR)检测方法和研究其生物学意义奠定基础。
1 材料与方法
1.1 材料
1.1.1菌株2010年7月~2012年12月于浙江省绍兴市人民医院、台州市中心医院和宁波市李惠利医院收集EM临床分离株。无菌操作采集患者血液、下呼吸道分泌物、尿液及创口分泌物等标本,35 ℃培养18~24 h,分离获得纯培养。剔除同一患者多次检出的重复菌株,共收集EM菌株65株,其中绍兴市12株、台州市7株、宁波市46株。所有菌株经法国生物梅里埃公司VITEK-2自动微生物鉴定分析系统鉴定为EM。
1.1.2仪器和试剂细菌基因组DNA抽提试剂盒购自天根生化科技(北京)有限公司;PrimeSTAR®Max DNA Polymerase和DNA Marker DL2000购自TaKaRa公司;C1000 Touch梯度PCR仪和Sub-Cell GT水平电泳槽为美国Bio-Rad公司产品;FluorChem E凝胶成像系统为美国Protein Simple公司产品。
1.1.3引物设计与合成本实验室已完成一株来自浙江省绍兴市人民医院的EM临床分离株FMS-007的全基因组测序(GenBank序列号:CP006576),根据FMS-007全基因组序列中PNGaseF基因和PNGaseF-Ⅱ基因进行引物设计。FMS-007的PNGaseF基因与ATCC 13253的同源性为77%,故针对ATCC 13253中的PNGaseF基因设计了第2对引物。16SrRNA引物为细菌检测通用引物[20]。 所有引物 (表1) 送生工生物工程(上海)股份有限公司合成。
表1PCR引物序列
Tab.1PCRprimers

PrimerSequenceSize (bp)16S rRNA F5'-AGAGTTTGATCCTGGCTCAG-3'1 500 R5'-AAGGAGGTGATCCAGCCGCA-3'PNGase FA F5'-GATATTGTATACGGGACACCGG-3'326 R5'-CTCTGATTATTGATAGGGTTTGCTG-3'B F5'-TGCTTTTCCATAAGGGACACC-3405 R5'-ACATTTACGCTTCCGGCTGAT-3'PNGase F-Ⅱ F5'-TGCAGCCTTATTACAGGTGCTC-3'528 R5'-CCCATTAAATAAAGACTGATCGTCC-3'
1.2 方法
1.2.1NCBI中EM菌株PNGaseF和PNGaseF-Ⅱ基因查找从美国国立生物技术信息中心(National Center of Biotechnology Information,NCBI)数据库中下载ElizabethkingiameningosepticumATCC 13253 (GCA_000401415.1)、ElizabethkingiameningosepticumNBRC 12535 (GCA_000367325.1)、Elizabethkingiameningosepticum502 (GCA_000447375.1)基因组序列文件,以FMS-007的全基因组序列作为模板,利用基本局部相似性比对搜索工具(Basic Local Alignment Search Tool,BLAST)进行序列比对。然后,应用Sanger Center开发的ACT工具[21]进行基因组比对。基于基因组比对的位置对应关系,根据FMS-007基因组PNGaseF和PNGaseF-Ⅱ位置定位另外3株菌株基因组中的PNGaseF和PNGaseF-Ⅱ基因,并在NCBI网站进行在线BLAST验证。
1.2.2DNA提取应用天根生化科技(北京)有限公司细菌基因组DNA抽提试剂盒提取EM基因组DNA,-20 ℃保存。抽提方法为DNA柱吸附洗脱法,按试剂盒说明书操作。
1.2.3PCR检测临床菌株PCR反应体系为50 μl,包括模板DNA(约50 ng/μl)1 μl、上游引物(10 μmol/L)2 μl、下游引物(10 μmol/L)2 μl、PrimeSTAR®Max DNA Polymerase(2×) 25 μl,用无菌去离子水补至50 μl。上述PCR反应引物包括16SrRNA基因、PNGaseF基因和PNGaseF-Ⅱ基因的引物。PCR反应程序:98 ℃ 10 s,55 ℃ 15 s,72 ℃ 5 s,循环35次。每次实验均设阳性对照(SX7菌株和ATCC 13253菌株)和阴性对照(H2O、阴沟肠杆菌和鲍曼不动杆菌临床分离株)。于PCR完成后,各取4 μl 16SrRNA基因、PNGaseF基因和PNGaseF-Ⅱ基因PCR产物混合,用于1.5%琼脂糖凝胶电泳,电压设定为120 V,电泳时间为1 h,并在凝胶成像系统中将结果拍照留存。阳性扩增产物测序后,利用NCBI数据库中BLAST工具进行序列比对。16S rRNA比对结果用于菌株类别的确认。
1.3 统计学方法
采用SPSS 16.0统计学软件处理数据,配对计数资料比较采用配对χ2检验,P<0.05为差异有统计学意义。
2 结果
2.1 NCBI中EM菌株PNGase基因的分布
除本实验室发表的FMS-007全基因组核酸序列外,在NCBI中检索到3株已发表全基因组核酸序列的EM,分别是EM (ATCC 13253)、EM (NBRC 12535)和EM (502)。根据FMS-007全基因组核酸序列中PNGaseF和PNGaseF-Ⅱ的位置查找另3株菌株中是否也存在PNGase基因时发现,这4株菌株均携带PNGaseF和PNGaseF-Ⅱ(表2)。其中FMS-007与502的PNGaseF核酸序列高度同源(100%);ATCC 13253与NBRC 12535的PNGaseF核酸序列高度同源(100%);FMS-007与ATCC 13253的PNGaseF核酸序列同源性为77%, 故将 FMS-007 和 ATCC 13253 菌株携带的PNGaseF分别命名为PNGaseF(A) 和PNGaseF(B)。以上4株EM菌株的PNGaseF-Ⅱ核酸序列同源性较好(81.8%~100%)。因此,检测EM临床分离株中PNGase基因时,分别针对PNGaseF(A) 和PNGaseF(B) 基因设计2对不同引物,利用PNGaseF-Ⅱ保守区设计1对引物(表1)。
表2NCBI中EM菌株PNGaseF和PNGaseF-Ⅱ的携带情况
Tab.2DistributionofPNGaseFandPNGaseF-ⅡamongEMsinNCBI
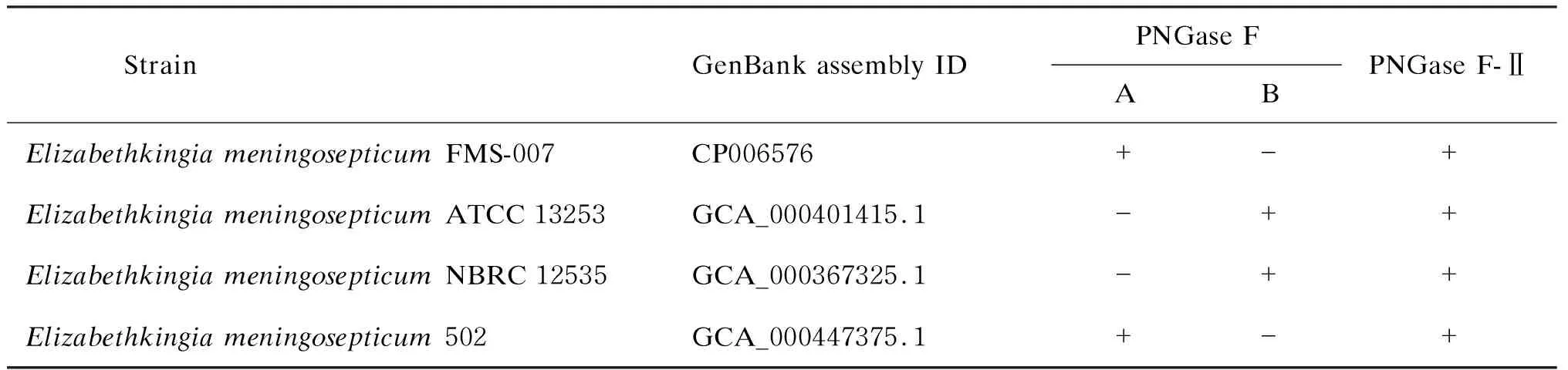
StrainGenBank assembly IDPNGase FABPNGase F-ⅡElizabethkingia meningosepticum FMS-007CP006576+-+Elizabethkingia meningosepticum ATCC 13253GCA_000401415.1-++Elizabethkingia meningosepticum NBRC 12535GCA_000367325.1-++Elizabethkingia meningosepticum 502GCA_000447375.1+-+
2.2 EM临床分离株中PNGase基因的分布情况
于浙江省采集EM临床分离株65株,应用组合PCR检测16SrRNA、PNGaseF和PNGaseF-Ⅱ基因。全部菌株16SrRNA基因扩增阳性(图1),经测序并与NCBI数据库中序列对比,扩增产物片段大小与引物设计一致,且均是EM特异性的16SrRNA。
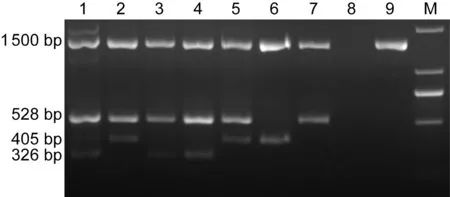
M, marker DL2000; 1, FMS-007; 2, ATCC 13253; 3-7, EM specimens; 8, H2O; 9,Enterobactercloacaespecimen.
图116SrRNA、PNGaseF和PNGaseF-Ⅱ的PCR产物电泳
Fig.1ElectrophoresisofPCRproductsof16SrRNA(1500bp),andfragmentsofPNGaseF(326bpor405bp)andPNGaseF-Ⅱ (528bp)genes
65株EM临床分离株中,PNGaseF扩增阳性41株,其中PNGaseF(A) 基因片段扩增阳性33株、PNGaseF(B) 基因片段扩增阳性8株;PNGaseF-Ⅱ扩增阳性62株(表3)。PNGase基因阳性扩增产物经测序并与NCBI数据库中序列对比,产物大小与设计大小一致,且均是PNGaseF和PNGaseF-Ⅱ特异性的。PNGase阳性菌64株,阳性率为98.5%;PNGaseF/PNGaseF-Ⅱ共阳性菌39株,共阳性率为60.0%;PNGaseF(A) 阳性菌33株,阳性率为50.8%;PNGaseF(B) 阳性菌8株,阳性率为12.3%;PNGaseF-Ⅱ阳性菌62株,阳性率为95.4%;PNGaseF-Ⅱ阳性率高于PNGaseF(配对χ2检验,P<0.01)(表4)。
表365株EM菌株中PNGase亚型分布
Tab.3DistributionofPNGaseamong65clinicalEMstrains
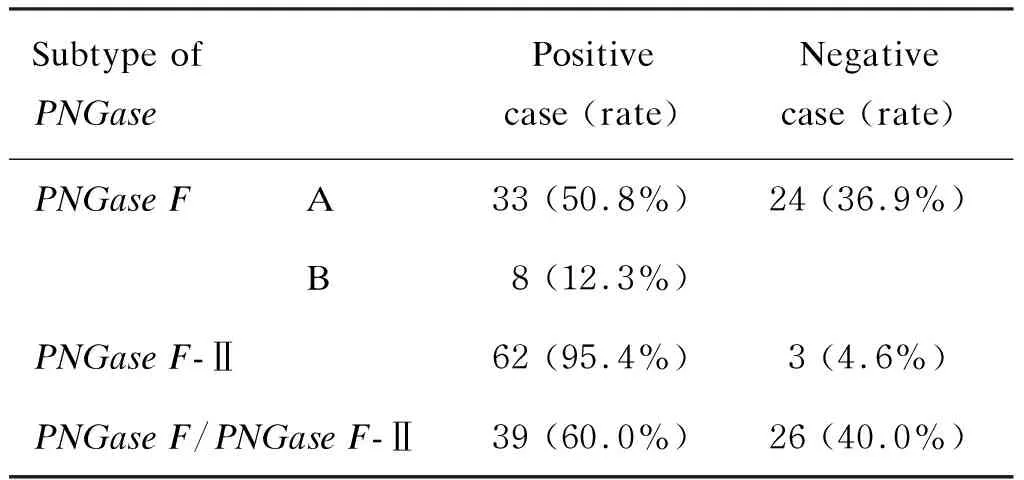
Subtype of PNGasePositive case (rate)Negative case (rate)PNGase F A33 (50.8%)24 (36.9%)B8 (12.3%)PNGase F-Ⅱ62 (95.4%)3 (4.6%)PNGase F/PNGase F-Ⅱ39 (60.0%)26 (40.0%)
表465株EM菌株中PNGaseF与PNGaseF-Ⅱ阳性率的比较
Tab.4ComparisonofPNGaseFandPNGaseF-Ⅱin65clinicalEMstrains
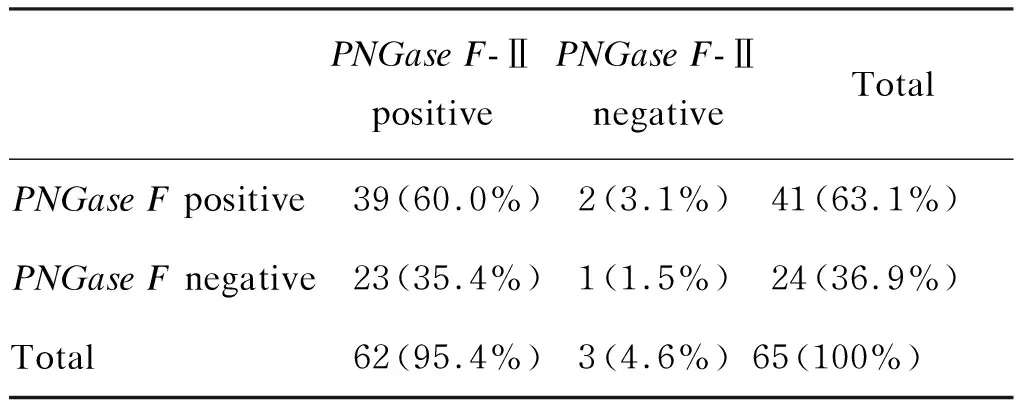
PNGase F-Ⅱ positivePNGase F-Ⅱ negativeTotalPNGase F positive39(60.0%)2(3.1%)41(63.1%)PNGase F negative23(35.4%)1(1.5%)24(36.9%)Total62(95.4%)3(4.6%)65(100%)
3 讨论
EM作为条件致病菌,其所致感染多为散发报道,未引起临床重视。但近年随着植入性医疗器械的广泛使用,EM感染在ICU中的发病率逐年升高;且因其具有天然耐多药的特性,给临床治疗带来困难[22]。因此,建立该菌快速检测方法有利于指导临床用药,对其特征性酶的分析有利于进一步深入研究。
EM具有非发酵,无动力,氧化酶、触媒、吲哚阳性等生化特征[23],现有临床手段是根据以上生化特征来鉴定,但这种方法费时费力,不利于及时指导临床用药,因此有必要建立核酸检测方法。PNGase在真核生物中广泛存在,但在原核生物中PNGase F仅报道存在于EM中[15],因此PNGase F可作为EM的特异性分子标记用于检测。本研究检测65株EM临床分离株,发现PNGase基因的携带率高达98.5%,提示将其作为EM检测的特异分子标记灵敏度高。其中,PNGaseF-Ⅱ的携带率(95.4%)高于PNGaseF(63.1%),提示PNGaseF-Ⅱ作为分子标记检测EM的灵敏度高于PNGaseF,更适用于EM的核酸检测。根据现有公开的4株EM全基因组核酸序列比对PNGaseF基因,发现存在PNGaseF(A)和PNGaseF(B),其序列同源性低于PNGaseF-Ⅱ,因此本研究设计的针对PNGaseF基因的引物可能只适用于PNGaseF(A)和PNGaseF(B),不适用于其他未知亚型的PNGaseF,从而导致假阴性的存在。
根据PNGase对EM进行分型未见报道。本研究首次根据PNGase F和PNGase F-Ⅱ这2种同工酶将其分为4型:PNGase F和PNGase F-Ⅱ共阳性、PNGase F阳性但PNGase F-Ⅱ阴性、PNGase F阴性但PNGase F-Ⅱ阳性及PNGase F和PNGase F-Ⅱ共阴性。本研究显示,经16S rRNA确认过的65株EM中,以PNGase F和PNGase F-Ⅱ共阳性菌株为主,占60.0%;其次是PNGase F阴性但PNGase F-Ⅱ阳性亚型,占35.4%;其余2种亚型所占比例较低:PNGase F阳性但PNGase F-Ⅱ阴性菌株占3.1%,PNGase F和PNGase F-Ⅱ共阴性菌株占1.5%。不同亚型PNGase在EM中的生物学意义未知,但同时携带PNGase F和PNGase F-Ⅱ的菌株占60.0%,提示EM中这2个同工酶的存在非常重要,可能在细菌感染、代谢或抵抗外环境中起作用,值得深入研究。
本研究的不足之处在于对PNGaseF和PNGaseF-Ⅱ基因进行PCR检测时仅以小样本(10株)阴沟肠杆菌和鲍曼不动杆菌(数据未显示)作为阴性对照,初步判断其特异性,还需以金黄杆菌属内的产吲哚金黄杆菌、吲哚金黄杆菌、黏金黄杆菌、金矿金黄杆菌和大菱鲆金黄杆菌作为阴性对照,并对检测方法的特异度进行评价。其中产吲哚金黄杆菌的阴性对照最重要,因其临床分离菌株数占金黄杆菌属的42.9%[7],与EM临床分离菌持平。本研究中的EM临床菌株全部来自浙江省,而不同地区来源的EM临床菌株中PNGase特征可能不同,这有待进一步研究。
综上所述,本研究建立了EM中PNGase检测和分型的方法,发现PNGase在EM中携带率高达98.5%,可作为分子标记用于EM检测。同时,EM以PNGase F和PNGase F-Ⅱ共阳性亚型为主,提示2个同工酶的存在可能具有重要的生物学意义,值得进一步研究。
[1] Thirumalaikolundusubramanian P. Flavobacterium meningosepticum in ICUs [J]. Ann Saudi Med, 1998,18(1): 84.
[2] Kim KK, Kim MK, Lim JH, Park HY, Lee ST. Transfer of Chryseobacterium meningosepticum and Chryseobacterium miricola to Elizabethkingia gen. nov. as Elizabethkingia meningoseptica comb. nov. and Elizabethkingia miricola comb. nov [J]. Int J Syst Evol Microbiol, 2005, 55(Pt 3): 1287-1293.
[3] Maraki S, Scoulica E, Manoura A, Papageorgiou N, Giannakopoulou C, Galanakis E. A Chryseobacterium meningosepticum colonization outbreak in a neonatal intensive care unit [J]. Eur J Clin Microbiol Infect Dis, 2009, 28(12): 1415-1419.
[4] Ceyhan M, Celik M. Elizabethkingia meningosepticum (Chryseobacterium meningosepticum) infections in children. Int J Pediatr, 2011, 2011: 215237.
[5] Lin PY, Chen HL, Huang CT, Su LH, Chiu CH. Biofilm production, use of intravascular indwelling catheters and inappropriate antimicrobial therapy as predictors of fatality in Chryseobacterium meningosepticum bacteraemia [J]. Int J Antimicrob Agents, 2010, 36(5): 436-440.
[6] Hsu MS, Liao CH, Huang YT, Liu CY, Yang CJ, Kao KL, Hsueh PR. Clinical features, antimicrobial susceptibilities, and outcomes of Elizabethkingia meningoseptica (Chryseobacterium meningosepticum) bacteremia at a medical center in Taiwan, 1999-2006 [J]. Eur J Clin Microbiol Infect Dis, 2011, 30(10): 1271-1278.
[7] 胡云建. Mohnarin 2006~2007年度报告:非发酵革兰阴性杆菌耐药性监测[J]. 中国抗生素杂志, 2008, 33(10): 597-601, S6.
[8] Lin XH, Xu YH, Sun XH, Huang Y, Li JB. Genetic diversity analyses of antimicrobial resistance genes in clinical Chryseobacterium meningosepticum isolated from Hefei, China [J]. Int J Antimicrob Agents, 2012, 40(2): 186-188.
[9] 熊旭明, 魏衍超, 刘卫江, 罗辉遇, 谢长江, 温得良. 重症监护病房内脑膜脓毒金黄杆菌医院感染肺炎临床及耐药分析[J]. 中华医院感染学杂志, 2005, 15(6): 708-710.
[10] Gosain A, Srivastava A, Saran S. Peptide: N-glycanase is expressed in prestalk cells and plays a role in the differentiation of prespore cells during development of Dictyostelium discoideum [J]. Indian J Exp Biol, 2014, 52(3): 197-206.
[11] Hossain MA, Nakano R, Nakamura K, Kimura Y. Molecular identification and characterization of an acidic peptide:N-glycanase from tomato (Lycopersicum esculentum) fruits [J]. J Biochem, 2010, 147(2): 157-165.
[12] Chang T, Kuo MC, Khoo KH, Inoue S, Inoue Y. Developmentally regulated expression of a peptide:N-glycanase during germination of rice seeds (Oryza sativa) and its purification and characterization [J]. J Biol Chem, 2000, 275(1): 129-134.
[13] Funakoshi Y, Negishi Y, Gergen JP, Seino J, Ishii K, Lennarz WJ, Matsuo I, Ito Y, Taniguchi N, Suzuki T. Evidence for an essential deglycosylation-independent activity of PNGase in Drosophila melanogaster [J]. PLoS One, 2010, 5(5): e10545.
[14] Xin F, Wang S, Song L, Liang Q, Qi Q. Molecular identification and characterization of peptide: N-glycanase from Schizosaccharomyces pombe [J]. Biochem Biophys Res Commun, 2008, 368(4): 907-912.
[15] Plummer TJ, Elder JH, Alexander S, Phelan AW, Tarentino AL. Demonstration of peptide:N-glycosidase F activity in endo-beta-N-acetylglucosaminidase F preparations [J]. J Biol Chem, 1984, 259(17): 10700-10704.
[16] Palm AK, Novotny MV. A monolithic PNGase F enzyme microreactor enabling glycan mass mapping of glycoproteins by mass spectrometry [J]. Rapid Commun Mass Spectrom, 2005, 19(12): 1730-1738.
[17] Krenkova J, Lacher NA, Svec F. Multidimensional system enabling deglycosylation of proteins using a capillary reactor with peptide-N-glycosidase F immobilized on a porous polymer monolith and hydrophilic interaction liquid chromatography-mass spectrometry of glycans [J]. J Chromatogr A, 2009, 1216(15): 3252-3259.
[18] Mauko L, Nordborg A, Hutchinson JP, Lacher NA, Hilder EF, Haddad PR. Glycan profiling of monoclonal antibodies using zwitterionic-type hydrophilic interaction chromatography coupled with electrospray ionization mass spectrometry detection [J]. Anal Biochem, 2011, 408(2): 235-241.
[19] Go EP, Hewawasam G, Liao HX, Chen H, Ping LH, Anderson JA, Hua DC, Haynes BF, Desaire H. Characterization of glycosylation profiles of HIV-1 transmitted/founder envelopes by mass spectrometry [J]. J Virol, 2011, 85(16): 8270-8284.
[20] Hiraishi A. Direct automated sequencing of 16S rDNA amplified by polymerase chain reaction from bacterial cultures without DNA purification [J]. Lett Appl Microbiol, 1992, 15(5): 210-213.
[21] Carver TJ, Rutherford KM, Berriman M, Rajandream MA, Barrell BG, Parkhill J. ACT: the Artemis Comparison Tool [J]. Bioinformatics, 2005, 21(16): 3422-3423.
[22] Balm MN, Salmon S, Jureen R, Teo C, Mahdi R, Seetoh T, Teo JT, Lin RT, Fisher DA. Bad design, bad practices, bad bugs: frustrations in controlling an outbreak of Elizabethkingia meningoseptica in intensive care units [J]. J Hosp Infect, 2013, 85(2): 134-140.
[23] Versalovic J, Carroll K, Funke G, Jorgensen J, Landry M, Warnock D. Manual of Clinical Microbiology [M]. 10th ed. Washington: ASM Press, 2011.
