条斑紫菜分子生物学研究现状
2014-06-26沈颂东
沈颂东
(苏州大学 医学部基础医学与生物科学学院 细胞生物学系,江苏 苏州 215123)
条斑紫菜(Porphyra yezoensis Ueda)隶属于红藻门(Rhodophyphyta),原红藻纲(Protoflorideophyceae),红毛菜目(Bangiales),红毛菜科(Bangiaceae),紫菜属(Porphyra)。紫菜属的种类分布广泛,目前已报道的种类约有 130多种[1]。我国记录定名的紫菜物种或变种有22种[2]。条斑紫菜为北太平洋西部特有的种类,主要分布于我国黄、渤海和东海北部沿岸,以及日本列岛和朝鲜半岛沿岸,是中、日、韩三国共同栽培的种类。紫菜是重要的经济海藻,目前全世界其栽培年产值为 20亿美元,约占整个海藻产业的2/3。我国是紫菜栽培生产的重要国家,2010年度的栽培量按国际标准计算达200亿枚(每枚为3g),其中江苏省生产出来的条斑紫菜就达到 32亿枚。世界各国的学者对紫菜的生物学进行了较多的研究,现综述如下。
1 条斑紫菜生活史
条斑紫菜(P.yezoensis)具有异形的世代交替,即宏观可见的叶状孢子体世代和微观的丝状孢子体世代交替出现完成其复杂的生活史转变过程。从仅有几百个细胞所组成的幼小叶状体开始,由顶端细胞不断转化为单孢子囊,形成、放散单孢子。单孢子为单倍体,所萌发生成的叶状体仍为单倍体。条斑紫菜雌雄同株,精子囊器淡黄色,呈条纹状混夹在深红色的果孢子囊中,整个生殖器官在叶的末梢端形成明显的深紫和淡黄色相间的条斑状。其中精子囊器有 128个或 64个精子囊,为♂A4B4C8,也有♂A2B4C8,果孢子囊为 16个果孢子,分裂式为♀A2B2C4。丝状体为二倍体阶段,成熟发生的壳孢子仍为二倍体,壳孢子萌发时经减数分裂形成叶状体,染色体数目n=3(图1)[2]。
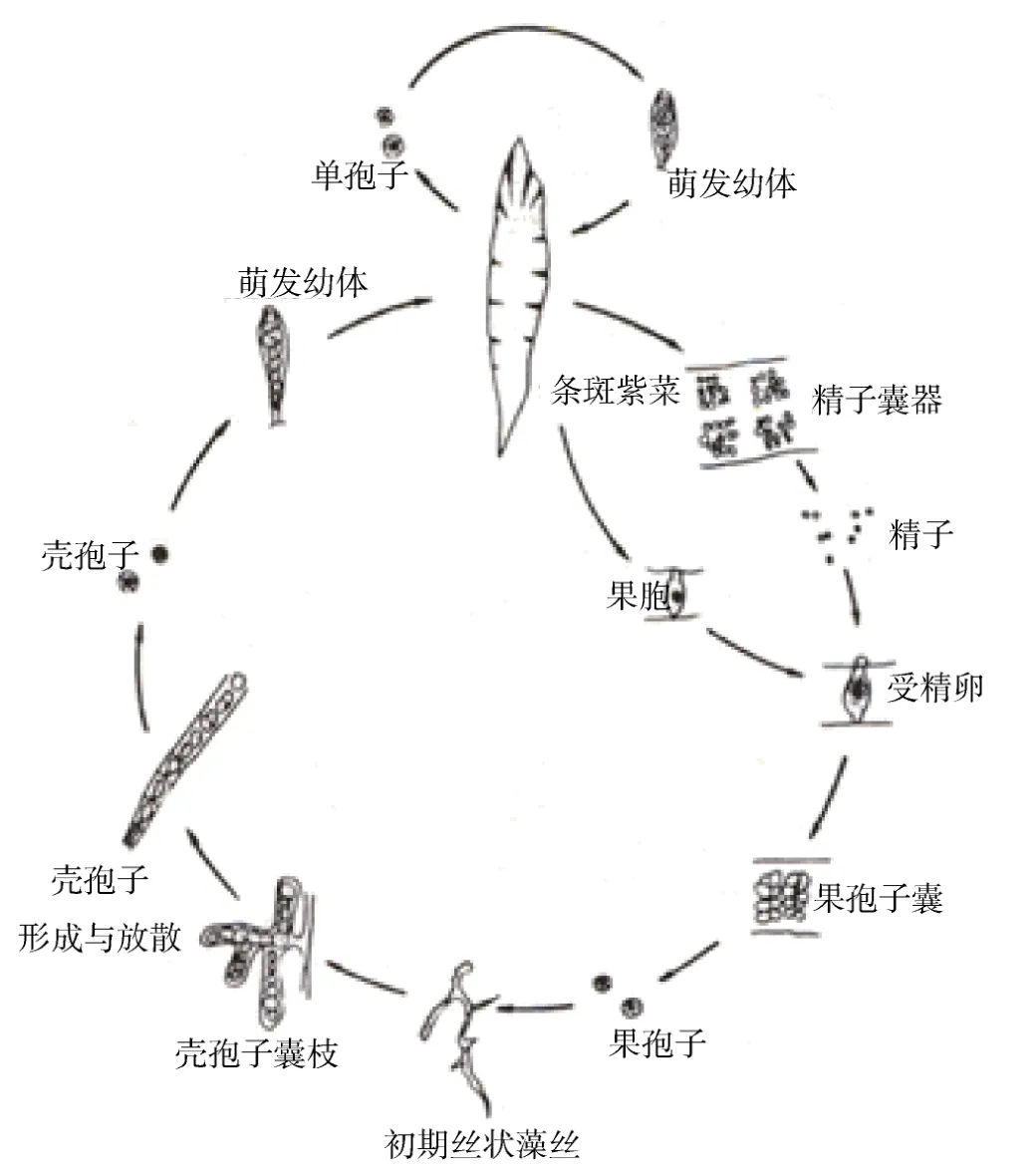
图1 条斑紫菜的生活史
2 紫菜的分子生物学研究
对紫菜属物种开展分子生物学研究早在1993年就已经开始了[3]。主要包括对DNA多态性进行的分子标记研究;对特定的功能进行的基因组学研究和功能基因组学研究。主要目的是测定基因序列以鉴定物种,确定物种之间的亲缘关系,关注这些研究的主要是遗传学和分类学研究者。Stiller等[3]对太平洋东北部一个新种P.rediviva的核糖体小亚基rRNA基因序列(small subunit rDNA,SSU rDNA)以及位于核糖体大小亚基 B/E-基因序列之间的间隔序列(internal transcribed spacer,ITS)进行 RFLP研究,并与其他几种紫菜进行比较分析后认为该新种与北大西洋的另一种紫菜P.purpurea有很近的亲缘关系。
通过国内外对紫菜属海藻分子标记技术研究获得的结果可以发现在紫菜属中具有丰富的遗传多样性,可以在今后的物种选育中作为很好的良种来源开展遗传育种工作。这些遗传多样性不一定表现于形态的多样性,不同种紫菜间具有很接近的外部形态往往是由于平行演化造成的非同源性相似,这对紫菜的系统分类乃至种系发生研究都颇具迷惑性。Niwa等[4]同时运用形态分类方法和 RFLP技术对 1种可能属于甘紫菜但尚未确定的野生个体进行鉴定分类,两种结果表明该野生个体属于甘紫菜,基于SSU rDNA和ITS序列的PCR-RFLP分析结果还表明与条斑紫菜相比具有较远的遗传距离。Niwa的研究表明紫菜系统分类仅依靠外部形态特征是不够的,而将形态与DNA分析两种手段相结合则可以较好的解决以上问题。He等[5]运用AFLP(amplified fragment length polymorphism)技术对来自同一个丝状体的两种叶片,由壳孢子萌发出来的40株和单孢子形成的88株,进行了电泳条带的比较,发现同一个来源的后代遗传相似度不高,说明它们之间发生了杂交,单孢子苗的多态性位点的比例高达 98.6%,超过壳孢子苗的 80.7%;而平均遗传相似度单孢子苗为 0.61,低于壳孢子苗的0.71。
通过测定核酸一级结构中核苷酸序列组成来比较同源分子之间相关性的方法称为核酸序列分析方法。分子系统学研究中常使用的基因组有三种: 叶绿体基因组、线粒体基因组、核基因组。Shivji[6]在20世纪 90 年代初构建了条斑紫菜叶绿体基因组图谱并对其结构作了报道,Reith[7]构建了P.purpurea的叶绿体基因组图谱,发现有 46%的基因为陆生植物所不具备。但由于技术要求以及耗费较大,目前测序主要集中在rbcL、ITS等一些小片段。近年来,随着DNA 测序技术的迅速发展和日益普及,DNA 测序在遗传多样性的研究中正在起着越来越大的作用。Yang 等[8]运用最新的 Solexa测序技术对条斑紫菜的转录组进行了大规模的测序,获得了大量的新基因;牛建峰等[9]对条斑紫菜(P.yezoensis)进行Solexa高通量测序,获得低覆盖度全基因组草图。该基因组草图大小约 220 Mbp,GC质量分数 53.08%;包含26 629 个预测基因,其中16 409个基因具有内含子,平均每个基因含 2.22 个内含子;基因结构分析表明具有内含子的基因平均长度为 2 214 bp,内含子平均大小 319 bp;代谢通路分析表明嘌呤代谢是含有蛋白数量最多的代谢途径。这些工作为以后开展功能基因的研究提供了丰富的研究素材。
2011年Shen等[10]利用基因抑制消减杂交(SSH)技术对条斑紫菜配子体和孢子体基因表达谱差异进行了研究。结果表明,选择果孢子替代丝状壳孢子作为配子体的材料,并从相同的叶状体上培养营养细胞(配子体)和果孢子(孢子体)细胞,这样能将样品间的差异减少到最低。测序后得到相应克隆的EST(Expressed sequence tag)序列,共有 360个序列已全部提交 NCBI。果孢子的 181个 EST序列号为HS572581- HS572762,营养细胞的179个EST序列号为HS572763- HS572942。将EST进行BLAST分析显示,共有196条基因,其中果孢子细胞有92条,营养细胞有104条。GO注释显示孢子体基因主要定位在细胞质和细胞核(58%);而配子体基因主要定位在质体、高尔基体、核糖体和内质网上。营养细胞主要的生物学功能与 DNA复制/修复、碳水化合物代谢、物质转运和转录,特别是对热激和对氧化性的应激方面;孢子体世代同样表达这些代谢过程,尤其在信号转导、DNA和蛋白质修饰和蛋白质和核苷酸代谢方面。根据 GO注释的结果发现孢子体世代倾向于生长和自我保护;而孢子体世代在发育上更积极。
He等[11]对条斑紫菜孢子体和配子体的 MicroRNAs进行了比较分析,测序得到的14个miRNAs中只有一个在两个世代中同时出现,几个新的miRNAs和其他生物没有同源性,说明紫菜中可能存在复杂的小RNA形成系统。
对于紫菜属的功能基因研究目前正处在蓬勃发展的阶段,由于在紫菜中转入外源基因来进行表达存在一定的困难,王金锋等[12]尝试将外源基因用玻璃珠法转入坛紫菜果孢子中,但是要将外源基因稳定表达出来还有很长的路要走。目前主要从紫菜中获得已知功能的基因在原核生物或者酵母、衣藻等真核生物中表达,以确定其功能。Wang等[13]就是从条斑紫菜中获得海藻糖 6磷酸合成酶基因转移到大肠杆菌中表达。Wu等[14]测定了条斑紫菜不同生活周期中的 7个管家基因的表达水平,通过检测这些基因的绝对表达量,发现 GAPDH的表达量最为稳定适合作为检测基因表达量的内参基因。张宝玉等[15]克隆了条斑紫菜alternative oxidase (AOX)的全长基因,共1650 bp,ORF为1332 bp,编码443个氨基酸。通过 qPCR检测了该基因在生活史个阶段的表达水平,发现丝状体阶段的AOX表达量显著高于叶状体阶段。
功能基因的研究急待加强
核糖体 RNA基因(rDNA)存在于所有的生物中,由一些高度重复序列组成、并有转录活性的多基因家族,它在真核细胞中有几十甚至数千个拷贝,在植物总DNA中约占10%。rRNA基因在真核生物中是高度保守的,它以18S rDNA(small subunit,SSU)、5.8S rDNA和28S rDNA(large subunit,LSU)顺序串联,组成一个基因簇,彼此被内转录间隔区(internal transcribed spacer,ITS)序列分开,而转录单元之间被基因间间隔区(intergenic spacer,IGS)序列间隔区分开。真核生物rDNA基因簇结构如模式图所示(图2)。在每个重复片段中,两个内部转录间隔区 ITS1和ITS2将18S、5.8S和28S分隔开。18S rDNA上游含有一个外转录间隔区(ETS),在ITS和ETS中含有 rDNA前体加工信息,相邻的重复片段之间为非编码区 IGS隔开, IGS中有促转录的增强子,在转录时有启动和识别的作用[16]。
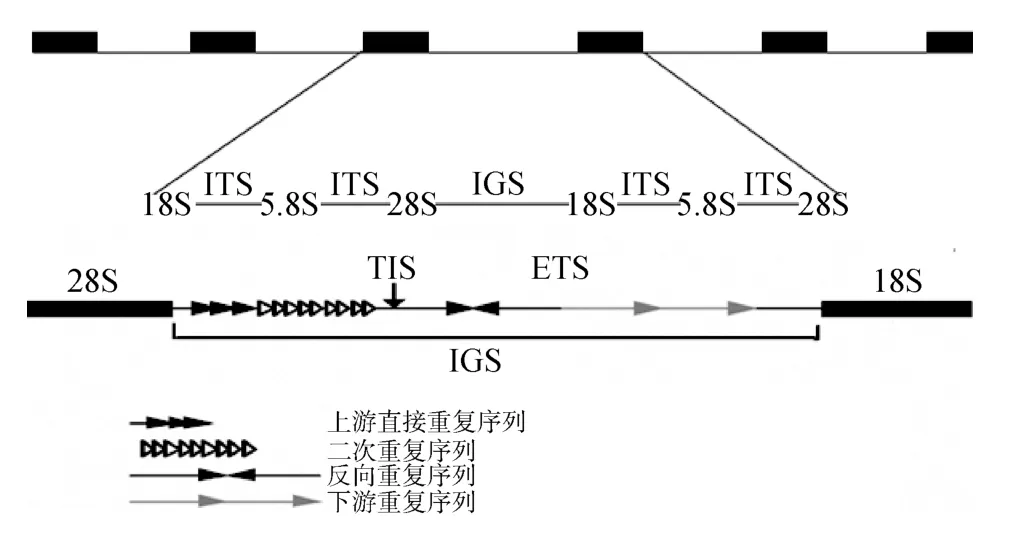
图2 植物18S~28SrDNA的基本结构示意图
rRNA的生物学功能在不同的物种中保持一致,成为分子进化的忠实尺度,消除了趋同进化和趋异进化所引起的系统误差。在进化速率上,编码区比较保守,可在种、属、科、目、水平上用于不同生物种的比较。18S rDNA和28S rDNA基因的3′端是高度保守的,即使在原核与真核生物之间也能建立位点同源性。其中,18S比28S基因更保守,5.8S基因仅有160个左右的碱基,最为保守。18S rDNA和28S rDNA基因是系统发育中种及种以上阶元的良好标记。rDNA序列由于在不同生物之间既有高度的保守性,又存在一定的序列变异性,已经成为生物分子鉴定和定量检测中最常用的分子指标[17-18]。
18S rDNA是唯一具有信息分子和功能分子两种作用的编码核糖体的基因,为高度重复序列,约800~10000拷贝,在细胞内连续排列。同时18S rDNA基因序列结构、功能十分保守,若它们的序列存在变异就能成为一种稳定的DNA标记,用于鉴别物种。由于18S rDNA区高度保守,主要适合科及以上阶元,而18S rDNA的全序列相对较小,更适合PCR扩增和测序,因此已被广泛的应用于藻类的分子系统学研究[19-20],解决了许多疑难海洋藻类的分类鉴定问题。
由于核糖体大亚基基因 28S rDNA具有高度可变区序列,能为设计 PCR引物或探针提供特异性强的序列,因此被用于区别分类物种之间的亲缘关系如属、种或地理株系等问题。在藻类中28S rDNA编码区的序列比较分析已多用于分类学和系统发生等方面的研究[21-22]。但目前国内利用核糖体大亚基来探讨紫菜属的系统发育的研究则未见报道。
Broom等[23]对分布于新西兰沿岸的49个紫菜核糖体的18S rRNA基因序列进行测序分析,结果表明,该区域的紫菜属植物存在较大的遗传变异和较远的种间遗传距离,这与它们的外部形态特征并不吻合,这很有可能是长期的生殖隔离所致。Kunimoto等[24]对 9种紫菜的 18S rDNA全序列进行了测序分析,SSU rDNA外显子序列在同种紫菜种内保持一致,体现出很高的保守性,而内含子序列在种内存在差异,并根据内含子的插入与缺失,将它们的rRNA基因结构分为 4种类型,结果表明不同地理居群的同种紫菜其18S 基因结构存在差异。
核糖体 DNA(rDNA)的内转录间隔区(ITS)和基因内间隔区(IGS)进化速度快,序列变异丰富,能够提供比较丰富的变异位点和信息位点。IGS是rDNA中进化速度最快的区域,在已经研究的IGS 序列中,发现 IGS 序列比 ITS序列存在着更多层次的差异,不仅在种间、种内居群间存在着差异性,甚至不同个体之间也存在着差异性[25]。在分子系统学研究中常以ITS或IGS序列作为构建系统进化树的基础。
Mizukami等[26]对野生种和栽培种条斑紫菜的ITS序列分析结果表明,野生种和栽培种ITS序列之间存在差异,并且野生种的不同个体间其 ITS序列也存在一定的差异。Niwa[27]对7种栽培品系条斑紫菜的 ITS1 rDNA序列进行了测序分析,结果表明,这7个品系的ITS1序列不存在差异性,因而不适合作为这7个品系的分类鉴定标记。
Kunimoto等[28]对甘紫菜的SSU rDNA和ITS测序结果表明,SSU rDNA外显子序列在甘紫菜种内保持一致,体现出很高的保守性,而内含子序列在种内存在差异,所以内含子以及ITS1序列可作为遗传标记进行种内遗传差异的鉴定,从而建立不同的甘紫菜品系。
在亲缘关系较近的物种分类鉴定研究中,当ITS序列存在争议时,IGS序列可以提供更有力的支持。对栽培稻种内ITS序列研究表明,利用ITS序列能将典型籼粳亚种分开,但亲缘关系较近的品种其 ITS序列完全相同。戴小军等[18]对栽培稻种内IGS序列研究表明,亲缘关系较近的品种其 ITS序列完全相同,由于 IGS序列比 ITS序列的信息位点多,基于IGS序列能将亚种内,特别是亲缘关系较近的品种区分开。因此 IGS序列更适于亚种内不同品种间遗传关系的分析。
本实验室2009年最早在藻类中克隆出了IGS的部分碱基序列,长度达到 1100 bp,在浙江宁波、福建莆田和广东汕头栽培的坛紫菜中发现25个碱基的变异,能够区分这些同一个物种不同地理群之间的品种差异。相关结果发表在 Journal of Applied Phycology上[29]。目前本实验室又对条斑紫菜的28S rRNA全序列进行了克隆,并获得了部分IGS碱基序列。2011年年底,Sutherland等[30]15位国际著名的藻类学家在美国的《Journal of Phycology》上发表了一篇文章,以18S rDNA序列和rbcL序列构建系统树,按照单系起源的原则,将广义的紫菜属分成了15个属,其中叶状体的 8个属包括 Porphyra,Pyropia,Wildemania和5个新属。产自中国沿海的22个物种都属于 Pyropia属,但是由于缺乏中国的研究者参与,采集的物种没有本地区的样品,需要更多的中国当地的研究工作来提供证据,特别需要 28S rDNA和IGS这样更易变化的分子序列来确定紫菜物种的亲缘关系。
[1]Yoshida T.Catalogus of species of Porphyra in the world,with special reference to the type locality and bibliography[J].Nat Hist Res Special Issue,1997,3:5-8.
[2]张学成,马家海,秦松,等.海藻遗传学[M].中国农业出版社,北京: 2005: 185-186.
[3]Stiller J W,Waaland J R.Molecular analysis reveals cryptic diversity in Porphyra (Rhodophyta)[J].Fish Sci,1993,62: 173-177.
[4]Niwa K,Kikuchi N,Aruga Y.Morphological and molecular analysis of the endangered species Porphyratenera (Bangiales,Rhodophyta)[J].J Phycol,2005,41:293-304.
[5]HeL,Zhu J,Lu Q,et al.Genetic similarity analysis within Pyropia yezoensis blades developed from both conchospores and blade archeospores using AFLP[J].J Phycol,2013,49: 517-522.
[6]Shivji M S.Organization of the chloroplast genome in the red alga Porphyra yezoensis[J].Cur Gene,1991,19:49-54.
[7]Reith M,Munholland J.A high resolution gene ma p of the chloroplast genome of the red alga Porphyra purpurea[J].Plant Cell,1993,5: 465-475.
[8]Yang H,Mao Y X,Kong F N,et al.Profiling of the transcriptome of Porphyra yezoensis with Solexa sequencing technology[J].Chinese Sci Bull,2011,56:2119-2130.
[9]牛建峰,高胜寒,骆迎峰,等.条斑紫菜低覆盖度基因组草图分析[J].海洋科学,2011,35(6): 76-81.
[10]Shen S D,Zhang G C,Li Y Y,et al.Comparison of RNA expression profiles on generations of Porphyra yezoensis (Rhodophyta),based on suppression subtractive hybridization (SSH)[J].BMC Research Notes,2011,4: 428.
[11]He L,Huang A,Shen S,et al.Comparative Analysis of microRNAs between sporophyte and gametophyte of Porphyra yezoensis[J].Comp and Func Genomics,2012,9(1): 28-43.
[12]Wang J F,Jiang P,Cui Y,et al.Gene transfer into conchospores of Porphyra haitanensis (Bangiales,Rhodophyta)by glass bead agitation[J].Phycol,2010,49: 355-360.
[13]Wang G L,Zhao G,Feng Y B,et al.Cloning and comparative studies of seaweed trehalose-6-phosphate synthase genes[J].Mar Drugs,2010,8: 2065-2079.
[14]Wu X,Huang A,Xu M,et al.Variation of expression levels of seven housekeeping genes at different life-history stages in Porphyra yezoensis[J].PLoS ONE,2013,8(4): 607-612.
[15]Zhang B Y,He L,Jia Z,et al.Characterization of the alternative oxidase gene in Porphyra yezoensis (Rhodophyta)and cyanide-resistant respiration analysis[J].J Phycol,2012,48(3): 657-663.
[16]Leskinen E,Pamilo P.Evolution of the ITS sequences of ribosomal DNA in Enteromorpha (Chlorophyceae)[J].Hereditas,1997,126: 17-23.
[17]Blomster J,Hoey E M,Maggs CA,et al.Species-specific oligonucleotide probes for macroalgae:molecular discrimination of two marine fouling species of Enteromorpha (Ulvophyceae)[J].MolEcol,2000,9:177-186.
[18]Sallares R,Brown T A.Phylogenetic analysis of complete 59 external transcribed spacers of the 18S ribosomal RNA genes of diploid Aegilops and related species (Triticeae,Poaceae)[J].Gene Res and Crop Evol,2004,51: 701-712.
[19]Muller K M,Sheath R G,Vis M L,et al.Biogeography and systematics of Bangia (Bangiales,Rhodophyta)based on the rubisco spacer,rbcL gene and 18S rRNA gene sequences and morphological analyses in North America[J].Phycologia,1998,37(3): 195-207.
[20]Sandra V T,Lindstrom C,Saunders G W.Phylogeny of the Dumontiaceae (Gigartinales,Rhodophyta)and associated families based on SSU rDNA and internal transcribed spacer sequence data[J].J Phycol,2001,37:184-196.
[21]Lellaert F,Rousseau F,Reviers B D,et al.Phylogeny of the Cladophorophyceae (Chlorophyta)inferred from partial LSU rRNA gene sequences: is the recognition of a separate order Siphonocladales justified?[J].Eur J Phycol,2003,38: 233-246.
[22]梅洪,刘国祥,胡征宇.基于28S rDNA序列的鞘藻目系统发育研究[J].水生生物学报,2007,31(4): 492-498.
[23]Broom J E,Nelson W A,Yarish C,et al.A reassessment of the taxomomic status of Porphyra suborbiculata,Porphyra carolinensis and Porphyra lilliputiana(Bangiales,Rhodophyta)based on molecular and morphological data[J].Eur J Phycol,2002,37:227-235.
[24]Kunimoto M,Kito H,Cheney D P,et al.Discrimination of Porphyra species based on small subunit ribosomal RNA gene sequence[J].J Appl Phycol,1999,11:203-209.
[25]戴小军,欧立军,李文嘉,等.栽培稻种内rDNA基因IGS序列分析及系统学意义[J].作物学报,2008,34(9):1569-1573.
[26]Mizukami Y,Kito H,Kaminishi Y,et al.Nucleotide sequence variation in the ribosomal internal transcribed spacer regions of cultivated (cultivars)and field collected thalli of Porphyra yezoensis[J].Fish Sci,1999,65: 788-789.
[27]Niwa K,Aruga Y.Rapid DNA extraction from conchocelis and ITS-1 rDNA sequences of seven strains of cultivated Porphyra yezoensis (Bangiales,Rhodophyta)[J].J Appl Phycol,2003,15: 29-35.
[28]Kunimoto M,Kito H,Mizukami Y,et al.Molecular features of a defined genetic marker for determination of the Porphyra tenera lineage[J].J Appl Phycol,2003,15: 337-343.
[29]Li Y Y,Shen S D,He L H,et al.Sequence analysis of rDNAintergenic spacer (IGS)of Porphyra haitanensis[J].J Appl Phycol,2010,22: 187-193
[30]Sutherland J E,Lindstrom S C,Nelson W A,et al.A new look at an ancient order: Generic revision of Bangiales(Rhodophyta)[J].J Phycol,2011,47,1131-1151.
