东北春大豆花荚脱落性状与SSR标记的关联分析
2014-06-12晓环燕平
, ,,晓环, ,燕平,,,,,,,
(1.中国科学院 大豆分子设计育种重点实验室,中国科学院 东北地理与农业生态研究所,黑龙江 哈尔滨 150081;2.中国科学院大学,北京 100049;3.黑龙江省农业科学院 牡丹江分院,黑龙江 牡丹江 157041)
0 引 言
植物器官脱落是自然界中的普遍现象,是植物自我保护的方式之一,但是在农业生产中,植物器官的脱落会带来严重的损失,如大豆的落花落荚。对植物器官脱落,特别是作物器官脱落进行研究,具有重要的意义。近年来,关于植物器官脱落的研究已深入到分子生物学水平,研究认为植物器官脱落的生理生化活动主要发生在器官基部的离区内。在植物离区发育和器官脱落相关基因的研究中,拟南芥和水稻的研究较为成熟。在拟南芥中,已鉴定出多个与离区发育和器官脱落相关基因,如影响花器官的离层发育的KNAT/BP基因[1]和引发花器官脱落的IDA和HAESE基因[2]。并且解析了由AS1、AS2、JAG、KNAT1/BP和FIL等组成的拟南芥果荚离层发育调控网络[3-5]。在水稻中,落粒性的分子机制研究也有了突破性进展,目前已成功克隆了落粒性基因SH4、SHA1、qSH1和OsSH1[6-9]。
大豆花荚脱落普遍存在于大豆生殖生长过程中,是影响产量提高的重要因素之一。实验和实践证明,通过降低花荚脱落率,提高大豆籽粒生产的潜力很大[10]。大豆花荚脱落率可达30%~80%[11],在不同品种间存在明显差异,这为研究调控大豆花荚离区发育和器官脱落相关基因提供了材料平台。长期以来,研究者对大豆花荚脱落的生理生化和激素水平进行了大量研究,在大豆花数和荚数相关QTL定位方面也取得了较大的进展[12-13],但在花荚脱落基因定位方面的研究相对较少。
关联作图是以自然群体为材料,以连锁不平衡为基础的一种分析方法,可用于鉴定群体内与目标性状密切相关的候选基因或遗传标记。关联作图自Thornsberry等首次应用于植物数量性状研究后[14],因其作图不需构建群体、定位精确、考察位点多[15]等优点,被广泛应用于作物数量性状基因定位研究。近些年利用关联作图解析与大豆产量性状相关SSR分子标记的研究已成为热点,2008年文自翔等[16]对大豆的16个农艺、品质性状与SSR标记进行了关联分析;2013年范虎等[17]对百粒重、开花期、成熟期等农艺性状与SSR标记进行关联分析。在范虎的研究中共获得51个关联位点,其中16个与连锁分析定位的QTL一致。
研究以东北春大豆种质资源组成的自然群体为实验材料,种植于黑龙江省农科院牡丹江分院大豆试验田,对其花荚脱落情况精确鉴定,2 a重复。利用分布于20个连锁群上的205个SSR标记进行基因分型。在遗传多样性、群体结构和连锁不平衡情况的分析基础上,利用关联分析方法解析与东北春大豆花荚脱落率显著相关的分子标记位点。研究结果可为后续相关基因克隆和分子育种提供参考。
1 材料与方法
1.1 试验材料及田间设计
以1960年-2011年东北育种单位育成的主要品种和部分祖先品种为材料,2011年为104个东北春大豆品种,2012年在2011年已有品种的基础上增加至314个。田间试验分别于2011年和2012年在黑龙江省农科院牡丹江分院大豆试验田进行,随机区组实验设计。2011年每个品种种植4行,行长3 m,行距70 cm,种植密度为每平方米10株,3次重复。2012年行长2 m,行距65 cm,种植密度分别为每平方米10株和20株。2 a使用同样的田间管理方法。
1.2 表型性状调查与分析
Fehr[18]提出的大豆生育时期的鉴定方法确定大豆生育时期。在R1期,每品种连续取5株,在垄上用纱布网将5株围起,垄上地面纱布网封闭,形成上开口的长方形箱,保证花荚脱落在封闭的长方形箱内。在花期和成熟期分别调查纱布网内花和荚的脱落数,R8期对纱网内5株进行考种,花荚脱落率为平均单株花荚脱落数占平均单株花荚总数的百分比。利用SAS 8.0软件分别统计花荚脱落率的平均值、标准差、变异系数等。
1.3 SSR标记全基因组扫描
利用改良的SDS法[19],从大豆种子中提取总DNA。依据2011年统计的花荚脱落率结果,由高到低选择10份有代表性的品种作为样本筛选引物,从大豆公共遗传图谱的1000对引物中筛选出均匀分布于在20个连锁群具有多态性的引物205对。PCR反应体系为10 μL,含1 μL DNA(100 ng·L-1)、1 μL 引物对(10 ng·L-1)、5 μL 2×Takara PCR master mix,3 μL dH2O,使用ABI Gene Amp PCR System 9700 PCR 仪扩增。PCR反应程序为95℃ 4 min;95℃ 30 sec,46℃ 30 sec,72℃ 30 sec,40个循环;72℃ 7 min,4℃保存。用12%的聚丙烯氨酰胺凝胶分离PCR扩增产物,EB替代染料染色。胶片在BIO-RAD Gel Doc XR System成像系统中扫描分析。
1.4 数据处理
1.4.1SSR标记分析。用Quantity One软件依据Takara 50 bp DNA Ladder Marker确定SSR等位变异的分子量,根据聚丙烯凝胶电泳的结果统计每个样品的等位变异类型。
用Power Marker V3.25 软件计算每个SSR位点的变异数目、变异频率和多态信息含量(polymorphism information content,PIC)。用STRUCTURE 2.3软件对2011年(104个品种)和2012年(314个品种)的群体进行类群划分。本文选取205个SSR位点进行分析,假设每个位点独立,并设定群体数目(K)为2~10每个K值运行5次,再跟据Evanno[20]等发表的方法确定合适的K值。公式为:Δk=m[|L(K+1)-2L(K)+L(K-1)|]/s[L(K)]。
用TASSEL 2.1软件中Link.Diseq.分析样本中成对SSR位点的r2和D′值,绘制LD配对情况矩阵图。从中筛选出共线(位于同一连锁群)SSR位点对,以D′值作为纵坐标,以SSR位点对的相对遗传距离为横坐标,利用SPSS 19绘制衰减散点图、并配置对数函数方程。
1.4.2表型性状与SSR标记的关联分析。用TASSEL 2.1软件中的GLM(general linear model)程序,将各个体的Q值作为协变量,对东北春大豆的表型数据和SSR标记的变异进行回归分析,分析结果中p<0.05的标记被认为与目标性状相关联。
2 结果与分析
2.1 表型性状分析
对2011年和2012年的田间表型数据统计分析可知,见表1。

表1 供试群体材料花荚脱落率的统计分析Tab.1 Statistic data of flower and pod abscission rate
2011年大豆花荚脱落率为22.63%~77.90%,平均花荚脱落率为49.26%。2012年,种植密度为每平方米10株的大豆花荚脱落率为10.86%~59.16%,平均花荚脱落率为32.84%;种植密度为每平方米20株的大豆花荚脱落率为15.93%~53.34%,平均花荚脱落率为33.36%。比较两年的统计数据可知,2011年的大豆花荚脱落率的平均值高于2012年两种种植密度,且差异较大。比较2012年不同种植密度的统计数据可知,种植密度为每平方米10株的大豆花荚脱落率低于种植密度为每平方米20株,差异也较大。2 a测得的相同品种大豆花荚脱落率虽然不同,但在不同年际、不同种植密度的群体中各品种的花荚脱落率高低趋势是一致的。
2.2 遗传多样性分析
利用205对SSR引物标记分析2011年和2012年的参试东北春大豆的遗传多样性,统计各位点等位变异数和多样性信息量(polymorphism information contents,PIC),见表2。2 a中所有标记位点的等位变异数与其PIC值都呈极显著正相关(2011年:r=0.551,p<0.01;2012年:r=0.528,p<0.01)。
在2011年104份大豆材料的205个SSR标记中共检测到等位变异659个,标记的等位变异数目变化范围为2~10个,每个标记平均有3.214个等位变异,标记PIC值变幅为0.054~0.766。其中,N连锁群的平均PIC值最高,为0.456;H连锁群的平均PIC值最低,为0.27。
在2012年314份供试材料的205个SSR标记中共检测到等位变异763个,标记的等位变异数目变化范围为2~12个,每个标记平均有3.722个等位变异,标记PIC值变幅为0.054~0.771。其中,D1b连锁群的平均PIC值最高,为0.453;H连锁群的平均PIC值最低,为0.280。
在所有SSR标记中,Sat_406(A2)和Satt592(O)均检测到5个等位变异,PIC值分别为0.703和0.357,差异很大。Satt592等位变异的分布不均匀,其中Satt592-1的频率高达76%,说明Satt592的选择倾向性较强,说明Satt592可能与控制某农艺性状的基因紧密连锁。

表2 2011年和2012年东北春大豆品种中205个SSR位点的等位变异和多态性信息含量(PIC)Tab.2 Allele number and polymorphic information content (PIC) of 205 SSR markers in 2011 and 2012 northeast spring sowing soybeans
2.3 群体遗传结构分析
群体结构是关联分析在植物遗传学中应用的首要限制因素,在进行关联分析之前,明确群体的遗传结构并在分析中加以控制,能够降低伪关联的概率。研究利用205对引物数据,采用基于数学模型的聚类方法对参试材料进行遗传结构分析,确定参试材料的亚群数目和群体结构。根据Evanno的方法,用ΔK确定K值。结果表明当参试种质的等位变异频率特征类型数K=2时,2011年104份和2012年314份材料的ΔK都为最大值。因此判断两样本亚群数都为2,群体结构分析结果如图1所示。按上述方法分类方法,将组成各亚群中品种的花荚脱落率进行统计,见表3。结果表明2011年不同品种间花荚脱落率差异为3.5倍,2012年种植密度为10株每平方米的不同品种间脱落率差异可达5.4倍,2012年种植密度为20株每平方米的不同品种间脱落率差异为3.3倍。分别对不同年际、不同种植密度两亚群进行单因素方差分析(Analysis of Variance,ANOVA)显示,两亚群间花荚脱落率差异显著,说明K=2与大豆花荚脱落率高低亚群的划分一致。

图1 2011年及2012年参试东北春大豆群体结构Fig.1 Population structure in Northeast spring sowing soybeans of 2011 and 2012注:2011年和2012年参试东北春大豆群体结构都为2个亚群,X轴为品种数目,Y轴上标记的数值代表该样品为该亚群的可能性。两种不同颜色代表STRCTURE软件预测被分为2个亚群。

2011年(10 plants·m-2)2012年(10 plants·m-2)2012年(20 plants·m-2)亚群SubpopulationPOP1POP2POP1POP2POP1POP2数量Number3470197116197116平均值Mean(%)51.3345.2529.2039.2631.6336.29标准差SD(%)11.3811.848.2910.066.957.67范围Range(%)32.66~77.9022.62~72.0910.86~51.9916.49~59.1615.93~52.9217.27~53.34F值 F value2.5991.6630.30p值 p value0.010.000.00
注:两个亚群的单因素方差分析:p<0.05表示两亚群间差异显著,p<0.01表示两亚群间差异极显著。
2.4 东北春大豆的连锁不平衡
图2显示了各个连锁群上SSR位点间连锁不平衡的情况,在205个SSR位点所有组合中,共线性的组合(同一连锁群)和非共线性组合(不同连锁群)都存在一定程度LD。LD成对位点数在2011年群体基因组中占所有位点组合的5.40%,在2012年群体基因组中占17.08%。从绝对数量上看,2012年群体拥有的不平衡成对位点数较2011年群体位点多,但从D′值次数分布及平均值来看,2011年群体间连锁不平衡程度更高,见表4。
LD衰减延伸的距离决定了关联分析所需使用分子标记数量以及关联分析精度。将共线性成对SSR位点的D′值随遗传距离的增加而发生的变化做回归分析,无论是在2011年还是2012年群体中,SSR位点D′值得衰减都很快,见图3,且两年D′值衰减都遵循方程Y=bln(x)+c。以D′最大值与最小值中点0.415(2011年)和0.492(2012年)为衰减临界,可分别求出2011年群体和2012年群体LD衰减延伸的最小距离分别为23.37cM和2.62cM。两年LD衰减延伸的最小距离不同且差别较大,可能是由于2012年的实验材料多,品种间亲缘关系复杂所致。

图2 2011和2012年东北春大豆20个连锁群205个SSR位点间连锁不平衡的分布Fig.2 Distribution of LD among 205 SSR loci on 20 linkage groups in northeast spring sowing soybeans of 2011 and 2012注:黑色对角线上方每一像素格使用右侧的色差代码表征成对位点间D′值大小,对角线下方为成对位点间LD的支持概率。

群体PopulationLD成对位点数Number of LD locus pairsD'值次数分布 Freq.dis.of D'(p<0.01)0-0.20.2-0.40.4-0.60.6-0.80.8-1D'平均值Mean of D'104份1123(5.40%)27593340113500.43314份3554(17.08%)1143193237186220.27

图3 共线SSR位点D′在2011年群体(A)及2012年群体(B)基因组随遗传距离衰减散点图Fig.3 Attenuation of D′value between synthetic marker pairs along with genetic distance increase in 2011 (A) and 2012 (B)
2.5 SSR标记与东北春大豆花荚脱落性状的关联分析
关联分析结果发现共有33个位点与大豆花荚脱落性状显著相关,其中Satt534(B2)、Satt452(E)、Satt244(J)和Satt478(O)在两年中都被检测到,见表5。
比较2011年与2012年重复的104个品种的花荚脱落率关联分析结果发现:①与2011年花荚脱落性状显著相关的SSR位点有7个,其中解释率最高的为Satt534(B1),其对表型变异的解释率为0.21。与2012年花荚脱落率显著相关的SSR位点有12个,其中解释率最高的为Sat_245(L),其对表型变异的解释率为0.20。② 两年均与花荚脱落率显著相关的位点有3个,分别为Satt534(B2)、Satt244(J)以及Satt478(O),平均解释率分别为0.19、0.15和0.14。
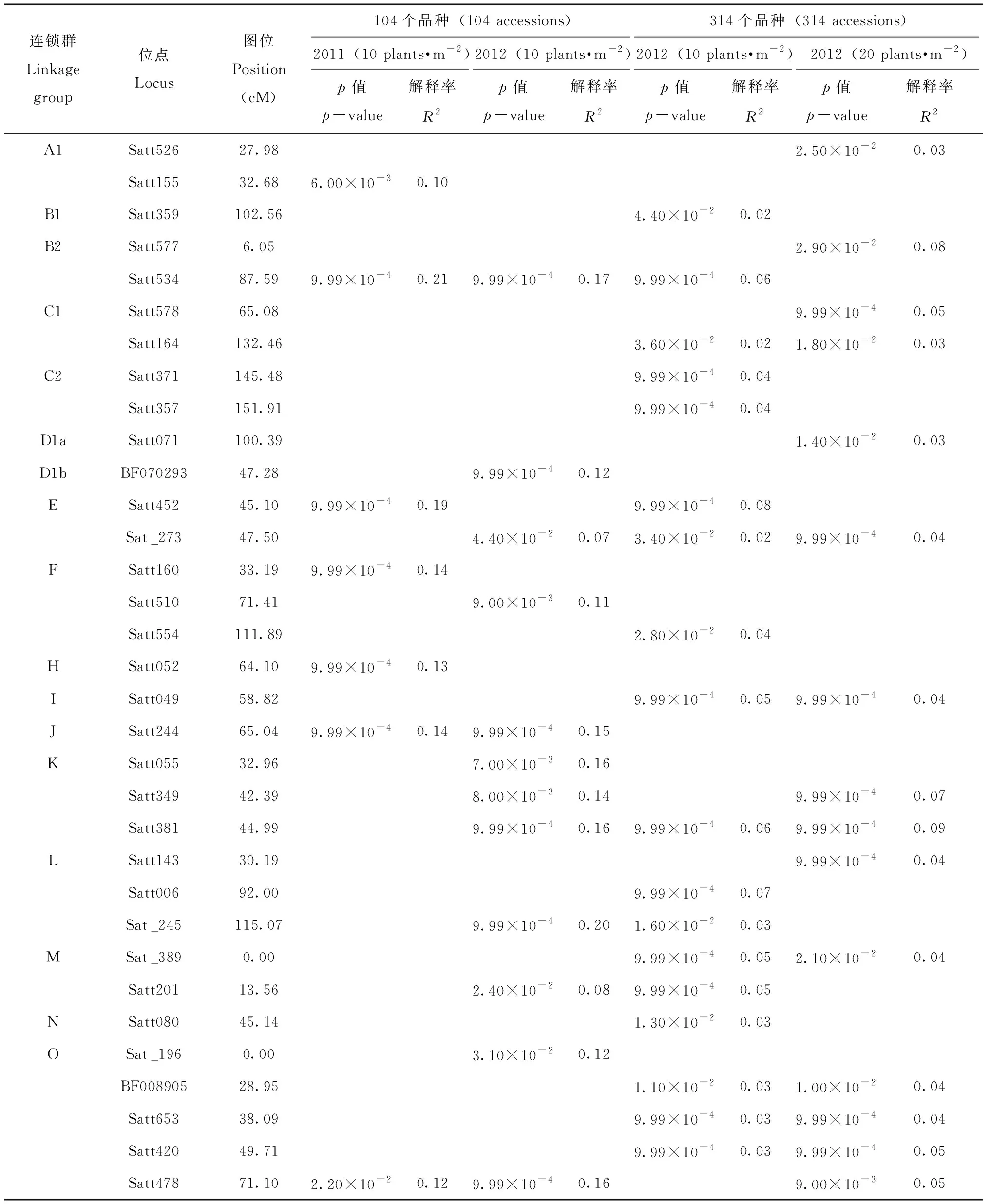
表5 与东北春大豆落花落荚率显著相关的标记位点及其对表型变异的解释率Tab.5 Marker loci associated with flower and pod abscission rate of northeast spring sowing soybeans and their explained phenotypic variation
比较2012年不同密度下种植的314个品种的花荚脱落率关联分析结果发现,①与每平方米10株的材料大豆花荚脱落率性状显著相关的位点有18个,其中解释率最高的为Satt452(E),其对表型变异的解释率为0.08。与每平方米20株的材料大豆花荚脱落率性状显著相关的位点有15个,其中解释率最高的为Satt381(K),其对表型变异的解释率为0.09。②两种种植密度条件下均与花荚脱落率显著相关的位点有8个,其中Satt381(K)的解释率最高为0.08。
比较2011年104个品种和2012年种植密度为每平方米10株314个品种的花荚脱落率关联分析结果发现,Satt534(B2)和Satt452(E)均与花荚脱落率显著相关。
3 讨 论
3.1 关联分析与连锁不平衡
关联分析是以连锁不平衡(LD)为基础的一种分析方法,可以用于分析表型性状与标记或候选基因遗传变异之间联系[21]。LD分析的是一个群体内的不同座位等位基因之间的非随机关联,可以是两个标记间或两个基因/QTL间的随机关联,也可以是一个基因/QTL与一个标记座位间的随机关联,所以又称配子不平衡、配子相不平衡或等位基因关联[22]。在影响群体LD水平的众多因素中,其中对LD程度具有决定性的影响是物种的交配方式,通常自交物种具有较高水平的LD,异交物种具有较低水平的LD。
大豆是自花授粉植物,其LD水平较高。有研究表明大豆的LD衰减距离大于50 kb[23],甚至超过10 cM[24]。研究中东北春大豆的LD衰减距离为23.37 cM(2011年)和2.62 cM(2012年),与前人研究相符。同时也说明LD具有明显的群体依赖性,研究中两群体品种虽然都为东北春大豆,但由于群体的组成不同,最终导致两群体LD衰减距离不同。
3.2 关联分析与功能标记的开发及应用
近年来,随着关联分析在植物研究中的成功应用,许多与大豆产量性状相关的功能标记被成功开发并应用。郝德荣等[25]利用1536个SNP标记和209个单倍型分析191个大豆地方品种,得到与产量和产量组成性状显著相关的标记。张丹等[26]应用关联分析鉴定大豆开花基因,将该基因定位在6号染色体上BARC-014947-01929和Satt365两个标记之间大约300 kb范围内,并找到3个候选基因。研究共检测到33个与东北春大豆花荚脱落性状显著相关(p<0.05)的SSR标记。其中,Satt534(B2)、Satt452(E)、Satt244(J)和Satt478(O)在两年中都被检测到是较为可信的标记位点。已有文献报道称Satt534与抗大豆花叶病毒RSC4有关[27],Satt452与成熟期基因E4/e4紧密连锁[28],Satt244与抗大豆灰斑病Rcs3有关[29],Satt478与大豆叶子导水率有关[30]。其中Satt452与soybase网站上发布的大豆裂荚基因的QTL有关,也可能是影响大豆花荚脱落重要的QTL。可以对这些标记进行发掘,找到具有等位变异的东北春大豆品种,应用于大豆育种工作中。
4 结 论
研究共发现33个与大豆落花荚脱落相关的QTL,不同年际和不同种植密度间与花荚脱落性状关联的QTL不同。其中有4个QTL(Satt534、Satt452、Satt244和Satt478)在两年中都与大豆花荚脱落率相关,是较为可靠的QTL。并且已有报道称Satt452与大豆裂荚性状相关,也可能是影响大豆花荚脱落重要的QTL,可以一步分析利用。
参考文献:
[1] Wang X Q,Xu W H,Ma L G,et al.Requirement of KNAT1/BP for the development of abscission zones inArabidopsisthaliana[J].Journal of Integrative Plant Biology,2006,48(1): 15-26.
[2] Butenko M A,Patterson S E,Grini P E,et al.Inflorescence deficient in abscission controls floral organ abscission in Arabidopsis and identifies a novel family of putative ligands in plants[J].The Plant Cell,2003,15(10): 2296-2307.
[3] Xu B,Li Z Y,Zhu Y,et al.Arabidopsis GenesAS1,AS2,andJAGNegatively Regulate Boundary-Specifying Genes to Promote Sepal and Petal Development[J].Plant Physiology,2008,146(2): 566-575.
[4] Shi C L,Stenvik G,Vie A K,et al.Arabidopsis Class I KNOTTED-Like Homeobox Proteins Act Downstream in the IDA-HAE/HSL2 Floral Abscission Signaling Pathway[J].Plant Cell.2011,23(7): 2553-2567.
[5] Bonaccorso O,Lee J E,Puah L,et al.FILAMENTOUSFLOWERcontrols lateral organ development by acting as both an activator and a repressor[J].BMC Plant Biology,2012,12(1): 176.
[6] Li C B,Zhou A L,Sang T.Rice domestication by reducing shattering[J].Science,2006,311(5679): 1936-1939.
[7] Lin Z W,Griffith M E,Li X R,et al.Origin of seed shattering in rice (OryzasativaL.)[J].Planta,2007,226(1): 11-20.
[8] Konishi S,Izawa T,Lin S Y,et al.An SNP caused loss of seed shattering during rice domestication[J].Science,2006,312(5778): 1392-1396.
[9] Lin Z W,Li X R,Shannon L M,et al.Parallel domestication of the Shattering1 genes in cereals[J].Nature Genetics,2012,44(6): 720-724.
[10] 汪宝卿,慈敦伟,张礼凤,等.同化物供应和内源激素信号对大豆花荚发育的调控[J].大豆科学,2010,29(5): 878-882.
[11] Heitholt J J,Egli D B,Leggett J E,et al.Role of assimilate and carbon-14 photosynthate partitioning in soybean reproductive abortion[J].Crop Science,1986,26(5): 999-1004.
[12] Zhang D,Cheng H,Wang H,et al.Identification of genomic regions determining flower and pod numbers development in soybean (GlycinemaxL.)[J].Journal of Genetics and Genomics,2010,37(8): 545-556.
[13] Zhang D,Liu C,Cheng H,et al.Quantitative trait loci associated with soybean tolerance to low phosphorus stress based on flower and pod abscission[J].Plant Breeding,2010,129(3): 243-249.
[14] Thornsberry J M,Goodman M M,Doebley J,et al.Dwarf8 polymorphisms associate with variation in flowering time[J].Nature Genetics,2001,28(3): 286-289.
[15] 杨小红,严建兵,郑艳萍,等.植物数量性状关联分析研究进展[J].作物学报,2007,33(4): 523-530.
[16] 文自翔,赵团结,郑永战,等.中国栽培大豆和野生大豆农艺品质性状与SSR标记的关联分析Ⅰ.群体结构及关联标记[J].作物学报,2008,34(7): 1169-1178.
[17] 范 虎,文自翔,王春娥,等.中国野生大豆群体农艺加工性状与SSR关联分析和特异材料的遗传构成[J].作物学报,2013,39(5): 775-788.
[18] Fehr W R,Caviness C E,Burmood D T,et al.Stage of development descriptions for soybeans(GlycinemaxL.Merr.)[J].Crop Science,1971,11(6):929-931.
[19] Kamiya M,Kiguchi T.Rapid DNA extraction method from soybean seeds[J].Breeding Science,2003,53(3): 227-279.
[20] Evanno G,Regnaut S,Goudet J.Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J].Molecular Ecology,2005,14(8): 2611-2620.
[21] Durrant C,Zondervan K T,Cardon L R,et al.Linkage disequilibrium mapping via cladistic analysis of single-nucleotide polymorphism haplotypes[J].American Journal of Human Genetics,2004,75(1): 35-43.
[22] 金 亮,包劲松.植物性状-标记关联分析研究进展[J].分子植物育种,2009,7(6): 1048-1063.
[23] Zhu Y L,Song Q J,Hyten D L,et al.Single-nucleotide polymorphisms in soybean[J].Genetics,2003,163(3): 1123-1134.
[24] Jun T H,Van K,Kim M Y,et al.Association analysis using SSR markers to find QTL for seed protein content in soybean[J].Euphytica,2008,162(2): 179-191.
[25] Hao D R,Cheng H,Yin Z T,et al.Identification of single nucleotide polymorphisms and haplotypes associated with yield and yield components in soybean (Glycine max) landraces across multiple environments[J].Theoretical and Applied Genetics,2012,124(3): 447-458.
[26] Zhang D,Cheng H,Hu Z B,et al.Fine mapping of a major flowering time QTL on soybean chromosome 6 combine linkage and association analysis[J].Euphytica,2013,191(1): 23-33.
[27] Wang D G,Ma Y,Liu N,et al.Fine mapping and identification of the soybeanRSC4resistance candidate gene to soybean mosaic virus[J].Plant Breeding,2011,130(6): 653-65.
[28] 宁慧霞,李英慧,刘章雄,等.大豆品种成熟期基因型推测的研究[J].作物学报,2008,34(3): 382-388.
[29] Mian R,Bond J,Joobeur T,et al.Identification of soybean genotypes resistant to Cercospora sojina by field screening and molecular markers[J].Plant Disease,2009,93(4): 408-411.
[30] Carpentieri-Pipolo V,Pipolo A E,Abdel-Haleem H,et al.Identification of QTLs associated with limited leaf hydraulic conductance in soybean[J].Euphytica,2012,186(3): 679-686.
