典型产纤维小体梭菌的遗传改造及其在纤维素乙醇中的应用研究进展
2014-05-04刘亚君崔古贞
刘亚君,崔古贞,洪 伟,张 杰,崔 球
(中国科学院 青岛生物能源与过程研究所,青岛 266101)
随着经济的高速发展,能源紧缺以及环境污染已成为世界范围内日益严峻和亟待解决的问题,我国也面临着相同的挑战。木质纤维素等生物质能源原料供应丰富且环境友好,是唯一可大规模再生且全面替代石化原料的资源[1-2]。因此,大力发展纤维素基能源及化工品的生产对缓解国家能源供需矛盾、减少化石能源消耗、保护生态环境具有重要意义,是加快发展循环经济、保障国家能源安全的一项重要战略任务[3-6]。
整合生物加工(consolidated bioprocessing,CBP)技术是从纤维素原料到乙醇的一锅法生产策略,能够极大简化其生产工艺,降低成本[3]。通过CBP生产乙醇,需要生产菌株同时具备纤维素酶生产、纤维素水解和下游产物发酵等能力,以解纤维梭菌(Clostridium cellulolyticum)和热纤梭菌(Clostridium thermocellum)为代表的产纤维小体厌氧梭菌初步具备以上特征,可以直接完成从木质纤维素原料到乙醇的生物转化,因此是可用于CBP纤维素乙醇生产的优良候选菌株[7-8],在工业应用中具有巨大的潜力。
然而,野生型产纤维小体梭菌的纤维素乙醇产率普遍较低,还不能满足工业化规模生产的要求[9],需要在合成生物学及系统生物学技术的指引下进行大量的遗传工程改造。这些厌氧革兰氏阳性菌的遗传改造技术目前尚不成熟,操作复杂、局限性大,难以实现高效、快速、精准的定向代谢工程改造。基于上述问题,包括笔者所在研究团队在内的国内外研究学者系统开发并完善了基于二类内含子的嗜中温及嗜高温遗传改造平台,可以快速高效实现靶向基因敲除、敲入和可控表达,并通过开发的靶向代谢工程改造技术,系统性地应用于产纤维小体梭菌的代谢工程改造中,实现了纤维素乙醇产量的显著提高[10-11]。本文中笔者全面综述以热纤梭菌和解纤维梭菌为代表的产纤维小体梭菌遗传改造平台的建立及其在纤维素乙醇生产应用方面的研究工作,包括DNA转化技术、报告基因系统及外源蛋白表达技术、靶向基因敲除等遗传工具平台方面的进展以及靶向代谢工程改造和代谢瓶颈分析等纤维素乙醇应用方面的内容。
1 嗜中温梭菌C.cellulolyticum的研究进展
1.1 遗传操作工具的开发和改良
1.1.1 转化方法
解纤维梭菌的外源DNA导入方法在2000年就有报道,后又发展出基于电转化的DNA导入方法,其遗传转化流程主要包括细胞培养、离心和洗涤、高压脉冲电场转化、电转化后的复苏及平板计数(图1)。然而,解纤维梭菌的转化效率一直处于较低水平,约为 102个/μgDNA[12-13],难以满足基于同源重组的遗传改造的需要。为提高转化效率,笔者所在实验室研究人员对其遗传转化条件进行了系统性优化,首先对电转化脉冲的电场强度、电阻、电容、电转化缓冲液和电转杯直径等因素进行了优化,发现利用常规电转化仪,电转化条件为2 000 V、1 000 Ω、25 μF、0.4 cm 的电转杯时可以获得较好的转化效率,比前人报导提高了约3倍(3.0×102个/μg DNA)[14];通过继续利用氨苄青霉素、异烟肼、苏氨酸及甘氨酸等细胞壁弱化剂对H10的感受态细胞进行孵育处理,实现了转化效率的进一步提高。结果表明,当使用10 mg/mL的甘氨酸时,转化效率可以再提高一个数量级[10];最后,通过进一步研发新型的基因导入装置MT01-3kV,使转化效率提高到5.3 ×104个 /μg DNA[15],可以基本满足同源重组实验的要求。

图1 解纤维梭菌遗传转化流程[14]Fig.1 Schematic representation of transformation of C.cellulolyticum[14]
1.1.2 外源蛋白的表达及报告系统
解纤维梭菌中外源蛋白的表达已通过转化表达质粒得以实现[16-17],但相关研究仍较少,这可能与表达体系不稳定或表达蛋白检测手段不完善有关。绿色荧光蛋白是监测基因表达、蛋白定位、蛋白功能及相互作用的强有力工具,但其发色团的形成需要 O2的参与,并不适用于厌氧环境[18-19]。PpFbFPs来源于恶臭假单胞菌(Pseudomonas putida),是依赖于黄素单核苷酸发光的厌氧荧光蛋白,可用于建立厌氧微生物中荧光蛋白报告系统[20-21]。笔者所在实验室研究人员根据解纤维梭菌的密码子偏爱性优化设计及合成了PpFbFpm基因,将PpFbFpm基因置于丙酮丁醇梭菌硫激酶基因启动子和转录终止子之间,用Pst I和Nar I双酶切后与经同样双酶切的pIMP1质粒连接,构建了厌氧荧光蛋白表达质粒pMTC6,并通过质粒转化,实现了荧光蛋白在 H10 胞内的表达(图 3)[10,14]。
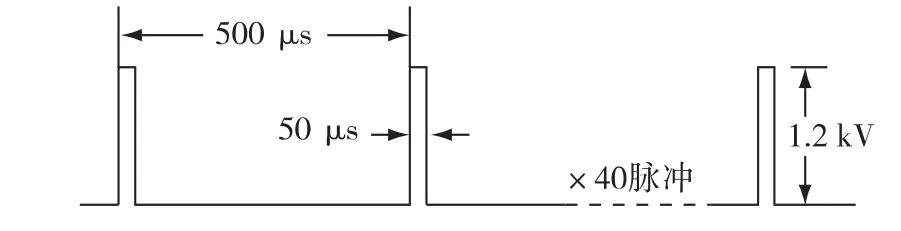
图2 MT01-3kV系统电脉冲参数[14]Fig.2 Parameters of custom MT01-3kV system[14]
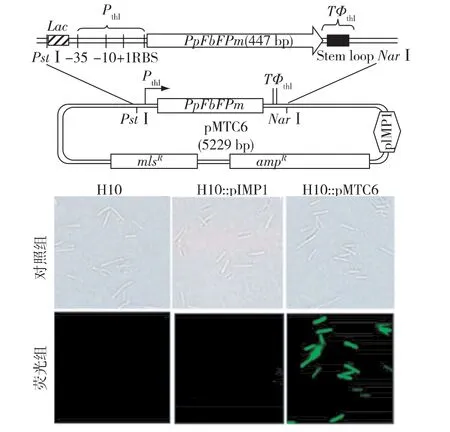
图3 PpFbFpm表达质粒pMTC6的构建及H10菌株的胞内荧光检测[14]Fig.3 Schematic diagram of plasmid pMTC6 and confirmation of PpFbFPm expression in H10 strains by in vivo fluorescent imaging[14]
1.1.3 基于ClosTron的靶向敲除方法
ClosTron是一种基于乳酸乳球菌来源的二类内含子Ll.ltrB构建的基因敲除系统。利用ClosTron方法进行基因靶向敲除主要包括targetron质粒构建、质粒转化、抗生素筛选转化子及PCR验证intron插入等步骤。与同源重组的方法相比,ClosTron方法具有高效快速的优势,并已有在线工具(http://clostron.com/)支持targetron的设计,因此在梭菌中已经被广泛应用[22-24]。已知的解纤维梭菌的靶向敲除主要也是采用这一技术[17,25-27]。然而,这种方法在解纤维梭菌中的应用还有如操作过程复杂、突变株筛选过程耗时长等不足,为此,笔者所在实验室研究人员对 ClosTron 系统进行了系统的优化升级[10,15,17]。
1.1.3.1 非甲基化质粒转化系统的构建
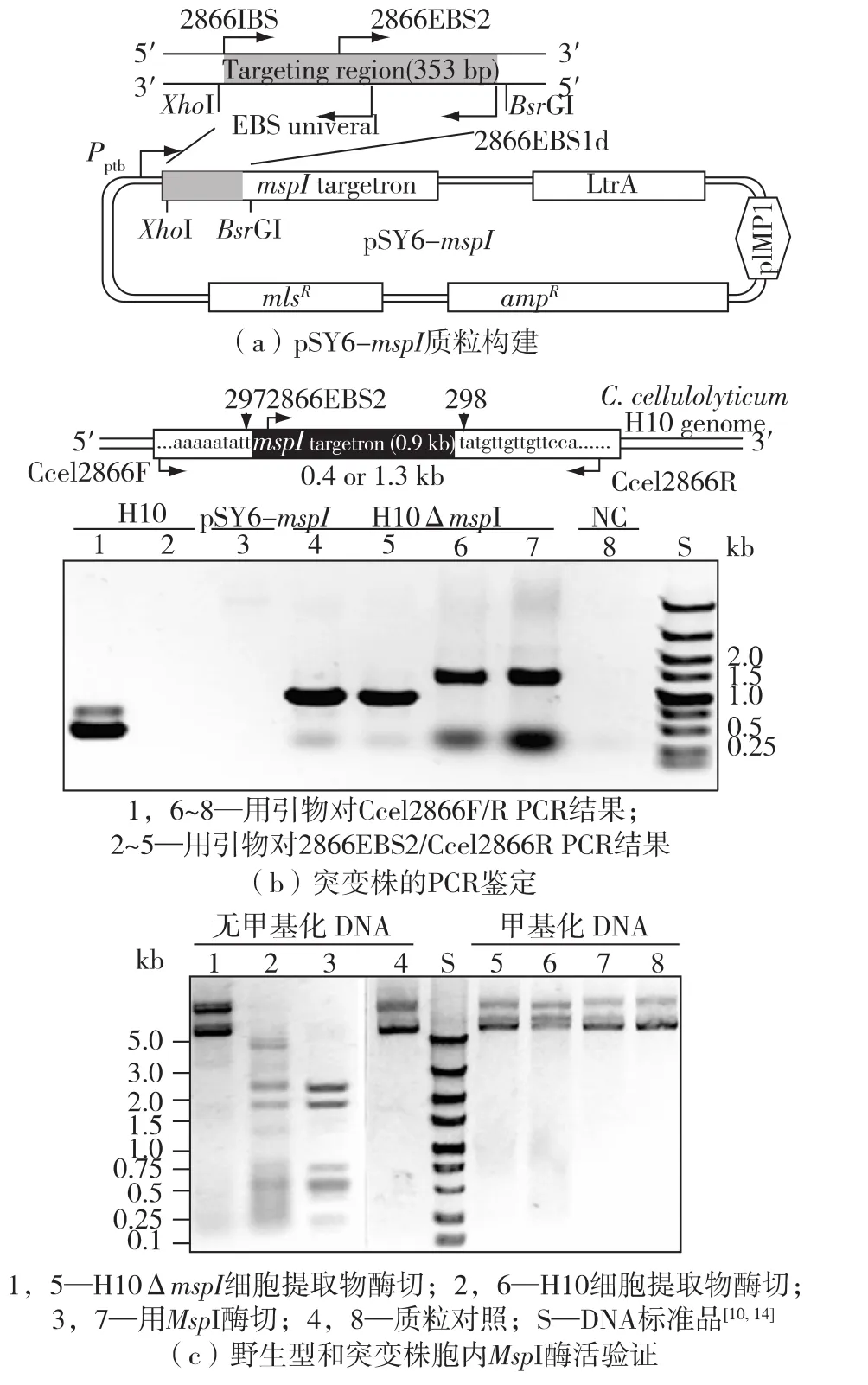
图4 基因ccel2866的靶向敲除与鉴定Fig.4 Disruption of gene ccel2866 and the confirmation
已知H10中具有限制性修饰酶MspI的表达基因ccel2866,MspI能降解外源DNA实现细菌的自我保护,因此,用于转化的外源DNA必须进行预甲基化以克服这一防御机制,从而使得遗传改造过程复杂化。为了实现不需预甲基化的遗传操作,简化操作步骤并减低成本,笔者所在实验室研究人员构建了基于pSY6的targetron质粒pSY6-mspI(图4(a))用于基因ccel2866的靶向失活。将该质粒转化到H10细胞中,通过红霉素筛选,获得阳性转化子,通过菌落PCR验证,验证targetron在靶位点的插入及ccel2866的敲除(图4(b)),随机挑取了48个菌落,均显示为阳性菌落,内含子插入的效率为100%。进一步通过MspI酶活性检测发现,突变株细胞提取物丧失了限制性内切酶活性,不能降解非甲基化的质粒(图4(c))。通过平板计数法检测甲基化和非甲基化的pSY6质粒分别转化野生型和ccel2866突变型菌株的转化效率,进一步证实,利用突变菌株能够获得非甲基化质粒的转化子,且转化效率同野生型菌株转化预甲基化质粒的效率相似。由此,获得了无需预甲基化操作的遗传操作系统,大大简化了基因工程改造的步骤[10]。
1.1.3.2 基于pyrF筛选系统的连续靶向敲除方法的构建
要实现解纤维梭菌的多基因连续操作,需要对前一步获得的工程菌株进行质粒丢失,否则无法进行后续实验。但是,目前在解纤维梭菌中能够使用的质粒的丢失效率非常低,每代的丢失率大约是4.7 ×10-3[12],因此,利用上述开发的遗传操作工具对C.cellulolyticum进行基因工程改造仍存在一个瓶颈,即如何有效地实现质粒的丢失。为解决这一问题,笔者实验室构建了基于pyrF的双向筛选系统。基因pyrF编码乳清核苷酸-5磷酸脱羧酶,是嘧啶生物合成的关键基因,同时也是抗代谢物5-氟乳清酸(5-fluoroorotic acid,5-FOA)的靶位点,可以作为有效的选择压力促进质粒的丢失[28-29]。
Cui等[15]首先通过同源重组的策略获得了H10的pyrF突变株,作为底盘细胞进行下游操作;然后将ClosTron系统中的targetron质粒系统进行改造,加入pyrF的表达框,并用其在底盘细胞中进行其他靶基因的敲除;在确定获得相应突变株后,将其接种到含有FOA的培养基中。由于质粒上含有pyrF的表达框,FOA的筛选压力就会促进质粒的丢失。最终,当突变株无法在添加有红霉素抗生素的培养基中生长时,就获得了质粒丢失的菌株,并可以采用同样的策略进行下一轮的基因操作,从而实现了基因的连续敲除。
1.2 通过基因工程改造提高乙醇产量
厌氧纤维素降解菌中,纤维素底物经过水解形成可溶性单糖或寡糖,在进入胞内经过糖酵解途径形成丙酮酸,再经过丙酮酸这一关键中间产物,进行乙醇合成途径,因此,要提高乙醇产量可以通过切断丙酮酸到乙酸、乳酸的合成途径,并且同时增强微生物催化丙酮酸合成乙醇的代谢途径,从而源于糖酵解的代谢流尽可能多的导向乙醇合成的方向,从而使得乙醇产量大幅度提高(图5)。

图5 厌氧梭菌纤维素乙醇代谢工程改造设计方案Fig.5 Metabolic engineering plan to enhance ethanol production of C.thermocellum
Guedon等[16]将来自运动发酵单胞菌的“丙酮酸-乙醛-乙醇”代谢途径引入到C.cellulolyticum中,从而使乙醇产量提高了 53%。Li等[26]实现了C.cellulolyticum中乳酸脱氢酶Ldh基因(ccel2485)和苹果酸脱氢酶基因(ccel0137)的敲除,从而切断了从丙酮酸到乳酸转化途径,使乙醇产量提高了8.5倍。然而,解纤维梭菌的乙酸产量也较高。因此要进一步提高其乙醇产量,还需要对乙酸合成途径进行阻断。
乙酸合成的最后一步则由乙酸激酶Ack(ccel2136)催化,因此,Cui等[10]同时构建了突变株H10Δldh和H10Δack,并利用已建立的遗传操作平台中的非甲基化系统,构建了突变株H10ΔmspIΔldh和H10ΔmspIΔack,分别实现 ccel2485和 ccel2136的靶向敲除,并对其生长状态和发酵产物进行了系统分析[10],发现mspI基因的敲除使得乙酸和乳酸的产量比野生型分别提高了38.2%和14.8%,但是乙醇的产量下降了36.5%,说明mspI基因的失活影响了细胞内代谢流的变化,因此,H10ΔmspI不适用于作为乙醇合成代谢工程改造的出发菌株。比较H10Δldh和H10Δack与野生菌H10发现,LDH或者ACK的敲除都可以显著提高乙醇的产量,比野生型乙醇产率分别提升了30%及50%[10]。这说明,在今后工作中应整合两种代谢工程改造策略,并结合发酵方法的优化,以实现H10的乙醇产量的进一步提高。
2 嗜高温梭菌C.thermocellum的研究进展
2.1 遗传改造平台的建立
热纤梭菌C.thermocellum等高温厌氧纤维素降解菌具有纤维素降解效率高、发酵过程污染几率小等独特的优点,是作为木质纤维素CBP糖化的优选目标微生物。然而,热纤梭菌是嗜热革兰氏阳性厌氧菌,遗传操作手段缺乏,相关技术主要掌握在多特蒙德大学Lynd实验室[30-32],这大大阻碍了热纤梭菌在纤维素乙醇中应用的研究。近年来,笔者所在实验室研究人员通过一系列的硬件和软件的开发,逐步建立及完善了针对这一嗜热微生物的遗传改造工具箱,能够实现与现有技术相比更加高通量高效的遗传操作。
2.1.1 外源DNA的导入方法
Lynd实验室在2004年就首次报道了热纤梭菌的电转化方法,然而一直难以重复[33]。为了解决这一难题,笔者在遗传转化软硬件方面进行了一系列研究,开发了新型基因导入装置MT01-3kV,并对电转化条件进行系统的优化,包括细胞生长时期、电转缓冲液组分、细胞壁弱化剂、电转化频率、脉冲数、电压、占空比等多个参数的优化,最终建立了较高效的热纤梭菌电转化方法(OD600约为0.8,1.5 kV,50 μs 1 次脉冲,10% 占空比,50 个脉冲),同时对质粒骨架进行优化,采用来源于地热芽胞杆菌的质粒复制子,最终将转化效率提高到 105个/μg DNA,可以满足后续的外源表达及靶基因敲除等遗传操作的需要。
2.1.2 外源蛋白的表达及报告系统
热纤梭菌中外源蛋白的表达鲜有报道,目前只实现了内源蛋白CipA和Bgl在热纤梭菌胞内的可检测表达[31,34]。这可能是由于厌氧微生物代谢水平低,不能支持蛋白的大量表达,或热纤梭菌中存在特殊的防御机制,阻止外源蛋白的表达。为实现热纤梭菌外源蛋白的可检测表达,并同时建立可靠的报告系统,笔者构建了厌氧荧光蛋白PpFbFpm的表达载体pHTM003,并转化至 C.thermocellum DSM1313细胞中,实现了厌氧荧光蛋白的表达(图6)。
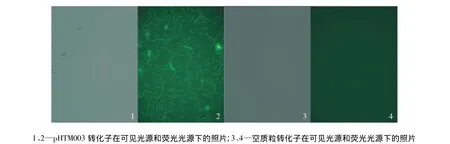
图6 PpFbFpm在热纤梭菌中的表达Fig.6 Expression of PpFbFpm in C.thermocellum
2.1.3 基因敲除方法
2.1.3.1 随机突变技术
Zverlov等[35]通过甲基磺酸乙酯对热纤梭菌野生菌株进行诱变,筛选到6株具有新型IS3家族的转座子IS1447的热纤梭菌突变株。然而这种随机的转座突变技术需要建立突变体库并针对研究目的进行筛选。这种方法通常受限于筛选手段且需要较大工作量,因此不能满足热纤梭菌高通量的系统代谢工程改造的需要。
2.1.3.2 同源重组技术
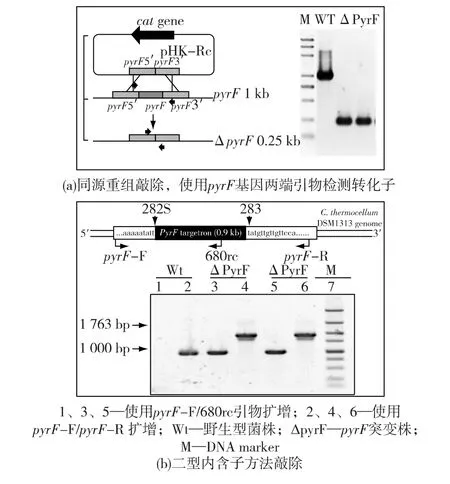
图7 C.thermocellum DSM1313中基因pyrF敲除的原理示意及PCR检测Fig.7 Schematic representation of pyrF disruption in C.thermocellum DSM1313 genomic DNA and the PCR confirmation
同源重组技术依赖于同源臂与基因组上靶基因序列的配对和交换,具有准确性高的优势,因此已经应用到热纤梭菌的靶基因敲除和敲入中(图7(a)),然而,该方法成功率较低。从转化方法建立到现在近10年的时间里,只得到了5种基因(ldh、pta、pyrF、cipA 和 cel48S)敲除的突变株[30,36-38]。此外,通过同源重组的方法对基因组靶位点的基因进行敲除或敲入需要引入抗生素抗性基因等筛选标记,且筛选标记会整合到基因组,不能循环使用,这严重限制了遗传改造的深度。Heap等[39]改进了同源重组方法,通过引入反向筛选标记实现了对筛选标记进行循环使用,但操作步骤仍较复杂。
2.1.3.3 基于嗜热targetron基因打靶技术
为了实现热纤梭菌高效快速的靶向基因敲除,笔者所在实验室与德克萨斯大学奥斯汀分校Lambowitz实验室合作,开发了基于耐热的二类内含子系统的嗜热 微 生 物 定 向 改 造 技 术 Thermotargetron[11]。Thermotargetron技术是源于1株嗜热蓝细菌Thermosynechococcus elongatus的二类内含子(图8(a))构建的高效、便捷的多基因靶向敲除系统。该耐热内含子在热纤梭菌基因组中具有较高的插入效率(67%~100%),这可能是由于高温条件促使了DNA的解链,使得内含子RNA更易识别DNA靶位点,提高了靶向敲除的效率[11]。利用该技术,笔者成功实现了pyrF的intron插入失活,从而构建了相应的突变菌株(图8(b))。研究人员利用该技术进一步对热纤梭菌纤维小体的重要脚架蛋白进行失活,共获得了8个突变株,用于分析不同脚架及脚架上的模块在纤维素降解过程中的作用。

图8 T.elongatus中Tel3c二类内含子及反转录酶Fig.8 T.elongatus TeI3c group II intron RNA and TeI4c RT components of the thermotargetron
2.2 乙醇合成代谢工程改造
Argros等[36]利用同源重组的方法将热纤梭菌中的乳酸脱氢酶和磷酸乙酰转移酶进行敲除(图5),获得了双突变株ΔldhΔpta,并通过与非纤维素降解菌的混养发酵得到38 g/L的乙醇产量,这是目前利用热纤梭菌所能获得的最高乙醇产量。笔者所在实验室研究人员利用Thermotargetron的方法,也获得了2株单突变株(Δldh,Δpta)和1株双突变株(ΔldhΔpta),并对其进行发酵分析,发现Δldh(乳酸脱氢酶突变株),Δpta(磷酸乙酰转移酶突变株)及双突变株ΔldhΔpta(乳酸脱氢酶与磷酸乙酰转移酶双突变株)的乙醇产量分别比野生型(WT)增加了37%、42%和56%[11]。此外,利用核磁共振的代谢物组学分析方法发现Δpta与ΔldhΔpta菌株中丙酮酸极其显著地积累,这一结果与文献中的报道一致[36]。这一现象说明丙酮酸到乙醇的代谢是整个乙醇生产的瓶颈。因此,要进一步提高热纤梭菌的乙醇产量,就需要对这一代谢途径进行进一步的改造和强化。
3 结论
以解纤维梭菌和热纤梭菌为代表的中温或高温产纤维小体微生物能够实现木质纤维素底物到生物乙醇的直接转化,在木质纤维素CBP应用中具有巨大的潜力。目前所开发的针对中温及高温梭菌的遗传改造平台使得对这些候选菌株进行系统的代谢工程改造成为可能,从而有望克服其现有的抗逆性低、碳谱窄或乙醇产量低等缺陷,以适应工业化生产的需要。因此,开发可靠的遗传改造平台具有广泛的应用价值,将有力地推动产纤维小体梭菌在木质纤维素生物转化的工业生产中的应用。
[1] Lynd L R.Overreview and evaluation of fuel ethanol from cellulosic biomass:technology,economics,the environment,and policy[J].Ann Rev Energy Environ,1996,21:403-465.
[2] 曹俐,吴方卫.液态生物质燃料发展对生态环境的影响研究进展与展望[J].国土与自然资源研究,2012(4):52-55.
[3] Lynd L R,Van Zyl W H,Mcbride J E,et al.Consolidated bioprocessing of cellulosic biomass:an update[J].Curr Opin Biotechnol,2005,16(5):577-583.
[4] Petiot E.On the road to cost-competitive cellulosic ethanol[J].Chimica Oggi-Chemistry Today,2008,26(1):20-22.
[5] 吴镇江.国内外纤维素乙醇技术进展研究[J].现代商贸工业,2011(8):269-270.
[6] 曲音波.纤维素乙醇产业化[J].化学进展,2007,19(7/8):1098-1108.
[7] Demain A L,Newcomb M,Wu J H.Cellulase,Clostridia,and ethanol[J].Microbiol Mol Biol Rev,2005,69(1):124-154.
[8] Lynd L R,Laser M S,Bransby D,et al.How biotech can transform biofuels[J].Nat Biotechnol,2008,26(2):169-172.
[9] Papoutsakis E T.Engineering solventogenic Clostridia[J].Curr Opin Biotechnol,2008,19(5):420-429.
[10] Cui G Z,Hong W,Zhang J,et al.Targeted gene engineering in Clostridium cellulolyticum H10 withoutmethylation[J].J Microbiol Methods,2012,89(3):201-208.
[11] Mohr G,Hong W,Zhang J,et al.A Targetron system for gene targeting in thermophiles and its application in Clostridium thermocellum[J].PLoS One,2013,8(7):e69032.
[12] Jennert K C,Tardif C,Young D I,et al.Gene transfer to Clostridium cellulolyticum ATCC 35319[J].Microbiology,2000,146:3071-3080.
[13] Tardif C,Maamar H,Balfin M,et al.Electrotransformation studies in Clostridium cellulolyticum[J].J Ind Microbiol Biotechnol,2001,27(5):271-274.
[14] 崔古贞.解纤维梭菌遗传体系的建立及其纤维素乙醇代谢工程研究[D].青岛:中国科学院青岛生物能源与过程研究所,2013.
[15] Cui G Z,Zhang J,Hong W,et al.Improvement of ClosTron for successive gene disruption in Clostridium cellulolyticum using a pyrF-based screening system[J].Appl Microbiol Biotechnol,2013,doi:10.1007/s00253-013-5330-y.
[16] Guedon E,Desvaux M,Petitdemange H.Improvementof cellulolytic properties of Clostridium cellulolyticum by metabolic engineering[J].Appl Environ Microbiol,2002,68(1):53-58.
[17] Celik H,Blouzard J C,Voigt B,et al.A two-component system(XydS/R)controls the expression of genes encoding CBM6-containing proteins in response to straw in Clostridium cellulolyticum[J].PLoS One,2013,8(2):e56063.
[18] Tsien R Y.The green fluorescent protein[J].Ann Rev Biochem,1998,67:509-544.
[19] Chudakov D M,Lukyanov S,Lukyanov K A.Fluorescent proteins as a toolkit for in vivo imaging[J].Trends Biotechnol,2005,23(12):605-613.
[20] Drepper T,Eggert T,Circolone F,et al.Reporter proteins for in vivo fluorescence without oxygen[J].Nat Biotechnol,2007,25(4):443-445.
[21] Drepper T,Gensch T,Pohl M.Advanced in vivo applications of blue light photoreceptors as alternative fluorescent proteins[J].Photochem Photobiol Sci,2013,12(7):1125-1134.
[22] Heap J T,Pennington O J,Cartman S T,et al.The ClosTron:a universal gene knock-out system for the genus Clostridium[J].J Microbiol Methods,2007,70(3):452-464.
[23] Heap J T,Cartman S T,Kuehne S A,et al.ClosTron-targeted mutagenesis[J].Methods Mol Biol,2010,646:165-182.
[24] Kuehne S A,Heap J T,Cooksley C M,et al.ClosTron-mediated engineering of Clostridium[J].Methods Mol Biol,2011,765:389-407.
[25] Chaves-Olarte E,Ferdinand P H,Borne R,et al.Are cellulosome scaffolding protein CipC and CBM3-containing protein HycP,involved in adherence of Clostridium cellulolyticum to cellulose?[J].PLoS One,2013,8(7):e69360.
[26] Li Y,Tschaplinski T J,Engle N L,et al.Combined inactivation of the Clostridium cellulolyticum lactate and malate dehydrogenase genes substantially increases ethanol yield from cellulose and switchgrass fermentations[J].Biotechnol Biofuels,2012,doi:10.1186/1754-6834-5-2.
[27] Fendri I,Abdou L,Trotter V,et al.Regulation of cel genes of C.cellulolyticum:identification of GlyR2,a transcriptional regulator regulating cel5D gene expression[J].PLoS One,2013,8(1):e44708.
[28] Boeke J D,Lacroute F,Fink G R.A positive selection for mutants lacking orotidine-5'-phosphate decarboxylase activity in yeast:5-fluoro-orotic acid resistance[J].Mol Gen Genet,1984,197(2):345-346.
[29] Suzuki H,Murakami A,Yoshida K.Counterselection system for Geobacillus kaustophilus HTA426 through disruption of pyrF and pyrR[J].Appl Environ Microbiol,2012,78(20):7376-7383.
[30] Waller B H,Olson D G,Currie D H,et al.Exchange of type II dockerin-containing subunits of the Clostridium thermocellum cellulosome as revealed by SNAP-tags[J].FEMS Microbiol Lett,2013,338(1):46-53.
[31] Olson D G,Giannone R J,Hettich R L,et al.Role of the CipA scaffoldin protein in cellulose solubilization,as determined by targeted genedeletion and complementation in Clostridium thermocellum[J].J Bacteriol,2013,195(4):733-739.
[32] Olson D G,Lynd L R.Transformation of Clostridium thermocellum by electroporation[J].Methods Enzymol,2012,510:317-330.
[33] Tyurin M V,Desai S G,Lynd L R.Electrotransformation of Clostridium thermocellum[J].Appl Environ Microbiol,2004,70(2):883-890.
[34] Maki M L,Armstrong L,Leung K T,et al.Increased expression of beta-glucosidase A in Clostridium thermocellum 27405 significantly increases cellulase activity[J].Bioengineered,2013,4(1):15-20.
[35] Zverlov V V,Klupp M,Krauss J,et al.Mutations in the scaffoldin gene,cipA,of Clostridium thermocellum with impaired cellulosome formation and cellulose hydrolysis:insertions of a new transposable element,IS1447,and implications for cellulase synergism on crystalline cellulose[J].J Bacteriol,2008,190(12):4321-4327.
[36] Argyros D A,Tripathi S A,Barrett T F,et al.High ethanol titers from cellulose usingmetabolicallyengineered thermophilic,anaerobic microbes[J].Appl Environ Microbiol,2011,77(23):8288-8294.
[37] Tripathi S A,Olson D G,Argyros D A,et al.Development of pyrF-based genetic system for targeted gene deletion in Clostridium thermocellum and creation of a pta mutant[J].Appl Environ Microbiol,2010,76(19):6591-6599.
[38] Olson D G,Tripathi S A,Giannone R J,et al.Deletion of the Cel48S cellulase from Clostridium thermocellum[J].PNAS,2010,107(41):17727-17732.
[39] Heap J T,Ehsaan M,Cooksley C M,et al.Integration of DNA into bacterial chromosomes from plasmids without a counterselection marker[J].Nucleic Acids Res,2012,40(8):e59.
