酿酒酵母人造纤维小体的研究进展
2014-05-04汤红婷鲍晓明
汤红婷,侯 进,沈 煜,鲍晓明
(山东大学 微生物技术国家重点实验室,济南 250100)
木质纤维素是地球上含量最为丰富的可再生资源之一[1]。高效、廉价地把木质纤维素资源转化为人类所需要的能源和化工原料,对于缓解目前存在的能源、资源等危机具有重要意义。燃料乙醇是在生物质能源中最先商业化的产品之一,按一定比例将其掺入汽油中,可以部分替代石油资源,降低油耗量,还可以促进汽油的充分燃烧,减少CO2的排放。与第一代以粮食类淀粉为原料生产的燃料乙醇相比,第二代燃料乙醇的生产是利用统合生物加工过程(consolidated bioprocessing,CBP)将纤维素酶和半纤维素酶产生、纤维素水解和乙醇发酵组合在一起,以木质纤维素为原料来高效生产燃料乙醇,这是实现木质纤维素转化生物乙醇的有效途径之一。CBP有利于降低生物转化过程的成本,简化生产工艺,因此受到研究者的普遍关注[2-4]。酿酒酵母(Saccharomyces cerevisiae)作为传统的乙醇生产菌株及重要的真核模式生物,是实现统合生物加工过程(CBP)极具潜力的底盘细胞。它的基因组简单,遗传操作体系完善,可根据细胞功能实现多种方面的设计和改造。同时,酿酒酵母也具有生长速率快、对抑制物耐受性强、食品级安全等优良生产特征[5-6]。
木质纤维素的高效降解是实现生物转化的关键点,降解木质纤维素的最基本酶有3种:断裂纤维素内部糖苷键的内切葡聚糖酶(endoglucanases,EG)、从一端水解产生纤维二糖的纤维二糖水解酶(cellobiohydrolases,CBH)和催化纤维二糖为葡萄糖的 β-葡萄糖苷酶(β-glucosidases,BGL)。在自然界中,能够生产纤维素酶的真菌和细菌是降解木质纤维素的主要力量。其中,某些厌氧菌利用细胞表面形成的纤维小体(cellulosome)实现纤维素的高效降解。纤维小体是由纤维素酶组装到细胞表面的支架蛋白(scaffoldin)上而形成的大分子纤维素酶复合物,支架蛋白的柔韧性保证了它与纤维素底物充分而紧密的结合,并且多种纤维素酶组分之间的协同作用和邻近效应增强了各种纤维素酶的水解功能,从而赋予了纤维小体高效水解纤维素的功能。另外,多个支架蛋白的嵌合和多样化的纤维素酶构成的纤维小体结构,这种复杂性也与其高效降解效率正相关[7]。与具有较高滤纸酶活的瑞氏木霉纤维素酶系相比,纤维小体对结晶纤维素(如微晶纤维素、棉花等)具有更高效的降解能力,纤维小体的比酶活大约是瑞氏木霉纤维素酶系的50倍[8-9]。目前,纤维小体被认为是所有微生物纤维素酶系中最为有效的纤维素降解系。因此,将纤维小体高效降解纤维素过程与酿酒酵母生产乙醇的特性有机地结合起来,是实现CBP途径构建的有效方式之一。近年来,纤维小体在酿酒酵母细胞表面的展示已成为CBP酵母新的研究热点。笔者主要综述纤维小体在酿酒酵母表面展示的研究进展,其中重点阐述纤维小体各元件的设计和改造,并针对酿酒酵母分泌途径的改造提出提高人造纤维小体分泌组装的可能性策略。
1 纤维小体的结构和功能
厌氧菌的纤维小体结构复杂(图1),具有催化活性结构域的纤维素酶系各组分上的对接模块(dockerins),分别与初级支架蛋白上串联的粘连模块(cohensins)相互作用,形成纤维小体基本结构单元。这种基本结构单元通过粘连模块-对接模块的相互作用固定在锚定支架蛋白上,再由锚定支架蛋白自身表层同源模块(S-layer homology module,SLH)或者分选酶基序(sortase)结合至细胞表面组装成纤维小体。
与游离的纤维素酶不同,纤维小体的酶组分都含有对接模块结构域。对接模块由2条约含有22个氨基酸残基的重复序列通过一个连接片段(linker)连接组成[10-11]。目前,根据系统发生关系分类,对接模块也至少包括Ⅰ、Ⅱ和Ⅲ型这3种类型[12]。纤维小体的催化模块种类极其繁多,如热线梭菌纤维小体组分中包含超过30种内切葡聚糖酶(endoglucanase),如 Cel8A、Cel9R、Cel5G 和 Cel9D等;4种外切葡聚糖酶(exoglucanase),如 Cel48S、CelO和 CbhA 等;2种 β-葡 萄 糖 苷 酶 (βglucosidase),如BglA;6种木聚糖酶(xylosidase)及几丁质酶(lichenases)、甘露糖苷酶(mannosidase)和果胶酶(pectate lyase)等[9]。
粘连模块串联而成的支架蛋白是纤维小体组装过程中关键的非催化亚基。根据粘连模块-对接模块间的作用特性,对接模块的系统发生图谱在很大程度上反映了粘连模块的对应图谱,所以粘连模块至少也包括Ⅰ、Ⅱ、Ⅲ这3种类型。某些嗜温梭菌仅含有6~9个Ⅰ型粘连模块的初级支架蛋白(图1(a)),如解纤维梭菌(Clostridium cellulolyticum)、嗜纤维梭菌(Clostridium cellulovorans)、约氏梭菌(Clostridium josui)和丙酮丁醇梭菌(Clostridium acetobutylicum)等[13-16],与纤维素酶组装成相对简单的纤维小体。而另一些细菌则含有多种类型的支架蛋白,复杂程度高(图1(b)、1(c))。如热纤梭菌(Clostridium thermocellum)纤维小体具有Ⅰ和Ⅱ型2类支架蛋白,Ⅰ型支架蛋白有只含有1个Ⅰ型粘连模块的OlpA和含有9个同源性极高的Ⅰ型粘连模块的CipA,其中两两之间的最高相似度可达到100%[17]。Ⅱ型支架蛋白含有1、2、4个Ⅱ型粘连模块的SdbA、Orf2和OlpB这3种锚定支架蛋白[9]。大部分的初级支架蛋白都含有与底物结合的碳水化合物结合结构域(carbohydrate-bindinging domain,CBD)。根据文献报道,支架蛋白上存在的CBD能够促进热纤梭菌CelS、CelD对微晶纤维素的降解能力[18-19]。Ⅰ和Ⅱ型支架蛋白的嵌合组装,不仅增加了纤维小体结构的复杂性,也增加了纤维小体中酶组分的种类和数量。黄色瘤胃球菌(Ruminococcus lf avefaciens)、解纤维醋弧菌(Acetivibrio cellulolyticus)的纤维小体结构更加复杂[20-21],如黄色瘤胃球菌17具有4种类型的支架蛋白ScaA、ScaB、ScaC和ScaE。此外,黄色瘤胃球菌17还具有2个可能结合底物结晶纤维素的蛋白CttA,该蛋白质的对接模块能与ScaE的粘连模块相互作用[22]。这样高度集成的超分子纤维小体,可以很好地组织、协调多种酶组分发挥协同作用,从而达到木质纤维素的高效降解。
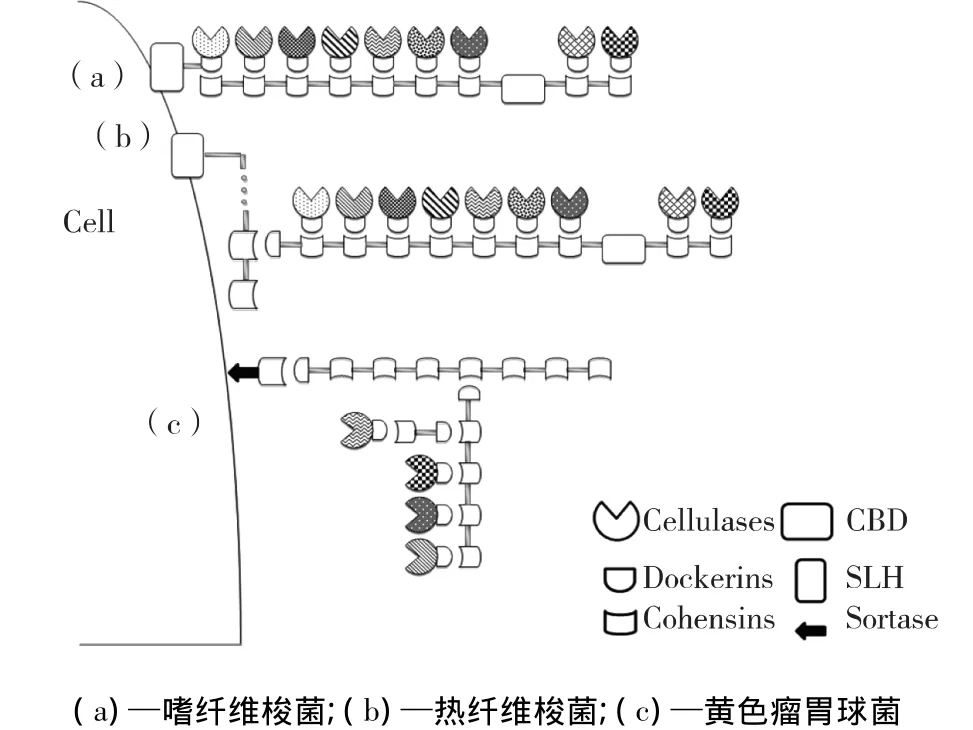
图1 厌氧细菌纤维小体基本结构示意Fig.1 Basic structure of cellulosome in anaerobic bacteria
天然纤维小体自组装是由细胞表面支架蛋白上的粘连模块与分泌到胞外的纤维素酶上的对接模块间通过疏水作用,并辅以较少的分子间氢键在胞外结合形成纤维小体结构的过程。同种类型的对接模块-粘连模块之间的相互作用具有非特异性,因此,不同纤维小体酶组分能与支架蛋白上的粘连模块随机结合,使形成的纤维小体的结构复杂多样化。同时,酶与酶之间的协同作用和邻近作用使纤维小体对微晶纤维素底物具有很高的降解活性[9]。不同来源厌氧微生物的同类型粘连模块序列相差很大,具有种间专一性;而不同类型的粘连模块间的序列同源性更低。根据种间专一性,不同来源的对接模块-粘连模块的杂合使用,纤维素酶的定位组装,使人工设计获得特定纤维小体成为可能。
2 酿酒酵母人造纤维小体的表面展示
人造纤维小体是根据天然纤维小体的结构,经过人工设计在异源细胞表面展示出的微型结构(图1)。展示于细胞表面至少有2个粘连模块的微型支架蛋白,与分泌表达带有对接模块的纤维素酶组分进行胞外自组装,形成细胞表面的微型复合物,即为人造纤维小体,也称为纤维小体嵌合体(cellulosome chimeras)[23]。随着对纤维小体认识的不断深入,纤维小体在木质纤维素资源转化中的巨大价值也越来越受人们的关注,更为细致复杂的人造纤维小体逐渐被设计研究,以期拓展其应用价值与研究领域。
2.1 纤维素酶的来源
纤维素酶在酿酒酵母细胞中表达构建人造纤维小体时,其来源并不一定局限于天然纤维小体本身的纤维素酶,真菌来源的纤维素酶与特异的对接模块融合,也可以实现纤维小体的组装。最初,分别通过厌氧细菌的部分支架蛋白,将1~2个纤维素酶在酿酒酵母细胞表面组装,构建了简单的人造纤维小体,进而根据纤维素酶系的复杂性,逐步展开多功能纤维小体在酿酒酵母表面展示的研究。
目前,大部分研究者都选择了全部或部分使用天然纤维小体纤维素酶[24-27],根据酶与酶之间的协同作用来优化纤维小体。Tsai等[24]比较了热纤梭菌的内切葡聚糖酶CelA、解纤维梭菌的外切葡聚糖酶CelE以及解纤维梭菌内切葡聚糖酶CelG组装单功能纤维小体,发现纤维素降解效率最高的是CelA,最低的是CelG,说明纤维小体酶降解纤维素能力具有一定的差异。同时,三功能纤维小体(CelA、CelE、CelG)酶之间的协同作用是游离酶的2.44倍,是两功能(CelA、CelE)的 1.65 倍。因此,增加酶组分的种类是优化纤维小体性能的有效途径。但是该纤维小体的降解产物积累了较高的寡聚糖,以热纤梭菌的β-葡萄糖苷酶BglA代替CelG时,促进了葡萄糖的产生,这说明了外切葡聚糖酶、内切葡聚糖酶和β-葡萄糖苷酶是纤维素彻底水解的3种基本酶。在基本酶的基础上,进一步增加酶组分种类,可能会在很大程度上提高纤维小体的降解能力。
除此之外,来源于真菌的游离纤维素酶融合相应的对接模块后,也能参与纤维小体的构建。Wen等[28]则将来源于瑞氏木霉(Trichoderma reesei)的内切葡聚糖酶EGⅡ和纤维二糖水解酶CBHⅡ以及棘孢曲霉(Aspergillus aculeatus)的β-葡萄糖苷酶BGL1与热纤维梭菌的外切葡聚糖酶CelS和内切葡聚糖酶CelA的对接模块融合后,通过EGⅡ和CBHⅡ对磷酸膨胀纤维素(PASC)降解产糖及BGL1水解纤维二糖测定,3种酶都能组装到支架蛋白上。真菌源的纤维素酶经过人为设计后,也可参与人造纤维小体的组装,这就大大丰富了纤维素酶的选择。游离酶一般都具有CBD结构域,不同的CBD对微晶纤维素的结合位点也不相同[29]。因此,游离酶的使用也能增加纤维小体对底物的结合能力。厌氧细菌本身也分泌一些游离酶,但是目前还没有研究使用此类酶进行纤维小体的组装。
2.2 人造纤维小体的自组装与支架蛋白的设计
纤维小体中的酶组分分泌到胞外后,与展示于细胞表面的支架蛋白匹配结合是人造纤维小体的自组装过程,2种组分在酿酒酵母表面形成这种匹配结合的来源方式有2种。一是纤维小体各元件在不同的细胞中甚至在不同物种的细胞中分别表达,再通过人工配比混合使纤维素酶结合到酿酒酵母细胞表面的支架蛋白上,形成纤维小体[24-25,30]。如Tsai等[24]将在大肠杆菌表达含对接模块的纤维素酶与表面展示有支架蛋白的酿酒酵母混合于缓冲液(50 mmol/L Tris-HCl pH 8.0、100 mmol/L NaCl、10 mmol/L CaCl2)中,4℃孵育1 h,使纤维素酶充分结合到支架蛋白上,结果显示:该酿酒酵母与单一纤维素酶组分混合,纤维素酶能够正确组合到支架蛋白上,且能不同程度地降解磷酸膨胀纤维素(PASC)。但该酿酒酵母与多个纤维素酶组分混合,各酶组分是否参与形成纤维小体并没有被证实。另一种则是与天然纤维小体的形成方式类似,将纤维小体各单元在同一个细胞表达[26-28],然后在胞外实现自组装。如Hyeon等[26]在酿酒酵母中共表达了支架蛋白和2种内切葡聚糖酶,非变性PAGE电泳和内切葡聚糖酶的羧甲基纤维素(CMC)酶谱分析结果表明:酿酒酵母表面形成了微型纤维小体,同时补加β-葡萄糖酶共同作用于非晶态纤维素CMC,乙醇产量在16 h能达到3.5 g/L。这种方式的优点是简化了纤维素酶的分离纯化和控制细胞培养比例的过程,且单个细胞的多功能构建也是CBP菌株的最终目的。
粘连模块与对接模块间的相互作用是自组装形成人造纤维小体的前提[31-32]。支架蛋白的人工设计是人造纤维小体复杂性的因素之一,目前人工设计支架蛋白的基本策略有2种:其一,串联同一来源的粘连模块形成纯合支架蛋白,不同纤维素酶组分则融合同样来源的对接模块,使纤维素酶随机在支架蛋白上自组装,可形成多样性的纤维小体;其二,串联不同来源的粘连模块组形成杂合支架,而不同纤维素酶组分也融合上对应来源的对接模块,根据对接模块与粘连模块的专一性结合特点,使纤维素酶定量定位地组装到纤维小体上。
直接选择同一来源同型粘连模块组成的支架蛋白,人造纤维小体能像天然纤维小体组装过程一样,纤维素酶的结合具有随机性。Wen等[28]将热纤梭菌带有CBD区域的前3个I型粘连模块作为支架蛋白,再把EG II、CBH II及BGL1与热纤梭菌纤维小体酶组分的对接模块融合,在酿酒酵母中共表达,赋予了酿酒酵母降解PASC的功能,但乙醇产量也只达到1.87 g/L。其中,三功能纤维小体构建过程中,3种纤维素酶的结合率都下降了,尤其是CBH II,荧光单位下降了160倍。原因可能是由于4种组分共表达给细胞带来了一定的代谢负担,导致支架蛋白的展示量和纤维素酶的分泌量下降;或者由于粘连模块-对接模块相互之间的亲和性不同,导致具有较弱亲和性的对接模块的纤维素酶处于组装弱势,形成纤维素酶种类不完全的纤维小体。人造纤维小体表达的纤维素酶种类单一,即使使用同一来源的同型支架蛋白,目前也不能实现人造纤维小体的多样化。
使用不同来源纤维小体的粘连模块串联的杂合支架蛋白,能使纤维小体酶的组装仅依赖粘连模块-对接模块作用特异性,不需要考虑亲和性的强弱。目前,多数人造纤维小体的设计,主要是依赖于粘连模块-对接模块间的相互作用。Tsai等[24]将来源于热纤梭菌、解纤维梭菌和黄色瘤胃球菌的粘连模块串联,各个纤维素酶则融合上相对应来源的对接模块。在酿酒酵母中表达这个杂合支架蛋白,纤维素酶能特异性地结合于支架蛋白上,如热纤梭菌纤维小体酶组分只能与热纤梭菌源的粘连模块组装,而不能与其他2种来源的粘连模块结合。重组酿酒酵母降解PASC可产生3.5 g/L的乙醇。杂合支架蛋白本身能控制纤维素酶的比例,而组装过程中纤维素酶的添加比例并没有得到控制,可能会导致某些纤维素酶的过剩。因此,Tsai等[25]在酿酒酵母中分别单独表达了纤维小体组成单元,通过对发挥不同功能的酿酒酵母共培养,并对共培养的细胞比例进行了优化,结果表明:当骨架蛋白、EG、CBH、BGL 的比例为7∶2∶4∶2时,发酵 PASC 生产乙醇的得率比等比例共培养时提高了2倍。除通过粘连模块与对接模块之间的相互作用外,Ito等[33]利用抗原抗体反应的原理,将金黄色葡萄球菌(Staphylococcus aureus)的表面抗原A的结合结构域Z区作为粘连模块与EG融合的免疫球蛋白A的Fc区对接,同时,噬纤维梭菌的粘连模块则与BGL融合的对接模块相互作用,将酶特异地组装在酿酒酵母表面。虽然,杂合支架蛋白的设计优化了纤维小体本身的纤维素酶组装比例,但纤维小体各组分在酿酒酵母中表达时,由于其天然结构发生了改变或其分泌量低,导致了人造纤维小体在酿酒酵母表面的组装效率很低。
2.3 人造纤维小体复杂化的设计
虽然人造纤维小体已经在酿酒酵母表面成功地自组装,但是其降解纤维素的能力仍然很低。与天然纤维小体相比,人造纤维小体的结构简单、酶组分单一,而纤维小体的复杂性是其降解高效性的重要影响因素之一。要实现高效CBP酵母的构建,还需要对酿酒酵母纤维小体进行进一步的复杂化和优化。首先,复杂化纤维素酶是较为直接简单的方式,可以通过在酿酒酵母中高效表达纤维素酶,优化酶与酶之间的协同作用来实现。Tsai等[24]比较热纤梭菌的内切葡聚糖酶CelA和解纤维梭菌的内切葡聚糖酶CelG发现,CelA水解CMC的能力高于CelG。Ilmén等[6]则在酿酒酵母中表达了14个来源的 CBH I与11个 CBH II,发现不同来源的CBH在酿酒酵母中表达活性相差很大,其中埃默森篮状菌(Talaromyces emersonii)CBHⅠ具有最高酶活,在其C端加上碳水化合物结合模块后,促进了其对微晶纤维素(Avicel)的水解能力以及其与具有高活性的Chrysosporium lucknowense的CBH II协同作用后,大幅度地增加了Avicel的降解作用。虽然,纤维素酶的来源和酶与酶之间的协同作用已经开始被研究,但是目前纤维小体的酶组分表达种类却不超过3种。另外,增加纤维小体支架蛋白的复杂度也是一种有效的方式,可以通过增加支架蛋白的长度和支架蛋白之间的嵌合使用来实现。Fan等[27]模拟热纤梭菌天然纤维小体的结构(图1(b)),构建了初级支架蛋白和锚定支架蛋白的两层支架蛋白。锚定支架蛋白(含有至少2个Ⅱ粘连模块结构域)由细胞壁蛋白展示于酵母细胞表面,分泌到胞外的初级支架蛋白通过其C-端的Ⅱ型对接模块对接到锚定支架蛋白的Ⅱ型粘连模块上,纤维素酶则由Ⅰ型对接模块-粘连模块组装到初级支架上。初级支架蛋白和锚定支架蛋白嵌合使用,不仅复杂化了纤维小体的结构,还增加了每个纤维小体中纤维素酶的数量。但重组菌株虽然能利用Avicel,但也只能产生1.4 g/L的乙醇。
2.4 酿酒酵母展示系统及分泌途径的优化
纤维素酶和支架蛋白的高效表达是实现人造纤维小体高效表达最基本的要求。影响异源蛋白分泌量的因素包括靶蛋白的特性、宿主细胞及其培养条件、表达载体、启动子选择、密码子偏好性、信号肽以及蛋白的加工分泌途径[34]。其中,蛋白的加工分泌途径是极其复杂且重要的影响因素。在胞质中翻译完成的多肽进入内质网(ER),经过ER中的分子伴侣、氧化还原酶和糖基化酶等折叠修饰后通过膜泡运输至高尔基体,而错误折叠的蛋白则被识别,或重新进行折叠或运回细胞质并通过内质网介导的降解体系(ER associated degradation,ERAD)降解。运至高尔基体(Golgi)中的蛋白进一步加工修饰形成成熟蛋白再由膜泡运至胞外。当错误折叠蛋白大量积累时就会激活非折叠蛋白响应(unfolded protein response,UPR),通过调控一系列分泌途径相关基因(包括分子伴侣、蛋白分泌、ERAD等基因)的表达水平,促进折叠错误的蛋白正确折叠或降解,减少细胞凋亡,促进蛋白分泌[35]。酿酒酵母表达不同来源的纤维素酶所引起UPR程度各不相同,且共表达多种蛋白将进一步加剧UPR[6],说明大量异源蛋白的表达可能导致参与酵母分泌途径中的重要因素如分子伴侣、膜泡运输、糖基化的蛋白因子等的不足。
在酿酒酵母中,由于蛋白折叠在ER中受多种基因的协同调控,往往是异源蛋白高效分泌表达的主要限制因素,因此,超表达一些分子伴侣、协助蛋白以及二硫键合成的相关酶类可能是一种提高异源蛋白分泌的简单有效策略[36]。如超表达分子伴侣Hsp70家族的ATPase Kar2p以及它的辅助蛋白(如Sis1p/Sil1p以及Lhs1p)可提高多种异源蛋白的分泌活性[37-38];超表达二硫键异构酶PDI使裂殖酵母的酸性磷酸酶和人类血小板源生长因子B分别提高了4倍和10倍,且使瑞氏木霉外切葡聚糖酶CBH I在酿酒酵母中分泌酶活提高了17.2%[4,39];UPR 激活途径中的转录因子 Hac1p超表达后,使酵母自身分泌蛋白蔗糖酶的表达量提高了2倍,异源 α-淀粉酶表达量提高了 2.4倍[40]。一般地,异源蛋白在酿酒酵母中表达都会因为过度糖基化,导致分泌酶活的降低。Suzuki等[41]敲除酿酒酵母分泌途径中多种糖基化修饰相关基因后,不同程度地提高了纤维素酶与支架蛋白的组装效率。然而,一些蛋白在ER中正确折叠了,却仍然有部分滞留在胞内,说明ER-Golgi或Golgi后期蛋白运输中的一些相关基因是限制性因素。超表达参与Golgi形成的囊泡与质膜融合的突出融合蛋白Sso1/2使蔗糖酶和淀粉酶的表达分别提高了6倍和3倍[42]。虽然对酿酒酵母的分泌途径进行改造能在一定程度上提高蛋白的分泌量,但分泌途径是一个完整的通路,如果要更高效地提高异源蛋白的分泌,就需要提高分泌途径的整体效率。
目前,酿酒酵母人造纤维小体的支架蛋白一般由凝集素Aga锚定于细胞表面,通过半乳糖诱导表达,但该表达体系有一定的缺陷。首先,支架蛋白的半乳糖诱导表达过程不能实现真正的CBP途径;其次,凝集素Aga蛋白由Aga1和Aga2双亚基组成,所以需要与EBY100细胞匹配使用,极大地限制了底盘细胞的改造。酵母细胞表面展示是把异源靶蛋白与酿酒酵母的载体蛋白融合后,通过GPI锚定机制使靶蛋白定位于细胞表面的表达系统。用于酿酒酵母表面展示的锚定蛋白有 Aga1p、Cwp1p、Cwp2p、Agα1p、Sed1p、Tip1p、Flo1p、YCR89w 以及Tir1p。以α-半乳糖苷酶为靶蛋白,比较不同展示系统的展示效率发现,Cwp2p、Aga1p和 Sed1p具有较高的锚定效率,但是Aga1p-α-半乳糖苷酶的总酶活分别比 Cwp2p-α-半乳糖苷酶和 Sed1p-α-半乳糖苷酶低6 倍和 8 倍[43]。Goyal等[30]将 Aga1p 表面展示系统更换为Agα1p表面展示系统,并且将半乳糖诱导型启动子换为酿酒酵母组成型强启动子PGK后,使支架蛋白的展示效率从60%增加到了85%。所以,优化支架蛋白的表面展示也是提高纤维小体展示效率的有效措施之一。
3 人造纤维小体在其他宿主中表达的研究
天然的纤维小体结构复杂,构建组分确定的纤维小体嵌合体,可简化纤维小体的结构,从而推动对天然纤维小体的研究。最初,为了研究纤维小体各组分的功能特性,Fierobe 等[31,44]在大肠杆菌中成功表达了微型纤维小体,每个酶组分能够特异性地结合至相应粘连模块上,并且将支架蛋白和酶组分按照化学计量数之比就可以实现完全对接。2002年Fierobe等[45]研究发现支架蛋白上CBM的出现能提高纤维素酶的总活力,且复合物酶组分的邻近效应能促进酶组分之间的协同作用。丙酮丁醇梭菌是生产具有重要工业价值产品——丁醇的菌株,其基因组内含有纤维小体基因,但是利用纤维素的能力非常弱。所以根据其自身带有的纤维小体相关基因进行理性设计,赋予其利用纤维素生产丁醇的能力也越来越受关注[46-47]。为了提高丙酮丁醇梭菌的纤维素利用能力,Perret等[46]在丙酮丁醇梭菌中表达了异源支架蛋白,但是还没有实现纤维素酶与支架蛋白的组装。此外,枯草芽胞杆菌自身能够表达半纤维素酶,并且能利用可溶性的戊糖和己糖(如葡萄糖、木糖以及纤维二糖),但是却不能以纤维素原料作为唯一C源生长。Murashima等[48]构建了来源于嗜纤维梭菌的微型支架蛋白miniCbpA,实现了内切葡聚糖酶EngB对接到枯草芽胞杆菌表面的支架蛋白上。Arai等[49]将嗜纤维梭菌的EngB、XynB及微型支架蛋白miniCbpA分别在枯草芽胞杆菌中单独表达,共表达过程自组装形成纤维小体。
4 展望
目前,酿酒酵母人造纤维小体的研究还处于初级阶段,很多问题并未得到解决,如支架蛋白的展示效率较低、纤维素酶组分单一、纤维小体各组分在酿酒酵母中表达量及表达活性不足、体外组装效率低等。为了提高酿酒酵母纤维小体对纤维素的降解能力,从提高酿酒酵母纤维素酶和支架蛋白的分泌能力以及优化微型纤维小体设计等方面着手,构建高效的纤维小体嵌合体。例如,筛选可在酵母中实现高活性表达的纤维素酶以及采用高效的表面展示系统提高支架蛋白的表面展示;或者通过酿酒酵母自身分泌途径的相关改造,提高纤维素酶和支架蛋白的分泌。另外,在3种纤维素酶协同作用的基础上,增加纤维素酶的种类及数量,构建多层支架蛋白,也可以达到优化纤维小体的设计的目的。高效CBP酵母的构建,是提高木质纤维素资源的转化能力的有效方式之一,因此,高效酿酒酵母纤维小体嵌合体的构建对于推动纤维素资源的利用有着重要意义。
[1] Lynd L R,van Zyl W H,McBride J E,et al.Consolidated bioprocessing of cellulosic biomass:an update[J].Curr Opin Biotechnol,2005,16:577-583.
[2] van Zyl W H,Lynd L R,den Haan R,et al.Consolidated bioprocessing forbioethanolproduction using Saccharomyces cerevisiae[M].Adv Biochem Eng Biotechnol,2007,108:205-235.
[3] Tang H,Hou J,Shen Y,et al.High beta-glucosidase secretion in Saccharomyces cerevisiae improves the efficiency of cellulase hydrolysis and ethanol production in simultaneous saccharification and fermentation[J].J Microbiol Biotechnol,2013,23(11):1577-1585.
[4] Xu L,Shen Y,Hou J,et al.Secretory pathway engineering enhances secretion of cellobiohydrolase I from Trichoderma reesei in Saccharomyces cerevisiae[J].J Biosci Bioeng,2014,117(1):45-52.
[5] Hahn-Hägerdal B,Wahlbom C F,Gárdonyi M,et al.Metabolic engineering of Saccharomyces cerevisiae for xylose utilization[J].Adv Biochem Eng Biotechnol,2001,73:53-84.
[6] Ilmén M,den Haan R,Brevnova E,et al.High level secretion of cellobiohydrolases by Saccharomyces cerevisiae[J].Biotechnol Biofuels,2011,4:30.doi:10.1186/1754-6834-4-30.
[7] Bayer E A,Belaich J P,Shoham Y,et al.The cellulosomes:multienzymemachines fordegradation ofplantcellwall polysaccharides[J].Annu Rev Microbiol,2004,58:521-554.
[8] Johnson E A,Madia A,Demain A L.Chemically defined minimal medium for growth of the anaerobic cellulolytic thermophile Clostridium thermocellum[J].Appl Environm Microbiol,1981,41(4):1060-1062.
[9] Demain A L,Newcomb M,Wu J H.Cellulase,clostridia,and ethanol[J].Microbiol Mol Biol Rev,2005,69:124-154.
[10] Tokatlidis K,Salamitou S,Béguin P,et al.Interaction of the duplicated segment carried by Clostridium thermocellum cellulases with cellulosome components[J].FEBS Lett,1991,291(2):185-188.
[11] Fierobe H P,Pagès S,Bélaïch A,et al.Cellulosome from Clostridium cellulolyticum:molecular study of the dockerin/cohesin interaction[J].Biochemistry,1999,38:12822-12832.
[12] Leibovitz E,Béguin P.A new type of cohesin domain that specifically bindsthe dockerin domain ofthe Clostridium thermocellum cellulosome-integrating protein CipA[J]. J Bacteriol,1996,178:3077-3084.
[13] Doi R H,Goldstein M,Hashida S,et al.The Clostridium cellulovorans cellulosome[J].Crit Rev Microbiol,1994,20(2):87-93.
[14] Gal L,Pages S,Gaudin C,et al.Characterization of the cellulolytic complex(cellulosome)produced by Clostridium cellulolyticum[J].Appl Environ Microbiol,1997,63(3):903-909.
[15] Kakiuchi M,Isui A,Suzuki K,et al.Cloningand DNA sequencing of the genes encoding Clostridium josui scaffolding protein CipA and cellulase CelD and identification of their gene products as major components of the cellulosome[J].J Bacteriol,1998,180:4303-4308.
[16] Sabathé F,Soucaille P.Characterization of the CipA scaffolding protein and in vivo production of a minicellulosome in Clostridium acetobutylicum[J].J Bacteriol,2003,185:1092-1096.
[17] Gerngross U T,Romaniec M P,Kobayashi T,et al.Sequencing of a Clostridium thermocellum gene (cipA)encoding the cellulosomal SL-protein reveals an unusual degree of internal homology[J].Mol Microbiol,1993,8(2):325-334.
[18] Kruus K,Lua A C,Demain A L,et al.The anchorage function of CipA (CelL),a scaffolding protein ofthe Clostridium thermocellum cellulosome[J].PNAS,1995,92:9254-9258.
[19] David WuJ H,Orme-JohnsonW H,DemainA L.Two components of an extracellular protein aggregate of Clostridium thermocellum together degrade crystalline cellulose[J].Biochemistry,1988,27(5):1703-1709.
[20] Xu Q,Barak Y,Kenig R,et al.A novel Acetivibrio cellulolyticus anchoring scaffoldin thatbearsdivergentcohesins[J].J Bacteriol,2004,186:5782-5789.
[21] Rincón M T,Cepeljnik T,Martin J C,et al.A novel cell surfaceanchored cellulose-binding protein encoded by the sca gene cluster of Ruminococcus flavefaciens[J].J Bacteriol,2007,189:4774-4783.
[22] Rincón M T,Martin J C,Aurilia V,et al.ScaC,an adaptor protein carrying a novel cohesin that expands the dockerin-binding repertoire of the Ruminococcus flavefaciens 17 cellulosome[J].J Bacteriol,2004,186:2576-2585.
[23] Mingardon F,Chanal A,Tardif C,et al.Exploration ofnew geometries in cellulosome-like chimeras[J].Appl Environ Microbiol,2007,73:7138-7149.
[24] Tsai S L,Oh J,Singh S,etal.Functionalassemblyof minicellulosomes on the Saccharomyces cerevisiae cell surface for cellulose hydrolysis and ethanol production[J].Appl Environ Microbiol,2009,75:6087-6093.
[25] Tsai S L,Goyal G,Chen W.Surface display of a functional minicellulosome by intracellular complementation using a synthetic yeastconsortium and itsapplication to cellulose hydrolysis and ethanol production[J].Appl Environ Microbiol,2010,76:7514-7520.
[26] Hyeon JE,Yu K O,Suh D J,etal.Production of minicellulosomes from Clostridium cellulovorans for the fermentation of cellulosic ethanol using engineered recombinant Saccharomyces cerevisiae[J].FEMS Microbiol Lett,2010,310:39-47.
[27] Fan L H,Zhang Z J,Yu X Y,et al.Self-surface assembly of cellulosomes with two miniscaffoldins on Saccharomyces cerevisiae forcellulosic ethanolproduction[J].PNAS,2012,109:13260-13265.
[28] Wen F,Sun J,Zhao H.Yeastsurface display of trifunctional minicellulosomes for simultaneous saccharification and fermentation of cellulose to ethanol[J].Appl Environ Microbiol,2010,76:1251-1260.
[29] Linder M,Teeri T T.The cellulose-binding domain of the major cellobiohydrolase of Trichoderma reesei exhibits true reversibility and a high exchange rate on crystalline cellulose[J].PNAS,1996,93:12251-12255.
[30] Goyal G,Tsai S L,Madan B.Simultaneous cell growth and ethanol production from cellulose by an engineered yeast consortium displaying a functional mini-cellulosome[J].Microb Cell Fact,2011,10:89.doi:10.1186/1475-2859-10-89.
[31] Fierobe H P,Pagès S,Bélaïch A,et al.Cellulosome from Clostridium cellulolyticum:molecular study of the dockerin/cohesin interaction[J].Biochemistry,1999,38:12822-12832.
[32] Yaron S,Morag E,Bayer E A,et al.Expression,purification and subunit-binding properties of cohesins 2 and 3 of the Clostridium thermocellum cellulosome[J].FEBS Lett,1995,360:121-124.
[33] Ito J,Kosugi A,Tanaka T,et al.Regulation of thedisplay ratio of enzymes on the Saccharomyces cerevisiae cell surface by the immunoglobulin G and cellulosomal enzyme binding domains[J].Appl Environ Microbiol,2009,75:4149-4154.
[34] Liu Z,Tyo K E,Martinez J L,et al.Different expression systems for production of recombinant proteins in Saccharomyces cerevisiae[J].Biotechnol Bioeng,2012,109:1259-1268.
[35] Travers K J,Patil C K,Wodicka L,et al.Functional and genomic analyses reveal an essential coordination between the unfolded protein response and ER-associated degradation[J].Cell,2000,101:249-258.
[36] IdirisA,Tohda H,Kumagai H,et al.Engineering of protein secretion in yeast:strategies and impact on protein production[J].Appl Microbiol Biotechnol,2010,86:403-417.
[37] Ji L,Shen Y,Xu L,et al.Enhanced resistance of Saccharomyces cerevisiae to vanillin by expression of lacA from Trametes sp.AH28-2[J].Bioresour Technol,2011,102:8105-8109.
[38] Payne T,Finnis C,Evans L R,et al.Modulation of chaperone gene expression in mutagenized Saccharomyces cerevisiae strains developed for recombinant human albumin production results in increased production of multiple heterologous proteins[J].Appl Environ Microbiol,2008,74:7759-7766.
[39] Robinson A S,Hines V,Wittrup K D.Protein disulfide isomerase overexpression increases secretion of foreign proteins in Saccharomyces cerevisiae[J].Biotechnology(N Y),1994,12(4):381-384.
[40] Valkonen M,Penttilä M,Saloheimo M.Effects of inactivation and constitutive expression of the unfolded-protein response pathway on protein production in the yeast Saccharomyces cerevisiae[J].Appl Environ Microbiol,2003,69:2065-2072.
[41] Suzuki H,Imaeda T,Kitagawa T,et al.Deglycosylation of cellulosomal enzyme enhances cellulosome assembly in Saccharomyces cerevisiae[J].J Biotechnol,2012,157:64-70.
[42] Ruohonen L,Toikkanen J,Tieaho V,et al.Enhancement of protein secretion in Saccharomyces cerevisiae by overproduction of Sso protein,a late-acting component of the secretory machinery[J].Yeast,1997,13:337-351.
[43] van der Vaart J M,te Biesebeke R,Chapman J W,et al.Comparison of cell wall proteins of Saccharomyces cerevisiae as anchors for cell surface expression of heterologous proteins[J].Appl Environ Microbiol,1997,63:615-620.
[44] Fierobe H P,Mechaly A,Tardif C,et al.Design and production of active cellulosome chimeras:selective incorporation of dockerincontaining enzymes into defined functional complexes[J].J Biol Chem,2001,276:21257-21261.
[45] Fierobe H P,Bayer E A,Tardif C,et al.Degradation of cellulose substrates by cellulosome chimeras:substrate targeting versus proximity of enzyme components[J].J Biol Chem,2002,277:49621-49630.
[46] Perret S,Casalot L,Fierobe H P,et al.Production of heterologous and chimeric scaffoldins by Clostridium acetobutylicum ATCC 824[J].J Bacteriol,2004,186:253-257.
[47] Mingardon F,Perret S,Bélaïch A,et al.Heterologous production,assembly,and secretion of a minicellulosome by Clostridium acetobutylicum ATCC 824[J].Appl Environl Microbiol,2005,71:1215-1222.
[48] Murashima K,Chen C L,Kosugi A,et al.Heterologous production ofClostridium cellulovorans engB,using protease-deficient Bacillus subtilis,and preparation of active recombinant cellulosomes[J].J Bacteriol,2002,184:76-81.
[49] Arai T,Matsuoka S,Cho HY,et al.Synthesis of Clostridium cellulovorans minicellulosomes by intercellular complementation[J].PNAS,2007,104:1456-1460.
