RNA干扰的研究进展
2014-04-29何洁凝田生礼
何洁凝 田生礼
摘要:RNA干扰(RNA interference,RNAi)是一种由双链RNA诱发的转录后水平的基因沉默现象,是近几年发展起来的基因表达调节新机制。RNAi广泛存在于真菌、植物和动物中,这种调控可以由siRNA、shRNA及miRNA等小分子RNA参与。在此主要对RNAi的研究进展如背景、分子调控机制、存在的问题和应用前景等进行了综述。
关键词:RNA干扰;小分子RNA;研究进展
中图分类号:Q819
文献标识码:A
文章编号:1007-7847(2014)03-0265-04
2006年,美国斯坦福大学的Andrew Z.Fire和美国马萨诸塞大学的CraigC.Mello分别被授予了诺贝尔生理学和医学奖,以表彰他们发现了RNA干扰(RNA interference,RNAi)现象,即用双链RNA使基因沉默(RNA interference gene silencingby double-stranded RNA)。自发现RNAi现象至2006年的8年间,在PubMed网站上能搜索到的有关RNAi的文章已经达到8900多篇,可见,从RNA干扰理论提出到现在已经历十余年时间,人们对其机制和应用的研究热情始终没有冷却。为了加深对该技术的了解与追踪,本文主要对RNAi的发现、作用机制、应用的研究进展予以综述。
1RNAi的发现
RNA干扰是一种在进化过程中存在的高度保守、由双链RNA分子(double-stranded RNA,dsRNA)诱发同源mRNA高效特异性降解的基因转录后沉默现象(post-transcriptional gene silenc-ing,PTGS)。1990年初,科学家在向矮牵牛花导入能使花卉变得更鲜艳的查尔酮基因后,结果却发现,一些花的颜色不但没有变鲜艳反而被“漂白”了。这种过度表达内源基因而引发的基因沉默的现象当时被称为共阻遏。1998年Fire等在向线虫中分别导入正义、反义和双链RNA同样干扰par-l基因表达,结果发现单链反义RNA和双链RNA均能特异性沉默靶基因的表达,双链RNA的效果明显比单链RNA的沉默效果强,于是他们将这种双链RNA抑制基因表达的现象称为RNA干扰(RNA interference,RNAi),把引发RNA干扰现象的RNA分子称为干扰RNA[1]。此后,RNA干扰这种现象被证明广泛存在于多种真核生物中。
2RNAi的作用机制
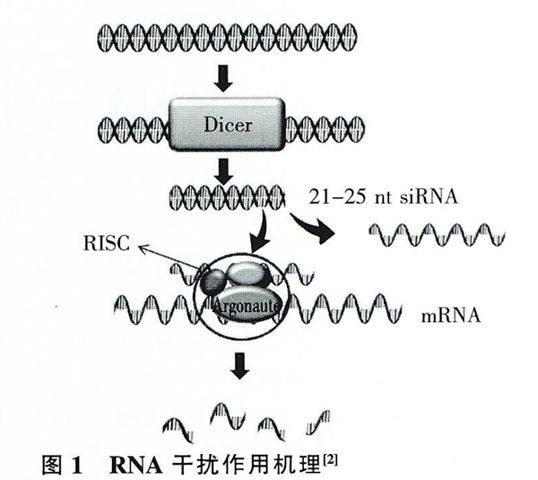
众所周知,RNAi的分子主要包括以下几种:siRNA、shRNA、miRNA,虽然它们在结构上各不相同,但是所有这些分子都具有诱导基因沉默的能力,它们在细胞中的作用机制主要有3个步骤[2]:
起始阶段:外源性或内源性双链RNA分子被核酸酶Dicer加工剪切成21~25个核苷酸长度的小分子双链。
RISC (RNA-induced silencing complex)的组装:产生的小分子双链RNA中其中一条链与相关的蛋白(主要是Argonaute蛋白)组装形成具有活性的沉默复合体RISC。
效应阶段:组装形成的具有活性的RISC作用于与其上的单链小分子RNA序列互补的mR-NA,并将该mRNA降解,使其翻译受到抑制,最终导致基因的沉默。siRNA、shRNA与miRNA对靶基因mRNA沉默时作用的位点有所不同,siRNA沉默目的基因主要作用于mRNA的编码序列,而miRNA主要作用于mRNA的3的非翻译区。
在RNAi过程中,研究得较清楚的蛋白主要是Argonaute蛋白家族:Argonaute(Ago)蛋白属于一个高度保守的蛋白家族,包括Agol、Ago2、A-go3、Ago4。它的大小约100kD,这4种蛋白都包含两个重要的结构域:PAZ、MID结构域,分别与miRNA3'与5'端相互作用。在所有的Ago蛋白中只有Ago2所包含的PIWI结构域具有切割活性,Agol、Ago3、Ago4没有此活性[3,4]。Argonaute能跟不同的小分子RNA(miRNA、siRNA)结合,根据小分子RNA结构的不同,它们所结合的Argonaute蛋白也有所区别,其中miRNA与Agol结合,而siRNA则与Ago2结合[5]。最近研究发现,Ago2能提高细胞中miRNA的稳定性及提高成熟miRNA在细胞中的水平[6];Ago3蛋白的PIWI与Piwi-in-teracting RNAs (piRNAs)相互作用产生沉默转座子的作用;Ago4在调控哺乳动物精细胞进入减数分裂和减数分裂性染色体失活(MSCI)中起作用[7]。
3RNAi的应用
3.1基因功能研究
在后基因组时代,基因功能的研究显得尤其迫切,可是,由于我们在进行研究基因功能实验时难以直接在人活体内进行,建立有效的转基因或基因敲除的动物模型显得格外重要,RNAi技术在此方面做出重大贡献。Kim等利用针对影响胚胎发育的两种内源性的Oct3/4和c-mos基因的siRNA,将其植入鼠卵母细胞前胚胎中,成功地清除了内源性的Oct3/4和c-mos产物,建立了敲除Oct3/4和c-mos基因的动物模型[8];其次利用RNAi文库也是基因功能研究的重要手段,RNAi文库是人工构建的一种混合文库,研究者能通过它诱导RNAi抑制众多种不同基因表达。目前,研究者利用RNAi文库已经发现了许多与生物功能相关的新基因和治疗疾病的新靶点。Buss等在秀丽隐杆线虫中通过全基因组RNAi筛选的方式,发现烟碱型乙酰胆碱受体(nicotinic acetylcholine re-ceptor subunit ACR-7)是抗精神病药物的靶点[9]。
3.2临床基础研究
RNAi技术具有简单、快速、特异性沉默任意基因的特点,使其被广泛用来研究肿瘤、肺动脉高压、先天性遗传病等疾病的治疗靶点,并取得了一定成果。最近Ku等证实通过脂质体的方式向T24膀胱癌细胞中导入针对生存素(survivin)的siRNA,该siRNA能特异性地干扰膀胱癌细胞中生存素的表达,同时加速了膀胱癌细胞的凋亡[10]。Wedg-wood等运用RNAi技术研究发现过氧化氢酶在新生儿肺动脉高压中血管的重塑中起着重要作用,有望成为该疾病治疗的靶点[11]。Leachman等对先天性厚甲遗传病患者通过皮下注射siRNA的方式针对突变的K6a角质蛋白进行沉默,3个月后成功对该蛋白进行沉默的同时发现患者皮肤起茧情况明显得到好转。此外,RNAi治疗并没有给患者带来其他负面影响[12],这是RNAi在药物治疗方法上的重要进展。RNAi技术还被应用于整形外科方面,整形外科的开展主要以手术为主,RNAi技术的应用为整形外科的开展注入了新的力量。众所周知,当人体皮肤深层受到创伤后,机体产生的炎症反应及应激反应会导致基质层许多生长因子异常增多,从而诱使皮肤深层基质细胞变性转化疤痕组织,产生疤痕,这是目前整形外科研究的热点和难点。Liu等将RNAi技术与胶原壳聚糖硅橡胶皮肤再生材料(collagen-chitosan/sili-cone membrane bilayer dermai equivalent)结合起来应用于背部皮肤严重缺损的猪的皮肤修复上,研究发现siRNA能明显抑制TGF-βi生长因子的表达并同时抑制皮肤修复过程中疤痕的产生[13]。在整形外科软骨移植中,经常会发现软骨外质细胞被大量降解,出现使细胞质中蛋白多糖与胶原水平失衡的现象,Wang等采用RNAi的手段通过抑制软骨细胞多聚蛋白聚糖酶1,2(aggrecanase)的表达,减少其对蛋白多糖的分解,维持软骨移植后软骨细胞外基质的平衡[14]。
3.3药物开发研究
RNAi技术还被应用于鉴定药物靶点和筛选药物方面。RNAi技术的应用明显地缩短了从筛选到鉴定药物靶基因功能的时间,有助于药物开发过程中对已知靶基因功能的高通量分析。在鉴定RNAi药物靶点上,Watanabe等研究发现通过siRNA干扰的方式在小鼠中沉默蛋白转移酶9(proprotein convertase subtilisin/kexin type 9,PC-SK9)能有效地减少血清中胆固醇水平,首次证明通过沉默PCSK9有望成为降低胆固醇的治疗靶点[15]。在筛选药物上,Liu-Sullivan[16]等通过pooledshRNA的方法发现维甲酸类药物能增强pkll激酶抑制剂药物GSK461364的抑制作用,进而阻止细胞有丝分裂,加速了癌细胞凋亡的速度。
4影响RNAi效率的因素及RNAi应用中存在的问题
4.1影响RNAi效率的因素
4.1.1RNAi序列的设计
RNAi技术的关键是如何有效发挥siRNA的干扰作用。在RNAi的实际应用中经常发现合理设计的siRNA不能发挥有效的干扰作用,分析影响siRNA干扰效果的因素包括dsRNA双链末端的稳定性、siRNA与RISC的结合情况及干扰载体的转染效率。在对siRNA特异性研究中,发现siRNA最好设计在19~25个碱基之间,且siRNA的GC含量应在30%~52%之间。此外,在siRNA特定的位置应设计特定的核苷酸,例如:siRNA的sense序列的5'端的第一个碱基最好是U,第3、10位碱基最好是A[17],因为A在热力学上结合力更低,因此会降低siRNA双链的稳定性使RISC更好解开双链siRNA,同时增加siRNA的基因沉默效率。相反,若siRNA序列中存在反向重复序列会降低siRNA的沉默效果。
4.1.2RNAi序列的稳定性
一般siRNA在血清的环境下,很容易被降解以致其难以在细胞中发挥其RNAi作用。因此对siRNA进行适当的化学修饰是必要的。
目前已经存在很多siRNA优化方法,研究发现siRNA经过tricyclo-DNA修饰后能明显提高siRNA在血清中的稳定性[18]。除了上述修饰外,还有一种化学修饰称LNAs修饰(locked nucleic acids),它是一种核酸类似物,其能使RNA/DNA增加耐受核酸酶的能力,此外,这种修饰能降低siRNA对细胞的毒性。Petri等[19]研究发现利用LNAs修饰后的siRNA更利于Ago2聚集和剪切,从而能明显提高siRNA的稳定性及减少siRNA的脱靶效应。
4.1.3RNAi序列的导入方式
RNAi技术被广泛的应用,成功将RNAi分子导入目的细胞,是实现RNAi的先决条件。RNAi分子存在易降解和不稳定的特点,为了保证RNAi分子能有效地被运送到目的组织细胞,需要特定的导入方式。文献报道的导入方式有多种,比如浸泡、注射、脂质体包裹转染、纳米材料导入、噬菌体包装转染或通过物理的方式直接导入等,其中,很多研究者会选择采用病毒载体,利用病毒高感染率、高表达的特性开展RNAi实验研究。但是它同时也存在着一些风险,比如病毒会诱发细胞中基因产生突变或会引起细胞中各种炎症反应等。
针对于病毒载体给细胞带来的各种影响,非病毒性的导入方式越来越受到研究者的青睐。目前,有一种著名的tkRNAi运输shRNA小分子的技术,这种技术主要是用非治病性的大肠杆菌内含产生特定shRNA的质粒DNA,并将其运送到细胞中以产生RNAi的效果。Kruhn等运用该技术将针对ABCB1的shRNA导入到人胃癌细胞中,shRNA能有效干扰ABCB1蛋白的表达,调节了癌细胞中由ABCB1蛋白介导的多药抗性的表型[20]。
4.2RNAi技术应用中存在的问题
4.2.1免疫反应
细胞中免疫系统对自身的核酸和外源核酸总是有区别对待的,这种反应主要体现在细胞水平上和分子水平上,其主要的机理是:外源的RNA或DNA会刺激细胞中IFN(type I interferon)因子和细胞炎症因子的产生。在RNAi过程中,依赖dsRNA的激酶(dsRNA dependent protein kinase,PKR)能激活2'-5'寡腺苷酸合成酶( 2'-5'oligoadenylate synthetase,OAS),OAS会刺激ATP形成2'-5'寡腺苷核苷酸,进一步激活细胞中核糖核酸酶(RNaseL)形成,进而降解细胞中的RNA,包括RNAi小分子,降低RNAi的沉默效率[21]。适当的化学修饰能改善siRNA引起的细胞免疫反应,如将siRNA用2'-O-Me,2'-O-F加以修饰足以改善siRNA的免疫刺激活性并能更好地优化siR-NA的沉默效果[22]。
4.2.2脱靶效应
众所周知,Ago-RISC对靶mRNA的剪切是具有高度特异性的,可是在siRNA的应用过程中也会出现许多脱靶效应从而诱发细胞毒性。siR-NA的脱靶效应主要与其序列中靠近5'端的2~8个碱基密切相关,因为它们与mRNA的3'UTR互补并与RISC结合紧密。为了减少siRNA的脱靶效应,提高siRNA的特异性主要通过以下方式:1)降低siRNA中5'端的热力学稳定性;2)在选择靶mRNA位置时要避免靠近mRNA的起始编码区;3)通过使用siRNA pools的方法,在既能保持siRNA的沉默效率下,又能减低siRNA的脱靶效应[23];4)文献报道,在siRNA的第7位加入一个不稳定的unlocked nucleic acid (UNA)修饰能减少siRNA的脱靶效应[24]。
4.2.3饱和现象
在正常细胞中,小RNAi分子与其靶基因之间始终保持着一种平衡的状态,这使细胞一直保持着正常的生理功能。大量外源的siRNA/shRNA被导入到细胞内,使细胞中正常RNAi水平达到一种不平衡的状态。大量外源的siRNA/shRNA进入细胞会与内源的miRNA竞争某些蛋白因子,如Dicer、Ago蛋白等,以致细胞内的miRNA不能行使正常的功能。在细胞中,即使使用弱启动子表达了大量shRNA也会诱发生物体内细胞毒性。为了避免上述问题的产生,Grimm等将Ago2、Exp5、L6-shRNA共同导入到小鼠内,结果发现shRNA的干扰效果明显增强,同时检测小鼠肝脏毒性明显降低[25]。为了避免内源miRNA与外源的shRNA竞争Dicer因子,Liu等最近研究出一种Ago2shRNA,该shRNA可以不通过Dicer而直接利用Ago2行使剪切功能,这种设计有望能在进一步提高RNAi的同时避免引起RNA干扰效果的脱靶效应[26]。
5展望
RNA干扰技术是一个生物学研究的亮点,它给许多疾病的治疗与药物的开发提供了新的思路,其中RNAi药物具有药效测试周期短,在理论上适合于任何基因治疗的特点,其中比如anti-sense RNAi药物,它的特异性非常强,有可能在RNAi药物开发中引来新的一场革命。目前,许多RNAi药物迅速地进入了临床研究阶段。虽然还存在一些问题,如出现脱靶效应、免疫反应等问题,但我们始终相信这些问题最终会被解决,使RNAi技术能更好地被广泛地应用在基础研究、临床研究及RNAi药物研发之中。
参考文献( References):
[1]FIRE A,XU S,MONTGOMERY M K,et al.Potent and specific genetic interference by double-stranded RNA in Caenor-habditis elegans[J]. Nature, 1998, 391(6669):806-811.
[2]SIOMI H,SIOMI M C.On the road to reading the RNA-inter-ference code[J]. Nature, 2009, 457(7228):396-404.
[3]JUVVUNA P K,KHANDELIA P,LEE L M,et al.Argonauteidentity defines the length of mature mammalian microRNAs[J].Nuclcic Acids Research, 2012, 40(14):6808-6820.
[4]WEI N,ZHANC L HUANG H,et al.siRNA has greatly ele-vated mismatch tolerance at 3'-UTR sites[J]. PLoS One, 2012,7(11):e49309.
[5]GHILDIYAL M,ZAMORE P D Small silencing RNAs: an ex-panding universe[J]. Nature Reviews Gcnctic, 2009, 10(2):94-108.
[6]WINTER J,DIEDERICHS S.Argonaute proteins regulate mi-croRNA stability: Increased microRNA abundance by Argo-naute proteins is due to microRNA stabilization[J]. RNA Biology,2011, 8(6):1149-1157.
[7]MODZELEWSKI A J,HOLMES R J,HILZ S,et al.AGO4regulates entry into meiosis and influences silencing of sexchromosomes in the male mouse germline[J]. Developmental Cell,2012, 23(2):251-264.
[8]KIM M H,YUAN X,OKUMURA S.et al.Successful inactiva-tion of endogenous Oct-3/4 and c-mos genes in mouse, preim-plantation embryos and oocytes using short interfering RNAs[J].Biochemical and Biophysical Research Communications, 2002,296(5):1372-1377.
[9]SAUR T,DEMARCO S E,ORTIZ A,et al.A genome-wideRNAi screen in Caenorhabditis elegans identifies the nicotinicacetylcholine receptor subunit ACR-7 as an antipsychoticdrug target[J]. PLoS Genetics, 2013, 9(2):e1003313.
[10]KU J H,SEO S Y,KWAK C,et al.Cytotoxicity and apoptosisby survivin small interfering RNA in bladder cancer cells[J].BJU International, 2010, 106(11):1812-1816.
[11]WEDGWOOD S,LAKSHMINRUSIMHA S,CZECH L et al.Increased p22(phox)/Nox4 expression is involved in remodelingthrough hydrogen peroxide signaling in experimental persistentpulmonary hypertension of the newborn[J]. Antioxidants&Re-dox Signaling, 2013, 18(14):1765-1776.
[12]LEACHMAN S A, HICKERSON R P, SCHWARTZ M E, etal. First-in-human mutation-targeted siRNA phase Ib trial ofan inherited skin disorder[J]. Molecular Therapy , 2010, 18(2):442-446.
[13]LIU X, MA L, LIANG J, et al. RNAi functionalized collagen-chitosan/silicone membrane bilayer dermal equivalent for full-thickness skin regeneration with inhibited scarring[J]. Biomateri-als, 2013, 34(8):2038-2048.
[14]WANG Z H, YANG Z Q, HE X J, et al. Lentivirus-mediatedknockdown of aggrecanase-l and -2 promotes chondrocyte-engineered cartilage formation in vitro[J]. Biotechnology and Bio-engineering, 2010, 107(4):730-736.
[15]DUFF C J,HOOPER N M. PCSK9: an emerging target fortreatment of hypercholesterolemia[J]. Expert Opinion on Ther-apeutic Targets, 2011, 15(2):157-168.
[16]LIU -SULLIVAN N, ZHANG J, BAKLEH A, et al. PooledshRNA screen for sensitizers to inhibition of the mitotic regulator polo-like kinase(PLKl)[J]. Oncotarget, 2011, 2(12):1254-1264.
[17]DYXHOOM D M, LIEBERMAN J. Running interference:prospects and obstacles to using small interfering RNAs assmall molecule drugs[J]. Annual Review of Biomedical Engineer-ing, 2006, 8:377-402.
[18]ITTIG D. LUISIER S, WEILER J, et al. Improving gene si-lencing of siRNAs via tricyclo-DNA modificacion[J]. ArtificialDNA. PNA & XNA, 2010, 1(1):9-16.
[19]PETRI S, DUECK A, LEHMANN G, et al. Increased siRNAduplex stability correlates with reduced off-target and elevatedon-target effects[J]. RNA, 2011, 17(4):737-749.
[20]KRUHN A, WANG A, FRUEHAUF J H, et al. Delivery ofshort hairpin RNAs by transkingdom RNA interference modu-lates the classical ABCBl-mediated multidrug-resistant phe-notype of cancer cells[J]. Cell Cycle, 2009, 8(20):3349-3354.
[21]SIOUD M. Recent advances in small interfering RNA sensingby the immune system[J]. New Biotechnology, 2010, 27(3):236-242.
[22]ROBBINS M, JUDGE A, MACLACHLAN I. siRNA and innateimmunity[J]. Oligonucleotides, 2009, 19(2):89-102.
[23]BRAMSEN J B, KJEMS J. Chemical modification of small in-terfering RNA[J]. Methods in Molecular Biology, 2011, 721:77-103.
[24]BRAMSEN J B, PAKULA M M, HANSEN T B, et al. Ascreen of chemical modifications identifies position-specificmodification by UNA to most potently reduce siRNA off-targeteffects[J]. Nucleic Acids Research, 2010, 38(17):5761-5773.
[25]GRIMM D, WANG L, LEE J S, et al. Argonaute proteins arekey determinants of RNAi efficacy, toxicity, and persistence inthe adult mouse liver[J]. The Journal of Clinical Investigation,2010, 120(9):3106-3119.
[26]LIU Y P, SCHOPMAN N C,BERKHOUT B. Dicer-indepen-dent processing of short hairpin RNAs[J]. Nucleic Acids Research,2013, 41(6):3723-3733.
