基于微卫星标记的不同壳型湖北钉螺小尺度景观群体遗传结构研究
2014-04-02李石柱
崔 斌,官 威,尤 平,李石柱
湖北钉螺(Oncomelaniahupensis) 是日本血吸虫唯一的中间宿主,我国大陆仅有一种,广泛分布于长江中下游地区[1-2]。由于其分布广泛、孳生环境复杂以及地理隔离的影响,湖北钉螺不同地理群体发生了显著的遗传分化和变异。近年来,随着湖北钉螺的群体遗传学、景观生态学等深入研究,我国大陆分布的钉螺地理种群主要划分为长江中下游的湖沼型景观种群、高山型景观种群、喀斯特地貌景观种群和滨海山丘型景观种群[3],其中湖北钉螺湖沼型景观种群主要分布在长江中下游的湖南、湖北、江西、安徽和江苏等省的湖沼、水网和丘陵等地区,这一地区景观范围广,水网密布,植被丰富,且雨量充沛,是遗传多样性最为复杂的一个种群,甚至同一地区不同微生境种群已表现出明显的遗传分化[4]。
湖北松滋地区位于湖北省西南部,地形西高东低,从西南海拔500~800 m较高区的山地向海拔50 m以下的江汉平原湖区呈四阶梯递降,境内河渠纵横,间有湖泊,其境内有螺环境多样,主要包括垸内沟渠型和山丘型等,且不同类型环境下钉螺随生境变化呈现出不同螺壳形态,这种在小尺度生境湖北钉螺种群中呈现明显表型差异的现象并不多见。微卫星DNA标记是广泛分布于真核生物基因组中的一类简单的短核苷酸串联重复序列,具有多态性高、重复性高、呈共显性表达、易检测和符合孟德尔分离等优点,因此被广泛应用于群体遗传多样性分析[5-6]。国内应用微卫星标记进行湖北钉螺遗传多样性的研究开展较晚,且仅有少量应用微卫星锚定进行群体遗传变异研究的报告[7-9]。本研究应用多态性微卫星DNA标记位点,分析了湖北钉螺松滋地区小尺度生境种群遗传结构,以探讨其遗传分化程度。
1 材料和方法
1.1钉螺样本来源 湖北钉螺采自松滋市境内的4个乡镇10个生境,采集地分布见表1。每个采集地钉螺在解剖镜下观察并记录螺壳形态,所有钉螺经室内饲养约1周后,用逸蚴法鉴定钉螺是否感染血吸虫,取阴性钉螺作为实验材料,75%乙醇浸泡,4 ℃保存待用。
1.2方法
1.2.1形态学鉴定 目测钉螺螺壳形态初步分类后,采用解剖镜下观察鉴定并拍照。
1.2.2分子检测
1.2.2.1DNA抽提 取单只湖北钉螺的腹足肌肉组织30 mg,采用软体动物DNA小量提取试剂盒(购自美国OMEGA生物技术公司)提取基因组DNA,-20 ℃保存待用,操作方法参考文献[10]。
1.2.2.2荧光标记引物 多态性微卫星位点的筛选参照文献[11-12],获得的多态性微卫星DNA位点的正向引物进行6-FAM、HEX和NED荧光标记(均购自上海生工生物技术有限公司)(表2)。
1.2.2.3PCR扩增 扩增反应体系为25 μL,包括20 ng模板DNA、0.5 μmol/L引物、1U的Taq酶、0.15 mmol/L的dNTPs、1.5 mmol/L的MgCl2和1×PCR缓冲液。PCR扩增条件为:95 ℃ 5 min;95 ℃ 1 min,50~55 ℃ 45 s,72 ℃ 1.5 min,共35个循环;72 ℃ 5 min。
1.2.2.4扩增产物检测 取1 μL PCR产物,0.6 μL ROX标准物和8.4 μL Hi-Di甲酰胺混匀,95 ℃变性5 min,随后用全自动遗传分析仪检测PCR产物(3730XL,USA, ABI )。

表1 湖北钉螺样本采集地分布

表2 湖北钉螺6个微卫星位点的扩增引物
1.2.3数据分析 基因扫描结果经Genescan和Genotype分析软件获得扩增片段的精确长度,结果以Excel表格形式输出,使用MS-Tools的Excel Microsatellite Toolkit转化原始数据为FSTAT2.9.3.2可识别文件,计算群体内的遗传分化指标平均等位基因数(mean number alleles,MNA)、期望杂合度(expected heterozygosity,He)和观察杂合度(observed heterozygosity,Ho)。应用POPGENE1.3.2软件分别检验哈代-温伯格平衡(Hardy-Weinberg equilibrium,HWE)、F-statistics固定指数(包括Fit、Fst和Fis)和遗传距离。应用ARLEQUIN3.11[13]软件计算成对群体间的基因流(Nm),利用AWOVA模块分析群体遗传变异的组成,再用POPGENE1.3.2软件以非加权组平均法(Unweighted Pair Group Method with Arith-metic means,UPGMA)进行聚类分析[14]。在GENEPOP中对遗传距离与地理距离的相关性进行回归的Mantel检验,根据Botstein等[15]公式计算多态信息含量(Polymorphism Information Content,PIC)。
2 结 果
2.1形态学鉴定结果 本研究共调查了松滋市10个生态环境,获得湖北钉螺样本400余只,解剖镜下鉴别不同生境下的钉螺螺壳形态可见,螺壳形态从山丘环境的光壳逐步向沟渠环境的浅肋、深肋演变,其中王家桥镇、陈店镇4个山丘环境采获的钉螺为光壳,新江口镇3个环境采获的钉螺同时存在光壳和肋壳(浅肋和深肋),老城镇3个沟渠环境为肋壳(图1)。

图1本研究中不同采集地湖北钉螺形态
Fig.1ShelltypesofO.hupensisinthisstudy
From left in line 1: SA, SB, SC, SD, SE; line 2: SF, SG, SH, SI, SK.
2.2基因扫描结果
2.2.1微卫星DNA位点多态信息含量 6个微卫星位点在10个群体中一共检测到141个等位基因,其中B14位点上最多,为34个,D11和T617位点最少,为20个,平均每个位点检测到23.5个。6个微卫星位点的平均有效等位基因数为1.575,各位点的有效等位基因数并不平衡,且数值差异较大,范围从0.445至3.060。
2.2.2群体内遗传变异 10个群体中,观测杂合度较高,在0.59~0.89之间,其中沟渠环境肋壳钉螺群体观察杂合度显著高于山丘型环境的光壳钉螺群体,最高者为老城镇镇SH群体(0.892),最低者为陈店镇SC群体(0.430)。各群体基因多态信息含量较高,PIC在0.528-0.857之间,平均PIC为0.732。其中沟渠环境肋壳钉螺群体显著高于山丘型环境的光壳钉螺群体,最高者为老城镇镇SH群体(0.857),最低者为新江口镇SF群体(0.528)。各群体内近交系数Fis较低,范围在0.056-0.638间,平均为0.301,山丘型光壳钉螺群体显著高于沟渠环境肋壳钉螺群体。各群体的遗传变异系数见表3。

表3 松滋钉螺遗传变异系数
2.2.3群体间遗传差异
2.2.3.1F-统计量 F-统计量中3 个固定指数群体内近交系数(Fis)、群体间基因分化系数(Fst)和总群体的近交系数(Fit)均是反映群体近交程度或群体间遗传分化的指标。本研究F-统计量结果见表4,总群体和群体内近交系数较高,分别为40.35%和30.32%,表明群体内近交严重;各亚群体间基因分化系数(Fst)为14.40%,表明群体间存在一定遗产分化,但分化程度并不平衡,其中SC与SG群体间Fst值最大,为25.25%,SF与SI群体间遗传分化最小,为-1.56%。群体SE与SK间的基因流最大,为19.78,群体SI和SF间基因流最小,为-16.23。

表4 所有样本F-statistics固定指数

表5 遗传分化程度(FST)(下)和基因流(Nm)(上)
Note: *P<0.05; **P<0.01。群体代号同表1。Population codes see Table 1.

表6 遗传距离和地理距离
注: 对角线右上:地理距离(KM);对角线左下;遗传距离。Diagonal right: geographical distance (KM); diagonal left; genetic distance.
2.2.3.2遗传距离和地理距离 对群体间遗传距离与地理距离的相关性进行回归的Mantel检验,结果显示,两者间呈现正相关关系,(R2=0.248,P<0.05)(图2),群体遗传分布符合距离隔离模型,相关性为15.75%。
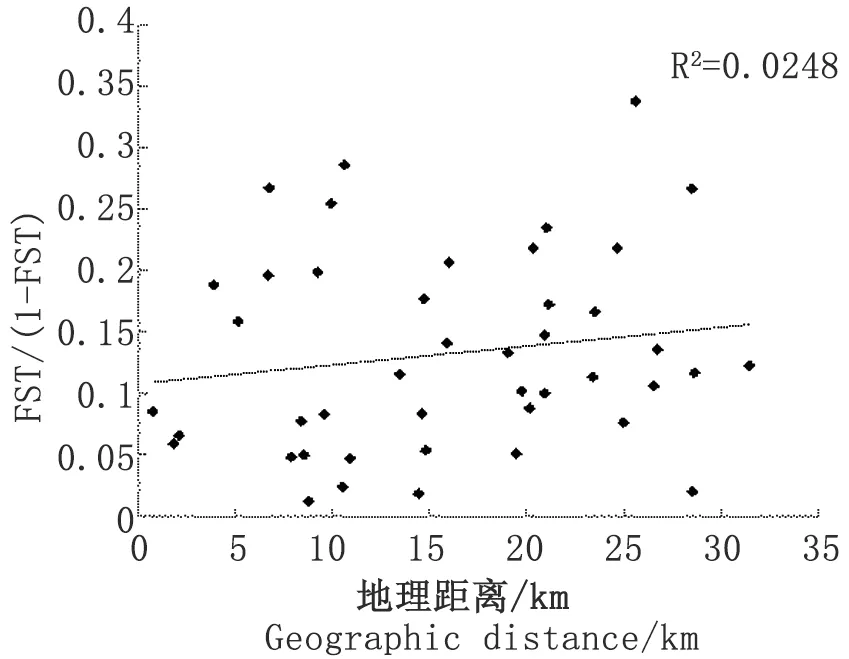
图2遗传距离与地理距离间的相关性分析
Fig.2Analysisontherelationshipbetweengeneticdistanceandgeographicdistance
分子变异等级分析的结果显示,松滋钉螺的变异主要存在于个体间,占总变异的86.48%,群体间变异和群体内个体间变异仅占总变异的7.02%和6.51%(表7)。群体的Fst为0.144,Nm为1.4857,显示群体间遗传差异小,分化不显著。
2.3聚类分析 基于10个群体间的遗传距离,用非加权平均法(UPGMA)聚类显示,肋壳(深肋)钉螺SH、SI、SK与光壳螺SE聚为一支后,再与光壳螺SA、SB形成的分支聚为一支;浅肋壳螺SF、SG先聚一起后,再与光壳SD、SC聚为第二分支。

图310个湖北钉螺群体的UPGMA聚类分析
Fig.3UPGMAclusteranalysisoftenO.hupensispopulations

表7 钉螺种群分子变异分析结果
3 讨 论
湖北松滋地区位于长江中下游地区,该地处巫山山系荆门分支余脉和武陵山系石门分支余脉向江汉平原延伸的过渡地带,地势西高东低,是山丘型和平原沟渠交界地带,生态景观多样,不同景观下湖北钉螺表现为不同的螺壳形态,即光壳和肋壳(浅肋和深肋)[16]。尽管如此,线粒体基因COI序列分析表明该地不同螺壳形态的湖北钉螺,并未表现明显的遗传差异[17]。由于单个线粒体基因序列标记的局限性和不能对全基因组遗传多态性进行分析[18],因此,有必要对该地钉螺的遗传多样性进行进一步深入分析。本研究调查了山丘型和垸内沟渠的10个地理群体,螺壳形态分别表现为光壳和肋壳,其中地处2种环境交界地带的新江口镇同时存在光壳和肋壳钉螺,肋壳则表现为过渡类型的浅肋型。为进一步探讨这种螺壳形态演变趋势的遗传本质,本研究选择6个微卫星DNA位点对该地区10个湖北钉螺群体进行遗传多样性分析,探讨松滋地区不同螺壳形态的湖北钉螺群体遗传分化情况。
本研究6个微卫星位点在10个群体中共检测到141个等位基因,平均每个位点检测到23.5个,表明各位点在不同群体间多态性较为明显,并据此计算各群体的多态信息含量(PIC)。PIC是描述群体在微卫星座位上的遗传变异程度的重要指标,PIC 值越大,说明群体内基因一致性差,变异越大,选择潜力也大;反之,PIC 越小,说明群体变异性越小,选择潜力也小[19]。本研究的10个群体中,PIC值为肋壳螺群体SH、SI、Sk大于光壳螺群体SA、SB、SC、SD、SE,光壳螺群体大于浅壳螺群体SF、SG,提示地处垸内沟渠环境下的肋壳钉螺群体的遗传变异较大,光壳钉螺群体遗传变异次之,而浅肋型钉螺群体遗传变异最小。尽管如此,10个群体的期望杂合度均大于0.5,表明该地区钉螺群体遗传分化明显,具有丰富的遗传多样性。
利用F统计量评估该地区遗传多样性表明,各位点的的FST平均值为0.1440,即14.4%的遗传变异来自群体间,85.6%的遗传变异来自群体内个体间,这表明松滋地区湖北钉螺虽然受到地理环境影响而分割成一些亚群,但各群体间遗传分化主要是由于钉螺个体间的遗传变异形成,而Fis、Fit值均为正值,且群体近交系数为30.32%,也表明钉螺群体间基因流不高[20]。AMOVA分析结果显示,10地钉螺个体间遗传变异(86.48%)远远高于群体间遗传变异(7.02%)和群体内遗传变异(6.51%),也说明遗传变异主要来自群体个体间,群体间遗传分化很小,其原因是由于采集点分布于同一水系,遗传分化并不明显[20]。因此,尽管在遗传距离与地理距离表现出一定的相关性,但由于本研究为小尺度下景观群体,因此,群体间遗传分化指数相差并不大,这可能是由于10个景观环境距离并不远,虽然壳型不同,但是都属于湖沼型群体的原因。利用UPGMA法聚类树显示,肋壳中的浅肋和深肋型钉螺群体各自形成明显的聚类后,分别与不同的光壳钉螺群体形成聚类,表明光壳钉螺群体中遗传多样性更为丰富。
综上所述,尽管松滋地区湖北钉螺螺壳形态表现出多种形态,但是微卫星DNA扫描各钉螺群体显示,虽然群体内遗传多样性较高,但遗传变异主要来自于群体个体间,各景观环境的钉螺群体间遗传分化不明显,这与石朝辉等人对庙河地区钉螺不同壳型间基因差异[17]、邱持平等对安徽地区钉螺不同生态环境下CoI基因差异的研究结果相一致[21]。目前推测可能是由于不同的生态环境的影响,如温度、湿度、海拔等,造成壳型有所不同。
参考文献:
[1]Wang L, Utzinger J, Zhou XN. Schistosomiasis control: experiences and lessons from China[J]. Lancet, 2008, 372(9652): 1793-1795. DOI: 10.1016/S0140-6736(08)61358-6
[2]Utzinger J, Zhou XN, Chen MG, et al. Conquering schistosomiasis in China: the long march[J]. Acta Trop, 2005, 96(2-3): 69-96. DOI: 10.1016/j.actatropica.2005.08.004
[3]Li SZ, Wang YX, Yang K, et al. Landscape genetics: the correlation of spatial and genetic distances ofOncomelaniahupensis, the intermediate host snail ofSchistosomajaponicumin mainland China[J]. Geospatial Health, 2009, 3(2): 221-231.
[4]Shi CH, Wilke T, Davis GM, et al. Population genetics, micro-phylogeography, ecology, and susceptibility to schistosome infection of Chinese Oncomelania hupensis hupensis (Gastropoda: Rissooidea: Pomatiopsidae) in the Miao River system[J]. Malacologia, 2002, 44(2): 333-347.
[5]Sun XW, Zhang XF, Zhao YY, et al. Development and application of microsatellite markers in aquatic species[J]. J Fishery Sci China, 2008, 15(4): 689-703. (in Chinese)
孙效文, 张晓锋, 赵莹莹, 等.水产生物微卫星标记技术研究进展及其应用[J]. 中国水产科学, 2008, 15(4): 689-703.
[6]Guo W, Shen ZR. Microsatellite marker and its application in entomology[J]. Biotechnology, 2004,14(2): 60-61. (in Chinese)
国伟,沈佐锐. 微卫星DNA标记技术及其在昆虫学上的应用[J]. 生物技术,2004,14(2):60-61.
[7]Niu AO, Xiong YW. Studies on the genetic variation ofOncomelaniahupensiswith SSR-PCR[J]. J Parasit Biol, 2002, 15(4): 230-233. (in Chinese)
牛安欧,熊衍文. 微卫星锚定PCR研究湖北钉螺的遗传变异[J]. 中国寄生虫病防治杂志,2002,15(4):230-233.
[8]Zhou YB, Zhao GM, Wei JG, et al. Genetic diversity in 19 Chinese populations ofOncomelaniahupensis(Gastropoda: Rissooidea) detected by simple sequence repeat-anchored polymerase chain reaction amplification[J]. Chin J Epidemiol, 2007, 28(9): 859-862. (in Chinese)
周艺彪,赵根明,韦建国,等. 微卫星锚定PCR分析19个湖北钉螺种群之间的遗传变异关系[J]. 中华流行病学杂志,2007,28(9):859-862.
[9]Guo JT, Zhou YB, Zhang ZJ, et al. Simple sequence repeat variation and small-scale spatial autocorrelatian analysis on smooth-shell populations ofOncomelaniahupensisin Sichuan province[J]. Chin J Epidemiol, 2009, 30 (5): 497-501. (in Chinese)
郭俊涛, 周艺彪, 张志杰, 等. 四川光壳钉螺微卫星遗传变异的小尺度空间自相关分析[J]. 中华流行病学杂志, 2009, 30 (5): 497-501.
[10]Ma L, Li SZ, Yang P, et al. Comparison of five different methods of extracting genomic DNA fromOncomelaniahupensis[J]. J Pathog Biol, 2011, 6(2): 129-132. (in Chinese)
马琳,李石柱,杨频,等. 湖北钉螺基因组5种抽提方法的比较[J]. 中国病原生物学杂志,2011,6(2):129-132.
[11]Li SZ, Wang YX, Ma YJ, et al. Isolation and characterization of polymorphic microsatellite markers ofOncomelaniahupensis[J]. Chin J Schistosomiasis Ctrl, 2010, 22(2): 122-125. (in Chinese)
李石柱,王艺秀,马雅军,等. 湖北钉螺多态微卫星DNA位点筛选和特征初步分析[J].中国血吸虫病防治杂志,2010,22(2):122-125.
[12]Zhang L, Li SZ, Wang Q, et al. Isolation and characterization of 15 new microsatellite markers inOncomelaniahupensis, the snail intermediate host ofSchistosomajaponicumin Mainland China[J]. Int J Mol Sci, 2012, 13(5): 5844-5850. DOI: 10.3390/ijms13055844
[13]Excoffier L, Laval G, Schneider S. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evol Bioinformat Online, 2005, 1: 47-50.
[14]Stefan Van Dongen T, Winnepenninckx B. Multiple UPGMA and neighbor-joining trees and the performance of some computer packages[J]. Mol Biol Evol, 1996, 13(2): 309-313.
[15]Khan HA, Arif IA, Bahkali AH, et al. Bayesian, maximum parsimony and UPGMA models for inferring the phylogenies of antelopes using mitochondrial markers[J]. Evol Bioinformat Online, 2008, 4: 263-270.
[16]Yang JY. Study on technical characteristics and regional applicability of rice Green control in Songzi City[J]. Hubei Plant Protect, 2010,(5):7-9. (in Chinese)
杨继云.松滋市水稻绿色防控技术特点与区域适用性研究[J]. 湖北植保,2010, (5): 7-9.
[17]Shi CH, Qiu CP, Xia MY, et al. Preliminary study on cytochrome C oxidase 1 gene ofOncomelaniahupensisfrom Miao River Area in Hubei Province[J]. Chin J Parasitol Parasit Dis, 2010, 19(1): 41-44. (in Chinese)
石朝辉, 邱持平, 夏明仪, 等. 湖北省庙河地区钉螺细胞色素C氧化酶1基因差异的研究[J].中国寄生虫学与寄生虫病杂志, 2010, 19(1):41-44.
[18]Hu Y, Li XM, Lin R, et al. Genetic variation of two mitochondrial DNA molecules from three isolates ofOncomelaniahupensis[J]. Chin J Parasitol Parasit Dis, 2007, 25(6): 474-477. (in Chinese)
胡缨, 黎学铭, 林睿, 等. 三地钉螺线粒体 DNA 两个分子的遗传变异研究[J]. 中国寄生虫学与寄生虫病杂志, 2007, 25(6):474-477.
[19]Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32(3): 314-331.
[20]Weir BS, Cockerham CC. Estimating F-statistics for the analysis of population structure[J]. Evolution, 1984: 1358-1370.
[21]Qiu CP, Chris S, Xia MY, et al. Study on genomic diversity among different isolates ofSchistosomajaponicumin China[J]. Chin J Parasitol Parasit Dis, 2002, 20(2): 79-82. (in Chinese)
邱持平, Chris S, 夏明仪, 等. 中国日本血吸虫地域株基因差异的研究[J]. 中国寄生虫学与寄生虫病杂志, 2002, 20(2):79-82.
