水貂肠炎病毒NS1基因进化分析
2014-03-24张海玲张蕾胡博白雪赵建军刘昊徐淑娟苗立光冯卓闫喜军
张海玲,张蕾,胡博,白雪,赵建军,刘昊,徐淑娟,苗立光,冯卓,闫喜军
(中国农业科学院特产研究所,长春 130112)
水貂肠炎病毒(Mink enteritis virus,MEV)是猫泛白细胞症病毒(FPV)的变异体,可在肠系膜淋巴结和隐窝上皮细胞内高度复制,是以肠黏膜脱落和腹泻为特征的急性、烈性、高度接触性传染病的病原体[1,2]。在自然条件下,不同品种和不同年龄的水貂均有感染性,但幼貂感染性极强。MEV引起的水貂病毒性肠炎最早于1949年由Schofield报道,1952年由Wills分离、鉴定并命名为水貂肠炎病毒[3,4]。在我国,最早于1981年,由姜廷秀报道。目前,该病蔓延全国。随着特种养殖业的发展,病毒性肠炎已经成为危害水貂养殖的三大疫病之一,给养貂业带来巨大的经济损失[5,6]。
水貂肠炎病毒为单链线性DNA病毒,长约5000bp[7,8],主要编码VP1、VP2、NS1、NS2 4种蛋白。其中,VP1、VP2为结构蛋白;NS1、NS2为非结构蛋白。NS1蛋白对MEV的复制具有重要调节作用。有报道称,NS1蛋白个别氨基酸的改变可能会影响到细小病毒的细胞嗜性。目前,国内尚无对MEV NS1基因的检测报导。本研究通过对国内4个地区的样品中MEV NS1基因的克隆测序,初步了解MEV NS1基因的变异特性,为以后对MEV NS1基因的研究及MEV的感染控制提供流行病学依据。
1 材料与方法
1.1 样品
将辽宁省、吉林省、山东省、河北省采集的发病致死水貂肠道组织低温冷冻处理后送至实验室,无菌加入10倍PBS,研磨,离心取上清液,冷藏待检。样品编号及详细信息见表1。
表111份水貂肠炎病毒阳性样品采集信息
Table1Informationsof11positivesamplescollectedfromminkenteritisvirus
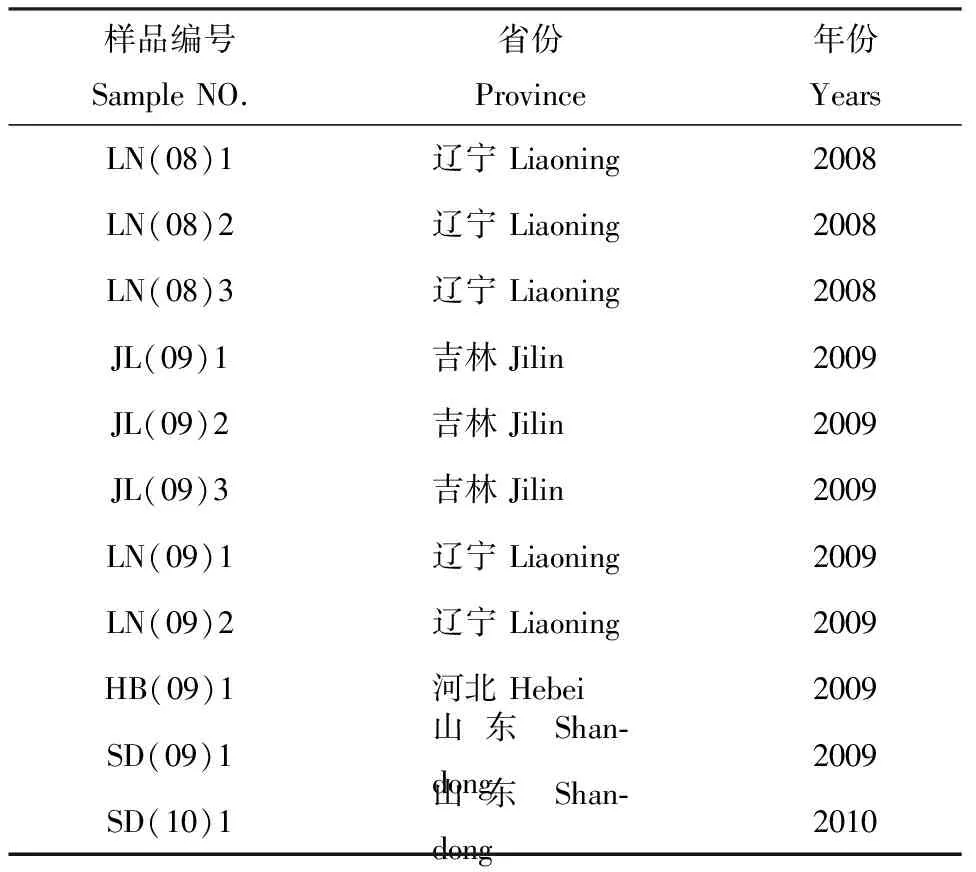
样品编号SampleNO.省份Province年份YearsLN(08)1辽宁Liaoning2008LN(08)2辽宁Liaoning2008LN(08)3辽宁Liaoning2008JL(09)1吉林Jilin2009JL(09)2吉林Jilin2009JL(09)3吉林Jilin2009LN(09)1辽宁Liaoning2009LN(09)2辽宁Liaoning2009HB(09)1河北Hebei2009SD(09)1山东Shan⁃dong2009SD(10)1山东Shan⁃dong2010
1.2 质粒与菌种
大肠杆菌DH5a感受态细胞和pMD18-TVector(大连宝生物工程有限公司)。
1.3 主要试剂
MiniBEST Viral DNA Extraction Kit Ver.3.0试剂盒、PCR 扩增试剂、Marker DL2000(TaKaRa公司);DNA凝胶回收试剂盒、质粒DNA小量提取试剂盒(Bioer公司)。
1.4 引物的设计与合成
参照GenBank MEV-B株序列(FJ592174)设计上、下游引物NS-1/NS-2、NS-3/NS-4,分2段测序,引物序列见表2。
表2水貂肠炎病毒NS1基因序列扩增引物
Table2AmplificationprimersofMinkenteritisparvovirusNS1gene

引物编号PrimerNo序列(5′-3′)Sequences退火温度(℃)ReturntemperatureNS-1CCATAGACCGTTACTGACATTCG53NS-2TACAGCTTGTGCTATGGCTTGAGNS-3AAATGATGGCACAACCAGGAGG56NS-4ACCTGGAGGCACAAGTCCTATAA
1.5 核酸的提取及PCR扩增
按照试剂盒提供的操作手册从组织样品研磨上清液中提取DNA。用PCR扩增目的片段。PCR反应体系:10×LATaqbuffer 5μL、dNTP(2.5mmol/L)4μL、引物NS-1/NS-2和NS-3/NS-4(50pmol/L)各1μL、模板DNA 1μL、LATaq酶0.25μL,加去离子水至50μL。PCR反应条件:94℃ 5min;94℃ 45min,54℃ 1min ,72℃ 2min,30个循环,72℃延伸7min。1.0%琼脂糖凝胶电泳鉴定PCR产物。
1.6 重组质粒的克隆与鉴定
取4.5μL纯化产物与0.5μL pMD18-T载体(50ng/μL)进行连接反应,将连接产物转化DH5α感受态细胞,涂布于Ampicillin抗性平板,37℃过夜培养后,对单个菌落进行PCR鉴定。阳性菌落接种5mL含Ampicillin的LB营养液中培养增殖,提取质粒。用BamHI和HandIII 双酶切鉴定后,送TaRaKa公司测序。
1.7 序列比对、同源性及系统发育分析
将测得的上、下2段序列用DNAstar分子生物学软件进行拼接,并用DNAstar与GenBank公布的其他细小病毒序列进行比对同源性及系统发育分析,引用序列见表3。
表3引用参考序列
Table3Referencesequencecitedinthisstudy

编号NO毒株StrainsGenBank登录号GenBankAccessionNo基因型Genotype国家Country1CPV-bM38245CPV-2USA2CPV-13EU659118CPV-2aUSA3Cpv-2aAJ564427CPV-2aIndia4Fpv-CU-4M38246FPVUSB5MEVBFJ592174MEVChina6cpv-15AY787926CPVUSA7fpv-TU4AB000067FPVJapan8fpv-TU8AB000069FPVJapan9MEVAbashiriD00765MEVJapan10CAENFRA3009FN6695071FPVGermany
2 结果与分析
2.1 PCR检测及NS1基因序列分析
在送检的32份样品中检测出21份阳性样品,在检测的几个地区中均有阳性样品分布。每个地区选取1~3份时间、地点、临床症状有差异的样品共计11份进行NS1基因的克隆和测序,结果显示,NS1基因全长2007bp,编码668个氨基酸。核苷酸序列和氨基酸序列同源性分别为98.8%~100.0%和98.5%~100%;本次测序序列和国内毒株MEVB基因的核苷酸和推导氨基酸序列同源性为98.7%~99.9%和98.7%~99.9%;与国外毒株MEVAbashiri基因的核苷酸和推导氨基酸序列同源性为99.0%~99.5%和98.7%~99.1%。通过对11株肉食兽细小病毒株和已知的9株参考毒株NS1基因氨基酸序列比对,发现有34个氨基酸位点存在变异,见表4。
表4NS1基因变异氨基酸位点
Table4AminoacidvariationsitesofNS1genes.

病毒株StrainsMEVNS1氨基酸变异位点VariationsitesofMEVNS1aminoacid10233340438892128194222227247248249282286350进化分支EvolutionarybranchJL(09)1V----------QI----1JL(09)2V----------QI----1LN(08)1V----------QI----1LN(08)2V--R-R----DQI----1HB(09)1V----------QI----1LN(08)3V-------R--QI----1SD(09)1--G---L--I-------2SD(10)1-----------------2JL(09)3-----------------2LN(09)2-------R---------2LN(09)1V----------QI----3Cpv-13V----------QI----3Cpv-15V----------QI----3Cpv-bV----------QI----3Cpv-2aV----------QIGGQ-3MEVAbashiriV---H-----------N4
续表4
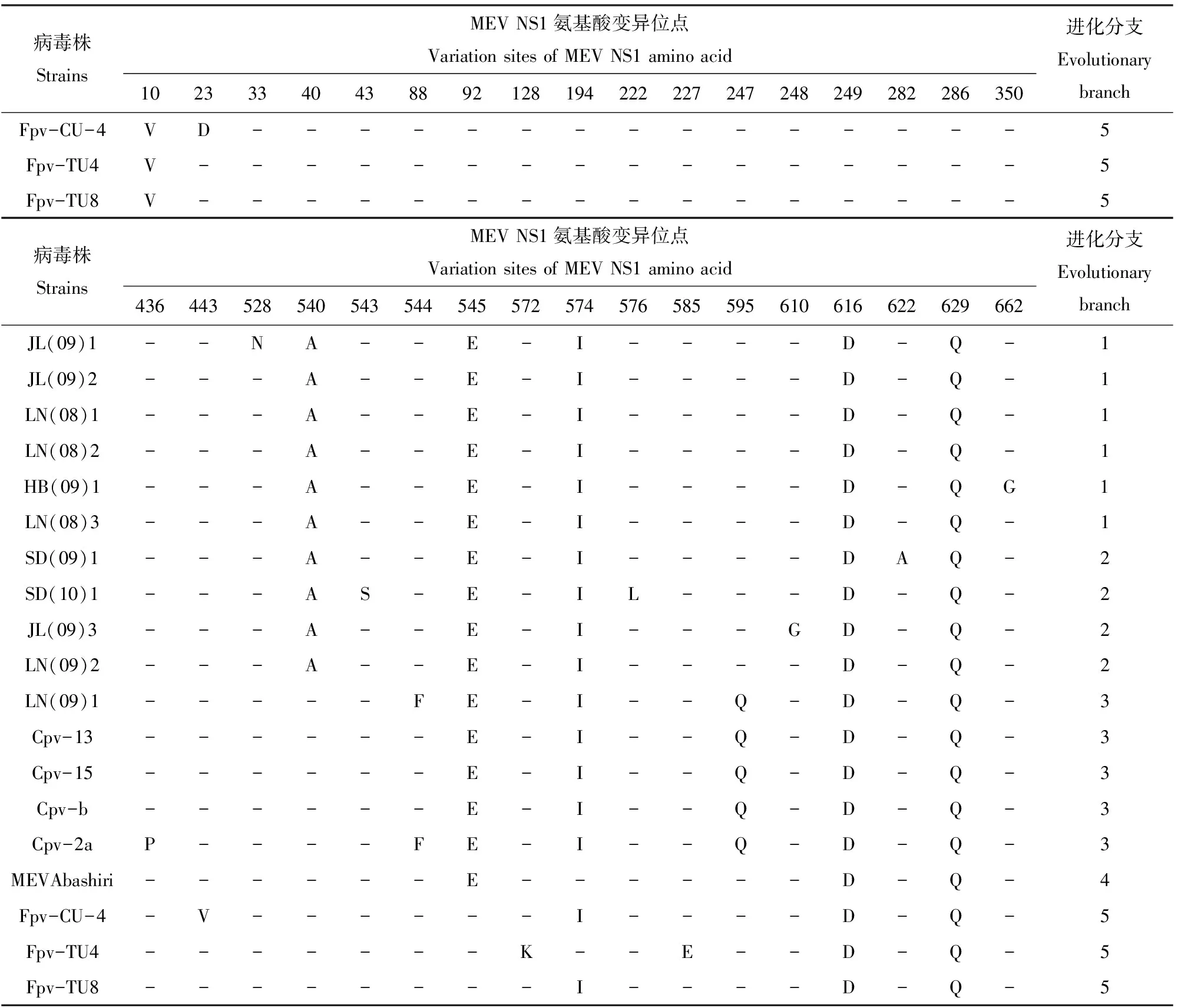
病毒株StrainsMEVNS1氨基酸变异位点VariationsitesofMEVNS1aminoacid10233340438892128194222227247248249282286350进化分支EvolutionarybranchFpv-CU-4VD---------------5Fpv-TU4V----------------5Fpv-TU8V----------------5病毒株StrainsMEVNS1氨基酸变异位点VariationsitesofMEVNS1aminoacid436443528540543544545572574576585595610616622629662进化分支EvolutionarybranchJL(09)1--NA--E-I----D-Q-1JL(09)2---A--E-I----D-Q-1LN(08)1---A--E-I----D-Q-1LN(08)2---A--E-I----D-Q-1HB(09)1---A--E-I----D-QG1LN(08)3---A--E-I----D-Q-1SD(09)1---A--E-I----DAQ-2SD(10)1---AS-E-IL---D-Q-2JL(09)3---A--E-I---GD-Q-2LN(09)2---A--E-I----D-Q-2LN(09)1-----FE-I--Q-D-Q-3Cpv-13------E-I--Q-D-Q-3Cpv-15------E-I--Q-D-Q-3Cpv-b------E-I--Q-D-Q-3Cpv-2aP----FE-I--Q-D-Q-3MEVAbashiri------E------D-Q-4Fpv-CU-4-V------I----D-Q-5Fpv-TU4-------K--E--D-Q-5Fpv-TU8--------I----D-Q-5
注:MEVB毒株(GenBank登录号:FJ592174)NS1基因氨基酸序列作为参考序列;“-”.与参考序列相同的氨基酸位点。
Note:Amino acid sequence of the strain MEVB NS1 gene(GenBank accession number:FJ592174)as the reference sequence;“-”.represents the same amino acid position with reference sequence.
2.2 NS1基因的系统发育分析
本次检测的11个样本主要分为3个分支,其中DL1与CPV-2a在同一分支(图1)。
3 讨论
水貂细小病毒、犬细小病毒(CPV)和猫细小病毒进化关系密切,其中,水貂细小病毒和犬细小病毒都是FPV的突变体[9]。三者在感染时都利用转铁蛋白受体[10],但是,只有CPV能够感染猫肾和犬肾细胞,而且FPV和CPV二者宿主范围的不同主要是由于衣壳蛋白VP2的变异及VP2和犬或猫转铁蛋白受体(TfR)的相互作用[11,12]。
NS1蛋白对细小病毒复制具有重要的调节作用,MEV的NS1基因含有1个ORF,全长2 007bp,共编码668个氨基酸。在细小病毒NS1蛋白中含有1个长约150个氨基酸的保守区域,这段保守区域从389位氨基酸开始,并含有391GKRN394保守序列,在不同细小病毒中,这一区域的氨基酸相似性远远高于其他区域。在这段区域中含有与ATPase或GTPase相关的ATP或GTP结合位点,并具有解旋活性,是病毒DNA复制所必需的。本实验中所有测序序列均含有391GKRN394保守序列,但JL(09)1株在保守区域内发生了改变,在528位的D(Asp)到N(Asn)的突变,该处改变是否对NS1的抗原性产生影响,有待于进一步的研究。

MEV最早报道于1949年,CPV最早报道于1978年,虽然报道的时间相差很大,但现在还不能说明他们之间的关系及进化顺序。关于细小病毒的进化推测最多的就是MEV和CPV由FPLV进化而来。但CPV与MEV之间的亲缘关系较远。本试验中检测的序列独自分为2支。其中DL1与CPV在同一进化分支,其与CPV的氨基酸同源性高达99.7%,氨基酸位点相对于CPV未发生特异的改变,其是否显示了MEV与CPV的NS1基因发生重组,还有待进一步证实。
[1]UTTENTHAL A,LARSEN S,LUND E,et al.Analysis of experimental mink enteritis virus infection in mink:in situ hybridization,serology,and histopathology[J].Journal of Virology,1990,64(6):2768-2779.
[2]PARK G S,BEST S M,BLOOM M E.Two mink parvoviruses use different cellular receptors for entry into CRFK cells[J].Virology,2005,340(1):1-9.
[3]WANG J,CHENG S,YI L,et al.Detection of mink enteritis virus by loop-mediated isothermal amplification(LAMP)[J].Journal of Virological Methods,2013,187(2):401-405.
[4]WILLS C G.Notes on infectious enteritis of MINK and its relationship to feline enteritis[J].Canadian Journal of Comparative Medicine and Veterinary Science,1952,16(12):419-420.
[5]HUNDT B,BEST C,SCHLAWIN N,et al.Establishment of a mink enteritis vaccine production process in stirred-tank reactor and Wave Bioreactor microcarrier culture in 1-10 L scale[J].Vaccine,2007,25(20):3987-3995.
[6]ZUO J,RAO J,XU H,et al.Analysis of the vp2 gene sequence of a new mutated mink enteritis parvovirus strain in PR China[J].Virology Journal,2010,(7):124.
[7]DECARO N,BUONAVOGLIA C.Canine parvovirus--a review of epidemiological and diagnostic aspects,with emphasis on type 2c[J].Veterinary Microbiology,2012,155(1):1-12.
[8]LANGEVELD J P,KAMSTRUP S,UTTENTHAL A,et al.Full protection in mink against mink enteritis virus with new Generation canine parvovirus vaccines based on synthetic peptide or recombinant protein[J].Vaccine,1995,13(11):1033-1037.
[9]STEINEL A,MUNSON L,VAN VUUREN M,et al.Genetic characterization of feline parvovirus sequences from various carnivores[J].The Journal of General Virology,2000,81(2):345-350.
[10]PARK G S,BEST S M,BLOOM M E.Two mink parvoviruses use different cellular receptors for entry into CRFK cells[J].Virology,2005,340(1):1-9.
[11]GOVINDASAMY L,HUEFFER K,PARRISH C R,et al.Structures of host range-controlling regions of the capsids of canine and feline parvoviruses and mutants[J].Journal of Virology,2003,77(22):12 211-12 221.
[12]HUEFFER K,GOVINDASAMY L,AGBANDJE-MCKENNA M,et al.Combinations of two capsid regions controlling canine host range determine canine transferrin receptor binding by canine and feline parvoviruses[J].Journal of Virology,2003,77(18):10 099-10 105.
