基于线粒体16S rDNA和COΙ基因探讨中国近海黄姑鱼类的分子系统进化关系*
2014-03-08张玉荣徐冬冬毛国民
辛 俭 张玉荣 徐冬冬 楼 宝 詹 炜 毛国民
(浙江省海洋水产研究所 浙江省海水增养殖重点实验室 舟山 316100)
黄姑鱼属(Nibea)鱼类是世界重要的海洋经济鱼类之一,主要分布于印度洋和太平洋海域。该属隶属于鲈形目(Perciformes)石首鱼科(Sciaenidae),首先由Jordan和 Thompson于 1911年在石首鱼属(Sciaena)内建立了黄姑鱼亚属(Nibea)(Jordanet al,1911),后由Trewavas(1977)将其提升为属(Trewavas,1977)。黄姑鱼属鱼类也是我国重要的海洋渔业资源,据文献报道,分布在我国的黄姑鱼属鱼类包括黄姑鱼(Nibea albifora)、浅色黄姑鱼(N.coibor)、半花黄姑鱼(N.semifascitata)、双棘黄姑鱼(N.diacanthus)、日本黄姑鱼(N.japonica)、状黄姑鱼N.miichthioides和尖头黄姑鱼(N.acuta)共7种(朱元鼎等,1963a,b;成庆泰等,1987)。此后,不断有学者对黄姑鱼属的分类进行修订:将双棘黄姑鱼改为双棘原黄姑鱼(Protonibea diacanthus),属原黄姑鱼属(Protonibea);日本黄姑鱼改为日本白姑鱼(Argyrosomus japonicus),状黄姑鱼更改为厦门白姑鱼(A.amoyensis),这两种鱼均属白姑鱼属(Argyrosomus);尖头黄姑鱼改为尖头黄鳍牙(Chrysochir aureus),属金鳍属(Chrysochir)(沈世杰等,1993;孟庆闻等,1995;庄平等,2006;伍汉霖等,2012)。尽管黄姑鱼属鱼类的形态分类已经得到学者的认同,但黄姑鱼类的同种多名、异名现象仍然较多,而且国内外对黄姑鱼类的分子系统关系的研究报道较少。
随着现代分子标记技术的发展,通过获得物种特定遗传标记的大量数据,构建分子系统树,可以对传统分类提供重要佐证以及补充或修正(Liuet al,2004;李琪等,2010)。线粒体DNA的分子结构简单,而且具有进化速率快、几乎不发生重组、严格母系遗传等优点,成为开展分子系统学研究的重要工具。目前,常用于鱼类分子系统分析的线粒体基因有:COI基因、16S rDNA、Cytb基因等(郭新红等,2004;程国宝等,2012 ;Zhanget al,2012)。利用线粒体DNA探讨石首鱼科鱼类的系统分化国内外已有一些报道(蒙子宁等,2004;童馨等,2007;Lakraet al,2009;柳淑芳等 2010;张永等,2011;马春艳等,2012),主要是探讨了石首鱼科的分子系统进化,但对黄姑鱼属的系统分类问题却鲜有涉及。
本研究测定了黄姑鱼属4个种30个个体的线粒体 16S rDNA和 COI基因的部分序列,并结合Genebank下载的其他石首鱼科鱼类相应的基因序列进行比较分析,构建分子系统树,以此探讨黄姑鱼属鱼类的分子鉴定及其系统进化关系。
1 材料与方法
1.1 试验材料
本研究所用的黄姑鱼(HG)、浅色黄姑鱼(QH)、日本黄姑鱼(RH)、状黄姑鱼(MH)等4种黄姑鱼类共计30尾,取样地点和样本数见表1。样品根据朱元鼎等(1963a,b)和成庆泰等(1987)的报道进行形态学鉴定,剪取鳍条保存于75%的酒精中带回实验室提取总 DNA,DNA提取方法采用试剂盒提取(北京天根)。

表1 四种黄姑鱼类的分类信息及采集地点Tab.1 Information of taxonomy and sampled localities for four Nibea fish species
1.2 16S rDNA和COΙ基因片段的扩增
实验用的16S rDNA和COI片段扩增的引物由上海生工公司合成。16S rDNA的序列为16S-F1-5′CGCCTGTTTATCAAAAACAT 3′和16S-F2 5′CCGGTCTGAACTCAGATCACGT 3′(徐冬冬等,2010);COI基因片段引物序列为:COI-F1 5′-TCAACCAACCACAAAGACATTGGCAC-3′和 COI-F2 5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′(Wardet al,2005;Lakraet al,2009)。PCR扩增采用50µL反应体系:1×PCR缓冲液,0.2mmol/L dNTP,2mmol/L Mg2+,1μmol/L正反向引物,2 UTaqDNA聚合酶(Takara),DNA模板约400ng。反应程序为:预变性94°C,5min;35个循环,每个循环包括94°C变性30s;退火(16S rDNA 56°C;COI 52°C)30s;72°C延伸lmin;最后72°C再延伸10min,4°C保存。PCR产物经电泳、割胶回收、产物纯化后获得目的片段,纯化后的目的片段采用ABI3730进行测序。
1.3 序列下载
从GenBank下载13种石首鱼科鱼类16S rDNA或COI基因序列,与本研究所检测的4种黄姑鱼类的基因同源序列一起分析。所分析物种的16S rDNA和COI基因序列及其相关信息如表2。

表2 实验用的石首鱼科鱼类的16S rDNA和COΙ基因信息Tab.2 Information of 16S rDNA and COI gene in Sciaenidae species
1.4 序列分析
采用NCBI的Blastn工具(http://www.ncbi.nlm.nih.gov/blast)对所获得的测序结果进行序列比对和分析,验证所得序列是否为目的片段。应用Clustal X 1.83软件对获得的16S rDNA和COI基因序列进行聚类和比对,并辅以手工校正(Thompsonet al,1997)。采用DNASP 5.0软件计算序列的碱基组成、转换(Transition)和颠换(Transversion)比例等参数。利用Mega 4.0 软件中的Kitumra-2-Parameter-Distance双参数模型计算遗传距离,并利用 Neibour–joining(NJ)方法构建分子系统进化树(Tamuraet al,2007)。
2 结果与分析
2.1 四种黄姑鱼类16S rDNA和COΙ基因序列特征
经PCR扩增、回收、测序等步骤得到4种黄姑鱼类的16S rDNA部分序列,采用Clustal X 1.83对所得序列进行排序和聚类,保留同源序列,长度为553—569bp,编码 184—189个氨基酸,其间存在插入/缺失位点,主要集中在137bp、192—205bp和550—551bp。四种黄姑鱼类的16S rDNA基因序列的平均碱基组成为A=22.3%,T=29.2%,C=21.7%,G=26.8%,其中(C+G)的含量为 48.5%,与张永等(2011)计算的13种石首鱼科鱼类的GC平均含量略有差异(47.3%),而与蒙子宁等(2004)统计的 8种石首鱼科鱼类的 GC平均含量相似(48.3%)。四种黄姑鱼类的 COI基因序列经排序和剪辑后得到637bp的同源序列,编码212个氨基酸,其间没有插入/缺失位点。四种黄姑鱼类的平均碱基组成为 A=22.4%,T=27.0%,C=31.6%,G=19.0%。其(C+G)的含量为50.6%,比柳淑芳等(2010)统计的30种石首鱼科鱼类的GC平均含量略高48.3%。
4种黄姑鱼类的16S rDNA序列中检测到的多态位点数为72,约占总序列的12.91%,简约信息位点数72个,其中,转换位点24个,颠换位点9个,转换颠换比(Ts/Tv)为2.6;COI基因检测到131个多态信息位点,约占总序列的20.56%,简约信息位点128个,包括转换位点45个,颠换位点23个,单个突变位点3个,转换颠换比2.0。由此可见,16S rDNA基因在所研究的4种黄姑鱼类较COI基因保守,这在其他海洋动物的遗传分析中也得到相似结果(刘君等,2009;Lakraet al,2009;Cuiet al,2010 ;Fenget al,2011)。这可能是由于线粒体基因组中不同区域的核苷酸突变速率不同或不同基因片段受到的选择压力不同,造成了不同基因片段的核苷酸变异存在差异(王巍等,2009;Teletchea,2009)。
2.2 四种黄姑鱼类的种内和种间遗传距离
根据4种黄姑鱼类的16S rDNA和COI基因序列变异采用双参数模型Kimura-2-parameter计算种内和种间遗传距离(表3,表4)。基于16S rDNA计算的种内的平均遗传距离为0.000—0.002,平均的种间遗传距离为 0.064;浅色黄姑鱼与状黄姑鱼的种间遗传距离(0.098)最大,黄姑鱼和浅色黄姑鱼的种间遗传距离(0.024)最小。基于COI基因计算的4种黄姑鱼类平均的种内遗传距离为 0.000—0.005,平均的种间遗传距离为 0.121;浅色黄姑鱼和日本黄姑鱼的种间遗传距离(0.192)最大;状黄姑鱼和日本黄姑鱼的种间遗传距离(0.002)最小。
线粒体DNA的COI基因已成为动物DNA条形码研究的首选对象(Hebertet al,2003;Wardet al,2005;李琪等,2010 ;Malakaret al,2012)。应用COI基因进行石首鱼科鱼类的物种鉴定已有报道。如Lakra等(2009)利用COI基因分析印度洋7种石首鱼科鱼类的种间遗传距离为 0.025—0.258;柳淑芳等(2010)分析了30种石首鱼科鱼类的种内遗传距离平均值为 0.006,种间为 0.210,种间遗传距离是种内的35倍;以上研究均表明COI基因可以作为上述石首鱼科鱼类物种鉴定的分子标记。但是,COI基因DNA条形码技术在应用过程中也引起很大的争议。Vences等(2005)通过对马达加斯加蛙类的研究,发现两栖类动物适合的条形编码基因是 16S rRNA基因。Elias等(2007)利用COI序列只能准确鉴定出77%的蝴蝶种类。
本研究采用 COI基因序列计算 4种黄姑鱼类的种内遗传距离为0.000—0.005,种间遗传距离为0.002—0.192,其中,浅色黄姑鱼与日本黄姑鱼、状黄姑鱼之间的遗传距离为 0.115—0.192,处于种间遗传分化的水平;而日本黄姑鱼和状黄姑鱼的种间遗传距离仅为 0.002,尚未达到 Hebert等(2003)所推荐的物种鉴定的最小种间遗传距离(0.02)。柴学军等(2009)比较日本黄姑鱼和状黄姑鱼的 cytb基因序列也发现二者具有很高的同源性(99.82%)。利用16S rDNA序列计算的日本黄姑鱼和状黄姑鱼的种间遗传距离为0.026。因此,16S rDNA可以用于日本黄姑鱼和状黄姑鱼的分子鉴定(Venceset al,2005);而COI基因能否作为这两种鱼分子鉴定的有效标记值得商榷。一般而言,采用单基因作为 DNA条形码进行物种鉴定可能存在一定的局限性,同时使用多个分子标记进行分析会增加结果的可靠性(郭新红等,2004 ;Eliaset al,2007 ;Zhanget al,2012)。
2.3 分子系统树分析
基于 Kimura双参数法分别根据 16S rDNA和COI基因序列计算了石首鱼科鱼类的遗传距离(表3,表4),并应用邻位相连法(Neighbor-joining,NJ)构建了分子系统进化树(图1,图2),枝上的数值是 1000次重复抽样检验的置信度值。由16S rDNA片段构建的NJ树来看,黄姑鱼与浅色黄姑鱼聚为一枝,状黄姑鱼首先与无味白姑鱼(A.inodorus)聚类,然后与日本黄姑鱼聚为一枝,具有很高的支持率。由COI基因片段构建的 NJ树来看,黄姑鱼与浅色黄姑鱼首先聚为一枝,日本黄姑鱼和状黄姑鱼聚为一枝。由拓扑结构来看,日本黄姑鱼和状黄姑鱼与黄姑鱼属代表种黄姑鱼和浅色黄姑鱼的亲缘关系较远。将日本黄姑鱼与状黄姑鱼以及黄姑鱼和浅色黄姑鱼划分为两个类群,基于16S rDNA和COI基因序列计算出两者间的遗传距离为0.102和0.180,表明日本黄姑鱼和状黄姑鱼与黄姑鱼属间存在较大的遗传分化,它们之间的亲缘关系已经超过了同属内的遗传分化水平(表3,表4)。因此,研究结果支持黄姑鱼属不包括日本黄姑鱼和状黄姑鱼的分类学观点。

表3 基于16S rDNA的石首鱼科鱼类的种内和种间遗传距离Tab.3 Intraspecific and interspecific genetic distance among Sciaenidae species based on 16S rDNA

表4 基于COΙ基因的石首鱼科鱼类的种内和种间遗传距离Tab.4 Intraspefic and interspefic genetic distance among Sciaenidae species based on COI gene

图1 基于16S rDNA构建的石首鱼科鱼类分子系统树(NJ法)Fig.1 Molecular phylogenetic tree of Sciaenidae fishes based on the fragment of the 16S rDNA(NJ method)
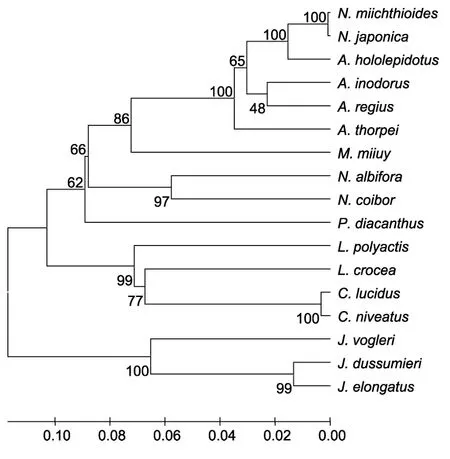
图2 基于COI构建的石首鱼科鱼类分子系统树(NJ法)Fig.2 Molecular phylogenetic tree of Sciaenidae fishes based on the fragment of the COI gene(NJ method)
综上,本研究结果从分子系统学角度为黄姑鱼属的重新修订提供科学依据,支持日本黄姑鱼和状黄姑鱼属白姑鱼属的分类学观点;利用16S rDNA可以对4种黄姑鱼类进行分子鉴定,但是,基于COI基因计算的日本黄姑鱼和状黄姑鱼的种间遗传距离尚未达到种间分化水平,应用该基因片段进行这两种鱼的分子鉴定值得商榷。然而,本研究所获得黄姑鱼属的种类及数量有限,希望以后能采集到尽可能多的种类及样品,利用多种分子标记从属间、种间以及种内不同水平进行黄姑鱼属的系统发育研究,进一步阐明中国石首鱼科鱼类的物种多样性及其进化关系,深化对石首鱼科鱼类的进化和中国近海海洋鱼类区系的认识。
马春艳,马凌波,倪 勇等,2012.基于RAG基因的中国近海13种石首鱼科鱼类系统进化关系.水产学报,36(1):9—16
王 巍,朱 华,胡红霞等,2009.五种鲟鱼线粒体控制区异质性和系统发育分析.动物学研究,30(5):487—496
成庆泰,郑葆珊,1987.中国鱼类系统检索.北京:科学出版社,317—324
朱元鼎,张春霖,成庆泰,1963a.石首鱼科.东海鱼类志.北京:科学出版社,268—293
朱元鼎,罗云林,伍汉霖,1963b.中国石首鱼类分类系统的研究和新属新种的叙述.上海 :上海科学技术出版社,47—56
伍汉霖,邵广诏,赖春福等,2012.拉汉世界鱼类系统名典.北京:北京出版社,105—535
庄 平,王幼槐,李圣法等,2006.长江口鱼类.上海:上海科技出版社,348—349
刘 君,李 琪,孔令锋等,2009.基于线粒体 COI的 DNA条形码技术在贻贝科种类鉴定中的应用.水生生物学报,35(5):874—881
李 琪,邹山梅,郑小东等,2010.DNA条形码及其在海洋生物中的应用.中国海洋大学学报(自然科学版),40(8):43—47
沈世杰,1993.台湾鱼类志.台湾 :国立台湾大学动物学系出版,383—392.
孟庆闻,陈力行,1995.石首鱼科.鱼类分类学.北京:中国农业出版社,713—728
柳淑芳,陈亮亮,戴芳群等,2010.基于线粒体 COI基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用.海洋与湖沼,41(2):223—232
柴学军,李鹏飞,吴祖杰,2009.日本黄姑鱼和状黄姑鱼细胞色素 b基因序列的比较分析.现代渔业信息,24(4):18—20
徐冬冬,尤 锋,楼 宝等,2010.8种鲆鲽鱼种间遗传距离与杂交亲和性的相关性分析.水产学报,34(2):178—184
郭新红,刘少军,刘 巧等,2004.鱼类线粒体DNA研究新进展.遗传学报,31(9):983—1000
程国宝,李三磊,徐冬冬等,2012.梭鱼和鲻鱼线粒体 16S rRNA和 COI基因片段的比较分析.浙江海洋学院学报:自然科学版,31(2):103—106
童 馨,杜 博,喻达辉等,2007.浅色黄姑鱼线粒体16S rRNA基因片段序列特征分析.海洋水产研究,28(3):85—91
蒙子宁,庄志猛,丁少雄等,2004.中国近海 8种石首鱼类的线粒体16S rRNA基因序列变异及其分子系统进化.自然科学进展,14(5):514—521
Cui Z,Liu Y,Liu Jet al,2010.Molecular identification ofPampusfishes(Perciformes,Stromateidae).Ichthyological Research,57(1):32—39
Elias M,Hill R I,Willmott K Ret al,2007.Limited performance of DNA barcoding in a diverse community of tropical butterflies.Proceedings of the Royal Society B:Biological Sciences,274(1627):2881—2889
Feng Y W,Li Q,Kong L Fet al,2011.DNA barcoding and phylogenetic analysis of Pectinidae(Mollusca:Bivalvia)based on mitochondrial COI and 16S rRNA genes.Molecular Biology Reports,38:291—299
Hebert,Paul D N,Sujeevan Ratnasingham,et al,2003.Barcoding animal life:cytochrome c oxidase subunit I divergences among closely related species.Proceedings of the Royal Society of London.Series B:Biological Sciences,270(S1):S96—S99
Jordan D S,Thompson W F,et al,1911.A review of the sciaenoid fishes of Japan.Proceedings of the United States National Museum,39(1787):241—261
Lakra W S,Goswami M,Gopalakrishnan A,2009.Molecular identification and phylogenetic relationships of seven Indian Sciaenids(Pisces:Perciformes,Sciaenidae)based on 16S rRNA and cytochrome c oxidase subunit I mitochondrial genes.Molecular Biology Reports,36(5):831—839
Liu Z J,Cordes J F,2004.DNA marker technologies and their applications in aquaculture genetics.Aquaculture,238(1):1—37
Malakar A K,Lakra W S,Goswami Met al,2012.Molecular identification of threeOmpokspecies using mitochondrial COI gene.Mitochondrial DNA,23(1):20—24
Teletchea F,2009.Molecular identification methods of fish species:reassessment and possible applications.Reviews in Fish Biology and Fisheries,19(3):265—293
Trewavas E,1977.The sciaenid fishes(croakers or drums)of the Indo-West-Pacific.Transactions of the Zoological Society of London,33(2):253—541
Thompson J D,Gibson T J,Plewniak Fet al,1997.The CLUSTAL–X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools.Nucleic Acids Research,25(24):4876—4882
Tamura K,Dudley J,Nei Met al,2007.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0.Molcular Biology Evolution,24(8):1596—1599
Vences M,Thomas M,Van der Meijden A,et al.2005.Comparative performance of the 16S rRNA gene in DNA barcoding of amphibians.Frontiers in Zoology,2(1):5
Ward,R,Zemlak T,Innes Bet al,2005.DNA barcoding Australia’s fish species.Philosophical Transactions of the Royal Society B:Biological Sciences,360(1462):1847—1857
Zhang J,Hanner R,2012.Molecular approach to the identification of fish in the South China Sea.PloS One,7(2):e30621
