酿酒酵母组蛋白H3 K4L和K36L突变对细胞生长和GAL1、SSA3、PHO5表达的影响
2014-03-08李芬杨敬辉吴秀丽张卫林王冠峰岳晓杰
李芬, 杨敬辉, 吴秀丽, 张卫林, 王冠峰, 岳晓杰
1.河南师范大学生命科学学院,新乡 453007;
2.河北农业大学海洋学院,秦皇岛 066003;
3.资源微生物与功能分子河南省高校重点实验室培育基地,新乡 453007
已知组蛋白可逆的乙酰化、甲基化修饰对基因表达具有重要影响。组蛋白甲基化多发生在赖氨酸(K)和精氨酸(R)残基上,组蛋白K甲基化由不同的组蛋白赖氨酸特异性甲基转移酶 (Histone lysine methyltransferases) 和赖氨酸特异性去甲基酶(Lysine-specific demethylase)共同维持,H3 的 K4、K9、K27、K36、K79和H4的K20是常见的甲基化位点,其氨基侧链可发生单、二和三甲基化,发生在不同组蛋白赖氨酸的甲基化修饰可对转录起激活或抑制作用。
H3-K4甲基化常与基因激活相关联。对H3-K4不同甲基化及其在不同生物体分布情况的分析显示,该位点的三甲基化局限于活性转录基因的5′端,二甲基化则分布于这些基因的主要部分[1]。酿酒酵母(Saccharomyces cerevisiae)H3-K4的3种甲基化均由Set1催化,H3-K36甲基化则由 Set2负责,两种酶均与延伸阶段的RNA多聚酶Ⅱ相关联[2]。但H3-K36的二、三甲基化都主要分布于活性转录基因的3′端,参与转录终止,是活性基因的重要标志[3,4]。组蛋白乙酰化常发生在赖氨酸残基,有些位点可发生多种转录后修饰(如甲基化、乙酰化修饰都可发生在H3-K4),且不同修饰之间还相互影响,因而使组蛋白不同转录后修饰位点的功能研究较为困难。已知H3、H4尾部多个赖氨酸突变都能引起特定的生长和转录缺陷[5~7]。但对不同组蛋白转录后修饰位点功能的比较研究及其与特定基因激活间的认识尚很缺乏。
为研究组蛋白不同转录后修饰位点的功能差异,丰富对“组蛋白密码”假说的认识,本文以酿酒酵母为材料,构建了H3-K4、H3-K36单独和二者共同突变为亮氨酸(L)的3种组蛋白突变株,并对其在高温、高盐等不同条件下的生长及诱导基因GAL1、SSA3、PHO5等的表达情况进行比较,结果表明不同转录后修饰位点突变对细胞生长和诱导基因表达的影响不同,其中H3-K4的修饰对于细胞快速适应不利环境条件可能更重要。
1 材料和方法
1.1 菌株、质粒
野生型酿酒酵母W303-1a:MATa ade2-1 ura3-1 his3-11,15,leu2-3,112 trp1-1 can1-100和缺失两拷贝组蛋白H3/H4,由LFH菌株:MATa ade2-1 ura3-1 his3-11,15, leu2-3,112 trp1-1 can1-100, hht1-hhf1Δ::TRP1 hht2-hhf2Δ::LEU2,pRS316: HHT1-HHF1(URA3)、pUC19、pRS313质粒。
1.2 质粒制备、大肠杆菌转化
参照 Sambrook等[8]的方法进行大肠杆菌转化。
1.3 定点突变及携带 H3 K4L和/或 K36L突变的H3/H4拷贝1的pRS313质粒的构建及鉴定
H3 K4L、K36L突变所需引物由上海生工生物工程技术服务有限公司合成,引物序列见表1。将H3 K4L和H3 K36L的引物分别与野生型组蛋白引物H3/4S和H3/4A相匹配,以pUCHHF1-HHT1为模板,经两轮PCR获得约300 bp和1100 bp两个短片段;再以二者等摩尔混合物为模板,以H3/4S和 H3/4A为引物进行第三轮PCR扩增得到含H3 K4L和K36L单突变的突变组蛋白H3/H4,将其分别克隆入pUC19并测序。以测序正确的含 H3 K4L的 pUCHHF1-hht1-K4L为模板,将H3 K36L引物分别与H3/4S和 H3/4A相匹配经两轮PCR获得约300 bp和1100 bp两个短片段,再将二者等摩尔混合物为模板,以H3/4S和 H3/4A为引物进行第三轮PCR扩增,得到含H3 K4L和H3 K36L双突变的组蛋白H3/H4,将其克隆入pUC19并测序。将测序正确的3种突变的组蛋白经EcoR I和XbaI双酶切,回收后分别亚克隆入经同样双酶切的 pRS313载体,得到的阳性克隆经EcoR I和XbaI双酶切验证。
1.4 酵母转化及基因置换实验(Plasmid Shuffle)实验
酵母转化及特定甲基化位点突变株制备、基因置换实验、组蛋白突变株筛选等参照文献[9~12]。
1.5 PCR鉴定3种组蛋白突变株
已知 pRS313 携带长度为 657 bp 的HIS3 基因,pRS316携带长度为801 bp的URA3基因,针对其序列设计引物,序列见表1。采用菌落PCR法鉴定突变株,挑取新鲜转化株单菌落(直径> 2 mm),在15 μL 2%SDS中涡旋15 s充分混匀,90℃ 孵育4 min,离心1 min,取上清-20℃保存备用。PCR扩增体系为50 μL:10 ×PCR Buffer 5.0 μL,50 mmol/ L MgCl21.5 μL,10 mmol/L dNTP 1.0 μL,25% Triton X-100 2 μL,引物 10 μmol/L各 0.5 μL,Taq酶 0.5 μL,上述 DNA 粗提液 1 μL ,加ddH2O至50 μL。PCR扩增程序:94℃预变性3 min;94℃变性30 s,53℃变性1 min,72℃延伸1min,35个循环;72℃延伸 10 min,4℃保存备用。扩增产物用1% 琼脂糖凝胶电泳检测。

表1 引物序列
1.6 突变株表型检测
参照李芬等[11,12]的方法,取鉴定正确的各组蛋白突变株和对照单菌落接种于适量培养基中,30℃、200 r/min 过夜后计数并调整各样品至同一浓度,对各菌株均作5个浓度梯度稀释,分别接种10 000,2000、400 、80 、16 个细胞于配制好的各种平板上,于 30℃和 39℃培养并观察各菌株生长,在 2 d、4 d 时拍照。
1.7 GAL1、PHO5和SSA3的诱导表达
GAL1、SSA3诱导表达主要参照李芬等[11,12]的方法。PHO5的诱导:在SD培养基中接种待测菌株,30℃、200 r/min培养至指数中期即OD值约1.0,取适量培养物作对照,剩余转入低磷培养基中,30℃、200 r/min继续振荡培养,分别于90 min、180 min取样,提取总RNA。
1.8 RNA的提取、RT-PCR和Real-time PCR
RNA提取采用热酸性酚法[13]。分别取 1 μg总RNA,依照试剂盒说明书42℃ 反转录1 h,取1/25 cDNA做为模板进行PCR扩增检测引物的特异性。扩增体系:Primix EX 25 μL,2 μmol / L 引物各 5 μL,无菌双蒸水补至 50 μL。扩增条件:94℃ 3 min;94℃1 min,53℃ 1 min,72℃ 1 min,25个循环;最后再72℃ 5 min。定量 PCR 采用 SYBR Green (TaKaRa,Osaka, Japan) 荧光染料法依照说明书进行扩增,扩增体系:SYBR Green Real-time PCR Master mix 12.5 μL,5 μmol/L 引物各 1 μL,模板 2 μL,无菌双蒸水补至25 μL。 扩增条件:95℃ 1 min,然后 95℃ 15 s和60℃ 1 min,45个循环。扩增GAL1、SSA3和ACT1基因的引物序列见李芬等[11,12]方法;扩增PHO5基因的引物序列见表1。上述引物进行PCR 扩增反应,ACT1为内参,2-ΔΔCt法计算结果[13,14]。
2 结果与分析
2.1 携带H3 K4L或/和K36L突变的H3/H4拷贝1的pRS313质粒的构建及鉴定
以pUCHHF1-HHT1为模板,经三轮PCR扩增获得含H3 K4L和K36L单突变的H3/H4全长序列。各PCR产物均与预期吻合,无非特异扩增现象。将其分别克隆入pUC19,酶切鉴定正确(图1A),测序无突变;以测序正确的含H3 K4L的pUCHHF1-hht1-K4L为模板,经3轮PCR扩增获得含H3 K4L/K36L双突变H3/H4全长序列,将其克隆入pUC19测序;酶切鉴定正确(图1C),3种突变拷贝测序结果表明,除引入的K4L和K36L外无其他突变(测序结果及比对略)。含 3种突变 H3/H4全长的 pUC19质粒经EcoR I/XbaI双酶切,回收后分别克隆入经上述两种酶消化的 pRS313载体,得到的阳性克隆经EcoR I/XbaI双酶切验证正确(图1:B和D )。
2.2 3个组蛋白特定甲基化位点突变菌株的获得及鉴定
采用乙酸锂转化法将分别携带突变 H3/H4的PRS313 质粒:pRS313 K4L、pRS313 K36L、pRS313 K4L/K36L分别转化LFH菌株,转化液涂SD/-His/+5-FOA平板,已知该菌株基因组H3/H4缺失,其生存所必需的组蛋白H3/H4 由携带野生H3/H4拷贝1和URA3筛选标记的 pRS316 提供,当培养基中添加5-FOA 时,含pRS316质粒提供野生H3/H4 的细胞会因表达URA3而死亡,丢失上述质粒未转入突变拷贝的细胞会因缺乏必需的H3/H4而死亡,只有该质粒被含突变H3/H4和HIS3标记的pRS313质粒替换的细胞方可存活。故利用SD/-His/+5-FOA筛选可获得H3-K4、H3-K36和二者共同突变为亮氨酸的S4、S36和D436菌株。
以HIS3特异引物进行菌落 PCR 结果显示,得到与预期相符的582 bp的扩增产物(图2),而URA3特异引物的PCR除对照得到预期的437 bp产物外,在3突变株中均无扩增条带;用抗H3-K4 3种甲基化和乙酰化抗体对 H3-K4 (S4) 突变株进行的免疫印迹实验进一步表明突变株该位点的甲基化和乙酰化修饰丧失,表明的确得到了正确的突变株。
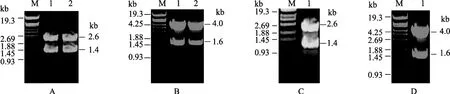
图1 携带H3 K4L或/和K36L突变H3/H4拷贝1的pRS313质粒的构建及鉴定
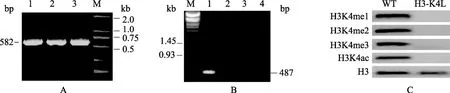
图2 组蛋白突变株的鉴定
2.3 S4、S36和D436菌株与野生型的表型比较
为研究H3-K4和K36两重要位点转录后修饰丧失对酵母生长的影响,将单突变株S4、S36和双突变株 D436与野生型(WT)在不同条件下的表型进行比较(图3)。由图3可以看出,3种组蛋白突变株的生长情况均不如野生型,其中双突变株D436生长最慢,高温、高盐条件下单突变株S4比S36稍慢,表明组蛋白H3-4K和36K的转录后修饰对细胞维持正常生存非常重要。在对高温、高盐等不利环境条件的快速适应方面,H3-K4的修饰比H3-36K更重要。
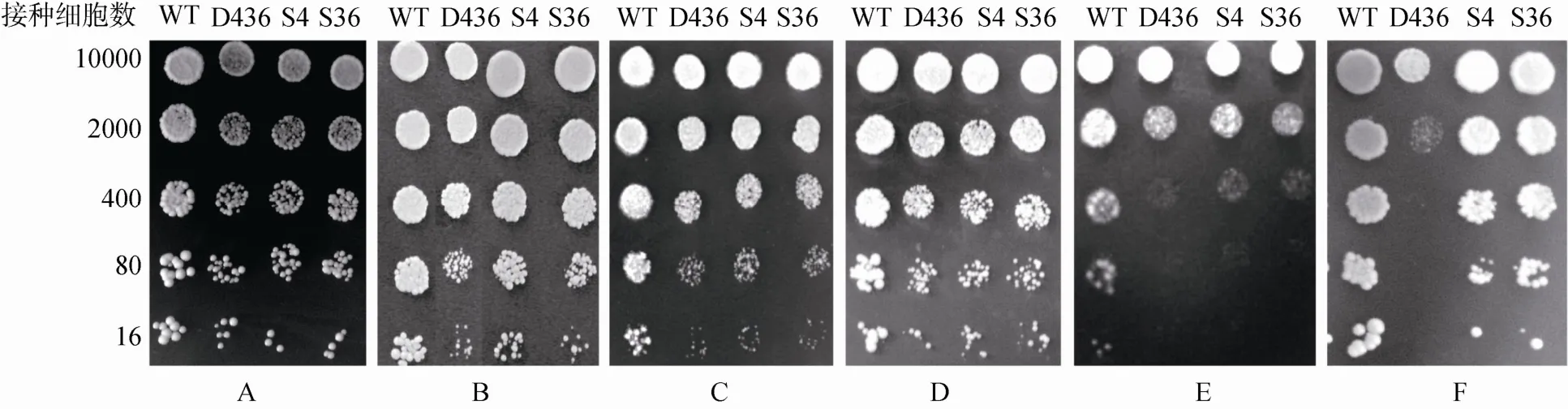
图3 组蛋白不同转录后修饰位点突变对酿酒酵母生长的影响
2.4 S4、S36和 D436菌株与野生型 GAL1基因表达的比较
因半乳糖为单一碳源时,双突变株生长明显较慢,可能是因为H3-K4和H3-K36同时突变影响了GAL基因的表达。本研究以 W303为对照,对 S4、S36 和 D436 菌株的GAL1基因表达进行了RT-PCR(图 4A)和 qRT-PCR检测(图 4B)。由图 4A 可知,PCR扩增获得与预期相符的特异性条带,qRT-PCR结果显示:未诱导时,各菌株GAL1基因的表达水平接近甚至趋于零,随诱导时间的延长该基因的表达均缓慢上升,但不同菌株上升速度明显不同;各突变株显著低于野生型(P<0.01),表明组蛋白不同转录后修饰位点突变影响了GAL1表达;双突变株D436的GAL1表达明显比单突变株S4、S36低(P<0.05),而单突变株 S4低于 S36(P<0.05)。表明上述两位点突变使GAL1激活延迟,突变株在半乳糖为单一碳源时的表型缺陷是因该条件下细胞生存所必需的GAL基因激活延迟所致。
2.5 S4、S36和D436菌株与野生型SSA3基因表达的比较
因双突变株对高温敏感,可能因组蛋白突变影响了热激蛋白基因的表达。本研究以野生型为对照,对3种组蛋白突变株的SSA3表达进行了RT-PCR(图5A)和 qRT-PCR检测(图 5B)。图 5A表明,PCR扩增获得了与预期相符的特异性条带。qRT-PCR结果显示:未热激时各菌株SSA3都有基础水平的表达,热激10 min后,其SSA3表达均有显著升高(10 min与 0 min相比较:WT、S36、S4:P<0.0001; D436:P<0.05),随热激时间延长各菌株SSA3表达呈下降趋势(30 min同10 min相比较:P>0.05),但不同菌株SSA3表达下降速率明显不同。其中组蛋白突变株SSA3表达均显著低于野生型对照,以双突变株D436最为明显(WT 同D436相比较:P<0.001;WT vs S4或S36:P<0.05)。单突变株S36与S4差别不大(P>0.05)。表明上述两位点突变可延迟SSA3激活,组蛋白双突变株的高温敏感表型是因该条件下细胞生存所必需的Hsp基因激活延迟所致。
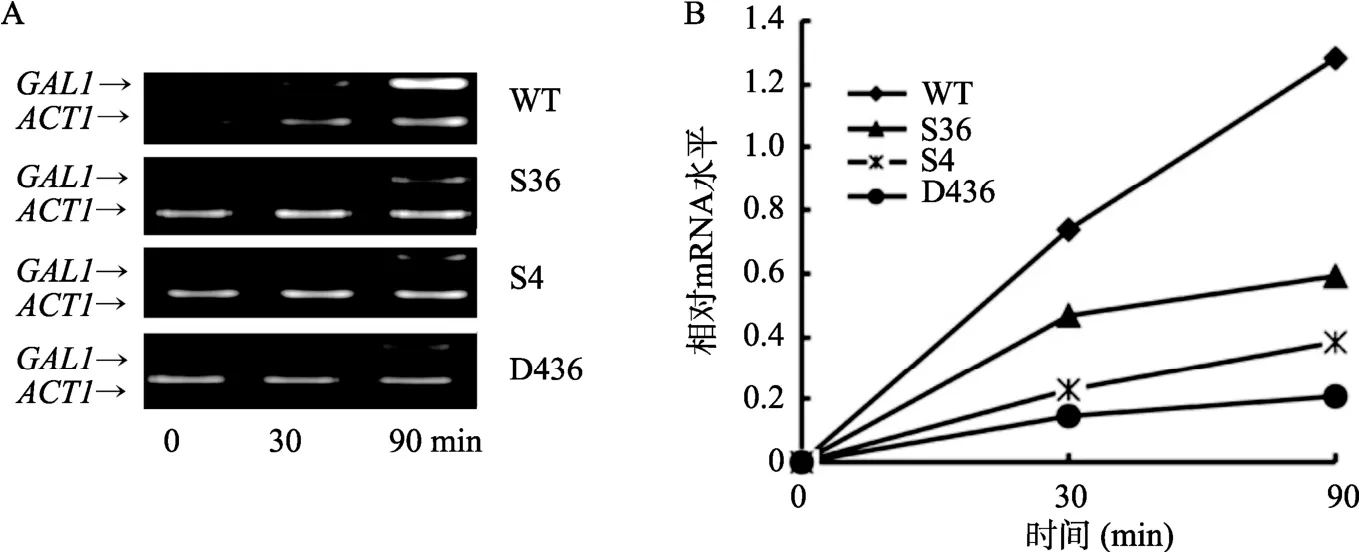
图4 组蛋白不同转录后修饰位点突变对酿酒酵母GAL1基因表达的影响
2.6 S4、S36和D436菌株与野生型PHO5基因表达的比较
低磷条件可诱导酵母PHO5基因表达。为检测组蛋白两位点突变可造成诱导基因激活延迟,本研究以ACT1为内参,对3突变株诱导基因PHO5进行 RT-PCR(图 6A)和 qRT-PCR(图 6B)分析。qRT-PCR结果表明,随诱导时间的延长各菌株该基因的表达均缓慢上升,但不同菌株上升速率明显不同;组蛋白突变株的该基因表达显著低于野生型对照(WT同 S4或 S36相比较:P<0.05),其中以组蛋白双突变株D436最为明显(WT 同D436相比较:P<0.01),表明两位点突变也显著延迟了诱导基因PHO5的激活。
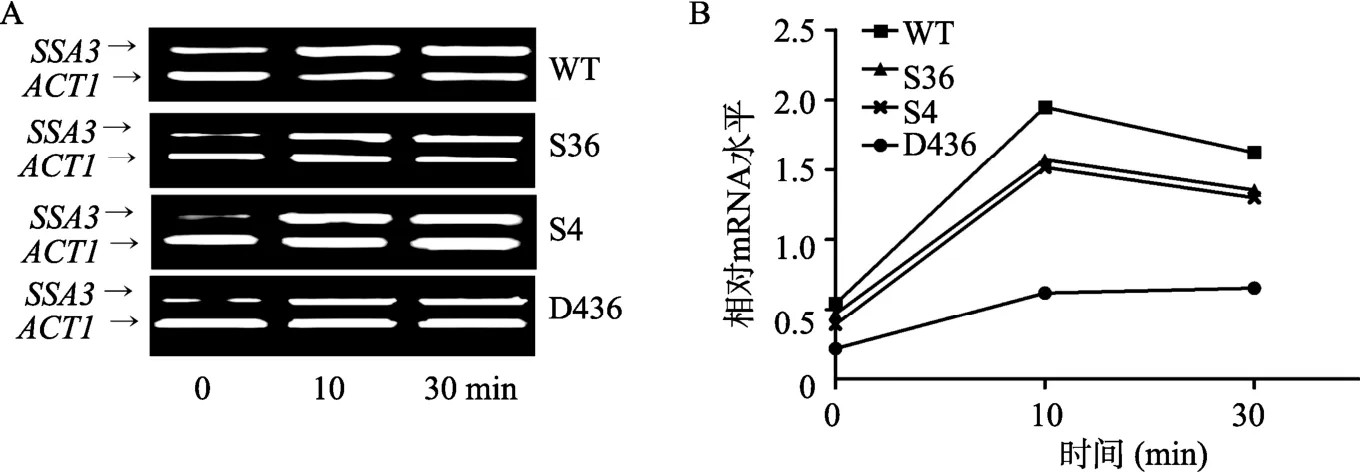
图5 组蛋白不同转录后修饰位点突变对酿酒酵母SSA3基因表达的影响
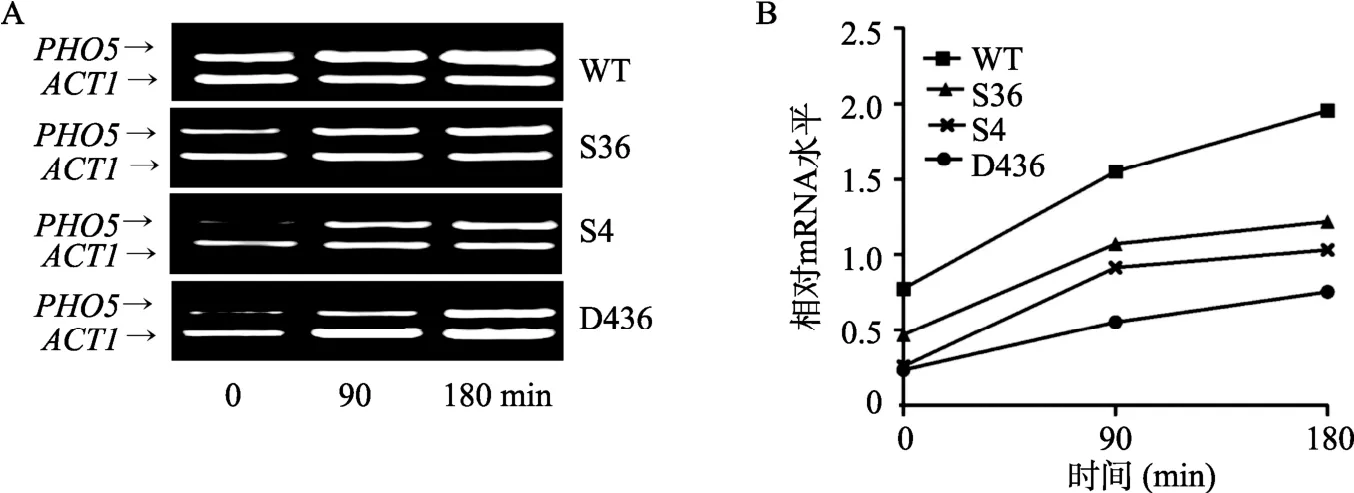
图6 组蛋白不同转录后修饰位点突变对酿酒酵母PHO5基因表达的影响
3 讨 论
发生在组蛋白特定氨基酸残基的乙酰化、甲基化、磷酸化、泛素化及ADP核糖基化等修饰对基因转录活性有重要影响,通过影响组蛋白之间、组蛋白与DNA之间的相互作用而改变染色质结构,进而调控基因表达。而且单一的组蛋白修饰常不能单独起作用,一种修饰常常可抑制或指导另一修饰的存在,形成不同组蛋白修饰的级联,而同时存在于组蛋白不同位点的多种修饰则作为一种语言或标志即“组蛋白密码”被细胞内多种非组蛋白所识别,并以此决定基因的表达,组蛋白密码的存在大大丰富了传统遗传密码的信息含量[4,15]。
为研究H3-K4和H3-K36两位点功能的差异,丰富对组蛋白密码假说的认识,本文以酿酒酵母为材料,构建了两位点可能存在的3种形式突变株即:4、36位单突变及两位点同时突变,并比较了高温、高盐等条件下突变株的生长及PHO5、SSA3和GAL1表达情况,结果表明:同野生型相比,供试条件下各突变株的生长及几个诱导基因的激活均有不同程度的减慢,双突变株D436缺陷表型最严重。表明组蛋白H3-K4和H3-K36的转录后修饰对细胞生长和适应不利环境条件极为重要。
同本实验室先前对 H3-K14 和 H4-K8 两乙酰化位点突变株的研究结果类似[12],单突变株S4和S36在半乳糖为单一碳源、30℃条件下无明显差别,但其GAL1激活则有显著差异(P<0.05)。可能因为组蛋白转录后修饰位点突变仅使某些诱导基因的激活延迟,并非关闭,因组蛋白转录后修饰功能重叠的影响,随诱导时间的延长,诱导基因经由其他途径被激活表达,从而使缺陷表型得到逐步缓解。细胞之所以能够在高温等不利条件下生存生长,是因能激活相应条件下细胞生存所必需的基因进行表达[12],故单突变株S4和S36在不同环境中的差异表型表明同一位点的修饰对不同基因的转录激活影响不同,H3-K4的修饰对于细胞快速适应高温等不利条件可能比H3-K36更重要。
组蛋白乙酰化位点 H3-K14和 H4-K8突变的3种突变株表型与本文的突变株表型及GAL1激活延迟均非常类似,只是程度不同[12],因此本研究认为该现象的产生是因组蛋白乙酰化、甲基化修饰相关联所致。已知人类Mll基因与许多不同种类的白血病的发病机理密切相关,包括 AML(Acute myeloid leukemia),Mll基因编码的蛋白有特异的 H3-K4甲基转移酶活性,H3的乙酰化可促进其活性增加[16]。酵母中组蛋白甲基转移酶Set1和Set2分别催化H3-K4和 H3-K36的甲基化[17],作为 NuA4组蛋白乙酰转移酶和 Rpd3组蛋白去乙酰转移酶复合物组分的Eaf3可识别基因5′端甲基化的H3-K4和H3-K36[18]。其中H3-K36的甲基化除了提供Rpd3S对转录基因编码区的去乙酰化信号外[19,20],还通过抑制组蛋白伴侣分子的作用而阻止乙酰化的组蛋白掺入转录基因的编码区[21],以去除转录延伸相关的乙酰化从而抑制基因内的转录起始,保证 RNAPII转录的精确性。Morillon等发现H3-K4和H3-K36的甲基化与特定基因(Met16和Rrs11B)5'端Esa1 (组蛋白乙酰转移酶复合物NuA4的催化亚基)依赖的H4的乙酰化相关[19],而Esa1可通过克服染色质的抑制状态而促进PHO5的转录[22]。 由此可见3个组蛋白突变株的表型缺陷和GAL1,SSA3及PHO5等诱导基因表达延迟缺陷可能是因为组蛋白上述转录后修饰位点突变,不仅造成了该位点甲基化、乙酰化等修饰的丧失,可能也影响了其它位点的乙酰化,进而改变了组蛋白密码,延迟该条件下细胞生存所必需的基因的激活导致的。
Carvin等[23]研究发现,Set1 缺失可导致PHO5和GAL1-10表达升高,该结果与本文H3 K4L突变引起的结果正好相反。我们认为该差异的产生可能是由于 Set1缺失仅影响 H3-K4的甲基化修饰,而H3 K4L突变则造成该位点各种转录后修饰的丧失(图2B);已知酵母中主要由Set1催化的H3-K4的甲基化修饰仅占H3总量大约34%[24],剩余的H3-K4还可发生乙酰化等转录后修饰;故严格说S4的表型实际上是H3-K4突变造成该位点转录后修饰丧失的综合效应,而非仅由突变位点甲基化丧失引起的效应,这也再次体现了组蛋白转录后修饰功能的复杂性。Ucar 等[25]运用 CoSBI(Cohernt and shifted bicluster identification)算法,在人 CD4+ T 细胞全基因组范围确定了包含 19 种转录后修饰的 873 种组蛋白修饰组合模式,其中 50% 以上的组合模式包含了H2BK5Ac、H4K8Ac、H2BK120Ac、H3K9Ac、3K18Ac、H3K27 Ac、H4K5Ac、H3K4Ac、H3K36Ac 和H3K4me3等10种修饰。Venkatatesh 等[21]对正常和Set2 缺失酵母菌株 6000 个基因的全部甲基化和乙酰化谱进行了比较,发现在正常细胞中,Set2 植入的抑制性甲基化标记(H3K36me)位于活跃转录的基因两侧,但在Set2 缺失突变株中,这些标记丧失,大部分基因的乙酰化标记反而比正常细胞增加。由此可见,组蛋白某个位点突变除造成该位点转录后修饰的丧失外,也影响其他位点的修饰,导致基因组组蛋白修饰模式改变,引发不同基因表达变化,最终影响表型。双突变株D436缺陷表型之所以更为严重,是因为H3-K4和H3-K36同时突变,两位点的乙酰化和甲基化修饰丧失,引发基因组组蛋白修饰模式发生比单突变更大的改变,从而影响更多基因的表达,导致它对各不利条件更敏感。正因为同时存在于组蛋白不同位点的多种修饰以组合模式的形式起作用,因此很难确定上述位点的乙酰化和甲基化修饰到底哪种与突变体的表型直接相关,本文的组蛋白突变株的缺陷表型严格上应是相应位点突变导致多种修饰改变而引发组蛋白修饰模式改变所造成的综合影响。
日益增多的证据表明组蛋白乙酰化、甲基化等转录后修饰不仅影响基因的表达,还与包括癌症等在内的多种人类重大疾病的发生发展密切相关,因此对不同组蛋白转录后修饰位点的功能差异进行深入的比较研究,不仅可以丰富并加深对组蛋白修饰功能和“组蛋白密码假说”的认识,对于某些因组蛋白转录后修饰异常引起的人类重大疾病发病机理的揭示和治疗均至关重要。
[1]Santos-Rosa H, Schneider R, Bannister AJ, Sherriff J,Bernstein BE, Emre NC, Schreiber SL, Mellor J, Kouzarides T. Active genes are tri-methylated at K4 of histone H3.Nature, 2002, 419(6905): 407–411.
[2]Krogan NJ, Kim M, Tong A, Golshani A, Cagney G, Canadien V, Richards DP, Beattie BK, Emili A, Boone C,Shilatifard A, Buratowski S, Greenblatt J. Methylation of histone H3 by Set2 in Saccharomyces cerevisiae is linked to transcriptional elongation by RNA polymerase II.Mol Cell Biol, 2003, 23(12): 4207–4218.
[3]Bannister AJ, Schneider R, Myers FA, Thorne AW,Crane-Robinson C, Kouzarides T. Spatial distribution of di- and tri-methyl lysine 36 of histone H3 at active genes.J Biol Chem, 2005, 280(18): 17732–17736.
[4]杜婷婷, 黄秋花. 组蛋白赖氨酸甲基化在表观遗传调控中的作用. 遗传, 2007, 29(4): 387–392.
[5]Grunstein M. Nucleosomes: regulators of transcription.Trends Genet, 1990, 6(12): 395–400.
[6]Smith MM. Histone structure and function.Curr Opin Cell Biol, 1991, 3(3): 429–437.
[7]Roth SY. Chromatin-mediated transcriptional repression in yeast.Curr Opin Genet Dev, 1995, 5(2): 168–173.
[8]Sambrook J, Frisch EF, Maniatis T. Molecular Cloning: A Laboratory Manual, 2nd ed. New York: Cold Spring Harbor Laboratory Press, 1989: 19−20, 55−56, 880−885.
[9]Hill J, Donald KA, Griffiths DE, Donald G. DMSO-enhanced whole cell yeast transformation.Nucleic Acids Res,1991, 19(20): 5791.
[10]李芬, 田树娟. 双元载体提供组蛋白 H3/H4拷贝 1的elp3Δ菌株的构建. 河南师范大学学报(自然科学版),2008, 36(2): 95–98.
[11]Li F, Lu J, Han QJ, Zhang GP, Huang BQ. The Elp3 subunit of human Elongator complex is functionally similar to its counterpart in yeast.Mol Genet Genomics, 2005, 273(3):264–272.
[12]李芬, 孔艳, 张帅, 马瑜, 胡岩岩. 组蛋白不同乙酰化位点突变对酵母细胞生长和SSA3、GAL1基因表达的影响. 中国生物化学与分子生物学报, 2010, 26(9): 822–827.
[13]Li F, Ma JX, Ma Y, Tian SJ, White RE, Han GC. hElp3 Directly modulates the Expression of HSP70 gene in HeLa cells via HAT activity.PLOS ONE, 2011, 6(12): e29303.
[14]Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method.Methods,2001, 25(4): 402–408.
[15]Strahl BD, Allis CD. The language of covalent histone modifications.Nature, 2000, 403(6765): 41–45.
[16]Tenney K, Shilatifard A. A COMPASS in the voyage of defining the role of trithorax/MLL-containing complexes:linking leukemogensis to covalent modifications of chromatin.J Cell Biochem, 2005, 95(3): 429–436.
[17]Gerber M, Shilatifard A. Transcriptional elongation by RNA polymerase II and histone methylation.J Biol Chem,2003, 278(29): 26303–26306.
[18]Xu C, Cui GF, Botuyan MV, Mer G. Structural basis for the recognition of methylated histone H3K36 by the Eaf3 subunit of histone deacetylase complex Rpd3S.Structure,2008, 16(11): 1740–1750.
[19]Morillon A, Karabetsou N, Nair A, Mellor J. Dynamic lysine methylation on histone H3 defines the regulatory phase of gene transcription.Molr Cell, 2005, 18(6): 723–734.
[20]Carrozza MJ, Li B, Florens L, Suganuma T, Swanson SK,Lee KK, Shia WJ, Anderson S, Yates J, Washburn MP,Workman JL. Histone H3 methylation by Set2 directs deacetylation of coding regions by Rpd3S to suppress spurious intragenic transcription.Cell, 2005, 123(4): 581–592.
[21]Venkatesh S, Smolle M, Li H, Gogol MM, Saint M, Kumar S, Natarajan K, Workman JL. Set2 methylation of histone H3 lysine 36 suppresses histone exchange on transcribed genes.Nature,2012, 489(7416): 452–455.
[22]Nourani A, Utley RT, Allard S, Côté J. Recruitment of the NuA4 complex poises the PHO5 promoter for chromatin remodeling and activation.EMBO J, 2004, 23(13): 2597–2607.
[23]Carvin CD, Kladde MP. Effectors of lysine 4 methylation of histone H3in Saccharomyces cerevisiae are negative regulators of PHO5 and GAL1–10.J Biol Chem, 2004,279(32): 33057–33062.
[24]Strahl BD, Ohba R, Cook RG, Allis CD. Methylation of histone H3 at lysine 4 is highly conserved and correlates with transcriptionally active nuclei in Tetrahymena.ProcNatl Acad Sci USA, 1999, 96(26): 14967–14972.
[25]Ucar D, Hu Q, Tan K. Combinatorial chromatin modification patterns in the human genome revealed by subspace clustering.Nucleic Acids Res,2011, 39(10): 4063–4075.
