水稻扩展蛋白家族的生物信息学分析
2014-03-08施杨徐筱李昊阳徐倩徐吉臣
施杨,徐筱,李昊阳,徐倩,徐吉臣
北京林业大学,林木育种国家工程实验室,北京100083
扩展蛋白(Expansin)是植物细胞壁的重要组成成分,通过打断细胞壁纤维素与半纤维素的非共价键,释放细胞壁多聚物的机械张力,以增加细胞壁的柔韧性[1,2]。众多研究表明,扩展蛋白除具有增大细胞的功能外,几乎参与了植物的整个发育过程,包括细胞生长[3~11]、种子萌发[8,12~15]、根毛形成[9,16,17]、根系生长[8,14,18~22]、叶原基形成[8,18,20,23]、叶子生长发育[20,24~26]、果实成熟[9,27,28]、器官脱离[9]、花粉管生长[9,20,29,30]等,在植物抗逆[31~37]方面也具有不可忽视的作用。
作为植物基因组中一类重要的基因家族,扩展蛋白在已检测的所有种子植物中都有广泛的分布,不同物种所含的家族成员在基因数量、序列组成、基因功能等方面都有很大的不同[38]。水稻(Oryza sativaL.)作为世界上最为重要的经济作物之一,已于2002年完成基因组测序[39]。统计分析显示,水稻基因组中存在多达58个扩展蛋白基因家族成员,这些基因蛋白在水稻的生长、发育、生殖、抗逆等重要农艺性状表现上执行重要的作用。本文依据水稻基因组测序的结果,对水稻扩展蛋白基因家族成员的组成、染色体分布、基因结构、基因表达等生物学信息进行了系统分析,以期为功能研究奠定基础。
1 材料和方法
1.1 材料
水稻扩展蛋白的基本数据来源于 Daniel Cosgrove实验室建立的网站(http://www.bio.psu.edu/expansins/),以及水稻基因组最新数据版本v3.0(http://rgp.dna.affrc.go.jp/)。
1.2 方法
1.2.1 扩展蛋白家族基因染色体的物理定位
依据Daniel Cosgrove实验室建立的网站提供的水稻扩展蛋白基因信息,确定每一个基因的染色体物理位置,使用MapDraw软件[40]绘制扩展蛋白基因在水稻染色体上的分布图。
1.2.2 扩展蛋白序列分析
利用生物信息学软件MEGA v5.0并采用Neighbor-joining算法[41]构建水稻扩展蛋白基因氨基酸序列的系统进化树;利用 Protein Homology/analog-Y Recognition EngineV2.0软件(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)进行蛋白二级和空间结构预测。利用在线软件 Mobyle 1.5(http://mobyle.pasteur.fr/cgi-bin/portal.py#welcome)计算水稻扩展蛋白各种氨基酸的个数及总数,特定氨基酸的比例为特定氨基酸的数量占所有氨基酸总数的百分比。采用生物信息学软件DNAMAN v7.0对水稻扩展蛋白家族成员进行序列间一致率比对;利用密码子分析软件CodonW v1.4.4[42]和EMBOSS(欧洲分子生物学开放软件系统)中的 CHIPS程序对水稻扩展蛋白密码子偏好性(Codon usage bias)进行统计分析;通过WebLogo 3[43]以及ProtParam[44]在线软件,对水稻中扩展蛋白的结构保守性、蛋白稳定性及相关的理化性质进行统计分析。
1.2.3 基因表达信息分析
将水稻扩展蛋白基因序列与水稻 EST数据库(RAP-DB)[45]进行比对(E-value<10–6),获得扩展蛋白基因在不同组织器官(根、叶、穗)不同发育阶段的表达信息,以文库中特定基因家族成员的EST数量所占的比率为信号,绘制基因表达图谱。
2 结果与分析
2.1 水稻扩展蛋白基因家族及染色体分布
扩展蛋白基因家族分为 A(或 α)、B(或 β)、LA(或γ)和LB(或δ)4个亚家族[24,46,48]。生物信息学分析显示,水稻基因组中共包含扩展蛋白基因家族成员58个,全部基因的基本信息见表 1。其中A类扩展蛋白34个,约占扩展蛋白基因家族成员总数的58.6%;B类扩展蛋白19个,约占扩展蛋白基因家族成员总数的32.8%;LA类扩展蛋白4个,LB类扩展蛋白1个,分别约占扩展蛋白基因家族成员总数的6.9%和1.7%。利用氨基酸序列建立水稻扩展蛋白家族成员亲缘关系图谱(图1),结果表明,亚家族的成员各自聚集成群,显现出亚家族间规律性的起源和进化关系。
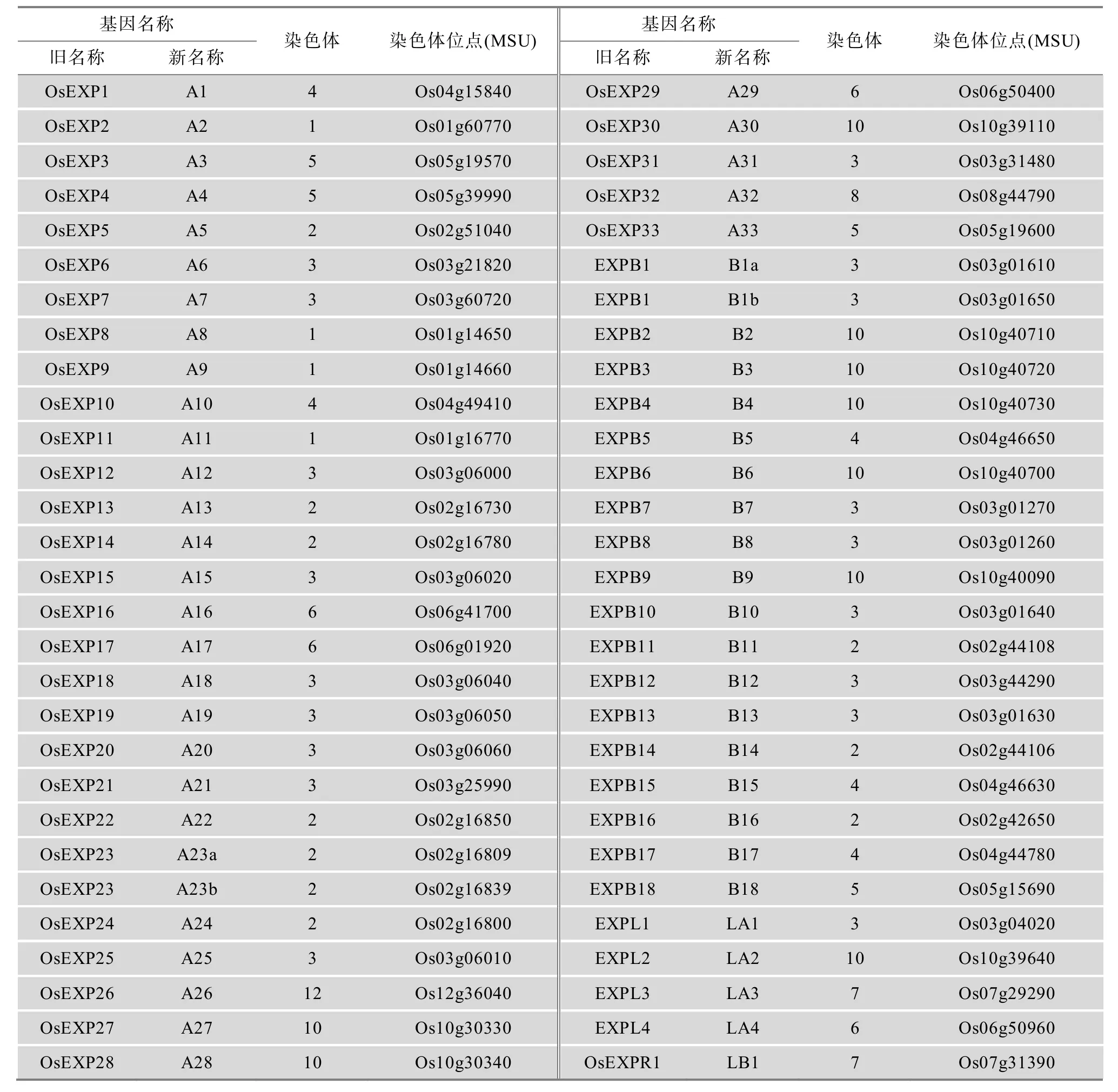
表1 水稻扩展蛋白基因的基本信息
水稻扩展蛋白基因在染色体上的分布见图 2。结果显示:第 3染色体扩展蛋白基因数目最多,包含 18个家族成员;其他基因分别位于第 1、2、4、5、6、7、8、10和第 12染色体上,分别包含 4、10、5、4、4、2、1、9和 1个家族成员;第 9和第 11染色体上不含有任何扩展蛋白基因。基因家族成员在染色体上有成簇存在的现象,同一亚家族的扩展蛋白基因更容易聚集在一起,如第 2染色体上存在由6个A类扩展蛋白基因组成的基因簇;第3染色体上包含一个由8个A类扩展蛋白基因组成的基因簇以及一个由5个B类扩展蛋白基因组成的基因簇;第10染色体上存在一个由5个B类扩展蛋白基因构成的基因簇等。
2.2 水稻扩展蛋白基因的组成结构分析
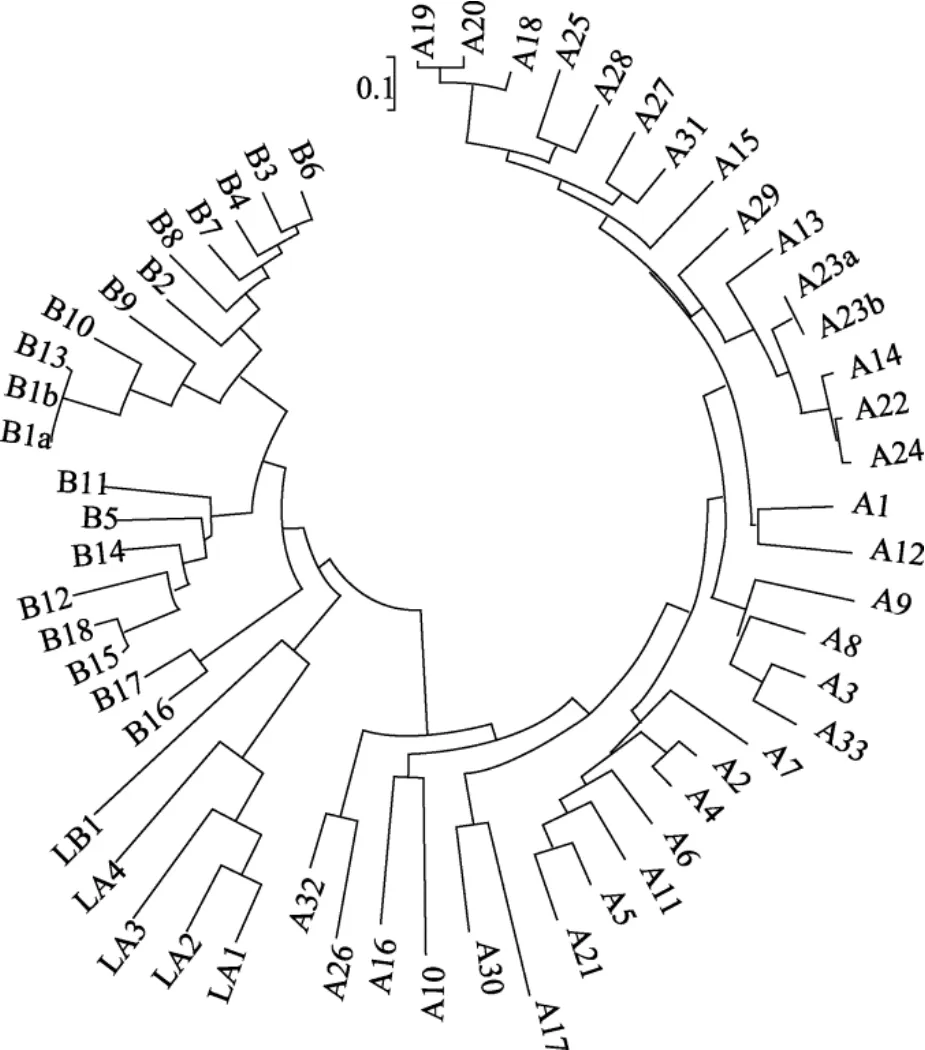
图1 水稻基因组中扩展蛋白家族成员的系统进化树
统计分析显示,编码水稻扩展蛋白基因的核苷酸序列长度在 687~1128 bp之间,不同亚家族基因之间序列大小略有差别。其中编码A类扩展蛋白基因的长度范围为705~912 bp,平均长度787 bp;编码B类扩展蛋白基因长度范围为687~1128 bp,平均长度 847 bp;其他类型扩展蛋白基因长度范围为771~942 bp,平均长度842 bp。
水稻扩展蛋白基因在基因组中的长度介于705~6464 bp之间,不同扩展蛋白基因包含数目、大小不等的内含子,但不同亚家族的外显子/内含子模式有一定的保守性。其中34个A类扩展蛋白基因亚家族中,17(50%)个基因分别含有 1个内含子,14(41%)个基因分别含有2个内含子;19个B类扩展蛋白基因亚家族中,9(47%)个基因分别含有 3个内含子;而LA和LB类扩展蛋白成员多含有4个内含子(表 2)。
扩展蛋白的氨基酸长度介于 228~375aa之间,平均长度269aa。不同亚家族扩展蛋白组成结构类似,均由信号肽、催化区、吸附区 3个部分组成[46,49],其空间结构也清楚显示出3个相对独立的结构域(图3)。N端的信号肽区大约有 18~30个氨基酸,可形成α螺旋,引导初生的多肽透过内质网,经切除修饰后的肽链由高尔基体转运分泌到细胞壁;位于序列中段的催化区(Catlysis domain, catdom)大小约15 kDa,其核心序列与内切葡聚糖酶EG45(纤维素酶系中的一个主要成分,可在葡聚糖链的随机位点降解底物产生寡糖)有较高的同源性;位于羧基端的与纤维素酶类似的纤维素吸附区(Cellulose-binding Domain,CBD)大小约10 kDa,长度较为保守,主要由3′端一个外显子编码。催化区、吸附区的氨基酸组合形成的二级结构多为β折叠和无规则卷曲。
水稻扩展蛋白不同亚家族的氨基酸组成分析见表3。整个家族氨基酸组成比例介于1.98%~11.89%之间,平均为 4.96%。不同亚家族之间的氨基酸组成比例跨度差异较大,其中A类蛋白的氨基酸组成比例介于1.31%~12.96%之间,平均为4.98%。B类蛋白的氨基酸组成比例介于1.93%~10.85%之间,平均为4.95%。
LA的氨基酸组成比例介于 0.07%~20.23%之间,平均为4.89%。LB的氨基酸组成比例介于1.17%~9.34%之间,平均为4.98%。部分氨基酸在不同亚家族间的比例相近,如缬氨酸(Val)在A、B、LA、LB中的数值分别为 6.84%、7.46%、8.46%和 7.39%;个别氨基酸在不同亚家族间的比例差异极为显著,如精氨酸(Arg)在 A、B、LA、LB中的数值分别为4.19%、10.06%、20.23%和4.28%。
2.3 水稻扩展蛋白基因序列相似性分析
2.3.1 序列一致率比较
DNA序列比较发现,水稻 58个扩展蛋白基因中,A23和B1分别有2个拷贝(A23a/b,B1a/b)。除个别基因外,水稻扩展蛋白亚家族基因之间序列一致率(或相似率)小于 50%,而同一亚家族不同成员之间一致率大于50%,如A19与A20之间一致率为94.7%,B1(a/b)和B13之间一致率为96.6%,而A19和B1(a/b)之间一致率只有45.8%。氨基酸序列比较发现,除个别基因外,亚家族基因之间一致率小于35%,而同一亚家族不同成员之间的一致率大于35%,如A19与A20之间、B1(a/b)和B13之间一致率均为 96.2%,而 A19和 B1(a/b)之间一致率只有27.6%。相对而言,A与B类、LA与LB类亚家族基因之间分别有较高的氨基酸一致率,而A/B类与LA /LB类亚家族之间的氨基酸一致率较低(表4)。
2.3.2 氨基酸保守性分析

图2 水稻中扩展蛋白基因的染色体分布
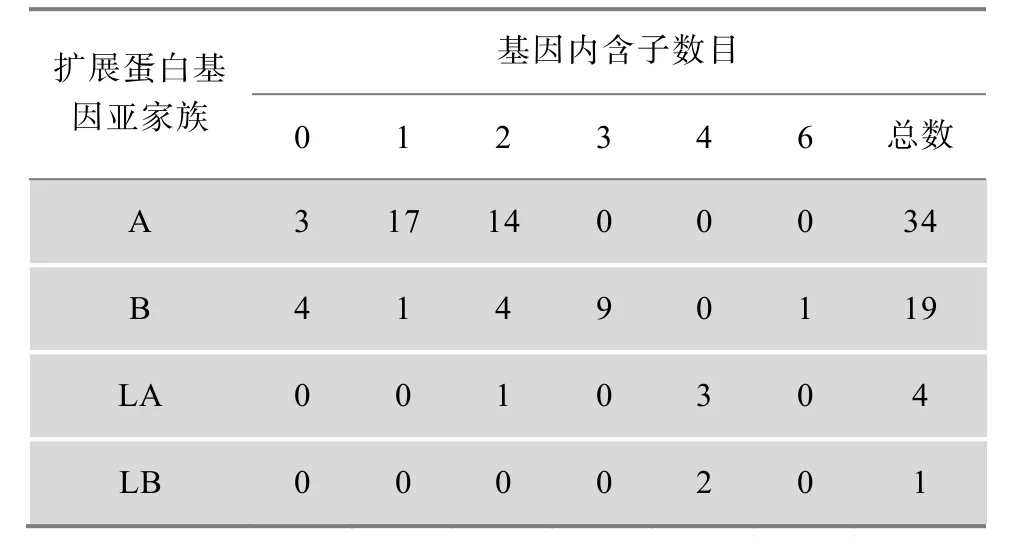
表2 水稻扩展蛋白基因的内含子数目比较
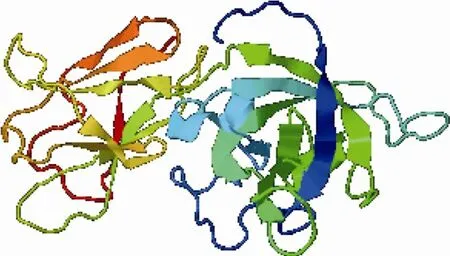
图3 扩展蛋白A1的空间结构预测
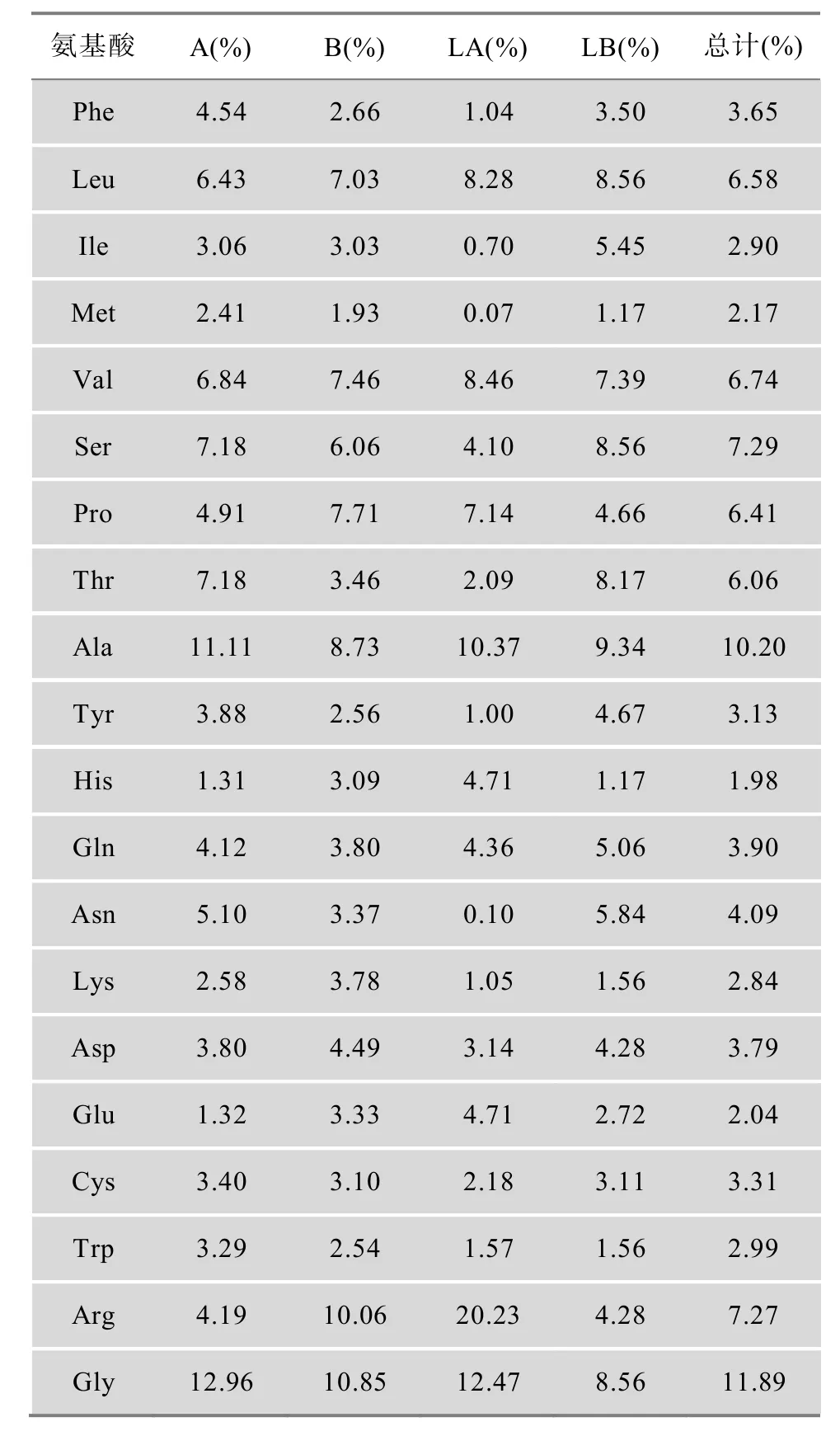
表3 水稻4类扩展蛋白亚家族成员氨基酸组成分析
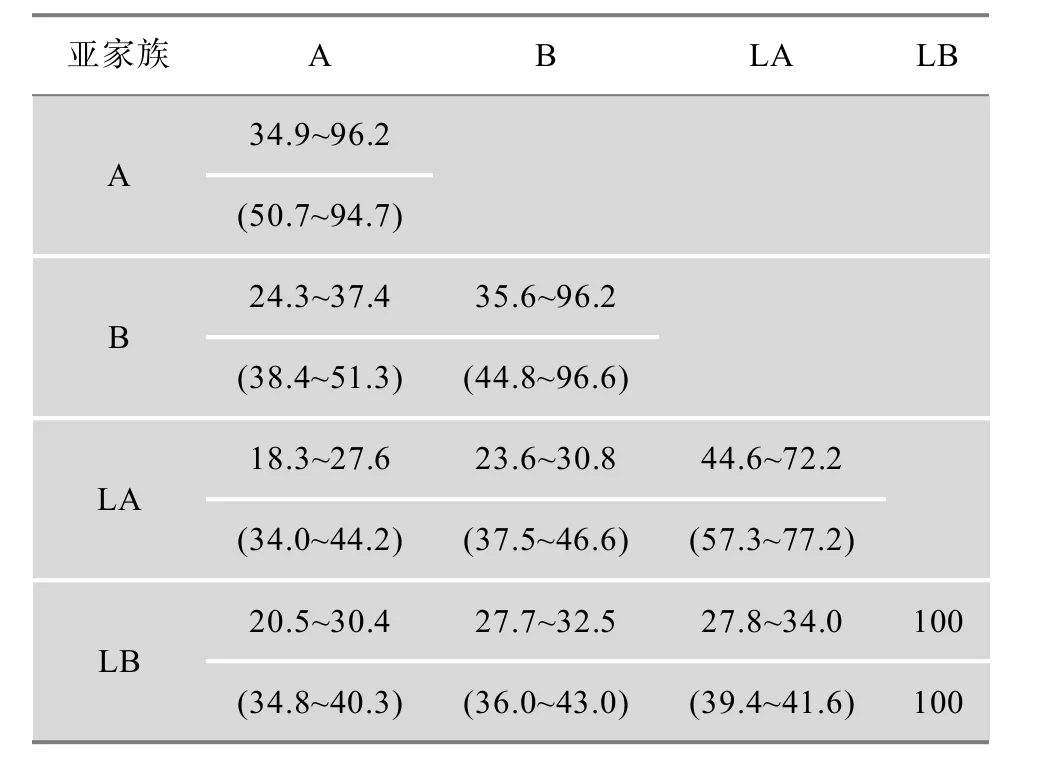
表 4 水稻扩展蛋白亚家族成员间氨基酸或核苷酸(括号内数字)序列一致率(%)比较
分别对A类和B类扩展蛋白氨基酸的保守性进行了分析(图4)。结果显示,扩展蛋白的信号肽部分序列差异较大,而催化区、结合区部分相对保守,并且含有几个丰富且异常保守的氨基酸,如催化区的半胱氨酸(C)和结合区的色氨酸(W)等。半胱氨酸被认为与二硫键的形成有关[50],但A类扩展蛋白的保守半胱氨酸残基有8个,B类只有6个。
2.4 水稻扩展蛋白密码子使用偏好性分析
对水稻 58个扩展蛋白基因家族成员的 15 635个密码子进行统计,发现氨基酸同义密码子的相对使用频率(Relative frequency of synonymous codon,RFSC)(某一密码子在样本中的实际观察数与该密码子对应的氨基酸在样本中的实际观察数的比值)具有明显的偏好性(表5)。如编码苯丙氨酸(Phe)的2个密码子UUU和UUC,前者的使用频率只有8.23%,后者的使用频率达到91.77%,因此UUC被定义为Phe的高频密码子(相对同义密码子使用频率单值超过 60%或超过该组同义密码子平均占有频率的 1.5倍的密码子)[51]。所有多密码子编码的氨基酸以及终止子均具有高频密码子,共计26个,Arg、Leu、Val、Thr、Ala等分别具有 2个高频密码子,Ser具有 3个高频密码子。
2.5 水稻扩展蛋白理化特征分析
统计分析(表6)表明,水稻扩展蛋白的分子量介于25.40~34.45之间,A类均值达到27.71,B类均值达到 29.51,总平均值为 28.51;家族成员蛋白质的理论等电点介于4.69~9.76之间,平均值达到7.85,近 70%的水稻扩展蛋白成员表现偏碱性;扩展蛋白成员的总平均疏水性(Grand average of hydropathicity, GRAVY) 介于–0.36~+0.18 之间,均属于亲疏性相当的两性蛋白(正值表示疏水性,负值表示亲水性,介于±0.5之间为两性蛋白)[52];扩展蛋白成员的不稳定性指数介于21.6~53.76之间,超过77%的扩展蛋白结构稳定性较好(>40为不稳定);扩展蛋白质的脂溶指数介于59.4~85.74之间,平均值达到70.61,较高的蛋白脂溶指数使得水稻扩展蛋白可以较好地适应各种环境。LA3、A10、B12、B17和LB1的脂溶指数甚至超过80,属于嗜热型蛋白[53]。
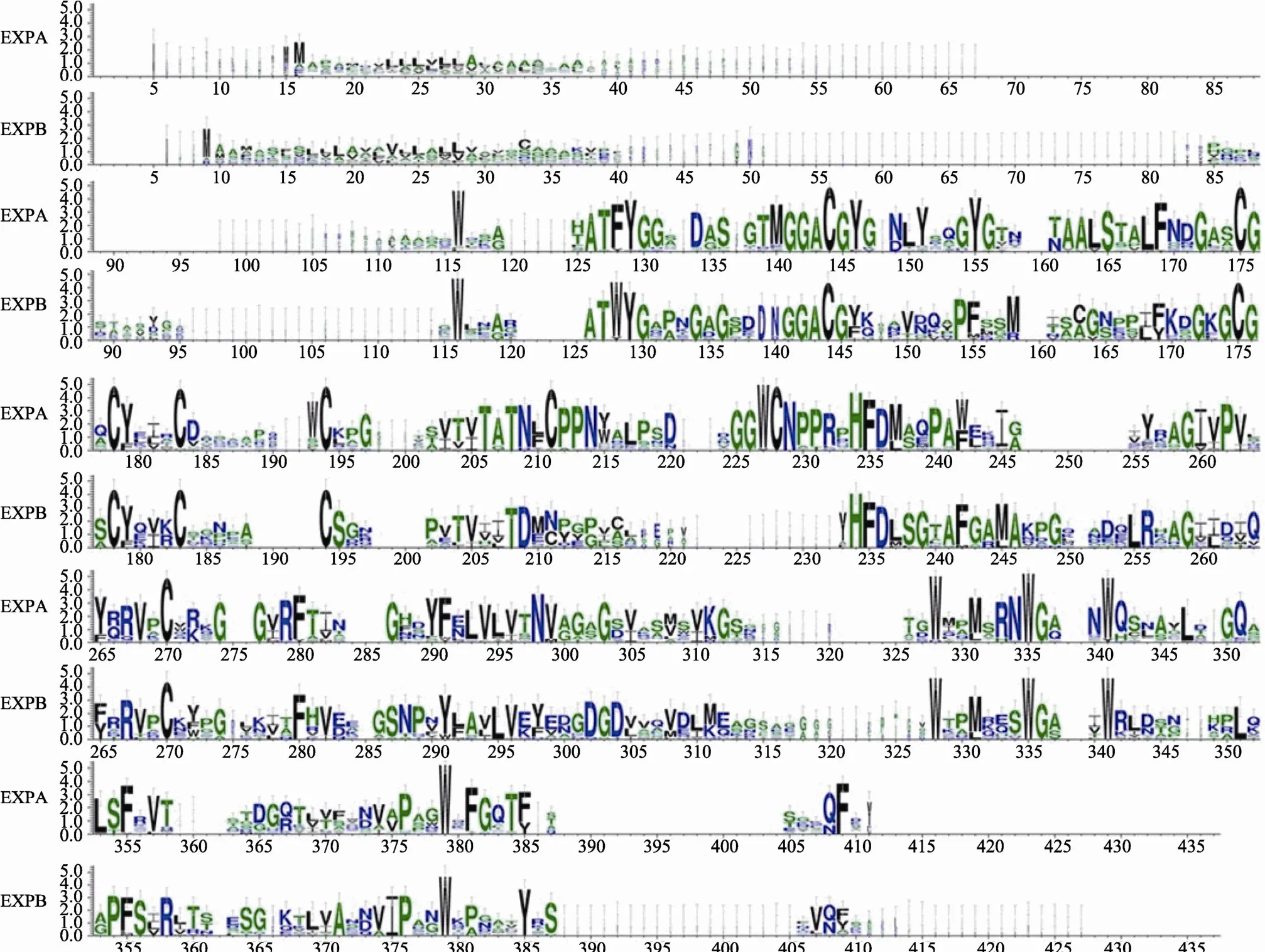
图4 水稻A、B类扩展蛋白亚家族成员氨基酸序列保守性分析
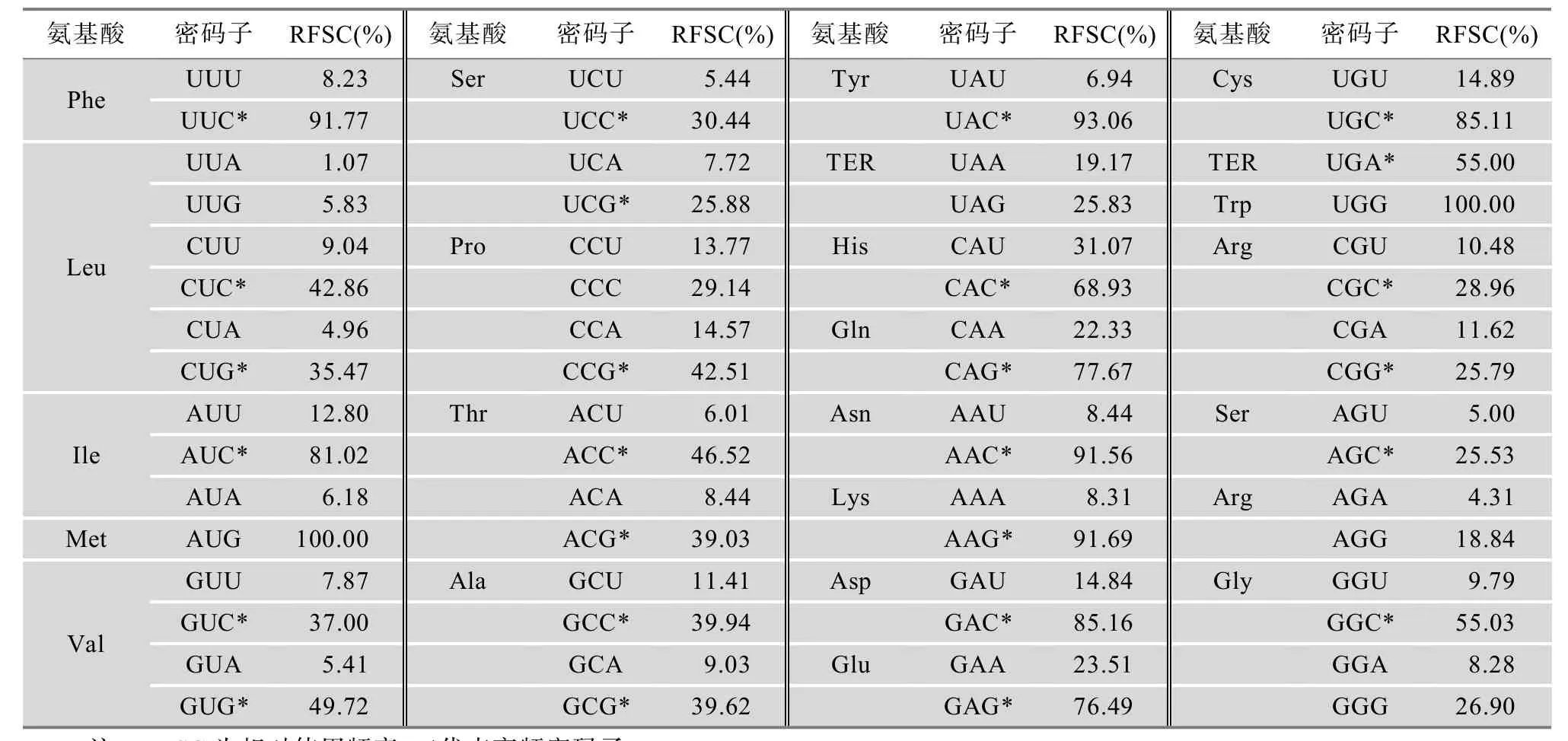
表5 水稻扩展蛋白基因密码子的相对使用频率(%)
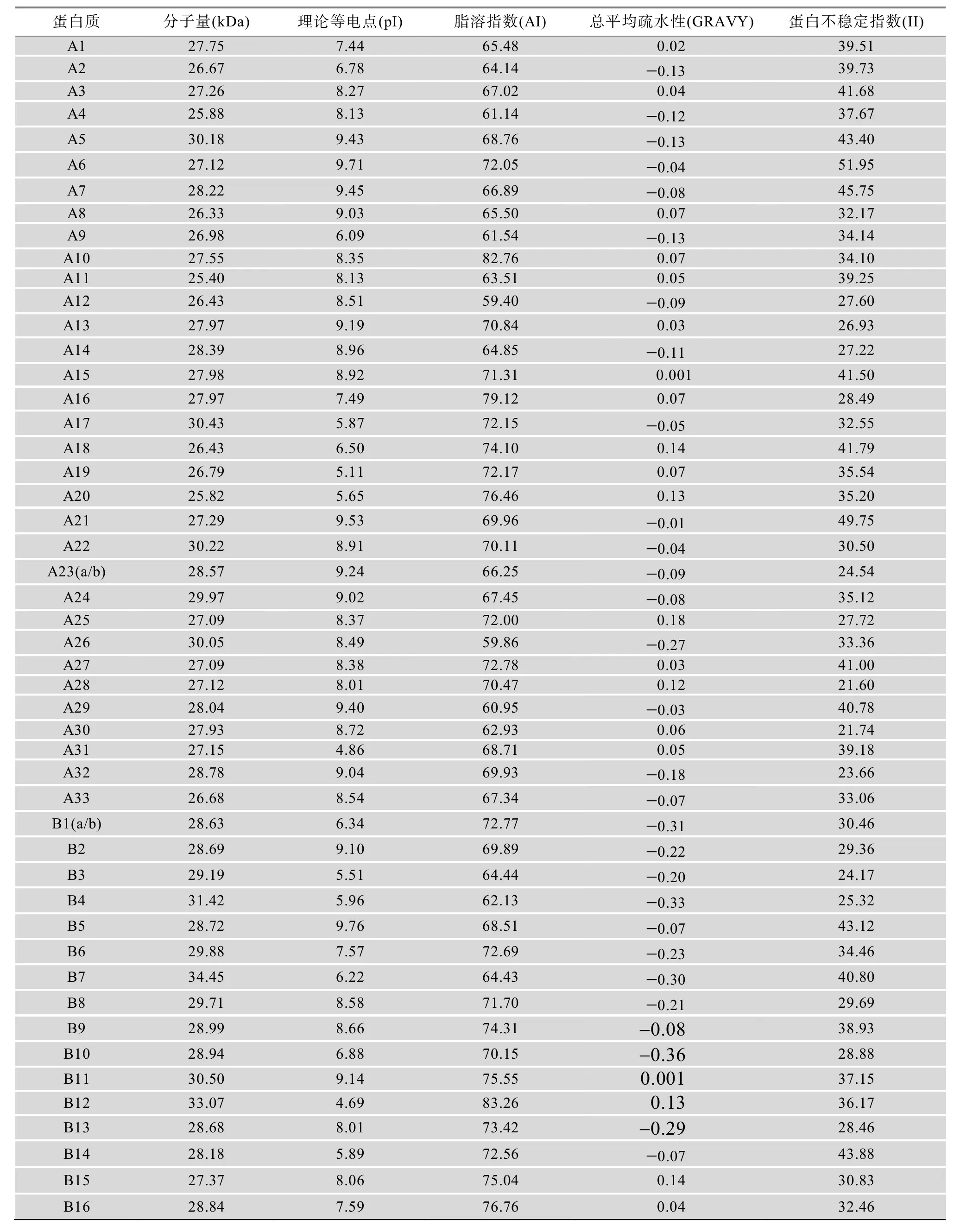
表6 水稻扩展蛋白分子量及理化性质
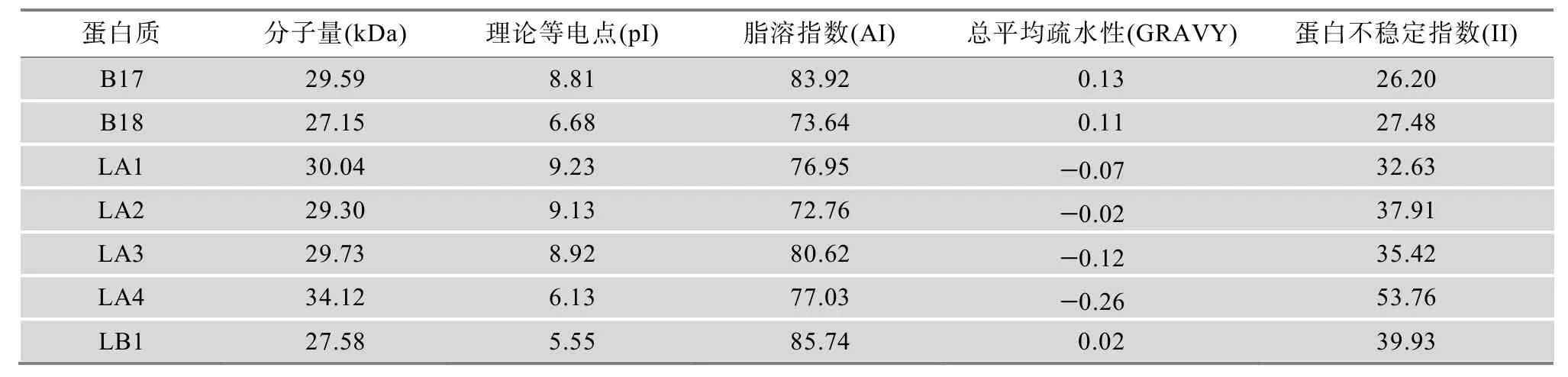
续表
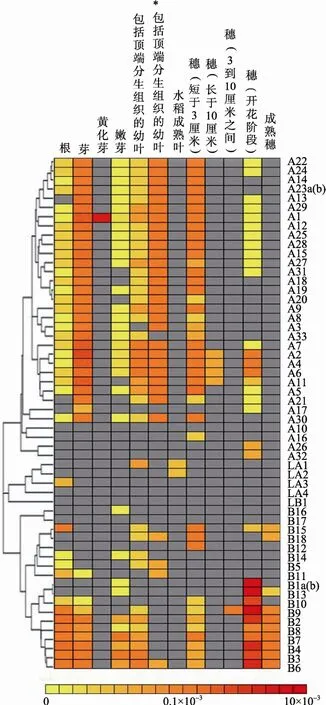
图 5 扩展蛋白家族成员在不同水稻组织器官中的表达分析
2.6 水稻扩展蛋白成员的表达模式分析
从水稻不同组织器官的 12个文库中检测到了扩展蛋白的表达信息,以文库中特定家族成员 EST数量所占的比率作为信号,绘制水稻扩展蛋白基因的时空表达图谱(图 5)。结果表明,A、B类亚家族基因表达的分布和丰度更高一些,而LA、LB类亚家族基因只在个别组织器官中表达。其中,水稻根中B亚家族表达量更高,幼嫩叶片和穗子中A亚家族表达更为丰富,但随着穗子的成熟,B亚家族的表达由弱变强。
3 讨 论
扩展蛋白广泛存在于植物基因组中,是植物细胞壁不可或缺的重要组成部分,也是物种进化过程中的重要链条之一。通过对扩展蛋白基因家族的系统生物信息学分析,有助于了解物种、亚家族、基因的基本特征,探索基因的起源和亲缘关系,分析基因在物种进化过程中的作用和地位,了解基因的功能。
水稻扩展蛋白基因家族包含4个亚家族,扩展蛋白在内含子/外显子模式、序列一致率、氨基酸组成以及基因表达模式上均呈现一定的亚家族特征,显示其进化上的规律,可以作为亚家族分类的重要依据,如除个别基因外,不同亚家族成员之间氨基酸的一致率低于 35%,而同一亚家族的成员之间大于35%;A类亚家族基因含有1或2个内含子(约占91%),而B类含有3个内含子(占47%),LA和LB类更趋向于4个内含子;A类蛋白含有较多苯丙氨酸(Phe),B类蛋白含有较多赖氨酸(Lys),LA类蛋白含有较多的精氨酸(Arg),LB类蛋白含有较多的丝氨酸(Ser);A类在芽中表达更高,B类在根和成熟穗中表达更高,LA和LB只在某些特定的部位表达。
另外,扩展蛋白也呈现一定的种属特征,主要表现在A/B类扩展蛋白比例、密码子偏好性等方面。如在已经完成测序的 3种模式植物水稻、拟南芥(Arabidopsis thaliana)和杨树(Populus)基因组中,A类扩展蛋白基因占总扩展蛋白基因的比例分别约为59%、72%和 75%,B类扩展蛋白的比例分别约为33%、17%和8%,水稻基因组中具有更多的B类扩展蛋白基因,而杨树和拟南芥基因组中含有更多的A类扩展蛋白基因,这一现象在玉米(Zea maysL.)、木瓜(Chaenomeles sinensis)等基因组中也有类似的特征,显现出扩展蛋白家族组成在单双子叶植物中的区别。
密码子的使用偏好性有利于基因的高效率表达[51,54,55],也是分类学上的一个重要指标。本研究对水稻扩展蛋白密码子使用频率分析发现,所有多密码子氨基酸及终止子均具有密码子使用偏好性,这一结果与水稻转录因子MADS密码子的偏好性非常吻合[56],高频密码子的数量和种类几近相同。同时,不同物种扩展蛋白的密码子使用频率表现不同,如在杨树、拟南芥以及水稻、玉米中,编码苯丙氨酸(Phe)的 UUC密码子的使用频率分别为 54.7%、55.0%、91.8%和 92.7%;扩展蛋白所有高频密码子的分布和数量也表现迥异,分别有 18、18、8和 5个氨基酸共 26、22、8和 6个高频密码子。3种终止密码子在几个物种中的使用频率也不尽相同(表7),特别是在UAA和UGA的选择上,水稻更趋向使用 UGA,而杨树和拟南芥更趋向使用 UAA。物种之间的密码子偏好性差异,不仅显示出种属特征,为基因的异源表达和基因工程育种工作提出了新的思路。
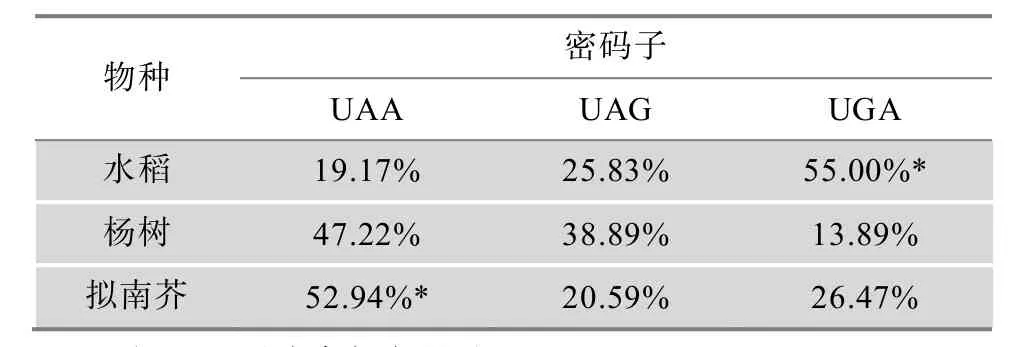
表7 不同物种扩展蛋白终止密码子的RFSC值
扩展蛋白是水稻基因组中不可或缺的组成部分,在植物生长、发育、生殖、抗性等许多生命活动中起着重要的作用。对不同扩展蛋白的表达分析(图5)可以看出,扩展蛋白的组成结构影响着基因的功能:A亚家族扩展蛋白更多作用于植物的生长发育和抗性,而B类亚家族蛋白则趋向于在生殖系统中起作用。如Yu等[17]发现A17为水稻根毛伸长所必需的基因;高英[57]认为A3基因在水稻中具有抗旱的作用;Choi等[12]发现水稻A4基因的正义表达有助于植株胚芽鞘和中胚轴的伸长;Ding等[31]研究发现,A1、A5、A10、B7基因的表达与水稻白叶枯抗性有关;Russell等[58]发现B类扩展蛋白基因与水稻花粉发育有关;Tabuchi等[59]发现玉米中 B类扩展蛋白与花粉管的生长发育有关。综上所述,目前有关水稻扩展蛋白功能的研究还比较浅显,结构与功能的关联的研究还比较少。本研究对水稻扩展蛋白系统的生物学信息分析,将为这一领域的研究提供一个基础平台。
[1]Cosgrove DJ. Loosening of plant cell walls by expansins.Nature, 2000, 407(6802): 321–326.
[2]Cosgrove DJ. Growth of the plant cell wall.Nat Rev Mol Cell Biol, 2005, 6(11): 850–861.
[3]Cho HT, Kende H. Expansins and internodal growth of deepwater rice.Plant Physiol, 2001, 126(4): 1471–1479.
[4]Cho HT, Kende H. Expansins in deepwater rice internodes.Plant Physiol, 1997, 113(4): 1137–1143.
[5]Cosgrove DJ. Characterization of long-term extension of isolated cell walls from growing cucumber hypocotyls.Planta, 1989, 177(1): 121–130.
[6]Lee Y, Kende H. Expression of β-expansins is correlated with elongation of internodes in deepwater rice.Plant Physiol, 2001, 127(2): 645–654.
[7]Mcqueen-Mason S, Durachko DM, Cosgrove DJ. Two endogenous proteins that induce cell wall extension in plant.Plant Cell, 1992, 4(11): 1425–1433.
[8]Cho HT, Kende H. Expression of expansin genes is correlated with growth in deepwater rice.Plant Cell, 1997, 9(9):1661–1671.
[9]Cosgrove DJ, Li LC, Cho HT, Hoffmann-Benning S,Moore RC, Blecker D. The growing world of expansins.Plant Cell Physiol, 2002, 43(12): 1436–1444.
[10]Liu WG, Tao J, Zhou XR, Yang WY. Characteristics of expansins insoybean (Glycine max) internodes and responses to shade stress.Asian J Crop Science, 2011, 3(1):26–34.
[11]Lee Y, Kende H. Expression of α-expansin and expansin-like genes in deepwater rice.Plant Physiol, 2002,130(3): 1396–1405.
[12]Choi D, Lee Y, Cho HT, Kende H. Regulation of expansin gene expression affects growth and development in transgenic rice plants.Plant Cell, 2003, 15(6): 1386–1398.
[13]Huang J, Takano T, Akita S. Expression of α-expansin genes in young seedlings of rice (Oryza sativaL.).Planta,2000, 211(4): 467–473.
[14]Lee Y, Choi D. Biochemical properties and localization of the β-expansin OsEXPB3 in rice (Oryza sativaL.).Mol Cell, 2005, 20(1): 119–126.
[15]Magneschi L, Kudahettige RL, Alpi A, Perata P. Expansin gene expression and anoxic coleoptile elongation in rice cultivars.J Plant Physiol, 2009, 166(14): 1576–1580.
[16]Cho HT, Cosgrove DJ. Regulation of root hair initiation and expansin gene expression inArabidopsis.Plant Cell,2002, 14(12): 3237–3253.
[17]Yu ZM, Kang B, He XW, Lv SL, Bai YH, Ding WN, Chen M, Cho HT, Wu P. Root hair-specific expansins modulate root hairelongation in rice.Plant J, 2011, 66(5): 725–734.
[18]Cho HT, Kende H. Tissue localization of expansins in deepwater rice.Plant J, 1998, 15(6): 805–812.
[19]Guo W, Zhao J, Li X, Qin L, Yan X, Liao H. A soybean β-expansin gene GmEXPB2 intrinsically involved in root system architecture responses to abiotic stresses.Plant J,2011, 66(3): 541–552.
[20]Shin JH, Jeong DH, Park MC, An G. Characterization and transcriptional expression of the α-expansin gene family in rice.Mol Cells, 2005, 20(2): 210–218.
[21]Yang L, Zheng B, Mao C, Qi X, Liu F, Wu P. Analysis of transcripts that are differentially expressed in three sectors of the rice root system under water deficit.Mol Genet Genomics, 2004, 272(4): 433–442.
[22]Wu YJ, Thorne ET, Sharp RE. Modification of expansin transcript levels in the maize primary root at low water potentials.Plant Physiol, 2001, 126(4): 1471–1479
[23]Fleming AJ, McQueen-Mason S, Mandel T, Kuhlemeier C.Induction of leaf primordia by the cell wall protein expansin.Science, 1997, 276(5317): 1415–1418.
[24]Cho HT, Cosgrove DJ. Altered expression of expansin modulates leaf growth and pedicel abscission inArabidopsis thaliana.Proc Natl Acad Sci USA, 2000, 97(17): 9783–9788.
[25]Keller E, Cosgrove DJ. Expansins in growing tomato leaves.Plant J, 1995, 8(6): 795–802.
[26]Madoka GM, Mellerowicz EJ, Abe H, Schrader J, Winzéll A, Sterky F, Blomqvist K, McQueen-Mason S, Teeri TT,Sundberg B. Expansins abundant in secondary xylem belong to subgroup a of the α-expansin gene family.Plant Physiol, 2004, 135(3): 1552–1564.
[27]Dotto MC, Pombo MA, Martínez GA, Civello PM. Heat treatments and expansin gene expression in strawberry fruit.Sci Hortic, 2011, 130(4): 775–780.
[28]Rose JKC, Lee HH, Bennett AB. Expression of a divergent expansin gene is fruit-specific and ripening-regulated.Proc Natl Acad Sci USA, 1997, 94(11): 5955–5960.
[29]Imin N, Kerim T, Rolfe B, Weinman JJ. Effect of early cold stress on the maturation of rice anthers.Proteomics,2004, 4(7): 1873–1882.
[30]Pezzotti M, Feron R, Mariani C. Pollination modulates expression of the PPAL gene, a pistil-specific β-expansin.Plant Mol Biol, 2002, 49(2): 187–197.
[31]Ding X, Cao Y, Huang L, Zhao J, Xu C, Li X, Wang S.Activation of the indole-3-acetic acid–amido synthetase GH3–8 suppresses expansin expression and promotes salicylate- and jasmonate-independent basal immunity in rice.Plant Cell, 2008, 20(1): 228–240.
[32]Ren H, Gu G, Longa J, Yin Q, Wu T, Song T, Zhang S,Chen Z, Dong H. Combinative effects of a bacterial type-III effector and a biocontrol bacterium on rice growth and disease resistance.J Biosci, 2006, 31(5): 617–27.
[33]Geilfus CM, Zoerb C, Muhling KH. Salt stress differentially affects growth-mediating β-expansins in resistant and sensitive maize (Zea maysL.).Plant Physiol Biochem, 2010, 48(12): 993–998.
[34]Han YY, Li AX, Li F, Zhao MR, Wang W. Characterizaition of a wheat (Triticum aestivumL.) expansin gene,TaEXPB23, involved in the abiotic stress response and phytohormone regulation.Plant Physiol Biochem, 2012,54(5): 49–58.
[35]Xu JC, Tian J, Belanger FC, Huang B. Identification and characterization of an expansin gene AsEXP1 associated with heat tolerance in C3 Agrostis grass species.J Exp Bot, 2007, 58(13): 3789–3796.
[36]Zhao MR, Han YY, Feng YN, Li F, Wang W. Expansins are involved in cell growth mediated by abscisic acid and indole-3-acetic acid under drought stress in wheat.Plant CellRep, 2012, 31(4): 671–685.
[37]王辉, 孙涛, 赵会君, 柴团耀. 商陆扩展蛋白基因PaEXP1在逆境胁迫下的表达. 中国科学院研究生院学报, 2010, 27(4): 448–455.
[38]Choi D, Cho HT, Lee Y. Expansins: expanding importance in plant growth and development.Physiol Plant, 2006,126(4): 511–518.
[39]Goff SA, Ricke D, Lan TH, Presting G, Wang R, Dunn M,Glazebrook J, Sessions A, Oeller P, Varma H, Hadley D,Martin C, Katagiri F, Lange BM, Moughamer T, Xia Y,Budworth P, Zhong JP, Miguel T, Paszkowski U, Zhang SP,Colbert M, Sun WL, Chen LL, Cooper B, Park S, Wood TC, Mao L, Quail P, Wing R, Dean R, Yu Y, Zharkikh A,Shen R, Sahasrabudhe S, Thomas A, Cannings R, Gutin A,Pruss D, Reid J, Tavtigian S, Mitchell J, Eldredge G,Scholl T, Miller RM, Bhatnagar S, Adey N, Rubano T,Tusneem N, Robinson R, Feldhaus J, Macalma T, Oliphant A, Briggs S. A draft sequence of the rice genome (Oryza sativaL. ssp. indica).Science, 2002, 296(5565): 92–100.
[40]刘仁虎, 孟金陵. MapDraw, 在Excel中绘制遗传连锁图的宏. 遗传, 2003, 25(3): 317–321.
[41]Gascuel O, Steel M. Neighbor-joining revealed.Mol Biol Evol, 2006, 23(11): 1997–2000.
[42]Peden JF. Analysis of Codon Usage. Nottingham: University of Nottingham, 1999, 101: 13951–13956.
[43]Crooks GE, Hon G, Chandonia JM, Brenner SE. WebLogo:A sequence logo generator.Genome Res, 2004, 14(6):1188–1190.
[44]Walker JM. The proteomics protocols handbook. New York City: Humana Press, University of Hertfordshire,Hatfield, 2005.
[45]Sakai H, Lee SS, Tanaka T, Numa H, Kim J, Kawahara Y,Wakimoto H, Yang CC, Iwamoto M, Abe T, Yamada Y,Muto A, Inokuchi H, Ikemura T, Matsumoto T, Sasaki T,Itoh T. Rice annotation project database (RAP-DB): An integrative and interactive database for rice genomics.Plant Cell Physiol, 2013, 54(2): e6.
[46]Sampedro J, Cosgrove DJ. The expansin superfamily.Genome Biol, 2005, 6(12): 242.
[47]牛艳梅, 沈文涛, 周鹏. Expansin超级家族的进化与命名. 广东农业科学, 2007, (8): 133–135.
[48]高英, 王学臣. 扩张蛋白(Expansin)研究进展. 农业生物技术科学, 2005, 21(7): 82–86.
[49]徐筱, 徐倩, 张楷, 徐吉臣. 植物扩展蛋白基因的研究进展. 北京林业大学学报, 2010, 32(5): 154–161.
[50]许成钢, 范晓军, 付月君, 梁爱华. 二硫键的形成与蛋白质的氧化折叠. 中国生物工程杂志, 2008, 28(S6):259–264.
[51]林涛, 倪志华, 沈明山, 陈亮. 高频密码子分析法及其在烟草密码子分析中的应用. 厦门大学学报(自然科学版), 2002, 41(5): 551–554.
[52]付海辉, 辛培尧, 许玉兰, 刘岩, 韦援教, 董娇, 曹有龙,周军. 几种经济植物UFGT基因的生物信息学分析. 基因组学与应用生物学, 2010, 30(1): 92–102.
[53]Ikai A. Thermostability and aliphatic index of globular proteins.J Biochem, 1980, 88(6): 1895–1898.
[54]刘庆坡, 冯英, 董辉. 20个物种同义密码子偏性的比较分析. 西北农林科技大学学报(自然科学版), 2004, 32(7):67–71.
[55]王艳, 马文丽, 郑文岭. SARS冠状病毒的密码子偏爱性分析. 生命科学研究, 2003, 7(3): 219–223.
[56]李娟, 薛庆中. 拟南芥及水稻转录因子MADS密码子的偏好性比较. 浙江大学学报(农业与生命科学版), 2005,31(5): 513–517.
[57]高英, 陈乃芝, 熊艳梅, 王学臣. 扩张蛋白在旱稻根的免疫组化定位及对旱稻抗旱性的表达分析. 自然科学进展, 2006, 16(4): 475–479.
[58]Russell SD, Bhalla PL, Singh MB. Transcriptome-based examination of putative pollen allergens of rice (Oryza sativassp. japonica).Mol Plant, 2008, 1(5): 751–759.
[59]Tabuchi A, Li LC, Cosgrove DJ. Matrix solubilization and cell wall weakening by β-expansin (group-1 allergen)from maize pollen.Plant J, 2011, 68(3): 546–559.
