一种基于高密度遗传标记的亲子鉴定方法及其应用
2014-03-08张哲罗元宇李晴晴贺金龙高宁张豪丁向东张勤李加琪
张哲,罗元宇,李晴晴,贺金龙,高宁,张豪,丁向东,张勤,李加琪
1. 华南农业大学动物科学学院,国家生猪种业工程中心,广东省农业动物基因组与分子育种重点实验室,广州 510642;
2. 中国农业大学动物科学学院,北京 100193
系谱是人类遗传及动植物育种研究的重要信息来源之一。系谱错误在遗传研究及育种生产中普遍存在,如英国奶牛群体的系谱错误率约为 10%[1],以色列为10.8%[2],丹麦为5%~15%[3],荷兰为12%[4],爱尔兰为7%~20%[5],国外奶牛系谱平均错误率约为11%[6]。我国天津及北京奶牛场的系谱错误率分别为12%[7]和17%~21%[8,9]。除奶牛外,系谱错误在其他畜种中也有研究报道[10]。系谱错误会减慢群体的遗传进展,比系谱缺失带来更大的育种损失[11],也会影响其他利用系谱信息的研究,如QTL(Quantitative trait locus)定位和基因组选择结果的可靠性。
用血型和血液蛋白型[12]及分子标记[13]可对疑似亲子关系进行亲子鉴定。但前者进行亲子鉴定准确率低,在实际应用中受到诸多限制[1]。近年来,随分子生物学的发展,尤其是测序及生物芯片技术的进步,小卫星[14,15]、微卫星[16,17]和单核苷酸多态(Single nucleotide polymorphism, SNP)[18]等分子标记逐渐用于畜禽系谱重建或校正[2,19]以及人类亲子鉴定[20]。其中,SNP标记因遗传稳定性高、突变率低、全基因组覆盖率高、分型准确性高和成本低等特点[21]已成为人类及动植物遗传研究中常用的一种分子标记。
目前可用于亲子鉴定的方法主要有排除法、似然法和基因重构法,它们主要用于自然群体的亲子鉴定,在家畜亲子鉴定中前两种方法使用较多[22]。基于这些方法的亲子鉴定软件主要有 Cervus[23]、KINSHIP[24]等。虽然各软件的特点和效果有一定差别[22],但它们均为亲子鉴定而设计,可解决不同情形下的亲子推断问题。为降低亲子鉴定的基因型检测成本,它们多采用相对复杂的算法,以保证用少量标记准确完成亲子鉴定。
近年来,很多研究通过利用高密度SNP标记进行基因组扫描或全基因组关联分析定位QTL[25,26],或通过全基因组选择预测动植物个体的育种值[27]或人类的患病风险[28]。这些研究的前期数据处理中,多数都需用到系谱以进行数据校正、基因型填充和单倍型推断等。因此,开展研究之前需先对系谱进行校正,以保证后续研究结果的准确性。然而,目前能直接使用全基因组高密度SNP标记的亲子鉴定方法尚缺。据此,本研究首先提出一种在群体中直接利用全基因组SNP标记进行亲子鉴定的新方法,进而在杜洛克猪及荷斯坦奶牛两个实验群体中进行验证,最后对该方法的应用效果及特性进行了详细讨论。
1 材料和方法
1.1 实验群体及基因型检测
本研究共使用2个实验群体。第一个群体是来自北京地区的2180头中国荷斯坦奶牛,包括87头公牛和2093头母牛。系谱记录显示所有母牛均来自14个公牛家系[29,30]。所有个体的基因组DNA从母牛凝血或公牛冷冻精液中提取,使用 Illumina BovineSNP 50 BeadChip[31]进行全基因组SNP标记检测,该芯片共包含54 001个SNPs。
第二个群体是来自福建省某种猪场的 191头纯种杜洛克猪,其中公猪18头,母猪173头,系谱记录完整。全部个体的基因组DNA均从耳组织提取,用 Illunima PorcineSNP60 BeadChip进行全基因组SNP标记基因型检测,该芯片包含61 565个SNPs[32]。系谱显示该基因型检测的群体中有73对亲子对,且均为后代与母亲的关系。
所有SNP数据均进行质量控制,去除SNP检出率<0.9、最小等位基因频率(Minor allele frequency,MAF)<0.01的 SNP位点和个体检出率<0.9的个体,然后进行后续分析。
1.2 系谱错误的检验方法
本检验方法基于孟德尔遗传定律,即每个遗传位点的等位基因均以孟德尔遗传方式由亲本传递给后代。据此,在疑似亲本和疑似后代个体所构成的待检测亲子对间,可对每一个双等位基因的遗传位点用如下规则进行孟德尔错误判定:(1) 若疑似亲本为纯合基因型,而疑似后代为另一种纯合基因型,如亲本为AA,后代为aa,则可判定为一个孟德尔错误;(2) 若疑似亲本为杂合基因型,则两种等位基因均有可能传递给后代,因此在另一亲本未知的情况下,无论后代为何种基因型均无法进行孟德尔错误判定。假定共有N个位点可用于进行该检验,其中疑似亲本为杂合基因型的位点数为Nh个,疑似亲子对间孟德尔错误次数为Nme,则有效检测次数为N - Nh,孟德尔错误率Re= Nme/(N - Nh)。需要注意的是,该方法基于如下假定:(1) 所有个体的DNA样品采集及基因型检测过程无误;(2) 基因型分型无错误发生或者有完全随机的分型错误发生,但错误率极低。
基于群体基因型数据,用上述规则对疑似亲子对及无关个体对进行孟德尔检测,即可获得孟德尔错误率的经验分布,划定错误率的阈值,即可对系谱的正确性进行判定。
1.3 系谱错误检验的程序实现及应用
利用1.2所述原理,使用R语言[33]进行程序开发,程序命名为EasyPC(Easy Pedigree Checking)。输入文件为群体基因型文件和待检测系谱文件。该程序可自动对群体孟德尔错误进行检测,并依据经验分布划定阈值,判断待检测系谱是否正确。程序最后自动输出系谱检测结果,并对错误率进行图形输出。EasyPC软件程序代码及测试数据已免费共享至https://github.com/SCAU-AnimalGenetics/EasyPC。
本研究将开发的程序应用于 1.1所述的两个群体中进行系谱错误检验。因群体结构不同,本研究在两个群体中使用了不同的检测策略。在奶牛群体中,检测全部公牛与全部母牛间的孟德尔错误率;在猪群体中,检测全部个体间的孟德尔错误率。
为进一步了解1.2所描述的方法的运行效率,本研究对比了EasyPC与Cervus 3.0[23]程序标准参数设置时的运行时间及资源占用情况。全部对比均在同一操作环境下进行,CPU 主频3.1GHz,内存4.0GB,Win7操作系统。
2 结果与分析
2.1 基因型数据
经过质控,奶牛和猪的数据集分别剩余2112和190个个体,45 738和40 999个SNPs用于系谱错误检测。本研究所用的奶牛群体中SNPs最小等位基因频率分布见图1。由图1可见,全部SNPs的MAF呈均匀分布,猪数据集的MAF分布与图1相似(结果未展示)。奶牛群体和猪群体的平均 MAF分别为0.26和0.25,杂合度均为0.35。
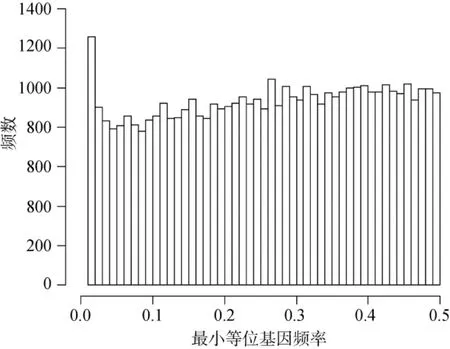
图 1 中国荷斯坦奶牛群体最小等位基因频率(MAF)分布图
2.2 孟德尔错误率分布
对质控后的奶牛群体中87头公牛及2025头母牛个体间进行孟德尔错误率计算,分布如图 2。孟德尔错误率整体呈正态分布,但在错误率接近0处有另一单峰。正态分布处为非亲子个体对间孟德尔错误率,0点单峰处应为真正的亲子对间孟德尔错误率。两部分分布的界限清晰,所以本研究选择1.0%作为对本数据集孟德尔错误率的判定阈值。孟德尔错误率最小值为0,最大值为0.18,平均值为0.11。
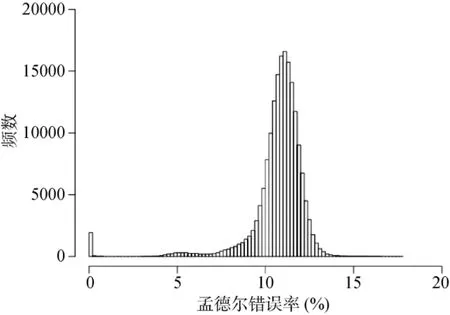
图2 87头公牛与2025头母牛配对的孟德尔错误率分布图
为获得杜洛克猪群体孟德尔错误率的经验分布,对该群体中经过质控后的全部 190个个体间进行孟德尔错误率计算,结果如图 3。孟德尔错误率整体呈正态对称分布,但在 0点处有一单峰。两部分分布界限清晰,所以本研究选择1.0%作为对本数据集孟德尔错误率的判定阈值。孟德尔错误率最小值为0,最大值为0.17,平均值为0.09。
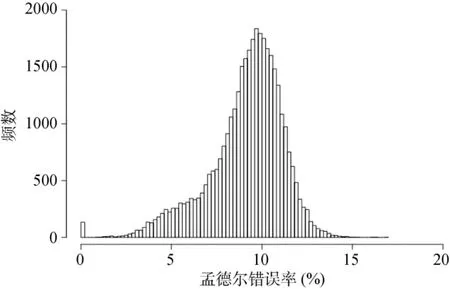
图3 190头杜洛克猪个体对间孟德尔错误率分布图
2.3 系谱错误分析
根据以上的孟德尔错误率分析可知,在孟德尔错误率的群体经验分布图中,两部分分布界限十分清晰。在本研究的两个群体中,均选定1.0%为鉴定系谱错误与否的孟德尔错误率阈值。用该阈值对奶牛群体进行系谱错误统计:在2025头母牛中,1937头母牛同时具有父亲基因型,可用于系谱错误检测。其中1 547头母牛系谱经基因型鉴定为系谱正确,剩余的 390条系谱中母牛与父亲公牛间孟德尔错误率超过阈值,可判定为错误系谱。因此,该群体中系谱错误率为 20%。通过该方法,390头系谱错误的母牛中有320头在公牛群体中配对发现孟德尔错误率低于阈值,参考牛的出生时间记录即可推测为正确的亲子关系。据此,可更正错误系谱并记录正确的亲子关系,有效地提升系谱正确率。
猪群体中的系谱错误分析表明:在73对系谱记录的同时进行基因型检测的亲子对中,4对亲子对因质控而被剔除,只剩下69对可用于后续研究。其中,有65对亲子对间孟德尔错误率小于阈值,即判定为系谱正确,其余 4对个体间系谱记录错误。因此,该杜洛克猪群体中系谱错误率为6%。此外,本研究在对所有个体进行孟德尔错误进行计算时,还发现了 4对个体间错误率小于阈值,可推断为亲子关系,用于系谱校正。
2.4 运行效率对比分析
本研究在奶牛数据集中随机筛选了不同标记数及不同个体数的组合,对比了EasyPC与Cervus的运行时间(表 1)。在所筛选的个体中,有 50头公牛作为其余母牛的候选父亲,两软件均在全部50头公牛与全部母牛间进行亲子鉴定。结果表明:相同情况下Cervus计算时间明显高于EasyPC,如2500标记,1000个体时两者相差约 20倍。随着标记数的增加,两个软件的运行时间均呈线性增加,但Cervus运行时间增加速度大于 EasyPC,且在标记数多于2500时无法运行。Cervus运行时间随个体数的增加无明显变化,EasyPC虽呈线性增长,但即使个体数增至2000时,运行时间仍远低于Cervus。
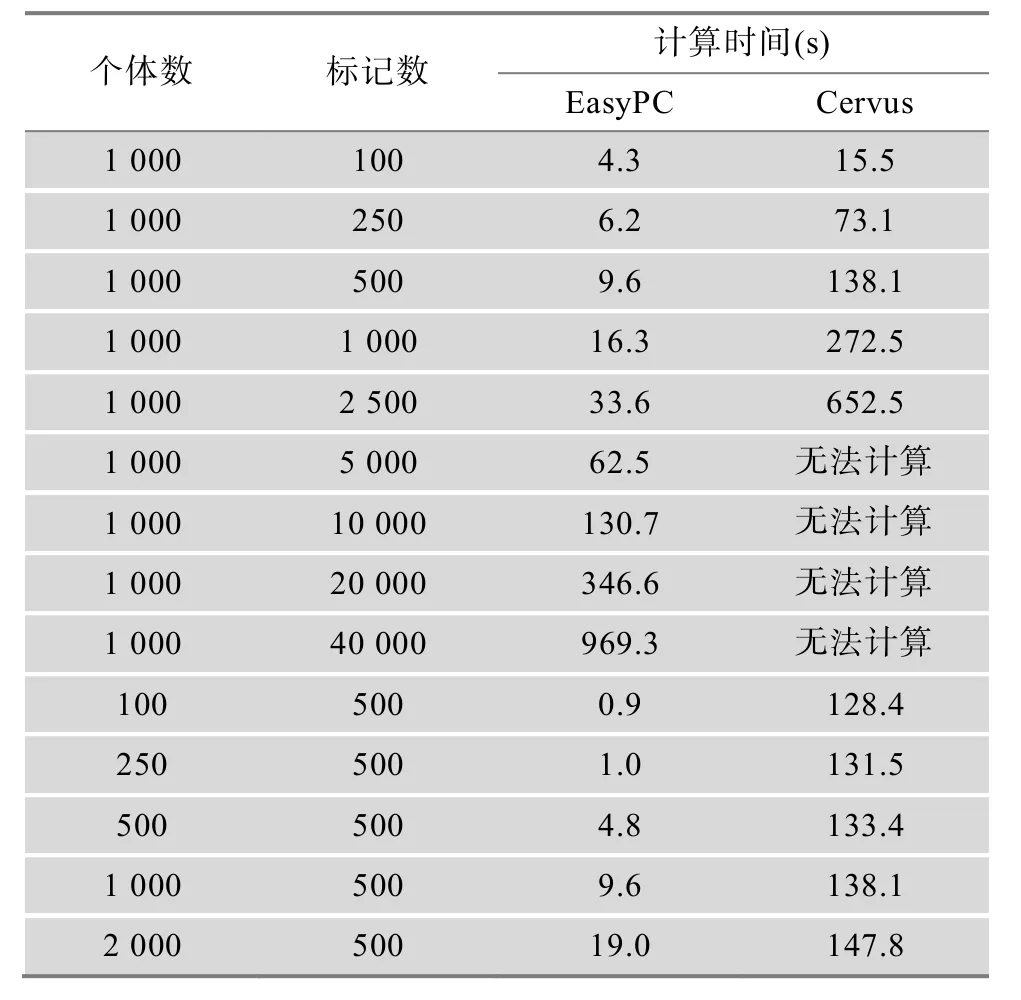
表1 不同个体数及标记数时两种方法运行时间对比
值得注意的是,除了本研究记录的运行时间以外,在应用两软件前的基因型数据及系谱整理方面也有较多差异。EasyPC仅需按格式提供完整的全基因组基因型文件和系谱文件即可,而Cervus则需按要求对全基因组基因型数据进行复杂的标记筛选和严格质控,这也降低了此种情况下软件应用的便利性。
3 讨 论
本研究提出了一种基于全基因组SNP标记,并以孟德尔错误率为判定依据来进行系谱错误检验的方法,并命名该方法为EasyPC。在奶牛及猪两个畜禽群体中的验证结果表明该方法能有效进行亲子间的系谱错误检验,更适用于全基因组基因型数据的亲子鉴定。
本研究所提出的系谱错误检验方法简单易行,尤其适用于全基因组高密度遗传标记数据。与Cervus的运行效率对比结果表明:常用亲子鉴定软件只能使用少量标记进行亲子鉴定,如在郭刚等人的研究[9]中对255个挑选的高多态性的SNP标记使用 Cervus进行计算。当我们尝试使用超过 2500个标记运行该软件时,该软件则无法正常获得结果。在全基因组高密度标记全部可用的前提下,若使用这些软件进行亲子鉴定,则要按照一定要求只筛选部分少量标记,进而按照操作步骤完成亲子鉴定。从信息利用的角度考虑,这显然不是最优化的方法,而且增加亲子鉴定操作的复杂度。但这些软件的算法更多是专为亲子鉴定或系谱重建工作而设计,以亲子鉴定为直接目的的研究或应用可使用这些软件以降低基因型检测的成本。相比之下,本研究所用的EasyPC方法简单易行。该方法依据孟德尔遗传定律及简单的假定条件,即可利用全基因组高密度遗传标记快速准确地实施系谱错误鉴定。在全基因组标记数据非常普遍的情况下,很多用到全基因组数据的研究都需首先进行系谱错误鉴定,进而开展其他研究,如全基因组关联分析、全基因组选择等。本文提出的这种简单易用的系谱校正方法必然会给相关研究的前期数据处理带来更多便利。
本研究所提出的系谱错误鉴定方法对系谱是否错误进行判定的依据是孟德尔错误率,而判定的标准则是根据研究群体孟德尔错误率的统计分布确定。在应用该方法进行判定时,应在群体内随机个体间进行孟德尔错误率计算。因随机个体间的孟德尔错误率计算结果可为本检测方法提供清晰的对照,为阈值的选取提供准确的依据。根据孟德尔遗传定律,亲子对间的孟德尔错误率极低,且在此仅受基因型检测错误的影响。因此,在标记数量很大时,孟德尔错误率极低。而随机个体对间的孟德尔错误率是群体MAF的函数,如某哈迪温伯格平衡位点的MAF为0.25时,据1.2的孟德尔错误率计算规则算得的错误率为11.25%。本文奶牛群体平均MAF为0.26,随机个体间平均错误率为 11%,与理论预期相符。据此,由较高的随机个体间孟德尔错误率作为对照,真实亲子对间因孟德尔错误率极低而很容易鉴定。本研究两个群体选择1.0%为阈值是根据错误率的群体经验分布数据而定(图2,图3)。但不同的应用中需要注意的是,MAF分布具有群体特异性。不同研究中,可能因基因型检测技术及群体遗传结构不同,选取的阈值也会有差异。但无论如何确定阈值,在孟德尔错误率的经验分布中,群体在错误率接近 0处会有一方差很小的正态分布峰,此处的个体对即为真实亲子对。此处孟德尔错误率不为0的主要原因是基因型检测错误导致孟德尔错误发生,但这种错误率在SNP芯片设计的容许范围内[24,34],而且不影响利用全基因组标记的亲子鉴定工作。
畜禽系谱错误在育种生产及科学实验过程中均会不可避免的发生。本研究所用的奶牛群体系谱来自生产现场,检测的错误率为 20%。而郭刚等[9]在同一群体内用 Cervus3.0软件进行亲子鉴定结果显示系谱错误率为 21%。两研究结果基本相符,差别可能是由于所用群体的筛选标准以及判别软件的不同所致,但都反映了生产现场系谱错误率较高的实际情况。本研究所用的杜洛克猪群体系谱错误率为6%,且其中有部分错误可能是实验采样或基因型检测过程中出错,因此实际群体系谱错误率可能更低。实际上该场是育种管理相对较为规范的育种场,从系谱错误率方面也反映了现场管理工作的规范程度。因此,系谱错误检验对生产管理也有重要意义。除此之外,及时剔除或纠正错误系谱可减少育种值估计误差,保证选种选配工作的准确性,提高育种工作的效率。
[1]Visscher PM, Woolliams JA, Smith D, Williams JL. Estimation of pedigree errors in the UK dairy population using microsatellite markers and the impact on selection. J Dairy Sci, 2002, 85(9): 2368–2375.
[2]Weller JI, Feldmesser E, Golik M, Tager-Cohen I, Domochovsky R, Alus O, Ezra E, Ron M. Factors affecting incorrect paternity assignment in the Israeli Holstein population. J Dairy Sci, 2004, 87(8): 2627–2640.
[3]Christensen LG, Madsen P, Petersen J. The influence of incorrect sire identification on the estimates of genetic parameters and breeding values. In: Proceedings of the 2nd World Congress on Genetics Applied to Livestock Production. Madrid, Spain, 1982: 200–208.
[4]Bovenhuis H, Van Arendonk JAM. Estimation of milk protein gene frequencies in crossbred cattle by maximum likelihood. J Dairy Sci, 1991, 74(8): 2728–2736.
[5]Beechinor JG, Kelly EP. Errors of identification Amongst cattle presented as progeny of some bulls used in the artificial-insemination service in Ireland. Ir Vet J, 1987,41(10): 348–352.
[6]Banos G, Wiggans GR, Powell RL. Impact of paternity errors in cow identification on genetic evaluations and international comparisons. J Dairy Sci, 2001, 84(11): 2523–2529.
[7]汪湛, 田雨泽, 刘和凤. 应用血型分析技术对奶牛亲子关系正确率的调查初报.中国畜牧兽医, 2005, 32(3):22–23.
[8]初芹, 张毅, 孙东晓, 俞英, 王雅春, 张沅. 应用微卫星DNA标记分析荷斯坦母牛系谱可靠性及影响因素.畜牧兽医学报, 2011, 42(2): 163–168.
[9]郭刚, 周磊, 刘林, 李东, 张胜利, 刘剑锋, 丁向东, 张毅, 王雅春, 张勤. 利用 SNP 标记进行北京地区中国荷斯坦牛亲子推断的研究.畜牧兽医学报, 2012, 43(1):44–49.
[10]韩春梅, 张嘉保, 高庆华, 陈庆波. 微卫星DNA在吉戎兔亲子鉴定中的应用研究.遗传, 2005, 27(6): 903–907.
[11]Sanders K, Bennewitz J, Kalm E. Wrong and missing sire information affects genetic gain in the Angeln dairy cattle population. J Dairy Sci, 2006, 89(1): 315–321.
[12]Stormont C. Contribution of blood typing to dairy science progress. J Dairy Sci, 1967, 50(2): 253–260.
[13]李东, 初芹, 王雅春. 单核苷酸多态性标记在牛亲子鉴定中的应用与展望.中国畜牧杂志, 2011, 47(7): 73–76.
[14]Kashi Y, Lipkin E, Darvasi A, Nave A, Gruenbaum Y,Beckmann JS, Soller M. Parentage identification in the bovine using “deoxyribonucleic acid fingerprints”. J Dairy Sci, 1990, 73(11): 3306–3311.
[15]Weir BS, Anderson AD, Hepler AB. Genetic relatedness analysis: modern data and new challenges. Nat Rev Genet,2006, 7(10): 771–780.
[16]Alford RL, Hammond HA, Coto I, Caskey CT. Rapid and efficient resolution of parentage by amplification of short tandem repeats. Am J Hum Genet, 1994, 55(1): 190–195.
[17]Glowatzki-Mullis ML, Gaillard C, Wigger G, Fries R.Microsatellite-based parentage control in cattle. Anim Genet,1995, 26(1): 7–12.
[18]Heaton MP, Harhay GP, Bennett GL, Stone RT, Grosse WM, Casas E, Keele JW, Smith TPL, Chitko-McKown CG,Laegreid WW. Selection and use of SNP markers for animal identification and paternity analysis in US beef cattle. Mamm Genome, 2002, 13(5): 272–281.
[19]Anderson EC, Garza JC. The power of single-nucleotide polymorphisms for large-scale parentage inference. Genetics, 2006, 172(4): 2567–2582.
[20]Pimenta JR, Pena SD. Efficient human paternity testing with a panel of 40 short insertion-deletion polymorphisms.Genet Mol Res, 2010, 9(1): 601–607.
[21]Werner FA, Durstewitz G, Habermann FA, Thaller G,Kramer W, Kollers S, Buitkamp J, Georges M, Brem G,Mosner J, Fries R. Detection and characterization of SNPs useful for identity control and parentage testing in major European dairy breeds. Anim Genet, 2004, 35(1): 44–49.
[22]Jones AG, Ardren WR. Methods of parentage analysis in natural populations. Mol Ecol, 2003, 12(10): 2511–2523.
[23]Kalinowski ST, Taper ML, Marshall TC. Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment. Mol Ecol,2007, 16(5): 1099–1106.
[24]Goodnight KF, Queller DC. Computer software for performing likelihood tests of pedigree relationship using genetic markers. Mol Ecol, 1999, 8(7): 1231–1234.
[25]Xu Z, Taylor JA. SNPinfo: integrating GWAS and candidate gene information into functional SNP selection for genetic association studies. Nucleic Acids Res, 2009, 37(Suppl. 2):W600–W 605.
[26]Cervino AC, Li G, Edwards S, Zhu J, Laurie C, Tokiwa G,Lum PY, Wang S, Castellani LW, Lusis AJ, Carlson S, Sachs AB, Schadt EE. Integrating QTL and high-density SNP analyses in mice to identify Insig2 as a susceptibility gene for plasma cholesterol levels. Genomics, 2005, 86(5): 505–517.
[27]Lee SH, van der Werf JHJ, Hayes BJ, Goddard ME, Visscher PM. Predicting unobserved phenotypes for complex traits from whole-genome SNP data. PLoS Genet, 2008,4(10): e1000231.
[28]Wray NR, Yang J, Hayes BJ, Price AL, Goddard ME,Visscher PM. Pitfalls of predicting complex traits from SNPs. Nat Rev Genet, 2013, 14(7): 507–515.
[29]Jiang L, Liu JF, Sun DX, Ma PP, Ding XDQ, Yu Y, Zhang Q. Genome wide association studies for milk production traits in Chinese Holstein population. PLoS ONE, 2010,5(10): e13661.
[30]Ding X, Zhang Z, Li X, Wang S, Wu X, Sun D, Yu Y, Liu J,Wang Y, Zhang Y, Zhang S, Zhang Y, Zhang Q. Accuracy of genomic prediction for milk production traits in the Chinese Holstein population using a reference population consisting of cows. J Dairy Sci, 2013, 96(8): 5315–5323.
[31]Matukumalli LK, Lawley CT, Schnabel RD, Taylor JF,Allan MF, Heaton MP, O'Connell J, Moore SS, Smith TPL,Sonstegard TS, Van Tassell CP. Development and Characterization of a High Density SNP Genotyping Assay for Cattle. PLoS ONE, 2009, 4(4): e5350.Ramos AM, Crooijmans RPMA, Affara NA, Amaral AJ,Archibald AL, Beever JE, Bendixen C, Churcher C, Clark R, Dehais P, Hansen MS, Hedegaard J, Hu ZL, Kerstens HH, Law AS, Megens HJ, Milan D, Nonneman DJ, Rohrer GA, Rothschild MF, Smith TPL, Schnabel RD, Van Tassell CP, Taylor JF, Wiedmann RT, Schook LB, Groenen MAM.Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technology. PLoS ONE, 2009, 4(8):e6524.
[32]R Core Team. R: A language and environment for statistical computing.R Foundation for Statistical Computing,Vienna, Austria, 2014.
[33]Worthington Wilmer J, Allen PJ, Pomeroy PP, Twiss SD,Amos W. Where have all the fathers gone? An extensive microsatellite analysis of paternity in the grey seal (Halichoerus grypus). Mol Ecol, 1999, 8(9): 1417–1429.
