重组大肠杆菌产青霉素酰化酶的发酵动力学研究
2014-02-21韦晓菊黎继烈朱晓媛
韦晓菊,黎继烈*,朱晓媛
(中南林业科技大学生命科学与技术学院经济森林培育与保护教育重点实验室,湖南长沙410004)
重组大肠杆菌产青霉素酰化酶的发酵动力学研究
韦晓菊,黎继烈*,朱晓媛
(中南林业科技大学生命科学与技术学院经济森林培育与保护教育重点实验室,湖南长沙410004)
对产青霉素酰化酶重组大肠杆菌M3进行分批发酵动力学研究。根据Logistic和Luedeking-Piret方程,拟合出重组大肠杆菌M3
青霉素酰化酶;分批发酵;发酵动力学模型
青霉素酰化酶(penicillinacylase)是一种具有重要应用价值的工具酶,既能广泛应用于半合成β-内酰胺抗生素,又能反向催化水解青霉素,生产合成β-内酰胺抗生素的中间体6-氨基头孢烷酸(6-amino-cephalosporanicacid,6-ACA)和7-氨基脱乙酰头孢烷酸(7-amino desacetoxy cephalosporanic acid,7-ADCA);同时在手性药物的拆分和多肽合成等方面也有不俗表现[1-5]。近年来抗生素生产市场已形成以DSM-GB公司为首(约占34%),中国、印度、韩国等亚洲公司(约25%)和荷兰Duchefa Biochemie B.V.公司(约18%)、上海Antibioticos国际贸易有限公司(约10%)等欧美公司并存的格局[6-7]。在市场需求的催生下,工艺流程简单,反应条件温和,清洁无污染的酶法合成抗生素应运而生。目前,青霉素酰化酶促合成β-内酰胺抗生素及制备6-ACA等中间体的新工艺和新技术不断涌现[8-10],使绿色环保的酶法合成抗生素替代化学法得以大规模推广,必将对抗生素产业发展产生深远影响。
发酵过程中菌体生长、产物生成、基质消耗的动态平衡及其内在规律构成的动力学模型即为发酵动力学模型的3个组成部分[11]。因而建立重组大肠杆菌产青霉素酰化酶的动力学模型,并对发酵过程的数据进行分析能更好的认识菌体的生长及产物形成的机制,为进一步放大实验以及工业化优化与调控提供理论依据。且重组大肠杆菌发酵原料廉价,产酶周期短[12],既适合多次重复验证试验以得出最优结果,又为大规模工业化生产提供了便利。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种
重组大肠杆菌M3:本实验室保藏。
1.1.2 培养基
种子培养基(LB培养基):蛋白胨1.0%,酵母浸粉0.5%,氯化钠1.0%,pH 7.0。121℃灭菌20 min。
发酵培养基:蛋白胨2.0%,酵母浸粉1.0%,氯化钠1.0%,甘油2.0%,卡那霉素50μg/mL,泡敌0.05%,pH 7.0。
1.1.3 试剂
蛋白胨(分析纯):北京奥博星生物技术有限责任公司;酵母浸粉(分析纯):上海研生生化试剂有限公司;氯化钠(分析纯):天津大茂化学试剂厂;甘油(分析纯):国药集团化学试剂有限公司;卡那霉素(分析纯):鹏程生物技术有限公司;异丙基硫代半乳糖苷(优级纯):上海励瑞生物科技有限公司。
1.2 仪器与设备
B型SL5L全自动控制发酵罐:上海保兴生物设备工程有限公司;SZK-730紫外可见分光光度计:深圳欧克仪表科技有限公司;S-250D型超声波细胞破碎仪:美国Branson公司;AvantiJ-E高速冷冻离心机:美国BeckmanCoulter公司。1.3实验方法
1.3.1 种子液培养
每瓶50mL分装于250mL,锥形瓶中,灭菌后加入100μg/mL卡那霉素,接入甘油保藏菌液,在30℃、160r/min条件下振荡培养24h。
1.3.2 分批发酵过程
分批发酵采用5L发酵罐,装料系数0.6,即加入3L发酵培养基,121℃实罐灭菌20min,冷却至30℃后接入100mL种子液。发酵过程中,保持罐内压为0.02~0.03MPa之间,并使用溶氧电极和pH电极实时监测溶氧和pH的变化情况。通过调节转速和通气量使溶氧保持在20%~60%。培养至10h,测定菌体浓度,降温至24℃,加入30μg/mL异丙基硫代半乳糖苷(isopropyl thiogalactoside,IPTG)进行诱导。
1.3.3 发酵过程中参数的测定
菌体浓度的测定:采用湿重法。取发酵液1mL用高速离心机离心10min弃上清液称质量,减去离心管的质量并求得菌体湿质量。
青霉素酰化酶酶活的定义:在37℃时,单位体积的青霉素酰化酶在单位时间内催化青霉素钾盐水解生成1μmol的6-APA所需的酶量定义为1个活力单位,U/L。根据定义采用碱滴定法[13]测定酶活力。
甘油含量的测定:用甘油铜比色法[14]。
1.3.4 数据的获取及处理
从发酵开始计时取样,再每3h取样测定不同时期的菌体湿质量、青霉素酰化酶酶活力以及甘油浓度,以上实验每组做3次平行实验,取平均值,并用Origin 8.0绘图软件对动力学方程进行非线性拟合,绘制动力曲线,以获得最佳动力学模型参数[15]。
2 结果与分析
2.1 重组大肠杆菌产青霉素酰化酶发酵过程中代谢变化规律
重组大肠杆菌M3代谢变化特征如图1所示。由图1可知,该菌体生长延迟期为0~3h,在该阶段菌体基质消耗较少,菌体浓度增长不明显,青霉素酰化酶产量为零。进入对数期后,菌体生长加快,基质消耗增加,甘油含量显著下降,青霉素酰化酶产量也随之增长。进入稳定期后菌体湿质量几乎不再增长,甘油消耗量也逐渐减少,但青霉素酰化酶酶活力持续升高,酶活力在27h时达到最高值33.5U/mL,由此判断该菌种产的青霉素酰化酶的合成方式属于生长部分偶联型。
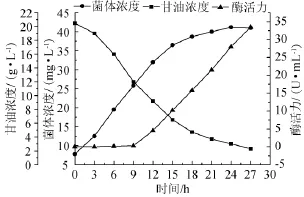
图1 重组大肠杆菌M3产青霉素酰化酶发酵进程曲线Fig.1 Fermentation curve of penicillin acylase produced by Escherichia coliM3
2.2 动力学模型
2.2.1 菌体生长动力学模型
细胞生长动力学普遍使用Logistic方程,其能较好描述分批发酵过程中因菌体浓度的增加对菌体自身存在的抑制作用。该方程被看作是一个表现细胞生长于营养物质之间非线性关系的经验方程[16-17]。本实验中大肠杆菌的生长曲线为较标准的S型,故采用Logistic方程作为研究重组大肠杆菌M3的菌体生长动力学模型。
Logistic方程:

式中:X为菌体浓度,mg/L;Xm为最大菌体浓度,mg/L;μm为菌体最大比生长速率,h-1;t为时间,h。
将式(1)积分为代数方程:

式中:X为菌体浓度,mg/L;X0为初始菌体浓度,mg/L;Xm为最大菌体浓度,mg/L;μm为菌体最大比生长速率,h-1;t为时间,h。
参照公式(2),使用Origin 8.0绘图软件对菌体生长实验数据进行非线性拟合,结果如图2所示。
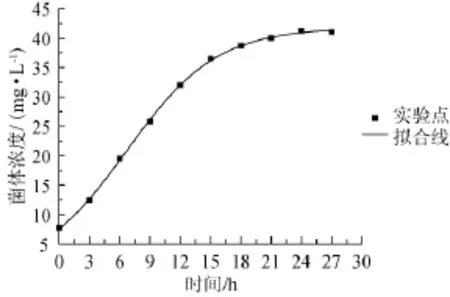
图2 菌体生长的试验值与模型计算值的比较Fig.2 Comparison of experimental data with calculated data on cell growth
菌体湿质量(X)随发酵时间变化的函数为:

式中:X为菌体浓度,mg/L;μm=0.224 42,X0=7.604 27mg/L,Xm=41.751 49mg/L;t为发酵时间,h。
由图2可知,实验数据与公式(3)的计算值基本吻合,R2=0.995 5,结果表明实验值与动力学模型基本相符,因而大肠杆菌产青霉素酰化酶优化调控可参考依据该动力学模型。
2.2.2 青霉素酰化酶合成动力学模型
由于微生物在发酵的动态过程中细胞内的生物具有十分复杂的合成途径,且其代谢调节机制又特点各异,于是依据产物生成速率与细胞生长速率之间的关系,将产物的形成与微生物细胞生长关系的动力学模型分为3类:Ⅰ生长偶联型;Ⅱ部分生长偶联型或称混合型;Ⅲ非生长偶联型[15,18],常用Luedeking-Piret方程[19-20]描述产物形成与细胞生长的关系如下:

式中:X为菌体浓度,mg/L;P为青霉素酰化酶酶活,U/mL;
α为生长偶联系数;β为非生偶联联系数。
α≠0、β=0表示第Ⅰ类发酵;α≠0、β≠0表示第Ⅱ类发酵;α=0、β≠0表示第Ⅲ类发酵。从大肠杆菌产青霉素酰化酶分批发酵试验结果可以推断该菌体的发酵过程属于第Ⅱ类,即产物形成与细胞生长呈部分偶联型。联合公式(2)、(4),Luedeking-Piret方程可积分为代数方程:
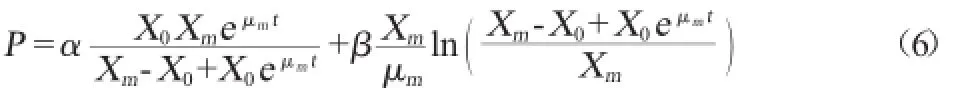
式中:X为菌体浓度,mg/L;X0为初始菌体浓度,mg/L;Xm为最大菌体浓度,mg/L;μm为菌体最大比生长速率,h-1;t为时间,h。P为青霉素酰化酶酶活,U/mL;P0为初始青霉素酰化酶酶活,U/mL;α为生长偶联系数;β为非生偶联联系数。

由于P0=0,X0对产物的影响可以忽略不计,式(5)可简化为:
式中:μm为菌体最大比生长速率,h-1;X0为初始菌体浓度,mg/L;Xm为最大菌体浓度,mg/L;t为时间,h。P为青霉素酰化酶酶活,U/mL;P0为初始青霉素酰化酶酶活,U/mL;α为生长偶联系数;β为非生偶联系数。
参照公式(6),采用Origin 8.0绘图软件对大肠杆菌产青霉素酰化酶发酵实验数据进行非线性拟合,结果如图3所示,得到青霉素酰化酶合成量(P)随发酵时间变化的函数:
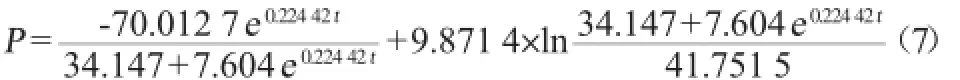
式中:P为青霉素酰化酶酶活,U/mL;t为时间,h。
其中α=-0.22052,β=0.05306,青霉素酰化酶活的实验值与模型计算值的比较见图3。由图3观察得出公式(7)较好的拟合了所得的实验数据,R2=0.993 0,也验证了该方程很好的描述出发酵过程中青霉素酰化酶的合成情况,但受到发酵液中蛋白酶以及其他限制性因素的影响,青霉素酰化酶酶活力在合成后期的增长受到了抑制。若及时补充底物,则可促进酶的继续合成,工业生产上可参考这一规律。

图3 青霉素酰化酶活的试验值与模型计算值的比较Fig.3 Comparison of experimental data with calculated data on penicillin acylase activity
2.2.3 基质消耗动力学模型
大肠杆菌产青霉素酰化酶发酵过程中,甘油的消耗一方面用于菌体生长,同时又用于其菌体细胞的维持和产物的合成及代谢,因此基质消耗速度可以由与Luedeking-Piret相似的方程表示:

式中:S为甘油浓度,g/L;X为菌体浓度,mg/L;t为时间,h;YX/S为碳源用于菌体生长得率常数;γ为碳源用于产物积累得率常数。
结合方程式(2)、式(8)积分可得:
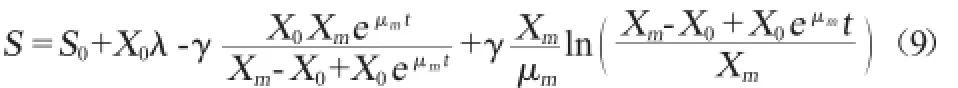
式中:S为甘油浓度,g/L;S0为初始甘油浓度,g/L;X为菌体浓度,mg/L;X0为初始菌体浓度,mg/L;Xm为最大菌体浓度,mg/L;μm为菌体最大比生长速率,h-1;t为时间,h;YX/S为碳源用于菌体生长得率常数,λ=1/YX/S;γ为碳源用于产物积累得率常数。
按照公式(9)对发酵过程中甘油消耗的实验值进行非线性拟合,结果如图4所示,得到基质消耗量随发酵时间变化的方程:

式中:S为甘油浓度,g/L;t为时间,h。
其中S0=21.085 37g/L,λ=0.449 28,λ=-0.004 67,由图4可以看出,式(10)能够很好的拟合了所得的实验数据,R2= 0.997 1,证明该模型能够很好的描述青霉素酰化酶合成过程中基质消耗情况。

图4 基质消耗的动力学模型与实验数据的比较Fig.4 Comparison of kinetic model data with experimental data on substrate consumption
3 结论
根据重组大肠杆菌M3发酵产青霉素酰化酶过程中各个参数的变化,采用Logistic方程和Luedeking-piret方程模拟出发酵的动态过程,建立了重组大肠杆菌M3产青霉素酰化酶的发酵动力学方程,分别为:
(1)菌体生长动力学模型

(2)青霉素酰化酶合成动力学模型

(3)基质消耗动力学模型
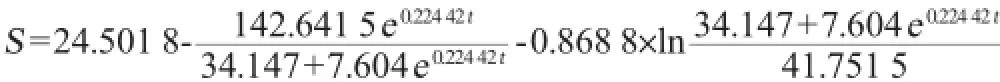
经过3个实验值与模型值的比对,发现所建立的动力学模型很好的拟合了实验数据,得出该菌株发酵动力学中菌体生长、青霉素酰化酶合成以及基质消耗的相关系数分别为:0.995 5、0.993 0、0.997 1。综上所述,3个模型的建立可以解释重组大肠杆菌M3产青霉素酰化酶的发酵动力学规律,为工业生产提供了可行的参考依据。
[1]杨正.青霉素酰化酶的分离纯化和固定化研究新进展[J].安徽化工,2010,36(3):7.
[2]崔鹏,万敏.青霉素酰化酶的生产与应用新进展[J].化学工业与工程技术,2005,26(6):42-45.
[3]任立华.青霉素G酰化酶工程菌培养条件的研究[J].齐鲁药事,2010,29(11):652.
[4]孔宪,李畅原,卢滇楠,等.青霉素酰化酶纳米凝胶的制备与性质[J].化工学报,2011,62(6):1641-1648.
[5]范超,黎继烈,吴浩,等.重组巨大芽孢杆菌产青霉素G酰化酶发酵条件的研究[J].中南林业科技大学学报,2011,31(7):124-129.
[6]BRUGGINK A,ROY P D.Synthesis ofβ-Lactam antibiotics[M].New York:Springer,2001.
[7]武英.全球抗生素市场概况分析[J].国外医药抗生素分册,2012,33(3):119-124.
[8]张业旺,谭强,刘瑞江,等.青霉素酰化酶制备6-APA的研究进展[J].中国抗生素杂志,2008,33(7):385-389.
[9]张业旺,刘瑞江,闻崇炜,等.青霉素酰化酶促合成β-内酰胺抗生素的过程优化研究进展[J].中国抗生素杂志,2010,35(10):721-724.
[10]朱科,王艳艳,丁海平,等.酶法制备阿莫西林的工艺优化研究[J].中国抗生素杂志,2011,36(1):44-47.
[11]陈丽.多拷贝毕赤酵母重组菌表达PIP生理特性和动力学研究[D].上海:华东理工大学硕士论文,2010.
[12]黄亚杰,王子辉,赵彦伟,等.重组大肠杆菌产谷氨酰胺酶培养基及发酵条件优化[J].中国酿造,2012,31(4):21-24.
[13]PRAMOD B MAHAJAN.Penicillin acylase[J].Appl Biochem Biotech,1984,12(9):537-554.
[14]阎杰,丘泰球.甘油铜比色法测定甘油含量的研究[J].中国油脂,2004,29(1):40-43.
[15]王挥,袁强,张敏,等.白地霉分批发酵产脂肪酶动力学模型的建立[J].中国粮油学报,2011,26(10):61-65.
[16]王莹,朴梅子,孙永海.基于遗传算法的羊肚菌液体发酵动力学模型的建立[J].生物工程学报,2008,24(8):1454-1457.
[17]万红贵,单咸旸,袁建锋,等.产胞外多糖芽孢杆菌的发酵动力学研究[J].中国酿造,2010,29(6):97-99.
[18]黎继烈,崔培梧,李忠海.柚苷酶产生菌Penicilliumsp.1523分批发酵动力学研究[J].中国食品学报,2012,12(1):32-37.
[19]ELIBOL M,MAVITUNA F.A kinetic model for actinorhodin production byStreptomyces coelicolorA3(2)[J].Process Biochem,1999,34 (6-7):625-631.
[20]朱博斐,原其朋.Blakeslea trispora发酵生产番茄红素动力学的研究[J].中国酿造,2009,28(6):105-107.
Fermentation kinetics of penicillin acylase produced by recombinantEscherichia coli
WEI Xiaoju,LI Jilie*,ZHU Xiaoyuan
(Ministry of Education Economic Forest Cultivation and Conservation of Education Ministry Key Laboratory,College of Life Science and Technology,Central South University of Forestry and Technology,Changsha 410004,China)
The fermentation kinetics for penicillin acylase production by the recombinantEscherichia coliM3 was studied in this paper.Based on the Logistic and Luedeking-Piret equation,the kinetic models of recombinantE.coliM3 about microorganism growth,penicillin acylase formation,substrate consumption and model parameter were proposed.The result showed that the fermentation process of penicillin acylase produced by recombinantE.coliM3 could be well expressed by the model.
penicillin acylase;batch fermentation;fermentation kinetics model
TQ920.1
A
0254-5071(2014)03-0032-04
10.3969/j.issn.0254-5071.2014.03.009
2014-01-25
国家林业局“948”项目(NO.2011-4-17)
韦晓菊(1987-),女,硕士研究生,研究方向微生物工程。
*通讯作者:黎继烈(1959-),女,教授,博士,研究方向农产品生物加工。
分批发酵过程中菌体生长、青霉素酰化酶合成、基质消耗的动力学模型及模型参数。结果表明,重组大肠杆菌M3产青霉素酰化酶的发酵动态过程能很好的被该模型所表达。
